结球甘蓝遗传图谱的构建及抗黑腐病QTL定位
2023-07-08乐祥庆钟雄辉宋旭朦颉建明康俊根
乐祥庆,钟雄辉,崔 建,韩 睿,宋旭朦,颉建明,康俊根
(1.甘肃农业大学 园艺学院,甘肃 兰州 730070;2.北京市农林科学院 蔬菜研究所,农业农村部华北地区园艺作物生物学与种质创制重点实验室,北京 100097)
结球甘蓝(BrassicaoleraceaL.var.capitataL.,2n=18)是2年生草本植物,为十字花科芸薹属[1],在我国种植广泛[2],年种植面积高达90万hm2[3],在蔬菜供应和出口中发挥重要作用。目前,在世界范围内黑腐病、枯萎病和根肿病是危害甘蓝产量和品质的三大主要病害,其中,黑腐病的危害最为严重。甘蓝黑腐病是一种细菌性病害,由野油菜黄单胞杆菌(Xanthomonascampestrispv.campestris)侵染所造成,发病后叶片出现“V”字型病斑[4],结球松散,叶球腐烂。由于黑腐病主要发生在叶片上,它对以叶片为主要产品和食用器官的作物影响都较大[5]。黑腐病是植物学家和昆虫学家哈里森于1898年在美国首次发现[6],随后在世界多个种植甘蓝的国家和地区陆续有发现。20世纪中后期,在我国的北京、天津、山西、内蒙古等地区有少面积的甘蓝黑腐病病害发生,但是随着甘蓝种植面积的增加,黑腐病对甘蓝品质和产量的威胁越来越大。
结球甘蓝的大多数农艺性状都属于数量性状,定位困难,研究效率低,其中最主要的因素是缺少适合数量性状研究的群体[7]。数量性状位点(Quantitative trait locus,QTL)指的是影响植物基因组上所有数量性状特征的基因的相对位置。许多重要的性状都是以数量遗传和持续变异为特点的多个基因所决定的。通过利用分子标记构建的遗传连锁图谱对相关性状进行QTL定位,可以在连锁群中鉴定控制数量性状的多个基因座及基因座间的相互作用关系。因此,分子标记遗传连锁图谱的构建在遗传育种学理论和功能基因克隆中具有重要的地位。遗传图谱广泛地应用于基因定位中,同时也为分子标记辅助选择和图位克隆提供了一个基础平台[8]。目前,国内外对甘蓝黑腐病QTL定位研究的报道比较少。Camargo[9]通过对甘蓝苗期及成株期的黑腐病抗性鉴定得到4个抗病QTL位点,分布在1,2,9号连锁群上,其中在2号连锁群有2个位点只与苗期的抗病性有关,其余2个位点与苗期及成株期的抗病性都相关。Tonu等[10]利用黑腐病感病材料GCP09和抗病材料Reiho P01构建的F2群体鉴定得到3个分别位于5,8,9号连锁群上的QTL位点,在8号连锁群上定位到1个主效位点,LOD值为7.7,最高可解释34%的贡献率。孔枞枞[11]利用抗病和感病材料构建F2群体,使用黑腐病1号生理小种并结合QTL-seq技术在3号连锁群上定位到抗病位点。
目前,国际上对甘蓝黑腐病菌致病力划分标准比较常用的是Vicente等[12]的划分标准。国内关于甘蓝黑腐病菌致病力划分标准的研究较少,有一些黑腐病致病力分化的研究。例如,翟文慧等[13]从11个地区采集黑腐病病样并进行分离鉴定,结果表明61株菌株均为野油菜黄单胞菌野油菜致病变种,其中1号生理小种占主导地位,为54.1%,4号生理小种占其次为36.1%,3号生理小种占比最少,没有发现2,5,6号生理小种。据统计表明,华北、东北等北方地区1,4号生理小种比例相当,1号生理小种主要在南方、西南、沿海地区[14]。本研究以结球甘蓝高抗黑腐病1号生理小种的自交系4674与高感黑腐病的自交系4673为亲本,构建F2分离群体为作图群体。通过苗期接种1号黑腐病生理小种对结球甘蓝进行抗病性鉴定,筛选多态性好的SSR和InDel标记,构建结球甘蓝遗传图谱,并在此基础上进行甘蓝抗黑腐病QTL定位。
1 材料和方法
1.1 供试材料
以结球甘蓝4674为父本,4673为母本杂交获得F1,F1自交得到F2,构建了包含152个单株的F2群体。2021年9月中旬将该群体种植于北京市农林科学院蔬菜研究中心农场。4674是黑腐病抗性较好的自交系,4673是农艺和品质性状优良但对黑腐病是高度感病的材料。本试验利用结球甘蓝4673和4674杂交获得F2群体进行遗传图谱构建和黑腐病抗性QTL定位。本研究所用病原菌为野油菜黄单胞杆菌(Xanthomonascampestrispv.campestris)1号生理小种。以上材料均来自北京市农林科学院蔬菜研究所甘蓝课题组。
1.2 病原菌培养
本试验所用黑腐病原菌为课题组在北京通州、海淀区四季青农场与中心基地采集的春季自然生长状态下甘蓝不同品种黑腐病感病叶片,随后对病原菌进行分离、纯化和鉴定。
用灭菌蒸馏水将黑腐病感病叶片清理干净,随后用75%酒精将叶片冲洗2遍,置于超净工作台中并用灭菌水将病叶再次清洗3~5次,将叶片表面水分吹干,用手术刀切割发病部位与正常组织间的部分,并将其切成小块。之后将切碎叶片转移到装有1.5 mL、pH=7.4的磷酸缓冲盐溶液的离心管中,置于28 ℃、150 r/min的摇床上进行黑腐病菌分离,分离时间为1 h。
菌种活化:振荡分离1 h后,取出离心管并将其置于超净工作台上,利用灭菌后的接菌环分别蘸取黑腐病菌的PBS缓冲液,在没有添加任何抗生素的LB固体培养基上进行划线。随后将其密封并置于28 ℃培养箱中,待培养基上出现黄色圆形黑腐病菌斑后,置于4 ℃冰箱中保存。
1.3 试验方法
1.3.1 人工接种 根据张鑫鑫等[15]研究报道,三叶一心期为甘蓝接病最佳时期,接种前需要根据单株数量确定病原菌的用量。利用分光光度计将菌液浓度稀释到1×108cfu/mL(OD600=0.2),供接种使用。为了增加菌液附着性,使菌液更有效地吸附到叶片上,每升菌液中加入2 mL吐温20,最后利用喷雾器均匀喷洒在F2群体的每个单株上。
1.3.2 病害鉴定 研究表明[15],甘蓝苗期和成株期抗病性基本相同,但苗期鉴定获得结果快,而且苗期鉴定可以在室内进行,以便更有效地控制生长所需条件。在苗期接种12~14 d后,按照甘蓝苗期鉴定方法及划分标准进行调查[16],逐一对每个单株的发病情况进行统计,并参照陈果等[17]的分级标准对F2群体进行病情分级和抗病性归类。发病指数(DI)=∑(病级叶数×该病级值)/(调查总叶数×最高级值)×100。按各株系发病指数确定其抗病类型,依据发病指数大小将各株系共分为以下5种类型:高抗:发病指数=0;抗病:0<发病指数≤10;中抗:10<发病指数≤30;感病:30<发病指数≤50;高感:50<发病指数。
1.3.3 DNA提取 取父本4674、母本4673及F2群体各单株大小约1 cm2的叶片放入2 mL离心管中,低温干燥器冻干后,采用改良后的CTAB法[18]提取各单株DNA,DNA浓度及质量通过Nanodrop 2000仪器检测,-20 ℃保存。
1.3.4 PCR链式反应体系与反应程序 PCR反应总体系为10 μL:DNA模板5 μL(30~50 ng/μL)、左右引物各0.5 μL、PCR mix 4 μL(包括TaqDNA聚合酶,dNTP,染料及PCR反应所需的缓冲液,由北京博迈德基因技术有限公司购入)。PCR反应程序步骤和参数设置参照朱洪运[19]的方法,反应产物利用10%的聚丙烯酰胺凝胶电泳分离。
1.4 数据统计
以父母本作对照,统计F2群体基因型数据,与甘蓝4673带型一致的记作a;与甘蓝4674带型一致的记作b;杂合记作h;无带或带型不清晰的记作—,将数据统计到Excel 2016表中。
1.5 甘蓝遗传图谱的构建
利用Joinmap 4.0构建遗传连锁图谱,遗传距离计算采用Kosambi函数进行并用厘摩(cM)表示,LOD大于5.0,其他为默认参数。使用Joinmap 4.0构建遗传图谱,具体操作步骤参照朱洪运等[20]的方法。连锁群的命名方法参照已公布的甘蓝遗传连锁图C1~C9依次进行命名。利用本课题组基于全基因组测序[21]自主开发的CA-CK系列的InDel标记,并结合本实验室已报道定位于染色体上的已知其他类型标记系列位置[22],将至少有3个自主开发或已知标记位于同一条连锁群上作为确定这一连锁群对应染色体的认定标准。
1.6 QTL定位及分析
利用定位软件MapQTL 6.0,结合各株系表型和遗传图谱对黑腐病群体进行定位,参照刘泽慈[23]将LOD值≥1.5作为QTL入选的标准,LOD值≥3.0则认为此处存在1个主效QTL,并依次在各染色体检测QTL的分布情况,并根据区间作图法对性状和基因型进行分析计算,估算出基因位点性状表型LOD值及加性效应等参数。与单标记作图法相比,其具有所需群体较小、能估计出QTL的位置与定位误差小等特点。参照朱洪运等[24]的命名方法进行命名。
2 结果与分析
2.1 引物筛选
选取本实验室基于全基因组测序自主开发的CA-CK系列的InDel标记及参考Wang等[25]、陈琛等[26]、Lü等[27]的文献设计的8C、M、BOSF、BOE等系列的InDel标记及本实验室前期设计的SSR标记,共404对分子标记。利用双亲4673和4674对404对分子标记进行PCR扩增与多态性筛选,对PCR扩增产物进行电泳检测,并进行2次重复对照,最终得到175对条带清晰,差异明显且重复性好的标记,标记多态性占比为43.3%。
2.2 高密度遗传图谱
利用筛选得到的175对差异显著的SSR和InDel标记和包含152个单株的F2群体构建遗传连锁图(图1)。有154对标记(Indel标记110对,SSR标记44对)分布到9条连锁群中,平均每条染色体上有17个标记。划分的连锁群数量与甘蓝基因组中9条染色体的数量一致。该图谱全长为714.29 cM(表1),9条连锁群长度为61.24~129.47 cM,分子标记数量为10~33个,遗传图谱的标记间平均图距为4.64 cM,其中参考Ensembl Plants(http://plants.ensembl.org/index.html)数据库得知甘蓝基因组大小为630 Mb,因此,构建的图谱上对应的平均图距与物理距离分别是4.64 cM,4.09 Mb。在这9条连锁群中,5号染色体最长,为129.47 cM,标记间平均图距为8.09 cM,标记间最大空隙为19.3 cM。8号染色体最短,为61.24 cM,标记间平均图距为3.60 cM。
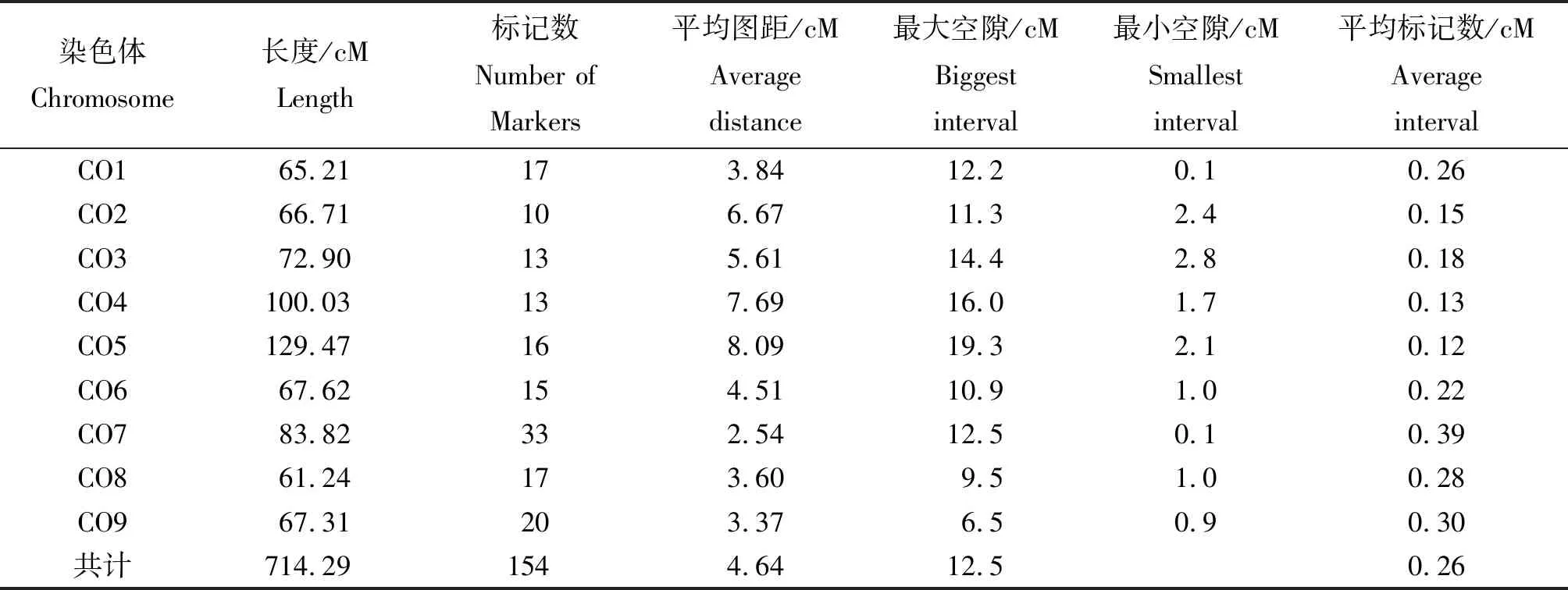
表1 SSR标记在遗传图谱上的分布Tab.1 Distribution of SSR markers on genetic map

图1 结球甘蓝遗传连锁图及黑腐病抗性定位区间Fig.1 Genetic linkage map and QTLs for resistance to black rot in cabbage
此外,在同一条染色体中也存在着标记分布不均匀现象,例如在分子标记分布最密集的C7染色体,相邻标记最小空隙为0.1 cM,最大空隙为12.5 cM,二者相差悬殊。其余几条染色体也同样存在标记分布不均匀特征。除了C8、C9连锁群外,其他连锁群均出现了大于10 cM最大相邻标记间隙。同时与许多其他作物一样,构建得到的图谱在连锁群上也出现了DNA分子标记聚集现象,由于开发标记时,在7号染色体上开发的标记多,所以在C7连锁群上可明显发现标记成簇状分布。
2.3 表型变异及遗传分析
利用Excel 2016对父母本及F2群体各单株病情进行分级统计分析,获得黑腐病亲本及F2群体抗性鉴定病情分级统计表(表2)及病情指数抗性分级柱状图(图2),从中可以看出两亲本中亲值为3.5,F2群体中病级平均值为3.23,介于两亲本之间,表现为中抗。F2群体病情等级为0~7级,其中抗黑腐病的病级多为1级,感黑腐病的病级多为5级,并且偏度为0.28,峰度为-0.98,这说明F2单株病级呈正态分布,符合QTL定位基本要求。由图3分析发现,F2群体的152个单株中黑腐病抗性为中抗的有51株,占比34%,因此,推测在抗病亲本4674中存在抗病的主效QTL。

表2 亲本及F2群体的黑腐病抗性鉴定病情分级Tab.2 Disease classification for identification of resistance to black rot of parents and F2 population
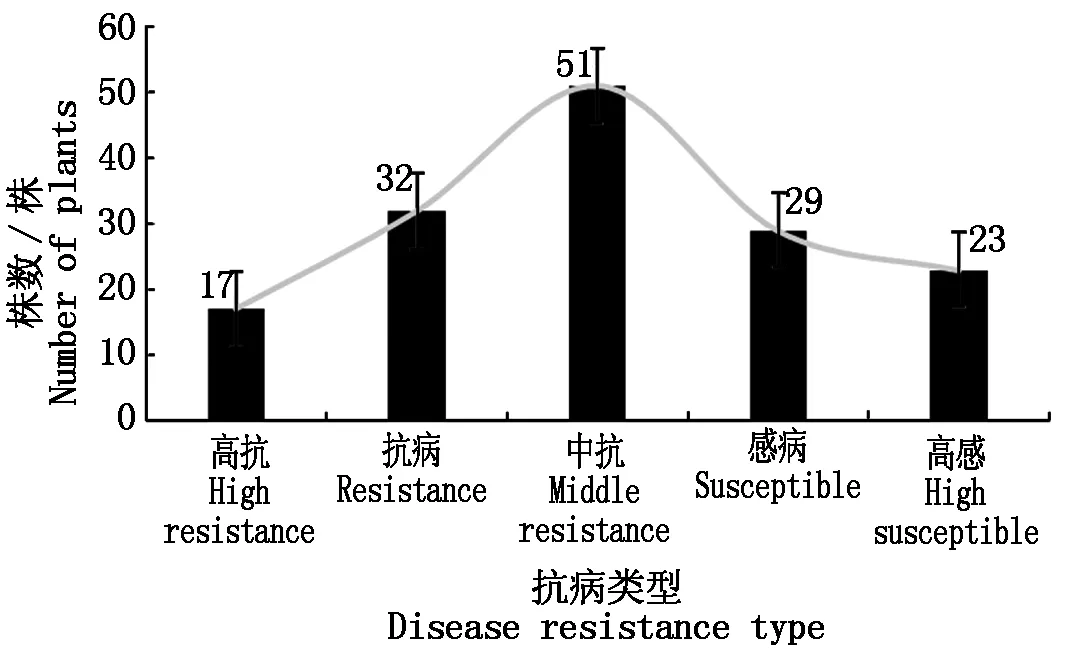
图2 F2群体苗期接种对黑腐病的抗性鉴定Fig.2 Identification of resistance of black rot in F2 individual young plants
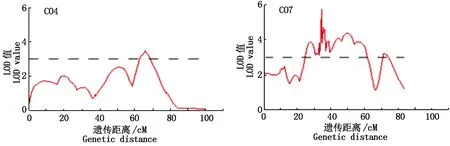
图3 F2群体黑腐病抗病QTL结果Fig.3 Results of QTL for resistance to black rot in F2 population
2.4 QTL定位分析
利用MapQTL 6.0软件进行黑腐病抗性QTL定位(图3),以LOD值≥1.5为QTL入选标准,LOD值≥3.0则认为此处存在1个主效QTL位点。共检测到7个抗黑腐病QTL位点(表3),分别位于4,6,7号连锁群上,LOD值为1.63~5.75,加性效应值为-0.54~0.92,可解释的表型贡献率为4.8%~16.0%。其中,在6号连锁群有1个QTL位点,遗传效应为负向;4号和7号连锁群各有3个QTL位点,遗传效应均为正向;LOD值大于3.0的QTL位点有3个,在4号连锁群的CD838151~BOE417区间内有1个位点,LOD值为3.47,可解释的表型贡献率为10.0%,7号连锁群上有2个位点,分别位于CG842110~CG842482和M29~M39,LOD值分别为5.75和3.20,可解释的表型贡献率为16.0%和9.2%。在检测到7个抗黑腐病位点中CG842110~CG842482区域内的LOD峰值最高,可解释的表型变异贡献率也最大。
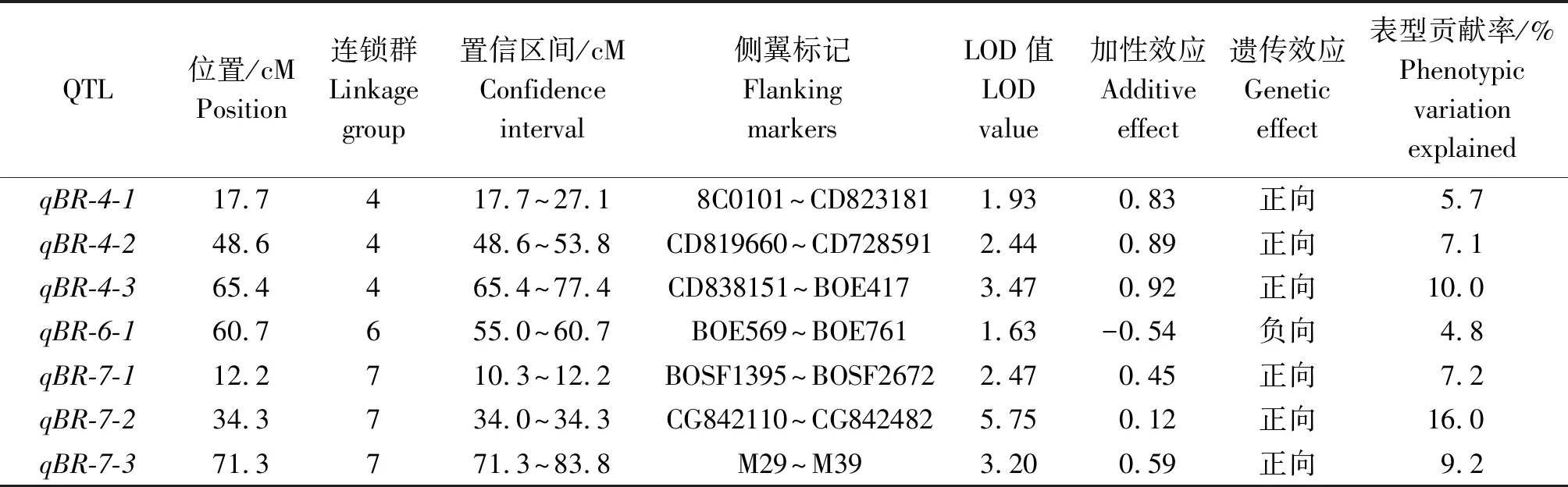
表3 甘蓝抗黑腐病性状QTL定位Tab.3 QTL mapping of resistance to black rot in cabbage
3 结论与讨论
3.1 分子标记遗传图谱的构建
QTL定位所用的群体一般有单双倍体(DH)、重组自交系(RIL)等永久性群体和F2、回交(BC)等暂时性群体。本试验所使用的为F2群体,虽然其具有可重复性较低,不易保存等缺点[28],但其优点是形成时间短,并且可以获得丰富的遗传信息;另外,F2群体内既存在纯合又有杂合基因型,所以F2群体既可以估算加性效应,又可以估算显性效应。永久性群体虽然具有稳定的遗传组成,可以重复试验,但是不能估算显性效应,而且构建费时费力,所以在图谱构建中大多采用F2群体。F2群体的大小对图谱的分辨率和准确性都有一定的影响,并且随着群体中个体数量的增加,其结果的可靠性也能得到一定的提高。F2群体在检测与重要性状紧密相连的基因位点时可以通过扩大群体降低误差来提高定位精度[29]。然而,如果数量太大,工作量和试验成本将大大增加。所以,在构建图谱过程中群体数量大小是至关重要的[30]。Li等[31]认为,对于100~200个单株定位群体,每个标记的间隔约为10 cM,就足以进行QTL初定位。本研究所用的F2群体包含152个单株,标记间平均密度为4.64 cM,表明该群体适合做定位。
分子标记遗传图谱的构建一般要求标记分布均匀且连锁群与染色体数量一致[32]。屠德鹏[33]认为,遗传图谱中分子标记的距离在10~20 cM为最佳,如果要用于数量性状基因定位,平均图距应小于10 cM。本试验是以构建甘蓝遗传连锁图为基础,目的是为了进行结球甘蓝抗黑腐病QTL定位。本研究构建的图谱平均图距为4.64 cM,其小于QTL定位所要求的平均图距10 cM,这表明该图谱可以用于甘蓝抗黑腐病QTL定位。在前人的报道中,遗传图谱大部分采用的是RFLP、ALFP等标记,而对SSR和InDel标记使用得较少,本研究所采用的SSR和InDel标记,大部分都是基于甘蓝重测序数据开发的标记,具有明确的物理图谱位置,可以直接把遗传图谱和物理图谱对应起来,为进一步的基因定位和克隆提供便利;而且这类标记数量多揭示的多态性高、具有多等位基因提供的信息量大、以孟德尔方式遗传共显性及成本低等优点,多用于QTL初步定位[34]。本研究构建的遗传图谱共有9条连锁群,总长度为714.29 cM,包含154个分子标记,标记之间的平均图距为4.64 cM,在图谱长度方面优于缪体云[35](图谱总长度为655.00 cM,包含458个标记)的研究;同时在标记数量和长度方面均优于李梅[36](图谱总长度677.70 cM,包含113个标记)的研究。本图谱虽然可以提高甘蓝遗传图谱的饱和度,但要进行精细定位和基因克隆还需要增加标记数量来提高该遗传图谱的密度。
3.2 结球甘蓝抗黑腐病QTL定位
在甘蓝抗黑腐病的研究中,苗期喷雾法常用于甘蓝黑腐病菌接种。刘泽慈[23]利用田间分离的黑腐病3号生理小种对结球甘蓝RILs与CSSLs群体进行喷雾法接病鉴定,共定位到分布在甘蓝第1,2,7和8号染色体上的11个抗黑腐病QTL,其中于7号染色体M35~M25区域在2次定位过程中表现出较强抗性,为稳定的QTL位点,可解释16.0%的表型贡献率。Doullah等[37]在2,3,7,9号连锁群上共鉴定到4个QTL位点,在2,9号连锁群上定位到的是2个主效的QTL位点,可以为选育抗黑腐病品种提供标记辅助选择,但由于构建图谱时只使用了92对分子标记,所以在定位的可信度上还缺乏说服力。其中,在7号染色体上检测到位于IPI~FLC3,LOD为2.25,解释了14.7%的表型贡献率。Kifuji等[38]利用甘蓝富士早生抗病品种和青花菜感病品种构建了1个F2群体,并新开发了161个EST-SNP标记,用于黄单胞菌黑腐病抗性的QTL分析,使得能够开发在甘蓝黑腐病抗性育种中有用的SNP标记提供了参考。鉴定得到3个位于2,4,5号连锁群上QTL位点,其中包含1个主效的位点QTL-1,位于2号染色体,最高可解释的表型贡献率为15.05%;同时在4号染色体的bocl4271s~bocl2635s标记间存在1个主效QTL位点,LOD值为4.86,可解释12.21%的表型贡献率。Lee等[39]通过在2个具有不同抗黑腐病能力的甘蓝自交系上开发dCAPS标记,并结合之前的图谱,在1,3,6号染色体上共检测到4个QTL位点,其中在3次的重复试验中BRQTL-Cl2位点都有被检测到,为稳定的QTL位点,可解释的表型贡献率为15.1%~27.3%,在6号染色体的sR12387~BnGMS353标记区间内有1个QTL位点,LOD值为3.847,可解释的表型贡献率为19.6%。本研究主要在4,6,7号染色体上检测到抗黑腐病QTL位点,前人的报道中,在这些染色体上也检测到了QTL位点,进一步说明了本研究结果的可靠性。但由于所用材料的不同,在相同染色体上定位的区间也有所不同,由此可以说明,影响QTL定位的因素有很多,双亲材料及群体类型、接病所用的黑腐病生理小种、分子标记、表型鉴定标准的不同及环境因素的影响都会导致定位结果存在异同。本试验在7号染色体CG842110~CG842482标记间定位到1个QTL位点qBR-7-2,这个位点是定位到的3个位点中效应最大的位点,LOD值为5.75,可解释的表型贡献率为16.0%,且这2个标记间距离只有0.3 cM,此区域有很高的研究价值,在后续的研究中可以以该区域为重点去开展试验,为主效QTL的精细定位奠定了良好基础。
本研究利用Joinmap 4.0构建结球甘蓝F2群体遗传连锁图谱,并对黑腐病抗性进行QTL定位。构建的遗传图谱有9条连锁群组成,共有154对分子标记(110对InDel标记,44对SSR标记),全长714.29 cM,平均图距为4.64 cM。利用MapQTL 6.0以LOD≥1.5作为入选标准,共检测出7个QTL位点,对黑腐病抗性的可解释表型贡献率为4.8%~16.0%。其中,有3个LOD值≥3.0为抗黑腐病的主效位点,有2个位点qBR-7-2和qBR-7-3分别在7号染色体上CG842110~CG842482及M29~M39标记间,另1个则位于4号染色体上的qBR-4-3在CD838151~BOE417标记间,3个主效QTL的LOD值分别为5.75,3.20和3.47,可解释的表型贡献率分别为16.0%,9.2%,10.0%,该研究结果可以为甘蓝抗黑腐病基因克隆及抗黑腐病品种的分子标记辅助选择提供理论指导。
