基于cyt b 基因的黄河上游高原鳅复合体的物种鉴定
2023-06-01伊日贵高强刘丹聂苗苗晁燕张存芳祁得林朱世海
伊日贵,高强,刘丹,聂苗苗,晁燕,张存芳,祁得林*,朱世海
(1.青海大学省部共建三江源生态与高原农牧业国家重点实验室,西宁 810016;2.青海大学生态环境工程学院,西宁 810016;3.青海大学农牧学院动物科学系,西宁 810016)
高原鳅属Triplophysa隶属于鲤形目Cypriniformes 条鳅科Nemacheilidae,广泛分布于青藏高原及其毗邻地区,是适应青藏高原寒冷、缺氧等气候特征的特殊类群(朱松泉,1989;武云飞,吴翠珍,1992;Prokofiev,2001)。约有150 种有效种被描述,有证据表明高原鳅属具有丰富的物种多样性,许多物种尚未被识别或描述(何春林等,2011)。
黄河上游高原鳅复合体是指由同域分布的、幼鱼阶段无法从形态特征分辨的高原鳅属鱼类组成。资料表明,黄河上游干流及支流湟水河流域主要分布有拟硬刺高原鳅T.pseudoscleroptera、硬刺高原鳅T.scleroptera、东方高原鳅T.orientalis、隆头高原鳅T.alticeps、斯氏高原鳅T.stoliczkae、粗壮高原鳅T.robusta、拟鲶高原鳅T.siluroides和黄河高原鳅T.pappenheimi等(武云飞,吴翠珍,1992)。在黄河上游高原鳅复合体中,硬刺高原鳅成体因背部和体侧上部具有黑褐色细斑纹,拟鲶高原鳅成体因头部和躯干宽阔而扁平,粗壮高原鳅因前躯呈圆筒形、背部在背鳍前后各有4~5 块深褐色斑块,隆头高原鳅成体因头背部高隆等典型特征而易于鉴定和区分,但其他高原鳅因形态特征不具典型性或因表型可塑性(或趋同进化),难以鉴定和区分(何春林等,2011)。更重要的是,在野外资源调查和物种多样性调研时,往往会采集到大量幼鱼,这些正值发育阶段的鱼类因不具典型特征而无法从形态进行分类和分析,为物种多样性和资源量的客观评估带来了一定的困难。
mtDNA 是研究分子系统学、生物地理学和亲缘物种遗传多样性的理想分子标记。在mtDNA 的13 个蛋白编码基因中,cyt b的结构和功能较清楚,且能用通用引物快速扩增,在不同科、属、种的研究中表现出明显的遗传差异(Guoet al.,2005),因此被广泛用于高原鱼类物种鉴定、分子进化、系统地理学和遗传结构研究(何德奎等,2006;Qiet al.,2007,2015;冯晨光,2018;Fenget al.,2019)。截止目前,针对黄河上游干流及支流湟水河流域高原鳅DNA 条形码和分子鉴定等方面的研究仍然滞后。本研究通过黄河上游干流及支流湟水河流域高原鳅样本的采集,利用cyt b基因标记开展分子鉴定,以期为黄河上游干流及支流湟水河流域的高原鳅物种分类、组成研究提供科学依据,为该地区高原鳅物种多样性的保护奠定一定的基础。
1 材料与方法
1.1 实验材料
高原鳅样本共95尾,采集于黄河上游干流及支流湟水河流域(表1)。野外采样时,取肌肉(成体)或幼体整体75%无水乙醇固定后,-80 ℃保存。
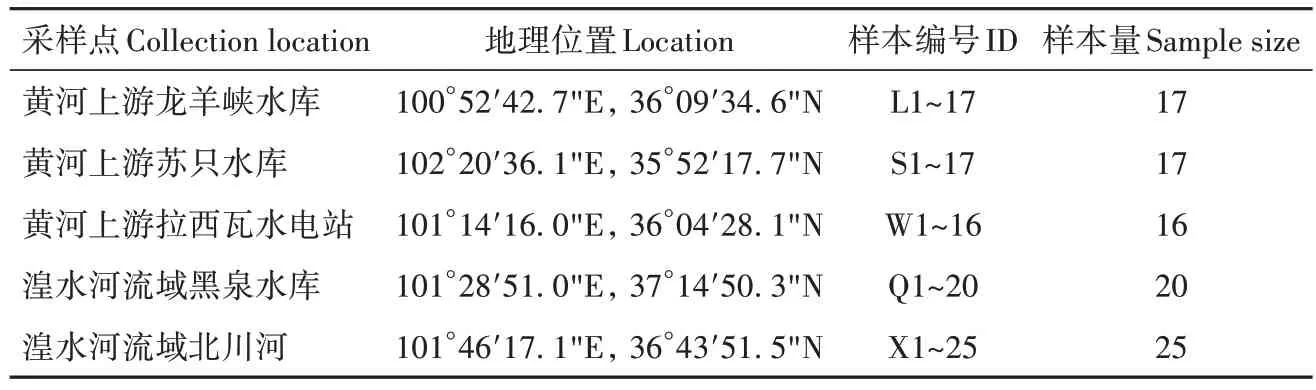
表1 样本采集信息Table 1 Sampling information of Triplophysa complex
1.2 分子生物学鉴定及系统发育分析
1.2.1 DNA 提取 取样本肌肉组织约30 mg,将组织剪碎后用ddH2O 处理为细胞悬液(处理2 次),按照动物组织基因组DNA 提取试剂盒(天根生化科技有限公司)操作说明提取样本基因组总DNA,使用NanoDrop 2000 超微量分光光度计进行DNA浓度和纯度检测,琼脂糖凝胶电泳检测DNA 的完整性,-20 ℃保存。
1.2.2cyt b基因片段扩增及测序 采用PCR 和通用引物L14724(5’-GACTTGAAAAACCACCGTT G-3’)和H15915(5’-CTCCGATCTCCGGATTACAA GAC-3’)(Xiaoet al.,2001)对mtDNAcytb基因全序列进行扩增。引物均由生工生物工程(上海)股份有限公司合成。采用20 μL 的PCR 扩增反应体系:1 μL 基因组总DNA,12.5 μL 2×Taq PCR MasterMix Ⅱ,Primer F/R 各1 μL(10 mmol·L-1),加ddH2O 至总体积20 μL;PCR 反应程序为:94 ℃4 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 1 min,30 个循环;72 ℃ 10 min。扩增产物经1%琼脂糖凝胶电泳检测,将合格的PCR 产物送生工生物工程(上海)股份有限公司双向测序。
1.2.3 数据分析 采用Clustal X(Larkinet al.,2007)和DNA STAR(Burland,2000)的Editseq 比对序列。采用DnaSP6(Rozaset al.,2003)分析单倍型数、单倍型多样性指数、核苷酸多样性指数、平均核苷酸差异数,并计算序列保守位点、变异位点和简约信息位点。采用MEGA 11(Tamuraet al.,2021)统计基因序列碱基组成、序列间的碱基变异频率和序列间的转换、颠换频率和转换颠换比。利用BLAST 在NCBI 对单倍型进行同源比对,并下载相似度高的序列:拟硬刺高原鳅(KX373844.1、MG697585.1)、硬刺高原鳅(KX373840.1)、麻尔柯河高原鳅T.markehenensis(MG725416.1)、东方高原鳅(MG725414.1)、隆头高原鳅(KX373837.1)、斯氏高原鳅(MG725382.1)、粗壮高原鳅(MG697581.1)、拟鲶高原鳅(MG697471.1、MG697474.1)和黄河高原鳅(MG697453.1)。利用PhyloSuite(Zhanget al.,2020)、IQtree v1.6.12(Nguyenet al.,2015)和MEGA 11 分别基于贝叶斯(BI)法、最大似然(ML)法和邻接(NJ)法构建系统进化树,以叶尔羌高原鳅T.yarkandensis(GenBank 登录号:KX373836.1、KT224367.1)为外群。构建BI 树时,4 条马尔科夫链同时运行1×106代,通过后验概率评估每个节点,每100代对系统树抽样;构建ML 树时,利用ModelFinder 选择最佳核苷酸替换模型(GTR+F+I),并采用启发式搜索算法;NJ 树采用K2P 双参数模型,重复取样次数为1 000。将所有单倍型提交到ABGD 网站(https://bioinfo.mnhn.fr/abi/public/abgd/abgdweb.html),选择K2P 模型,P 设为0.001~0.01,X 设为1.5,其余参数设置为默认值。最后利用MEGA 11 计算基于K2P双参数模型的遗传距离。
2 结果与分析
2.1 cyt b基因的碱基组成及序列变异分析
95 尾个体均获得cyt b基因序列全长,长度为1 140 bp,所测cytb基因序列中未发现碱基的插入或缺失。经检测,95 条cyt b基因序列隶属于46 个单倍型,所有序列均已提交至GenBank 数据库(登录号:OP616066~OP616111)。在46个单倍型的1 140 个位点中共存在317 个变异位点,约占27.8%。其中,单一突变位点50 个、简约信息位点267个(23.4%)。单倍型多样性为0.958,核苷酸多样性为0.092。cytb基因序列的A、T、G 和C碱基平均含量分别为25.9%、30.9%、16.5%和26.6%,其中A+T 含量56.8%,明显高于G+C 含量(43.1%),G 含量最低,表现出明显的反G 偏倚性,与其他硬骨鱼类cyt b基因碱基组成的基本特征一致(祁得林等,2009);且cyt b基因第一密码子的GC 平均含量为50.0%,第二和第三密码子的平均含量分别为38.6%和39.8%;转换位点和颠换位点总数分别为86 个和19 个,碱基变异均匀的分布于该序列的各个区域,但有明显的密码子偏倚,第3 密码子的转换和颠换位点最多,分别为73 个和17 个,碱基转换与颠换的比值为4.2,转换明显多于颠换。
2.2 高原鳅复合体的分子鉴定
2.2.1 基于BLAST 比对的物种鉴定 46 个单倍型与GenBank 数据库中的参考序列具有较高相似度(97.63%~100.00%),涉及高原鳅属鱼类9 个物种。15 个单倍型(H32~46)与拟硬刺高原鳅、硬刺高原鳅、麻尔柯河高原鳅3 个物种序列高度相似;8个单倍型(H23~29、31)与东方高原鳅序列高度相似;5个单倍型(H10~14)与隆头高原鳅序列高度相似;7 个单倍型(H3~9)与斯氏高原鳅序列高度相似;1个单倍型(H30)与粗壮高原鳅序列高度相似;10 个单倍型(H1~2、H15~22)与拟鲶高原鳅、黄河高原鳅序列高度相似(表2)。
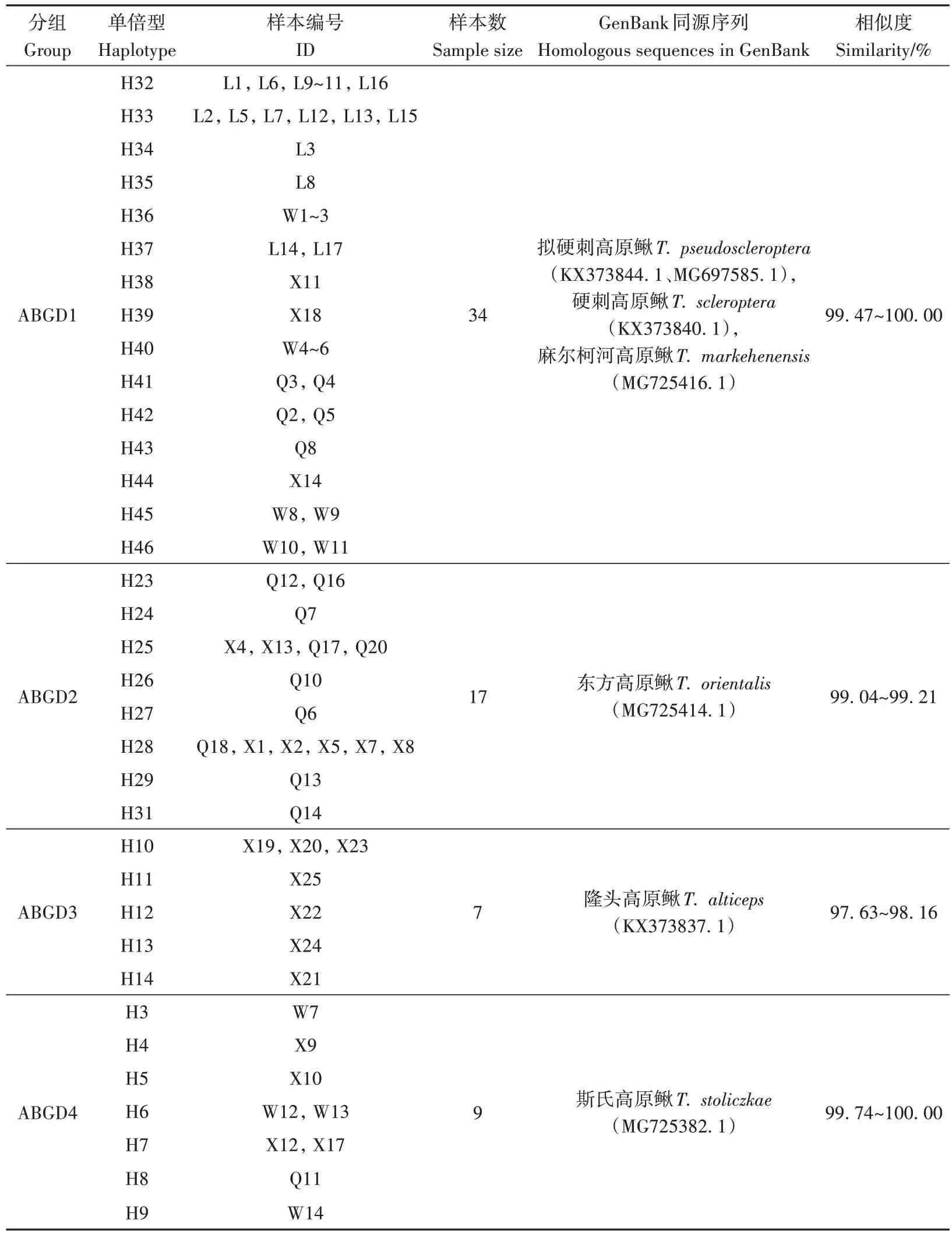
表2 高原鳅复合体cyt b基因的采样编号、单倍型及BLAST比对结果Table 2 Sample number,haplotype and BLAST results of cyt b gene of Triplophysa complex
2.2.2 基于cytb基因的系统进化树的物种鉴定以叶尔羌高原鳅的2条序列为外群,结合本研究所有单倍型序列,采用BI、ML 和NJ 法构建系统发育树,3 种系统发育分析方法得到拓扑结构高度一致的系统发育树(图1)。待鉴别的所有样本单倍型序列与其近缘种共聚为8 个明显的独立单系群(A~H),但个别聚类支如A 支和H 支的支持率不高。其中,高原鳅复合体的11 个单倍型(H36~46)与拟硬刺高原鳅和麻尔柯河高原鳅的已知序列聚为一个单系群A,4 个单倍型(H32~35)与硬刺高原鳅的序列聚为一支B,8个单倍型(H23~29、H31)与东方高原鳅已知序列组成为单系群C,5 个单倍型(H10~14)与隆头高原鳅已知序列聚为单系群D,7 个单倍型(H3~9)与斯氏高原鳅已知序列聚为单系群E,1 个单倍型(H30)与粗壮高原鳅已知序列聚为1 个单系群F,3 个单倍型(H2、H21~22)与黄河高原鳅已知序列形成1 个单系群G,其余7 个单倍型(H1、H15~H20)与拟鲶高原鳅2 条已知序列表现出高度的序列同源性,聚在同一支上(H)。
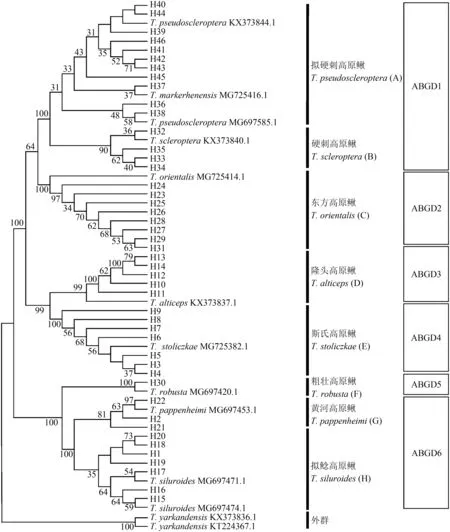
图1 利用邻接法构建的高原鳅复合体cyt b序列的系统发育树Fig.1 Neighbor-Joining phylogenetic tree based on mitochondrial cyt b sequence for Triplophysa complex
2.2.3 ABGD 划分 利用ABGD 网站对高原鳅复合体的46 个单倍型序列和GenBank 数据库下载的已知同源序列进行进一步的划分检验,结果包括原始划分和递归划分。其中,原始划分把46 个单倍型和已知同源序列划为6 组,划分结果较稳定,而递归划分把46个单倍型和已知同源序列划分为7~8 组,划分结果有波动。因此,选择较稳定的原始划分为参考,在P 为0.001~0.01 的范围内,自动检测出6 组,ABGD 划分结果与系统进化分析结果在ABGD1和ABGD6出现偏差(图2)。
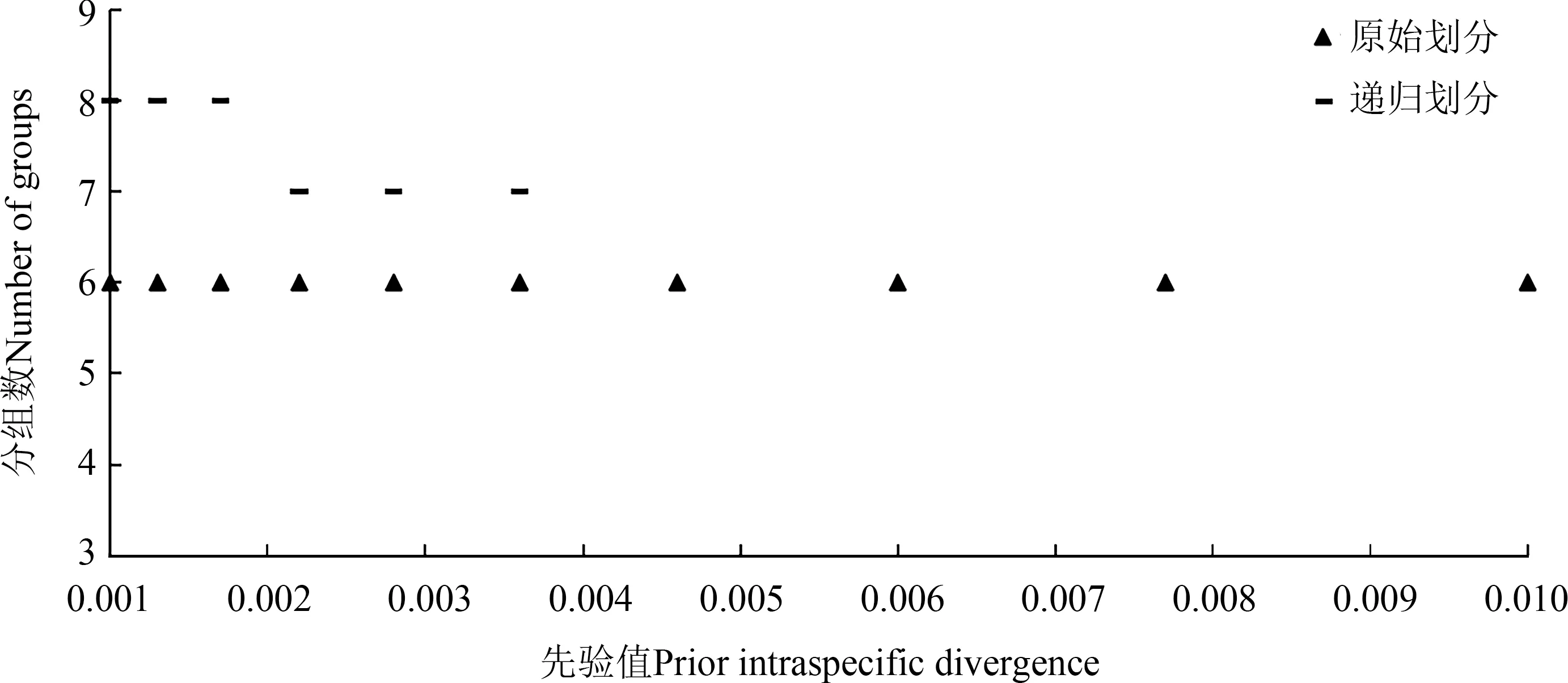
图2 ABGD软件对高原鳅复合体的划分结果Fig.2 Automatic partition results of Triplophysa complex based on ABGD
2.2.4 种间、种内遗传距离分析 采用K2P 双参数模型统计高原鳅复合体的种内、种间的遗传距离(表3):种内遗传距离为0.000 6~0.019 2(平均为0.004 4),种间遗传距离为0.005 1~0.159 8(平均为0.105 6)。系统发育树上显示的A、B 支的单倍型之间和G、H 支的单倍型之间具有较低的遗传距离(0.005 1、0.005 3),同时A 支和B 支与已知拟硬刺高原鳅、麻尔柯河高原鳅、硬刺高原鳅序列间遗传距离很低(0.002 3、0.001 8、0.004 2和0.004 8、0.003 5、0.000 9);同样,G 支和H 支与同源序列拟鲶高原鳅、黄河高原鳅也提示为种内距离标准(0.000 6、0.004 6和0.004 8、0.002 5)。

表3 高原鳅复合体基于cyt b基因序列的种内、种间遗传距离Table 3 Genetic distance within-species and between-species of Triplophysa complex based on cyt b gene sequences
3 讨论
3.1 cyt b 基因用于高原鳅属鱼类物种鉴定的有效性
mtDNA 在进化中表现为选择中性,能够解决由于趋同进化、可塑性引起的传统形态分类方法难以界定的物种鉴定(Xiaoet al.,2001;Qiet al.,2015;Fenget al.,2019)。以往动物cyt b基因研究表明,遗传距离超过6%的个体间已有明显的亚种或种的分化(杨学干等,2001;Yanget al.,2002;王义权等,2004)。裂腹鱼亚科Schizothoracinae 鱼类的研究显示,各指名种间基于cyt b基因序列的遗传距离在1.33%~9.80%,种间平均遗传距离明显低于6%,这可能与高原鱼类因青藏高原快速隆升所致的物种分化较晚有关(祁得林等,2006)。本研究中,综合BLAST 同源比对、系统发育分析和ABGD 划分结果显示,尽管利用cyt b基因无法完全厘清ABGD1组和ABGD6组的物种,但大部分物种能够准确鉴定,且被鉴定的物种间平均遗传距离(0.105 6)是种内遗传距离(0.004 4)的24 倍,符合Hebert 等(2004)提出的“10×规则”。因此,利用cyt b基因进行高原鳅属鱼类物种鉴定是可行的。
3.2 黄河上游高原鳅复合体的物种组成
种内、种间遗传距离是进行物种鉴别的主要标准(周建设等,2019)。本研究结合遗传距离结果和系统进化关系进行分析,进一步证实单系群C中的8 个单倍型(H23~29、H31)与相应已知序列(东方高原鳅)的遗传距离为0.007 3,单系群D 的5 个单倍型(H10~14)与已知序列(隆头高原鳅)的遗传距离为0.019 2,单系群E 中的7 个单倍型(H3~9)与已知序列(斯氏高原鳅)的遗传距离为0.001 3,单系群F 的1 个单倍型(H30)与已知序列(粗壮高原鳅)的遗传距离为0.000 9,均能够明显呈现出种内个体间遗传距离水平(Hellberget al.,2014),由此可确定单系支C 的8 个单倍型(H23~29、H31)隶属于东方高原鳅,单系支D 的5 个单倍型(H10~14)属于隆头高原鳅,单系支E中的7个单倍型(H3~9)为斯氏高原鳅,单系支F的1个单倍型(H30)为粗壮高原鳅。这些单系群之间的平均遗传距离为0.117 9,符合种间水平。在所采集样本中,X3、X19 和X22 接近成体发育阶段,X3 具有粗壮高原鳅的典型特征,而X19和X22具有隆头高原鳅的典型特征,这与本研究中基于cyt b基因序列的物种鉴定相符。
资料表明(武云飞,吴翠珍,1992),麻尔柯河高原鳅仅分布在大渡河流域,鉴于本次样本来自于黄河流域,因此麻尔柯河高原鳅可以从ABGD1组中排除。硬刺高原鳅和拟硬刺高原鳅在肠道和鳔后室结构上存在明显差异,且拟硬刺高原鳅的背鳍最后1 根不分枝鳍条的粗硬部分少于硬刺高原鳅(武云飞,吴翠珍,1992)。成体阶段,硬刺高原鳅因背部和体侧上部具有黑褐色细斑纹而容易区分,如本研究中L5 和L16 均为成体,被鉴定为硬刺高原鳅,但在幼鱼阶段,这2 个物种很难区分。鉴于,单系群A 的11 个单倍型(H36~46)与其聚为一个支的拟硬刺高原鳅的遗传距离(0.002 3)、以及与硬刺高原鳅的遗传距离(0.001 8)很低,加之这2个物种在黄河上游广泛分布(武云飞,吴翠珍,1992),本研究认为ABGD1 组可能是:由1 个物种组成,应将早期形态学分类中的硬刺高原鳅和拟硬刺高原鳅合并为硬刺高原鳅;由2个形态模糊种组成,但在今后的工作中需要结合分子手段和传统形态学进一步厘清分类学地位。
对于ABGD6 组而言,样本X15、X16 和W15 均为成体,具有拟鲶高原鳅头部和躯干宽阔而扁平的典型特征,7 个单倍型(H1、H15~H20)可进一步明确为拟鲶高原鳅。黄河高原鳅支中H2、H21 和H22 样品均为幼体,采样时无法从形态学鉴定物种,但这3个单倍型与黄河高原鳅和拟鲶高原鳅序列的遗传距离分别为0.000 6 和0.004 6,均显示出种内水平。黄河高原鳅头部特征与拟鲶高原鳅趋同,具有扁平的特征,但是黄河高原鳅成体躯干呈圆筒状,因此,在幼鱼至成体发育的过渡阶段两者的物种鉴定容易出现偏差。本研究认为GenBank中黄河高原鳅序列MG697453.1 可能属于这种情况,实质上应该是拟鲶高原鳅。
高原鳅属鱼类在外形特征等方面表现出相似性,尤其是在幼鱼阶段很难用传统的分类学方法进行鉴定,这可能与同域分布物种的趋同进化有关(Muschicket al.,2011;Qiet al.,2012)。无论如何,利用cyt b基因序列进行黄河上游高原鳅复合体进行分子鉴定,在一定程度上是可行的。本研究在黄河上游干流及支流湟水河流域5 个采样点共95 个样本中至少共鉴定到6 个物种,但cyt b基因作为核外遗传物质,其信息量有限,特别是在近缘物种的分类研究中要慎重对待。
