小篷竹叶绿体基因组特征及比较分析
2023-05-26伍明理晏融融代朝霞苟光前
伍明理, 晏融融, 代朝霞, 徐 雪, 苟光前
(1.贵州大学生命科学学院/农业生物工程研究院,山地植物资源保护与保护种质创新教育部重点实验室, 贵阳 550025;2.贵州大学林学院, 贵阳 550025)
小篷竹(AmpelocalamusluodianensisT. P. Yi &R. S. Wang)为禾本科(Poaceae)竹亚科(Bambusoideae)悬竹属(Ampelocalamus)植物,是1984年在贵州省罗甸县发现的一竹种,其俗名为藤竹[1]。小篷竹为贵州省特有种,主要分布于罗甸县、长顺县、册亨县、望谟县、紫云县等喀斯特(石漠化)地区,生长于海拔600~1 000 m的石灰岩裸露山地[2]。小篷竹多分布于喀斯特地区,对土层浅薄的喀斯特石山(甚至悬崖)环境具有较高的适应性和较强的抗逆性,对石灰岩有破碎化作用,可改善土壤的理化性质;此外,其复杂的地下竹鞭可有效减少水土流失,为喀斯特地区保水固土的优良物种[3-4]。小篷竹外形美观,沿岩壁下垂如簾,有很好的栽培观赏和园林绿化功能,具有一定栽培价值。小篷竹在生态、经济以及观赏方面具较高的价值,与贵州特有的爬竹、乳纹方竹、多毛箬竹一样[5-7],该物种目前种群数量锐减,野外分布受到威胁,在《中国物种红色名录》和《IUCN红色名录》中均被列为极危种[2]。
叶绿体是半自主细胞器,具有独立的遗传信息传递体系,是环状的DNA双螺旋结构,其可编码与光合作用有关蛋白[8-9]。随着高通量测序技术的发展,为叶绿体基因组的测序提供了极大的便捷,叶绿体基因组研究得以快速发展。叶绿体基因组是单性遗传,主要通过母系遗传,保守性较高[10-11]。植物的叶绿体基因组具有典型四分体结构,由一个大的单拷贝区(Large Single Copy,LSC)、一个小的单拷贝区(Small Single Copy,SSC)和两个反向重复区(Inverted Repeats,IRa和IRb)组成[10,12]。目前,叶绿体基因组广泛运用于物种鉴定、分子标记、遗传多样性分析和种质资源保护等方面,尤其在系统发育与进化方面的研究更为显著[13-14]。
本研究利用高通量测序技术对小篷竹进行测序,获得了小篷竹的叶绿体全基因组,对其进行组装和注释。随后,对叶绿体基因组(cpDNA)进行结构特征分析与系统发育研究,以期为小篷竹的遗传育种与分子进化研究提供理论依据,还可为悬竹属的系统发育与进化研究提供分子数据信息。
1 材料与方法
1.1 植物材料与DNA的提取
小篷竹鲜叶片样本于2020年10月采自贵州省罗甸县(海拔837 m,106°45′43″E,25°32′42″N),将新鲜的叶片样本放入硅胶中保存,凭证标本保存于贵州大学自然博物馆标本室(标本号:GB 303)。利用植物DNA提取试剂盒(天根生化科技有限公司)提取叶片DNA。
1.2 小篷竹cpDNA的测序、组装与注释
依托商业化平台(北京擎科生物科技有限公司)进行文库构建,在Illumin NovaSeq 6000测序仪上测序,获得质控过滤后的Clean data 4.55 Gb数据。在GetOrganelle v1.7.5软件进行组装[15],在PGA程序对组装好的序列进行注释[16],随后将注释好的序列于Genious 9.0.2软件中进行手动校正[17]。
1.3 小篷竹cpDNA的密码子偏好性、散在重复序列与SSR位点分析
使用CodonW 1.4.4软件检测小篷竹叶绿体基因组中用于蛋白质编码基因的密码子使用频率和相对密码子使用频率。
利用REPuter[18]在线软件检测散在重复序列,包括正向重复(Forward Repeats,F)、反向重复(Reverse Repeats,R)、回文重复(Palindromic Repeats,P)和互补重复(Complement Repeats,C),参数设置如下:最大重复长度(Maximum Computed Repeats)为100,最小重复长度(Minimal Repeat Size)为30,汉明距离(Hamming Distance)为3,未设置编辑距离(Edit Distance)。
简单重复序列(Simple Sequence Repeats, SSR)是利用在线软件MISA[19]进行检测,参数设置为:单核苷酸重复单元不少于10个,二核苷酸重复序列不少于5个,三核苷酸以上重复序列不少于4个,四核苷酸以上重复序列不少于3个,五核苷酸以上重复序列不少于3个,六核苷酸以上重复序列不少于3个。
1.4 小篷竹cpDNA的比较基因组学
使用IRscope工具对小篷竹和悬竹属其他11个物种的叶绿体基因组进行比较,分析叶绿体基因组结构中的IR边界变化情况。在Genious软件中利用Mauve进行比对比较。使用在线软件mVISTA对小篷竹和悬竹属其他11种的叶绿体全基因组序列进行差异性比较。
1.5 小篷竹cpDNA的系统进化分析
本研究选取禾本科叶绿体基因组序列22条,其中悬竹属11种(MH 410123、MK 393365、MK 393366、MK 393368、MK 393374、MK 393369、KX 372537、MK 393370、MK 393371、MK 393372、MK 393373)、簕竹属4种(HQ 337797、KJ 722536、FJ 970915、MH 410121)、牡竹属3种(MK 679785、NC 050753、MK 679780)、箬竹属2种(JX 513421、JX 513422)和单枝竹属1种(MK 679779),以稻属的野生稻(NC 017835)作为外类群。使用MAFFT v 7.503软件进行多序列比对,基于IQtree的最大似然法(Maximum Likelihood,ML)构建系统发育树,同时在CIPRES网站的MrBayes on XSEDE v 3.2.7 a基于贝叶斯法(Bayesian Inference,BI)构建系统发育树,ML树和BI树在TreeGraph软件进行合并。
2 结果与分析
2.1 小篷竹cpDNA结构与一般特征
小篷竹叶绿体基因组结构呈典型的四分体结构,包括大单拷贝区(Large Sinsle Copy,LSC)、小单拷贝区(Small Single Copy,SSC)和两个反向重复区(Inverte Repeat,IR)。基因组序列全长139 547 bp,GC含量为38.9%,其中LSC区长83 145 bp,GC含量为37.0%;SSC区长12 808 bp,GC含量为33.2%;IRb和IRa区长21 797 bp,GC含量均为44.2%,IR区的GC含量明显高于LSC区与SSC区(图1)。
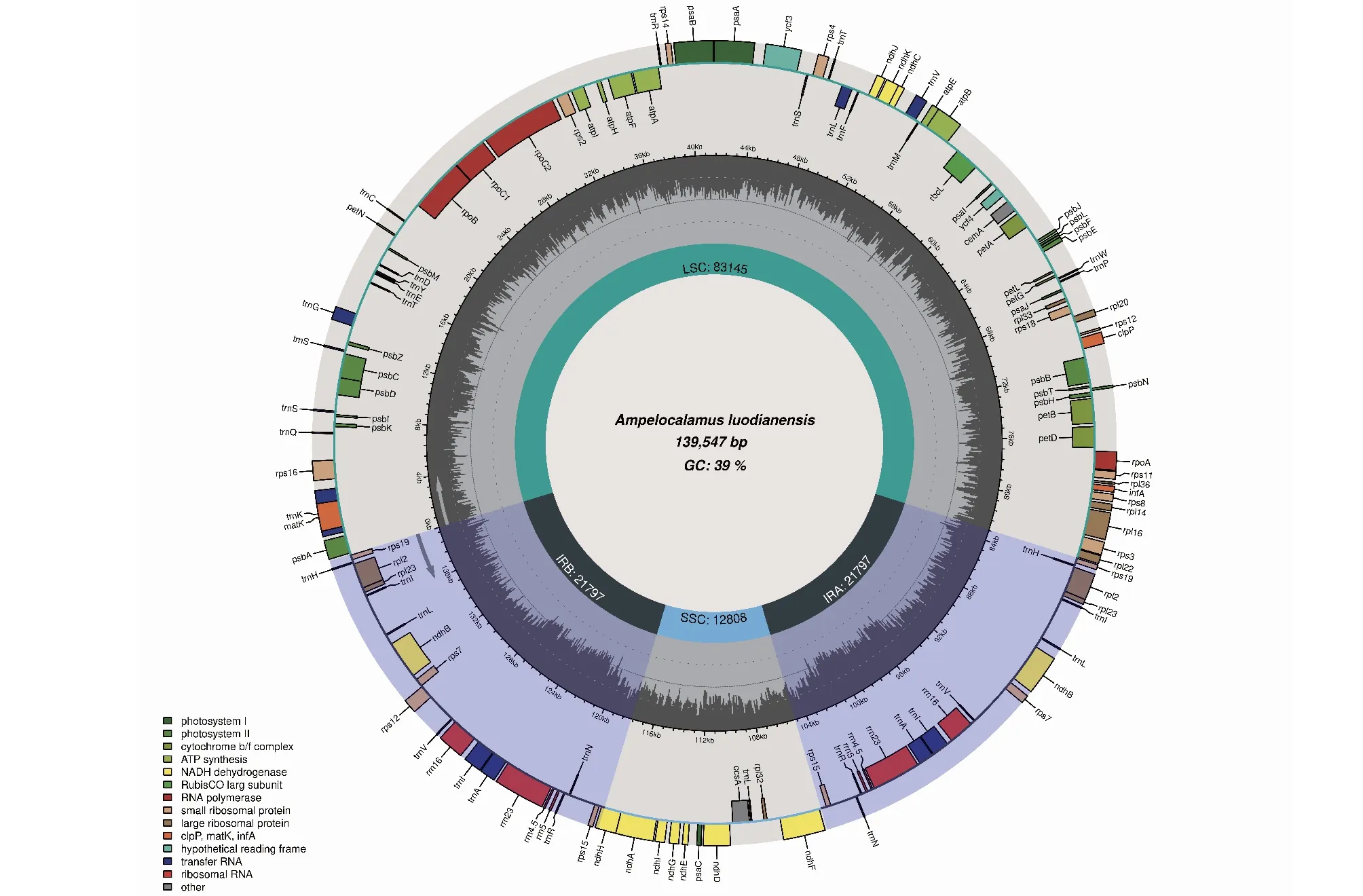
图1 小篷竹叶绿体全基因组图谱Fig.1 The whole genome map of the A. luodianensis chloroplast
对小篷竹叶绿体基因组进行注释,并对其基因信息进行统计,去掉重复基因,注释到129个独立基因,包括83个编码蛋白基因、8个rRNA基因,38个tRNA基因。其中,14个基因(ndhB、ndhA、rpl16、rpl2、petB、atpF、petD、rps16、trnK-UUU、trnI-GAU、trnA-UGC、trnG-UCC、trnV-UAC、trnL-UAA)有1个内含子,2个基因(rps12、ycf3)有2个内含子(表1)。

表1 小篷竹叶绿体基因组注释基因Table 1 Genes annotated in the chloroplast genome of A. luodianensis
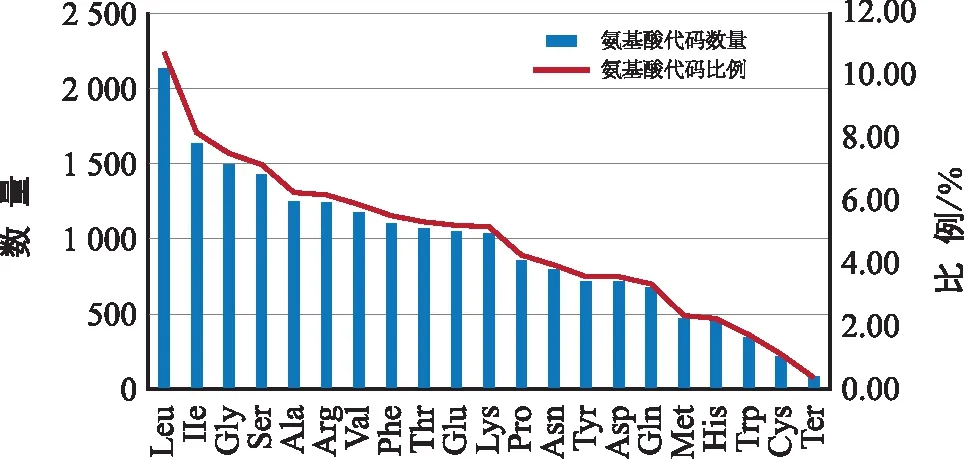
注:柱形图表示氨基酸代码的数量,红色线表示氨基酸代码的比例。图2 叶绿体基因组蛋白质编码序列中的氨基酸频率Fig.2 Amino acid frequencies in A. luodianensis chloroplast genome protein coding sequences
2.2 小篷竹cpDNA的密码子偏好性分析
小篷竹叶绿体基因组密码子使用频次统计结果显示,所有蛋白编码序列共有19 956个密码子,由64种不同类型组成,包含20种氨基酸和3种终止密码子(UUA、UAG、UGA)(表2,图2)。其中,亮氨酸(Leu)是20种氨基酸中使用频率最高的氨基酸,共有2 136个(10.70%)编码亮氨酸,半胱氨酸(Cys)使用频率最低,密码子使用数量为220个(1.10%),分别为编码最多和最少的氨基酸。甲硫氨酸(Met)和色氨酸(Trp)仅有一种类型的密码子,密码子分别为472个和345个。所有密码子中,AUU最多,UGC最少,分别为817个和49个,RSCU值分别为1.5和0.45。小篷竹叶绿体基因组的64种密码子中,RSCU(相对同义密码子)值超过1.0的有31种,除UUG(Leu)和UCC(Ser)两种密码子外,其余29种均以A/U结尾,31种密码子具有明显偏向性;而AUG(Met)和UGG(Trp)的RSCU值为1.0,这两种密码子无偏向性。
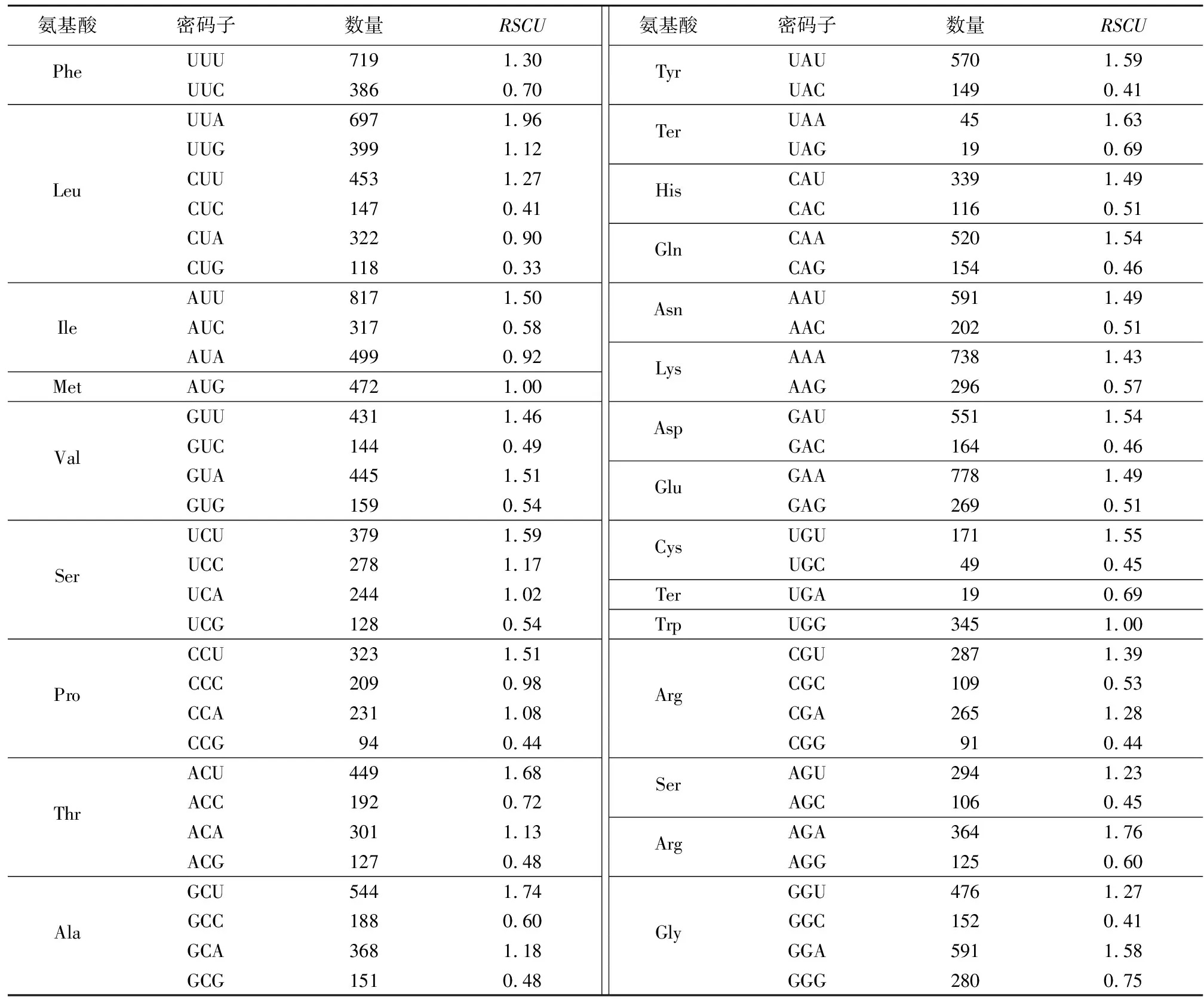
表2 小篷竹叶绿体基因组的密码子使用度Table 2 Codon usage in chloroplast genome of A. luodianensis
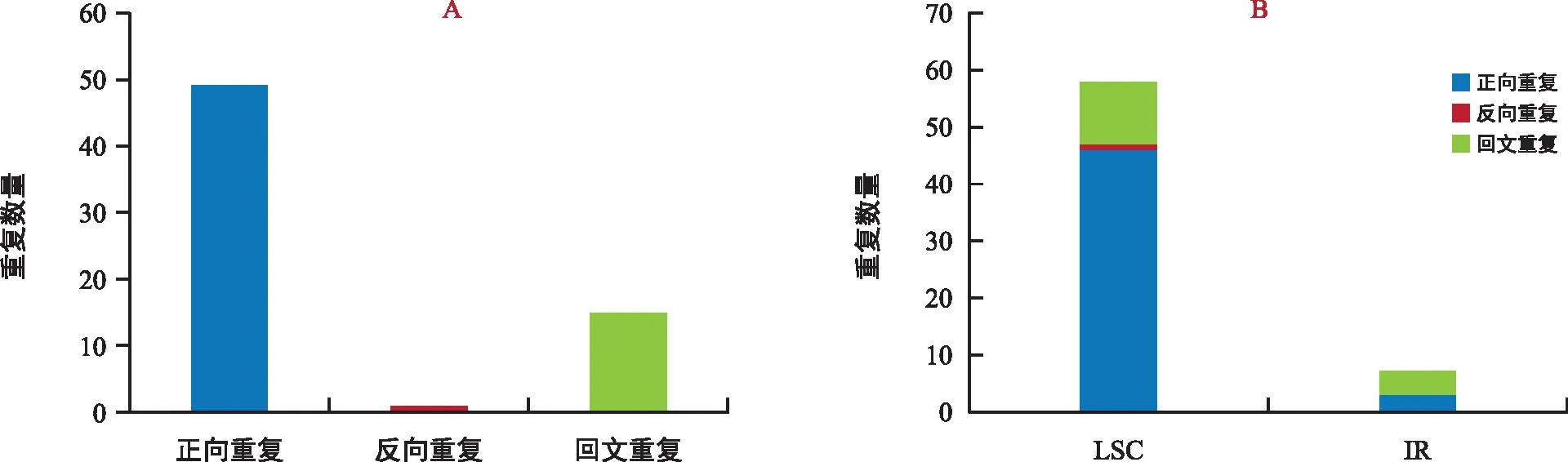
注:A为不同重复类型数量;B为不同区域的重复数量。图4 小篷竹叶绿体基因组重复序列分析Fig.4 Repeat sequences analysis on A. luodianensis chloroplast genome
2.3 小篷竹cpDNA的重复序列和SSR位点分析
对小篷竹的叶绿体基因组进行SSR位点筛选分析,共鉴定出了84个SSR位点,其中单核苷酸重复类型28个,二核苷酸重复类型4个,三核苷酸重复类型41个,四核苷酸重复类型11个(图3)。SSR位点主要分布于LSC区(64,76%),其次是分布在IR区(14,17%),SSC区最少(6,7%)。所检测到4种类型的SSR位点中,数量最多的是A/T,占比31.0%。SSR位点绝大多数位于基因间隔区(IGS)和编码区(CDS),占89.0%,而位于内含子(Intron)仅有9个,占11.0%。
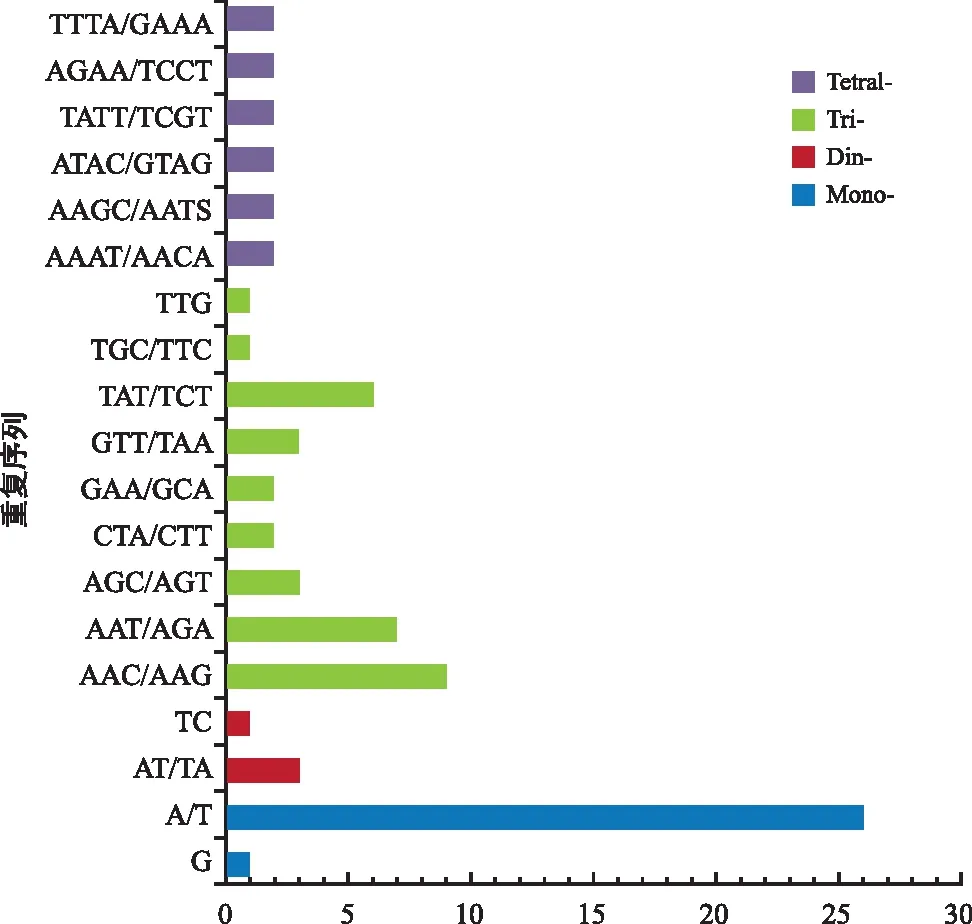
图3 小篷竹叶绿体基因组SSRs类型及数量Fig.3 The SSRs types and numbers of A. luodianensis chloroplase genome
利用REPuter软件对小篷竹叶绿体基因组进行重复序列分析。结果显示,共检测到正向重复、反向重复、回文重复3种类型,未检测出互补重复;正向重复序列有49个,反向重复序列有1个,回文重复序列有15个;重复序列长度在30~100 bp之间,重复序列全部位于LSC区和IR区(图4)。
2.4 小篷竹cpDNA的IR边界收缩与扩张分析
对悬竹属12种植物的叶绿体基因组IR边界进行比较分析。结果(图5)显示,所有类群IR大小范围在21 792~21 822 bp之间;rpl22基因LSC/IRb边界右侧,距边界24~37 bp之间;IRb/SSC边界无基因跨越;SSC/IRa边界位于ndhH基因内,基因大小为1 181 bp,间隙为187~192 bp;rps19基因较为保守,全位于IRa区域,距离IRa/LSC边界长度大小仅相差1 bp;psbA基因全位于IRa/LSC边界右侧,A.breviligulatu向IRa/LSC边界扩张,距边界27 bp。
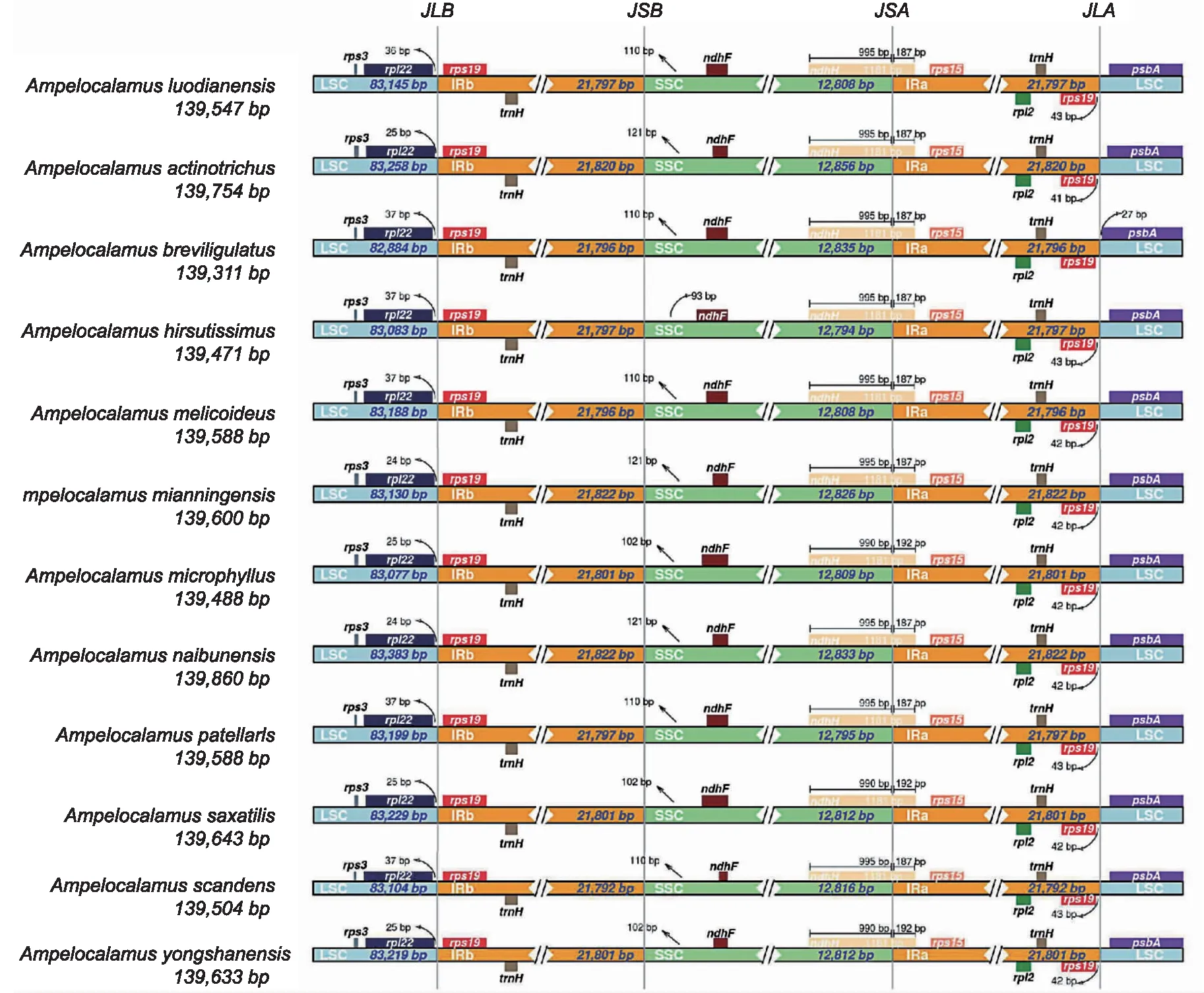
图5 悬竹属12种植物叶绿体基因组的LSC、IRs和SSC区边界的比较Fig.5 Comparison of LSC, IRs, and SSC border regions for chloroplast genome of twelve Ampelocalamus species
2.5 小篷竹cpDNA比较分析
将小篷竹与同属的钓竹(A.breviligulatus)、永善悬竹(A.yongshanensis)进行叶绿体基因组比较。
使用DnaSP 6.12软件分析悬竹属3个种的核苷酸多态性位点(图6)。结果表明,悬竹属3个种的核苷酸多态性(Pi)值在0~0.008 89之间变化,Pi平均值为0.001 04。共检测到10个高变异区(Pi>0.005),包括8个基因(trnS、trnT、trnL、rbcL、ycf4、rpl14、ndhF、rpl32)和两个基因间隔区(psbE-petL、rps15-ndhF);在这些高变区中,有7个位于LSC区,3个位于SSC区,IR区没有发现高变区;rpl14是最容易异变的区域。
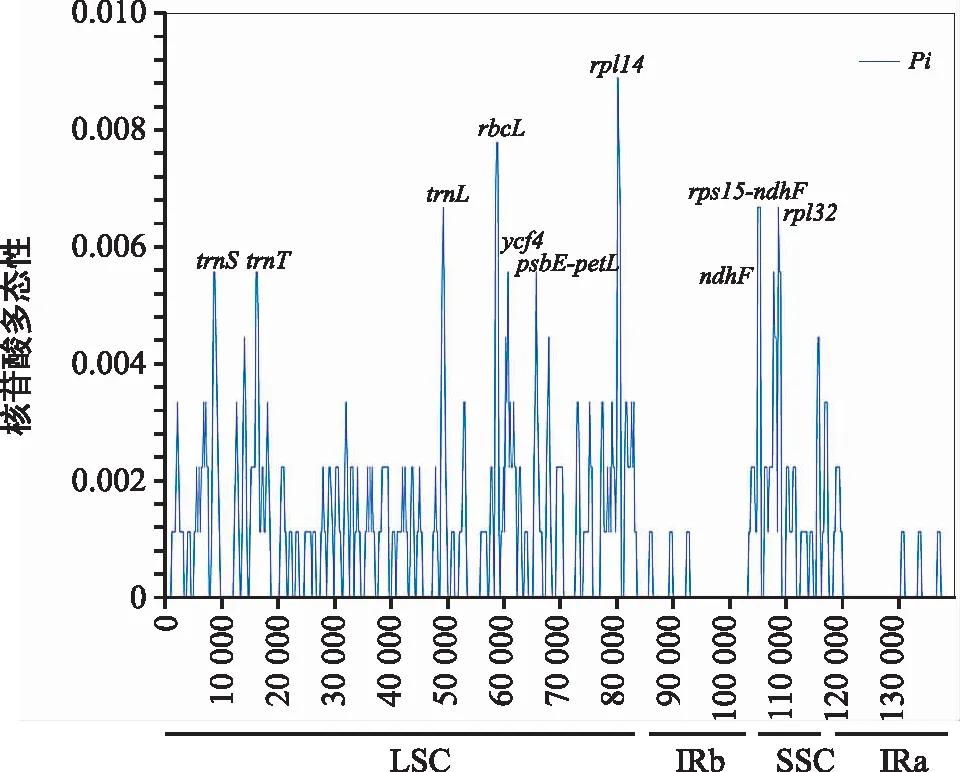
图6 悬竹属3种植物叶绿体全基因组的核苷酸多样性滑动窗图Fig.6 Sliding window plots of nucleotide diversity across the complete chloroplast genome of three Ampelocalamus species
采用Mauve对悬竹属3个竹种进行比对比较,以小篷竹为参考序列(图7)。结果表明,所有序列都呈共线性关系,未发生易位和倒位。
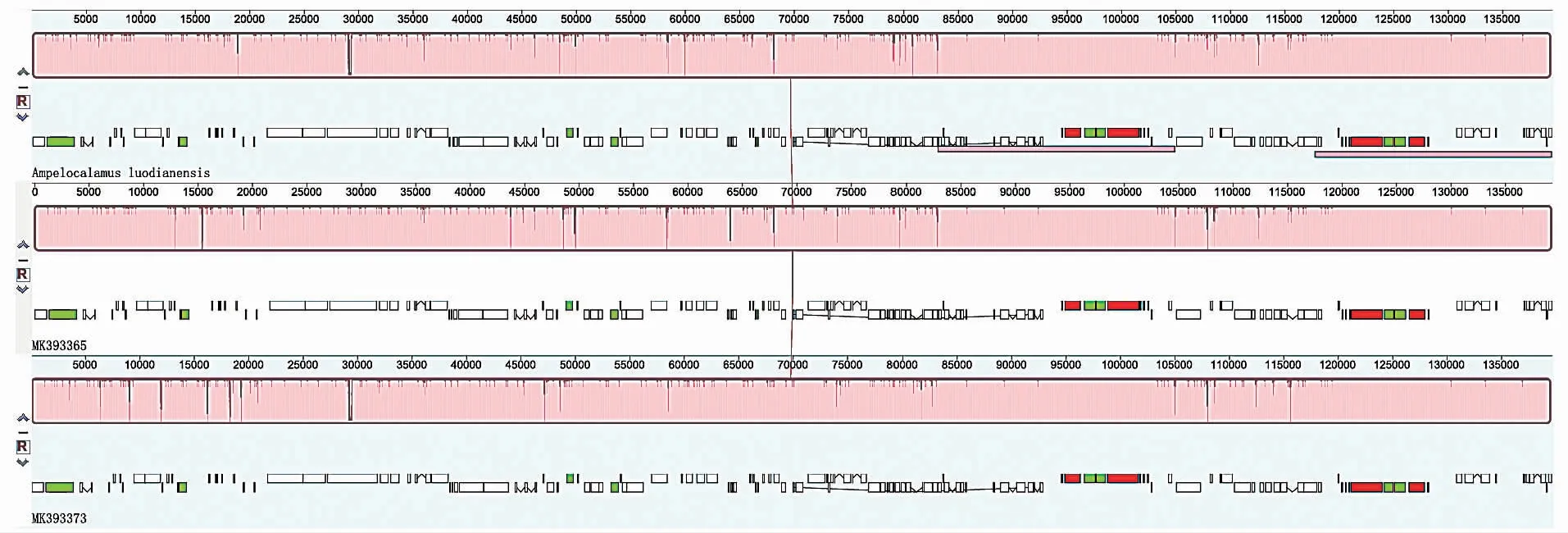
图7 悬竹属3种植物叶绿体全基因组的排列比较Fig.7 Ranging comprisons of chloroplast genom of three Ampelocalamus species
采用在线基因组比对工具mVISTA对悬竹属3个种序列进行全局比对(图8)。结果显示,3条序列叶绿体基因组具有较高的相似性,其大部分区域较为保守,IR区比LSC和SSC更为保守,非编码区变异性高于编码区,rpoC2、rps19等基因在蛋白编码区存在差异。
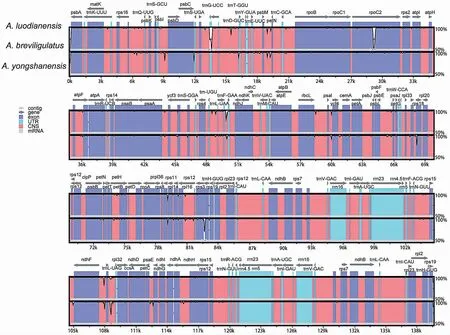
图8 悬竹属3种植物叶绿体全基因组全局比对Fig.8 Global alignment on chloroplast genomes of three Ampelocalamus species
2.6 小篷竹cpDNA系统发育分析
为确定小篷竹的系统发育位置,从NCBI获取禾本科6属22种的完整叶绿体基因组序列,以稻亚科稻属的野生稻作为外类群,采用最大似然法(ML)和贝叶斯法(BI)对其叶绿体基因组进行了系统发育分析(图9)。ML和BI建树所得系统树的拓扑结构一致,故将两者合并,同时标注支持率。系统发育结果显示,小篷竹与钓竹(A.breviligulatus)和南川竹(A.melicoideus)的亲缘关系最近(支持率均为100%),互为姐妹类群。
3 讨 论
本研究对小篷竹的叶绿体基因组进行测序、组装和注释,并进行一系列的结构特征分析。结果表明,小篷竹叶绿体基因组为一环状DNA分子,呈典型的四分体结构,由一个大单拷贝区(LSC)、一个小单拷贝区(SSC)和两个反向重复区(IRb和IRa)组成,与大多数被子植物的叶绿体基因组结构相似[20]。小篷竹叶绿体基因组全长139 547 bp,其GC含量为38.9%,AT含量为61.1%,共编码129个基因,其16个编码蛋白基因含有内含子,与爬竹(A.scandens)叶绿体基因组结构相似[21]。分析编码区密码子的使用偏好性,对研究物种的基因功能和系统进化具有重要意义[22]。小篷竹叶绿体基因组的31种密码子具有明显的偏向性,大部分以A/U结尾,这与大多数被子植物密码子使用偏好类似。
叶绿体基因组的SSR是一种高效标记工具,广泛应用于物种鉴定、属间分类、群体遗传以及系统发育关系等方面的研究[23]。在小篷竹叶绿体基因组中检测到84个SSR,包含4种重复类型,主要由A/T组成,大部分位于LSC区。重复序列主要来源于叶绿体基因组中的重复、缺失和重排,分析重复序列对研究基因组的重组和重排具有重要意义[24-25]。在小篷竹中叶绿体基因组中,共鉴定出65个重复序列,最常见的是正向重复(49个),重复序列长度在30~100 bp之间。小篷竹的重复序列与毛环方竹类似[26]。将小篷竹与同属的钓竹(A.breviligulatus)、永善悬竹(A.yongshanensis)进行比较,核苷酸多态性位点结果显示,总检测到10个高变异区(Pi>0.005),rpl14是最容易异变的区域。这些高变区的序列在其他种也有报道,且在IR区没有发现高变异区,是高度保守的区域[27-28]。mVISTA全局比对结果也表明,IR区比LSC和SSC更为保守。
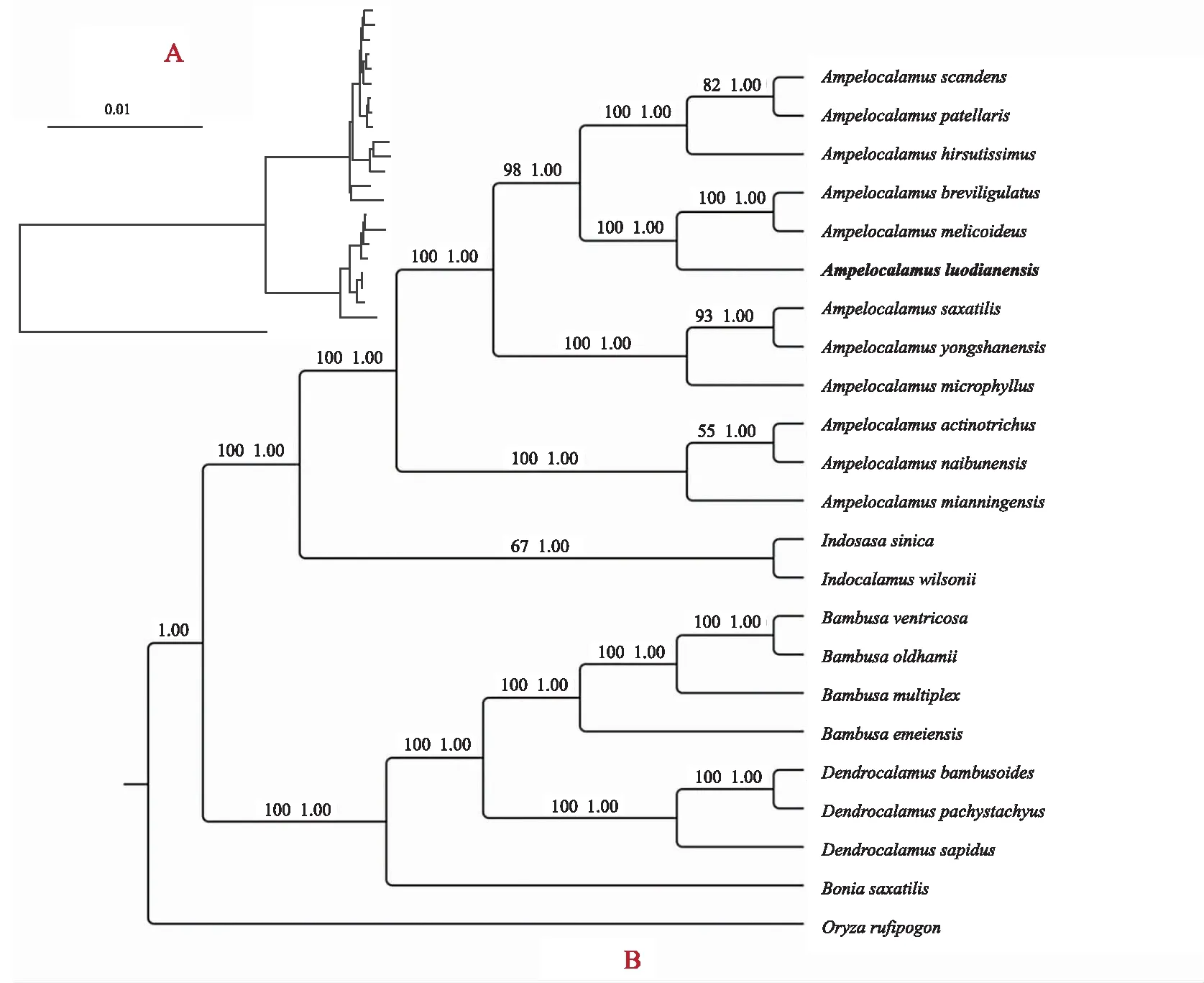
注:A为具有时间标尺的系统发育树;B为无时间标尺的系统发育树。图9 基于叶绿体全基因组用ML和BI构建的系统进化树Fig.9 Phylogenetic tree inferred from maximum likelihood and bayesian inference based on complete chloroplast genome
目前,依据形态特征分类,小篷竹的系统位置有争议[29-30]。已有研究表明,叶绿体全基因组比叶绿体片段构建的系统发育关系较好[27],本研究使用ML和BI法对叶绿体全基因组进行建树。结果显示,小篷竹与钓竹(A.breviligulatus)和南川竹(A.melicoideus)的亲缘关系最近,互为姐妹类群。此外,悬竹属与箬竹属互为姐妹类群,表明两者的关系较近,这与Liu等[22]和Fan等[8]的结果一致。
本研究通过对小篷竹叶绿体进行测序,并对其叶绿体基因组进行密码子偏好性、长重复序列、SSR位点、IR边界收缩与扩张、核苷酸多态性位点、系统发育的分析。研究结果可为小篷竹种质资源保护、分子标记和物种进化等提供基础资料,还对石漠化地区植物遗传多样性研究具有重要意义。
