葫芦科作物抗氧化基因的全基因组鉴定及分析
2023-05-23钱玉磊闫晋强刘文睿吴智明谢大森江彪
钱玉磊 闫晋强 刘文睿 吴智明 谢大森 江彪
摘要:抗氧化(oxidation resistance,OXR)基因是含有TLDc结构域的一类基因,能有效清除活性氧,在抗氧化过程中发挥着重要作用。基于葫芦科基因组数据库,从6个葫芦科作物中共鉴定出37个OXR成员,其中冬瓜、西瓜、甜瓜、葫芦和黄瓜均有6个成员,南瓜有7个成员。通过系统发育以及保守结构域分析,将这些OXR成员分为 5个亚族:OXR1、OXR2、OXR3、OXR4和OXR5。OXR基因在染色体上呈不均匀分布,同一亚族OXR成员所编码的氨基酸数、分子量、外显子数、内含子数以及保守基序的排列模式都极为相似,表明OXR基因具有高度保守性。顺式作用元件分析结果显示,OXR基因的启动子区域含有多种顺式作用元件以及MYB转录因子的结合位点。基因复制分析发现,葫芦科OXR2成员通过片段复制或全基因组复制发生了一定扩张,这些复制基因对均受到纯化选择。共线性分析结果显示,在亲缘关系较近的葫芦科作物之间,OXR基因所在的共线性区块较大。不同组织的表达谱分析结果显示,葫芦科OXR4成员有相似的表达模式,在叶片中表达量均较高;OXR2亚族内复制基因出现了非对称表达的现象。
关键词:葫芦科;OXR基因;保守结构域;顺式作用元件;生物信息学;表达谱;亲缘关系
中图分类号:S188;S642.01 文献标志码:A
文章编号:1002-1302(2023)08-0051-10
基金项目:国家自然科学基金(编号:31972403、32102406);广东省农业科学院中青年学科带头人项目(编号:R2020PY-JG003);广东省农业科学院优秀博士项目(编号:R2021YJ-YB2004)。
作者简介:钱玉磊(1997—),男,河南郑州人,硕士研究生,研究方向为蔬菜分子生物学。E-mail:qylei2022@163.com。
通信作者:江 彪,博士,研究员,研究方向为蔬菜遗传育种与分子生物学。E-mail:jiangbiao@gdaas.cn。
植物受到胁迫刺激时,细胞内的电子传递和氧化还原的动态平衡被破坏,导致细胞内的氧由氧化态转变为还原态,从而产生过量的活性氧,造成蛋白质、膜脂、DNA以及其他细胞组分严重损伤[1]。然而,活性氧也有对植物有利的一面,其可作为一种信号分子,协调细胞内许多重要的信號传导,如蛋白质的修饰、基因表达和激素调节、协调适应或传递防御反应等[2-4]。植物体内具有复杂的活性氧清除系统,主要包括保护酶系和抗氧化物质,使体内活性氧产生与降解处于动态平衡,不对自身造成损伤[5]。
抗氧化(oxidation resistance,简称OXR)基因所编码的蛋白质含有保守结构域TLDc[TBC (Tre2/Bub2/Cdc16),LysM (lysine motif),domain catalytic],可以影响氧自由基清除基因的表达,从而降低细胞中活性氧水平[6]。近年来,在拟南芥基因组中已鉴定出6个具有TLDc保守结构域的OXR基因家族成员,其中AtOXR2是一种线粒体蛋白,过表达AtOXR2可以提高拟南芥的光合作用效率,增强对氧化胁迫的耐受能力[7]。在蔬菜作物中,张梦云等基于十字花科基因组数据库,对7种十字花科植物基因组中的抗氧化基因进行了鉴定和生物信息学分析[8]。
葫芦科是世界上最重要的植物科之一,包含许多常见的蔬菜和瓜果,具有重要的经济价值。随着基因组测序技术的发展,黄瓜(Cucumis sativus)[9]、甜瓜(C. melo)[10]、西瓜(Citrullus lanatus)[11]、葫芦(Lagenaria siceraria)[12]、南瓜(Cucurbita moschata)[13]、冬瓜(Benincasa hispida)[14]等葫芦科作物先后完成了全基因组测序,为系统研究葫芦科作物的抗氧化基因家族奠定基础。本研究通过系统鉴定6个葫芦科作物抗氧化基因家族,并开展生物信息学分析和不同组织的表达谱分析,为深入阐明葫芦科作物OXR家族的基因功能奠定理论基础。
1 材料与方法
1.1 葫芦科OXR成员的鉴定
从葫芦科基因组数据库(http://cucurbitgenomics.org/)下载黄瓜、甜瓜、西瓜、葫芦、南瓜和冬瓜的基因组信息文件,Pfam网站(https://pfam.xfam.org/)下载TLDc保守结构域的隐马尔可夫模型(PF07534),使用hmmsearch指令,搜索阈值<0.000 01,在葫芦科6种作物的蛋白序列文件中进行搜索。以已报道的6个AtOXR基因家族成员的蛋白质序列作为查询序列[7],与葫芦科作物的所有蛋白质序列进行BLASTP(检索阈值<0.000 01)。将上述2种方法所鉴定到的候选葫芦科OXR成员的蛋白序列提交至NCBI、Pfam和SMART数据库,查询结构域特征,最后下载结果文件,并人为鉴定TLDc结构域是否存在,最终确定葫芦科OXR成员。
1.2 系统发育分析、保守结构域分析和物理化学性质分析
利用MEGA11中的ClustalW(默认设置)比对鉴定出的37个葫芦科OXR成员和6个AtOXR成员的蛋白质序列,构建Neighbor-Joining树,使用 1 000 个重复进行 bootstrap分析。根据拟南芥的分类方法将葫芦科OXR成员分类[7],利用Evolview v3将分类结果及每个OXR成员的蛋白序列和TLDc 结构域所在位置可视化[15],利用在线工具ProtParam(https://web.expasy.org/protparam/)预测OXR成员的分子量(MW)和等电点(PI)。
1.3 葫芦科OXR成员的染色体定位
根据相应的基因组注释文件,提取37个OXR成员的基因位置信息,然后使用MG2C(http://mg2c.iask.in/mg2c_v2.1/)将其定位到相应的染色体上。
1.4 保守基序和基因结构分析
从基因组注释文件中获得OXR基因的外显子和内含子位置信息,将葫芦科OXR成员和拟南芥OXR成员的蛋白质序列提交MEME在线网站(https://meme-suite.org/meme/doc/meme.html),最大基序(motif)检索数设置为15,利用CFVisual(https://github.com/ChenHuilong1223/CFVisual)将结果可视化。
1.5 启动子分析
提取每个OXR成员起始密码子前2 500 bp序列,提交PlantCare网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),获得顺式作用元件信息,然后以 R 4.1.3的tidyverse和ggplot2函数绘制启动子热图。
1.6 复制基因分析及选择压力分析
使用MCScanX[16]中的detect_collinearity_within_gene_families.pl分析6个葫芦科物种内的OXR复制基因对以及非OXR复制基因对,然后提取所有复制基因对在基因组中的位置信息,并利用Circos软件[17]将复制基因对可视化。
利用Muscle软件分别比对复制基因对的氨基酸序列,然后用ParaAT[18]将氨基酸序列比对引导对应的编码序列比对,进一步使用KaKs_Kaculator 2.0[19]计算复制基因对的Ka(非同义替换率)/Ks(同义替换率),通过Ka/Ks的值来判断基因在进化过程中受到的选择压力。
1.7 共线性分析
利用葫芦科作物的基因组文件及基因组注释文件,使用JCVI[20]进行6个葫芦科物种间的OXR基因共线性分析。
1.8 葫芦科OXR成员在不同器官组织的表达谱分析
从葫芦科基因组数据库下载冬瓜、南瓜(PRJNA385310)、甜瓜(PRJNA383830)、黄瓜(PRJNA80169)和葫芦(PRJNA387615)的不同组织(根、茎、叶、花)的表达谱数据,分别以OXR1在根中的FPKM(fragments per kilobase of exon model per million mapped fragments)值为基准,计算OXR成员在根、茎、叶、花的相对表达量,使用TBtools中的HeatMap[21]繪制热图,将结果可视化。
2 结果与分析
2.1 葫芦科OXR基因家族成员的鉴定
根据TLDc的隐马尔可夫模型(PF07534),使用hmmsearch指令在葫芦科6种作物全基因组中共搜索到37个候选OXR成员基因。同时,利用同源BLAST方法鉴定到38个候选OXR成员基因。随后,将2种方法鉴定到的候选成员的蛋白质序列提交数据库进行确认,结果表明,hmmsearch指令鉴定到的成员都含有TLDc保守结构域,同源BLAST检测出的38个成员包含hmmsearch指令鉴定到的37个成员,另外1个候选OXR成员不含有TLDc保守结构域,因此将其去除。最终,在葫芦科6种作物中共鉴定出37个OXR成员,其中冬瓜、黄瓜、西瓜、甜瓜和葫芦均有6个成员,南瓜有7个成员。
2.2 系统发育、保守结构域和物理化学性质分析
以葫芦科37个OXR成员和拟南芥6个OXR成员的蛋白质序列构建系统发育树,可将葫芦科OXR基因家族分为5个OXR亚家族,分别为OXR1亚族、OXR2亚族、OXR3亚族(AtOXR6归入OXR3亚族)、OXR4亚族、OXR5亚族。葫芦科6个作物在OXR1亚族、OXR3亚族、OXR4亚族和OXR5亚族均仅有1个成员。在OXR2亚族中,拟南芥仅有1个成员,而冬瓜、黄瓜、西瓜、甜瓜和葫芦有2个成员,南瓜有3个成员。因此,相较于拟南芥,葫芦科OXR2成员在进化史中有一定的扩张。
根据葫芦科OXR成员所在亚家族,按照“物种拉丁名缩写+亚家族+小写字母”的形式命名。同一亚族OXR成员的TLDc结构域的长度和位置相似,不同亚族的OXR成员的TLDc结构域的位置和长度有差别(图1),如OXR4亚族的TLDc结构域的长度短于其他OXR族,且位于蛋白质的中部,并不靠近C端。
理化性质分析结果(表1)表明,葫芦科作物OXR家族成员的氨基酸数量在232~1 059之间,等电点在4.65~7.07之间。分子量最小的是LsiOXR4,仅有25.64 ku,其开放阅读框长度为 699 bp,编码232个氨基酸;而分子量最大的CmaOXR4,有118.62 ku,其开放阅读框长度达到 3 180 bp,编码1 059个氨基酸。通过对比葫芦科和拟南芥的OXR基因家族成员的氨基酸数发现,同一亚家族内,除南瓜CmaOXR1和CmaOXR4外,其他葫芦科OXR成员和拟南芥OXR成员的氨基酸数量相似。
2.3 染色体定位
将37个葫芦科OXR基因家族成员定位到相应的染色体上,结果显示OXR基因在染色体上呈不均匀分布。南瓜OXR基因分布于6条染色体上,葫芦OXR基因分布于5条染色体上,冬瓜和甜瓜分布于4条染色体上,黄瓜和西瓜OXR基因位于3条染色体上。不同染色体上分布的OXR基因数量不同,最多有4个(黄瓜3号染色体)(图2)。
2.4 基因结构和保守基序分析
为了研究葫芦科作物OXR基因家族成员的进化关系,对37个葫芦科OXR成员和6个拟南芥OXR成员的外显子和内含子的数量及排列模式进行了分析。结果表明,位于不同亚家族的所有OXR成员的外显子和内含子的数量以及排列模式差异显著,而同一亚族的葫芦科OXR成员的内含子-外显子排列模式基本一致(图3)。
根据MEME网站分析,在葫芦科OXR成员和拟南芥OXR成员中共检测到15个保守基序,每个OXR成员大致含有5~9个不等的保守基序,同一个亚族内的OXR成员所含的保守基序的数量和保守基序的分布模式大致相同,推测同亚族的OXR可能发挥相似的功能。不同亚族的OXR成员的保守基序的分布模式有所不同,可能是造成其功能差异的主要原因。
2.5 启动子分析
提取葫芦科OXR基因起始密码子前2 500 bp序列,提交至Plantcare网站,预测其顺式作用元件(图4)。共发现49种顺式作用元件,主要分为4类:调控生长发育、光反应、激素响应和逆境胁迫相关。其中调控生长发育的元件包括对栅栏叶肉细胞分化、胚乳表达、昼夜节律、分生组织表达等调控元件,而在BhiOXR2a和CmeOXR2a中还发现有参与类黄酮生物合成的MYB结合位点。光反应元件包含24个不同的元件,说明葫芦科OXR基因家族可能广泛参与了光调控途径。多个葫芦科OXR基因中有许多与脱落酸、赤霉素、水杨酸和生长素等植物激素响应相关的顺式作用元件。另外,OXR3亚族成员都具有参与脱落酸响应的ABRE顺式作用元件,数量1~4个不等,推测葫芦科OXR3可能参与脱落酸的响应。逆境胁迫相关元件主要包含多种非生物胁迫响应元件(干旱、低温、缺氧和创伤响应元件)以及参与干旱诱导的MYB结合位点。
2.6 复制分析及基因选择压力分析
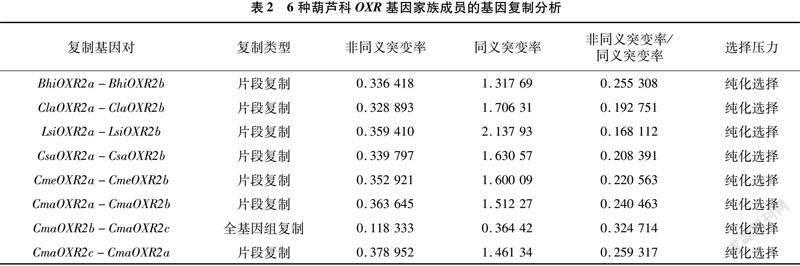
6个葫芦科作物物种内的共线性分析发现,在葫芦科OXR基因家族中,只有OXR2亚族发生了复制(图5)。其中,南瓜OXR2复制基因对较多,而冬瓜、黄瓜、甜瓜、西瓜和葫芦由于近期没有发生全基因组复制事件,其复制基因对较少。
通过葫芦科OXR2复制基因对的Ka/Ks分析,发现Ka/Ks的值均小于1,表明这些复制基因对受到纯化选择(表2)。在同义突变率Ks分析中,CmaOXR2b-CmaOXR2c复制基因对的Ks值为0.364 422,南瓜近期发生特异的全基因组复制事件(specific whole-genome duplication,sWGD)的Ks值为0.32[13],二者相差不大。此外,CmaOXR2b-CmaOXR2c共线对所在的共线区块所含的基因对数(183对)远多于CmaOXR2a-CmaOXR2c共线对所在的共线块所含的基因对数(72对),证实CmaOXR2b-CmaOXR2c是由南瓜的近期发生sWGD而产生的。
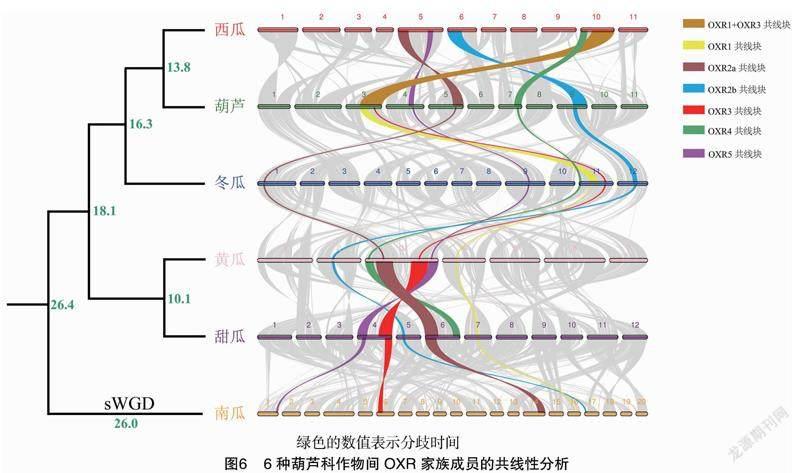
2.7 共线性分析
为研究OXR基因家族在葫芦科内的进化历史,根据之前报道的物种分化树将葫芦科作物两两开展共线性分析。结果发现 亲缘关系越近 OXR所在的共线性区块越大,如黄瓜和甜瓜、西瓜和葫芦。ClaOXR1和ClaOXR3位于西瓜的10号染色体上,LsiOXR1和LsiOXR3位于葫芦的3号染色体上,都在同一个较大的共线块(OXR1+OXR3 block)中。此外,葫芦科不同作物同一亚族成员间存在一定的共线性。不同作物OXR2a之间、不同作物OXR2b之间存在共线性,不同作物OXR2a和OXR2b间不存在共线性,说明在葫芦科祖先物种中可能发生了古老的复制事件,产生了OXR2a和OXR2b(图6)。
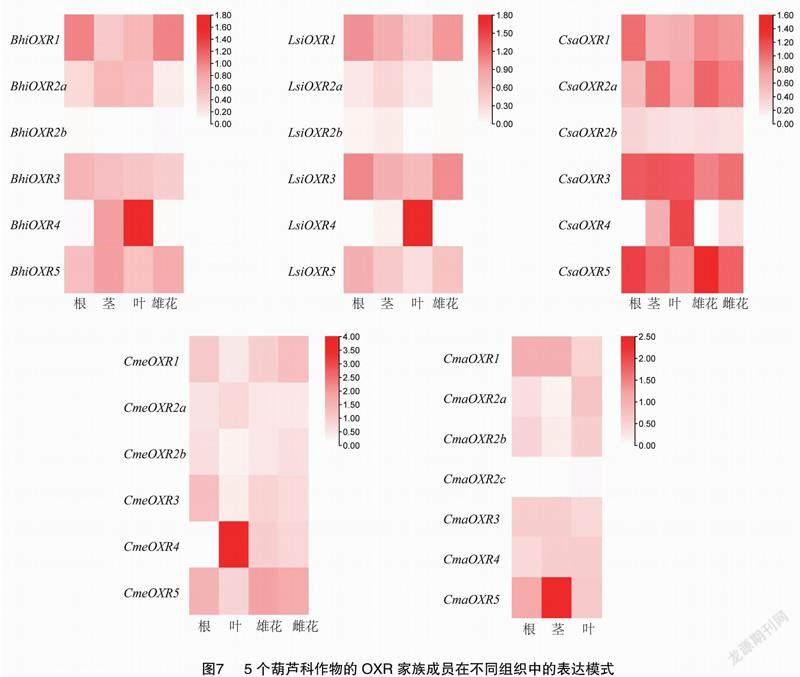
2.8 葫芦科OXR基因家族的组织表达谱分析
基于葫芦科基因组数据库中已发表的表达谱数据,分析OXR基因在不同组织中的表达模式。结果(图7)显示,不同亚族基因的表达模式有所差异,如冬瓜BhiOXR3在根、茎、叶、花中表达量无明显差异,而BhiOXR4在叶片中表达量明显高于其他组织。此外,不同作物相同亚族基因表达也存在一定的差异,冬瓜、葫芦、甜瓜、黄瓜OXR4基因在叶片中表达量明显高于其他组织,而南瓜OXR4基因则无明显的叶片高丰度表达。值得注意的是,复制基因对(BhiOXR2a-BhiOXR2b、LsiOXR2a-LsiOXR2b、CsaOXR2a-CsaOXR2b、CmaOXR2b-CmaOXR2c)在不同组织中的表达存在较大差异,出现表达分化现象。
3 讨论与结论
抗氧化(OXR)基因有助于保护真核生物免受活性氧的伤害[22]。目前,OXR基因家族已在拟南芥、小麦、向日葵和某些十字花科植物的基因组中被鉴定出来,并深入研究了部分基因的功能[7-8,23-25]。
然而,关于葫芦科OXR家族基因的研究还未见报道。本研究基于葫芦科基因组数据库,从6个葫芦科作物中共鉴定出37个OXR基因,其中冬瓜、黄瓜、西瓜、甜瓜和葫芦均有6个OXR基因,而南瓜有7个OXR基因。
根据氨基酸序列和保守结构域的相似性,葫芦科OXR基因家族可分为5个亚家族,这一结果同拟南芥、向日葵和十字花科植物的分类结果相似[7-8,25]。同一亚家族的拟南芥 OXR基因和葫芦科OXR基因编码的氨基酸个数、开放阅读框和分子量的大小都相似,其内含子-外显子的数量及保守基序相似,说明同一亚家族内的OXR基因可能具有相似的生物学功能。
OXR基因的啟动子含有多种顺式作用元件,表明OXR基因可能参与多种植物代谢调控途径。过表达拟南芥OXR基因,可通过影响水杨酸代谢而清除多余的活性氧,从而能够提高对氧化胁迫的耐受性[7,24]。葫芦科OXR基因的启动子区域包含调控生长发育、光反应、激素响应和非生物胁迫响应相关元件,这一结果与之前报道的十字花科植物OXR基因和小麦OXR基因的顺式作用元件[8,23]相似,说明葫芦科OXR基因可能在抵御生物和非生物胁迫时发挥重要作用。
基因复制是植物基因组进化的主要驱动力,为遗传革新、表型多样性以及物种分化提供了大量的原始遗传物质[26-27]。葫芦科OXR2亚族成员通过片段复制或全基因组复制发生了扩张,并且OXR2复制基因对的Ka/Ks 比值均小于 1。Ka/Ks<1 表示基因受到纯化选择[28],说明葫芦科OXR2亚族在进化上相对保守,有助于维持OXR2亚族成员功能的稳定性。通过探寻OXR基因家族在葫芦科内的进化历史,发现 6个葫芦科作物的OXR2a和OXR2b相互之间都具有共线性关系,推测OXR2亚族的成员在这6个葫芦科作物的祖先物种中通过古老的复制事件,产生了OXR2a和OXR2b等2个同源基因对,伴随着葫芦科物种的分化,OXR2a和OXR2b也被保留在了6个葫芦科物种的基因组中。南瓜基因组中有3个OXR2基因,其中CmaOXR2b和CmaOXR2c是同源基因,这一发现进一步证实南瓜近期发生了sWGD事件[13]。
为了研究OXR基因的表达特征,本研究从葫芦科基因组数据库下载冬瓜、南瓜、甜瓜、黄瓜和葫芦不同组织的表达谱数据。表达谱分析发现不同亚族OXR基因的表达量有差异,表明它们在不同组织器官中的抗氧化作用机制不尽相同。葫芦科OXR4亚族的基因的表达模式较为相似,暗示OXR4亚族的基因可能具有相似的抗氧化功能。乔鑫通过对梨等141种植物基因组中的不同类型的复制基因的鉴定和表达分析,发现复制基因广泛存在非对称表达现象(2个同源基因拷贝的表达水平己经偏离了1 ∶1的对称性表达模式)[27],然而葫芦科OXR2亚族内复制基因存在显著的表达差异,也出现了非对称表达的现象。这可能是由于基因复制发生后,2个同源基因拷贝在启动子区域的分化导致它们之间的表达分化[29],但其表达产生分化的机制还需进一步解析。
综上所述,本研究利用多种生物信息学分析网站和软件,系统鉴定了6种葫芦科作物的OXR基因,并综合分析了葫芦科OXR基因的系统发育关系、物理化学性质、染色体定位、基因结构、保守基序、顺式作用元件、在葫芦科作物中的进化历史和在不同组织中表达模式等。鉴定和分析结果为进一步研究葫芦科作物OXR基因家族在响应非生物胁迫方面的生物学功能奠定基础。
参考文献:
[1]李 格,孟小庆,蔡 敬,等. 活性氧在植物非生物胁迫响应中功能的研究进展[J]. 植物生理学报,2018,54(6):951-959.
[2]Czarnocka W,Karpiński S.Friend or foe?Reactive oxygen species production,scavenging and signaling in plant response to environmental stresses[J]. Free Radical Biology and Medicine,2018,122:4-20.
[3]Foyer C H,Ruban A V,Graham N. Viewing oxidative stress through the lens of oxidative signalling rather than damage[J]. The Biochemical Journal,2017,474(6):877-883.
[4]Mittler R. ROS are good[J]. Trends in Plant Science,2017,22(1):11-19.
[5]赵 滢,王振兴,许培磊,等. 山葡萄双丰和左优红叶绿素荧光特性及活性氧代谢与低温伤害的关系[J]. 园艺学报,2018,45(4):650-658.
[6]Finelli M J,Oliver P L.TLDc proteins:new players in the oxidative stress response and neurological disease[J]. Mammalian Genome,2017,28(9):395-406.
[7]Colombatti F,Mencia R,Garcia L,et al. The mitochondrial oxidation resistance protein AtOXR2 increases plant biomass and tolerance to oxidative stress[J]. Journal of Experimental Botany,2019,70(12):3177-3195.
[8]張梦云,袁凌云,朱世东,等. 十字花科植物抗氧化基因(OXR)的鉴定与生物信息学分析[J/OL]. 分子植物育种,2021:1-11.(2021-08-26)[2022-12-20]. http://kns.cnki.net/kcms/detail/46.1068.S.20210825.1923.021.html.
[9]Huang S W,Li R Q,Zhang Z H,et al. The genome of the cucumber,Cucumis sativus L.[J]. Nature Genetics,2009,41(12):1275-1281.
[10]Garcia-Mas J,Benjak A,Sanseverino W,et al. The genome of melon (Cucumis melo L.)[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(29):11872-11877.
[11]Guo S G,Zhang J G,Sun H H,et al. The draft genome of watermelon (Citrullus lanatus) and resequencing of 20 diverse accessions[J]. Nature Genetics,2013,45(1):51-58.
[12]Wu S,Shamimuzzaman M,Sun H,et al. The bottle gourd genome provides insights into Cucurbitaceae evolution and facilitates mapping of a Papaya ring-spot virus resistance locus[J]. The Plant Journal,2017,92(5):963-975.
[13]Sun H H,Wu S,Zhang G Y,et al. Karyotype stability and unbiased fractionation in the paleo-allotetraploid Cucurbita genomes[J]. Molecular Plant,2017,10(10):1293-1306.
[14]Xie D S,Xu Y C,Wang J P,et al. The wax gourd genomes offer insights into the genetic diversity and ancestral cucurbit karyotype[J]. Nature Communications,2019,10:5158.
[15]Subramanian B,Gao S H,Lercher M J,et al. Evolview v3:a webserver for visualization,annotation,and management of phylogenetic trees[J]. Nucleic Acids Research,2019,47(1):270-275.
[16]Wang Y P,Tang H B,de Barry J D,et al. MCScanX:a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research,2012,40(7):49.
[17]Krzywinski M,Schein J,Birol I,et al. Circos:an information aesthetic for comparative genomics[J]. Genome Research,2009,19(9):1639-1645.[HJ2mm]
[18]Zhang Z,Xiao J F,Wu J Y,et al. ParaAT:a parallel tool for constructing multiple protein-coding DNA alignments[J]. Biochemical and Biophysical Research Communications,2012,419(4):779-781.
[19]Wang D,Zhang Y,Zhang Z et al. KaKs_Calculator 2.0:a toolkit incorporating gamma-series methods and sliding window strategies[J]. Genomics,Proteomics & Bioinformatics,2010,8(1):77-80.
[20]Tang H,Bowers J E,Wang X,et al. Synteny and collinearity in plant genomes[J]. Science,2008,320(5875):486-488.
[21]Chen C J,Chen H,Zhang Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[22]Blaise M,Alsarraf H M A B,Wong Jaslyn E M M,et al. Crystal structure of the TLDc domain of oxidation resistance protein 2 from zebrafish[J]. Proteins,2012,80(6):1694-1698.
[23]李 寒,夏鵬亮,唐艳红,等. 小麦OXR基因家族全基因组生物信息学及表达分析[J/OL]. 分子植物育种,2022:1-10.(2022-04-11)[2022-05-20]. http://kns.cnki.net/kcms/detail/46.1068.S.20220411.0854.004.html.
[24]Mencia R,Céccoli G,Fabro G,et al. OXR2 increases plant defense against a hemibiotrophic pathogen via the salicylic acid pathway[J]. Plant Physiology,2020,184(2):1112-1127.
[25]Torti P,Raineri J,Mencia R,et al. The sunflower TLDc-containing protein HaOXR2 confers tolerance to oxidative stress and waterlogging when expressed in maize plants[J]. Plant Science,2020,300:110626.
[26]Flagel L E,Wendel J F. Gene duplication and evolutionary novelty in plants[J]. The New Phytologist,2009,183(3):557-564.
[27]乔 鑫. 梨等植物基因组中重复基因的鉴定与进化分析 [D]. 南京:南京农业大学,2018:72-76.
[28]周至铭,杨佳宝,张 程,等. 向日葵LACS家族鉴定及响应非生物胁迫表达分析[J]. 园艺学报,2022,49(2):352-364.
[29]Hahn M W. Distinguishing among evolutionary models for the maintenance of gene duplicates[J]. Journal of Heredity,2009,100(5):605-617.
