无辅助因子放线菌抗生素合成关键酶Dpgc的生信分析
2023-05-12马红梅杨东伟黄海宋宇
马红梅,杨东伟,黄海,宋宇
无辅助因子放线菌抗生素合成关键酶Dpgc的生信分析
马红梅1,2,3,杨东伟1,黄海1,2,3,宋宇1,2,3
1. 海南热带海洋学院, 海南 三亚 572022 2. 海南省热带海洋渔业资源保护与利用重点实验室, 海南 三亚 572022 3. 热带海洋生物资源利用与保护教育部重点实验室, 海南 三亚 572022
为研究万古霉素等抗生素合成关键酶的特征,通过多种生信工具对Dpgc的序列进行分析、预测其理化性质及结构功能。结果显示:Dpgc分布于细胞质中,无信号肽,为亲水性可溶性蛋白,有7个潜在的磷酸化位点。预测到一个结构域,属于巴豆酸酶/烯酰辅酶A(CoA)水合酶超家族,其二级结中主要以螺旋和无规则卷曲为主;三级结构预测到5种模型,其中模型4的误差小,以它为基础,预测到一个体积为171.52 ų的催化口袋。采用swiss-model进行同源建模,结果表明:Dpgc建模结构可靠,可为该酶活性的进一步分析提供理论参考。Dpgc关键酶的生信分析佐证了该酶结构的一些实验研究的结果,为万古霉素等抗生素合成产量的提高提供了参考。
放线菌; 抗生素合成酶; 生物信息
放线菌是产抗生素的主要菌种,能产生多种在临床应用的抗生素,其中万古霉素被誉为“抗生素的最后一道防线”[1],是临床上治疗由甲氧西林耐药金黄色葡萄球菌(MRSA)引起的严重感染的一线抗生素[2]。该抗生素是抗革兰氏阳性菌的一种糖肽类抗生素,最初从东方拟无枝酸菌()的发酵液中分离。对万古霉素生物合成的研究中发现,聚酮体酶DpgA以丙二酰CoA为底物,经过水合酶(DpgB或DpgD)、脱氢酶(Dpgc)、转氨酶(Pgat)的作用,合成3,5-二羟基苯基甘氨酸(Dpg)[3],整个反应过程中,Dpgc是放线菌合成抗生素代谢链中非蛋白质氨基酸生物合成途径中的关键酶,属于加氧酶中的一种[4]。大多数特征化的加氧酶利用结合的过渡金属,如利用铁、铜、黄素或蝶呤辅因子来激活三重态氧进行氧化反应,而Dpgc是一类不依赖有机辅助因子或金属离子即可结合并利用分子氧的双氧化酶[5],是万古霉素和太古霉素合成中的关键酶[6]。目前该抗生素的生产主要通过微生物发酵后提纯发酵液中的成分,在提取工艺优化的情况下,其产量和品质的提高主要取决于Dpgc酶的活性。论文利用生信分析中的不同工具对Dpgc的生化性质与结构进行了预测,为该酶作用的充分发挥提供了理论研究基础。
1 材料与方法
1.1 菌种序列
从GenBank中获取已公布的放线菌和建模外源菌的Dpgc氨基酸序列Dpgc氨基酸序列登录号:登录号:G4V4T6;HCCB10007登录号:AEI58890;sp.登录号:QUQ69271;登录号:AMO93225;登录号:BAU09325;登录号:AAM80546;登录号:AYA22307;登录号:VBA40025;登录号:PPR39952;登录号:ABN80424。
1.2 分析方法
利用Mega软件对序列比对后作进化树。利用在线网址https://web.expasy.org/protparam/分析其理化性质;https://services.healthtech.dtu.dk/services/SignalP-5.0/及6.0预测Dpgc的信号肽;https://services.healthtech.dtu.dk/service.php?TMHMM-2.0对其进行跨膜分析;https://psort.org/documentation/index.html对Dpgc进行亚细胞定位。https://services.healthtech.dtu.dk/中的NetPhos 3.1对其磷酸化位点分析;https://web.expasy.org/protscale/对其亲水性和疏水性分析,利用https://heliquest.ipmc.cnrs.fr/对其双亲性螺旋分析;https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html对其二级结构分析;http://bioinf.cs.ucl.ac.uk/psipred对其二级结构预测;https://robetta.bakerlab.org/对其三级结构进行预测,并利用在线工https://proteins.plus/#dogsite对预测的最佳模型进行催化口袋分析;https://swissmodel.expasy.org/进行同源建模。
2 结果与分析
2.1 不同菌的Dpgc同源性分析
选用放线菌与细菌的Dpgc序列作进化树分析,由图1可知,与HCCB1007聚为一支,进化距离最小,同源性最高。与sp.聚为一支,与聚为一支,进化距离小,同源性较高,、、三种细菌与其他放线菌(除外)的进化距离大,同源性低。
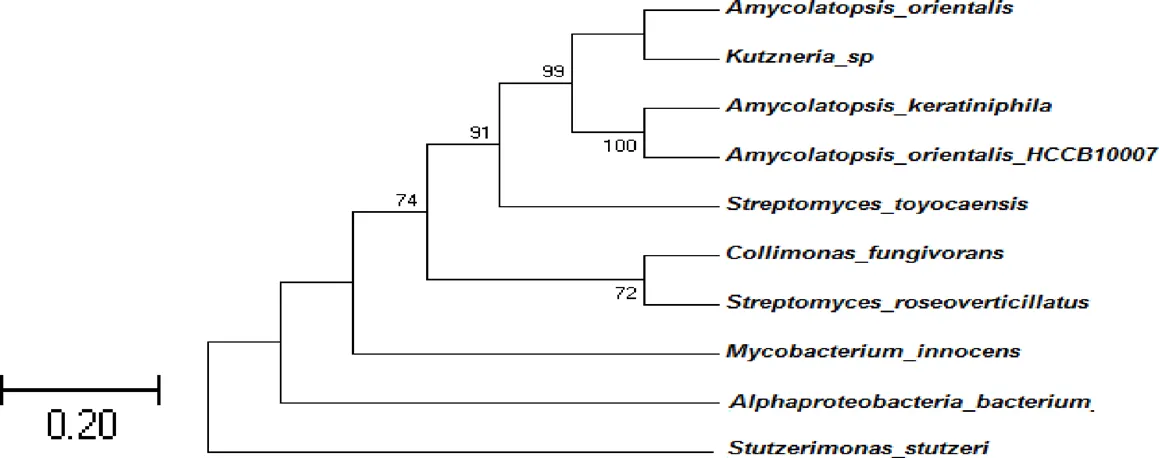
图 1 10种微生物Dpgc的同源性分析
2.2 Dpgc酶序列的基本信息分析
2.1.1 理化性质由ProtParam预测结果显示:Dpgc中氨基酸数目为434个,理论相对分子质量为47088.68,理论等电点为5.50,原子总数为6647,分子式为C2062H3337N611O624S13,不稳定系数为44.26,为不稳定蛋白,脂溶系数为96.36,酸性氨基酸残基总数(Asp+Glu)为62,碱性氨基酸残基总数为54。不同氨基酸的数目及比例见表1。由表1可知:Ala的数目最多,占比15.2%,其次为Leu (L)和Arg (R),其他氨基酸的占比均小于10%,其中Cys (C)和Trp (W)的数目少,比例相差不大,分别为0.7%和0.5%。总平均疏水性为(GRAVY)为-0.127,初步分析表明该酶为亲水性可溶性蛋白。
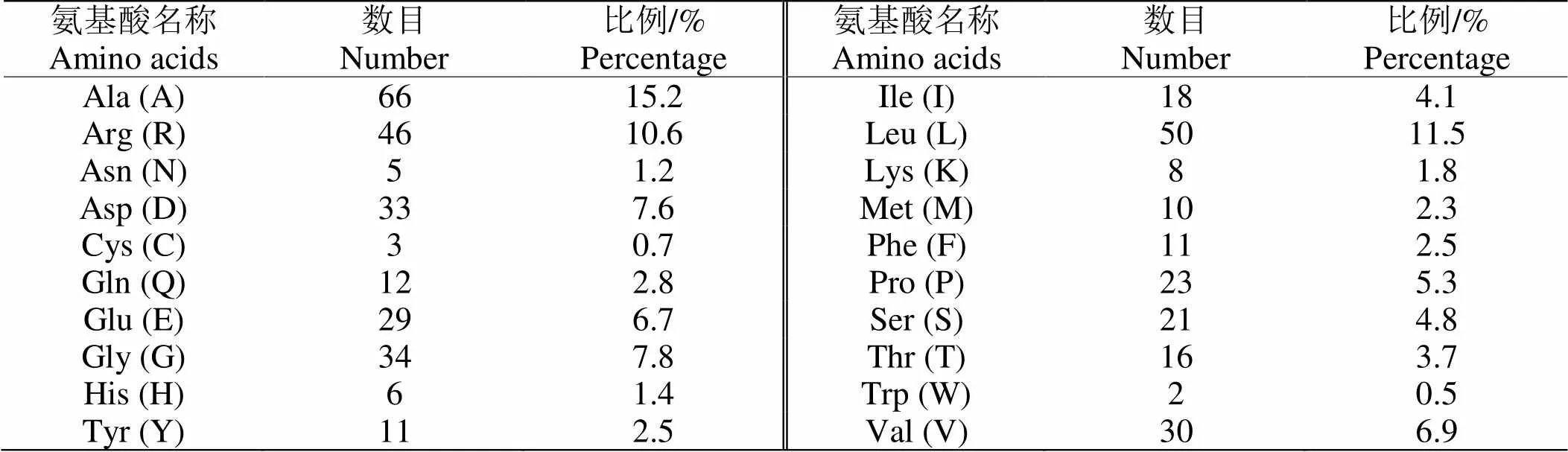
表 1 氨基酸组成分析
2.1.2 疏水性分析因氨基酸的亲水性与疏水性是构成蛋白折叠的主要驱动力之一,因此蛋白质亲水性分布图可反映蛋白质的折叠情况。使用ProtScale软件中的Hydropath. / Kyte & Doolittle标度分析酶的疏水性。当氨基酸的打分值大于0,表示疏水,正值越大,疏水性越强;负值表示亲水,负值越小,表示亲水性越强。值在-0.5~0.5间的氨基酸为两性。由图2可知,Dpgc多肽链中第301处苯丙氨酸的值最大为2.233,表明此处氨基酸疏水性最强,多肽链上第111和第112位点处分别为精氨酸和谷氨酸,两者得分均为-2.989,表明此两种氨基酸的亲水性最强。0~86处氨基酸密集式亲水,总得分小于0,故Dpgc酶蛋白为亲水性蛋白。
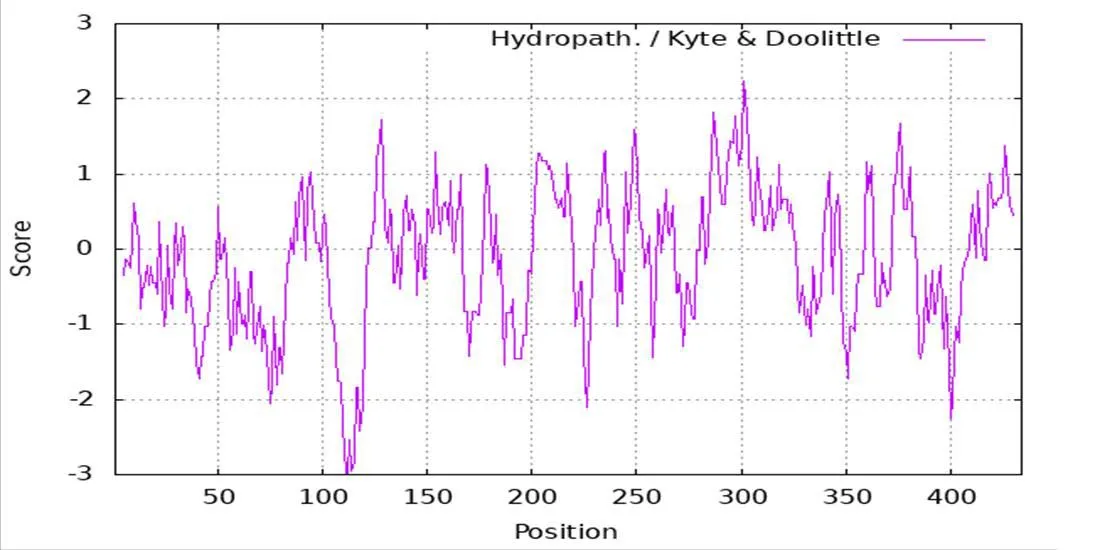
图 2 Dpgc疏水性图谱
2.3 Dpgc酶序列的特征信息分析
2.3.1 磷酸化分析磷酸化是蛋白质翻译后的一种修饰,是一种动态的生物调节过程,与DNA损伤修复、转录调节、信号传导、细胞凋亡的调节等生物学功能密切相关。磷酸化多肽主要指肽链中的Ser、Tyr和Thr残基的侧链羟基被修饰成酸式磷酸酯多肽。由NetPhosbac预测Dpgc结果见图3。

图 3 Dpgc磷酸化位点
由图3可知,酶蛋白中共有7个潜在的磷酸化位点得分大于0.5,6个是serine,1个是threonine,其中得分最高的为78T,其次为272S,表明此两处位点的磷酸化置信度高,磷酸化置信度最低处为222S,9S,35S,37S和131S处的得分介于最高与最低之间,说明这4个位点被磷酸化修饰的置信度介于中间。
2.3.2 Dpgc双亲性螺旋分析采用HeliQuest中的anlysis预测Dpgc的双亲性螺旋,它通过螺旋的氨基酸序列(-螺旋、3-10螺旋、3-11螺旋或π螺旋)计算其物理化学性质和氨基酸组成,并使用结果筛选数据库以识别具有此特征的蛋白质片段相似的特征。文中采用HeliQuest中的模板analysis确定α螺旋类型的疏水性、疏水力矩、净电荷(z)和氨基酸组成等特性,windows size选36,预测分析结果显示:1-36位构成一个双亲性螺旋,疏水性为0.341,疏水力矩<µH>为0.256,极性残基占52.78%,不带电荷的残基+GLY为GLN 1、HIS 1、SER 4、THR 3、GLY 2;带电残基为LYS 1、ARG 2、GLU 1、ASP 4,非极性残基占47.22%,非极性残基中无芳烃残基,特殊残基为CYS 0, PRO 4,预测结果见图4,其疏水面为L V M L P L。
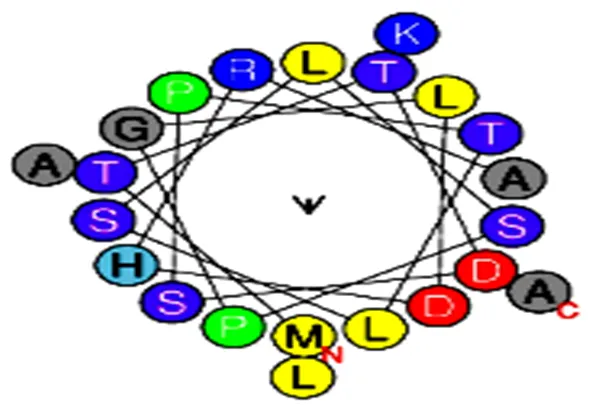
氨基酸序列:1MTTDSPTLSLSPGLDHRALAKAAQRVDELLDGLPSP36
2.3.3 Dpgc信号肽分析信号肽位于蛋白质的N端,一般由16-26个残基组成,可将蛋白质引导定位至不同的细胞器。采用signal 5.0中的gram-positive预测信号肽,结果见图5(A)。Dpgc蛋白具有信号肽的可能性为0.0122,具有双精氨酸转移信号肽(TAT)的可能性为0.0123,具有脂蛋白信号肽(Sec/SPII)的可能性为0.0021,为其它类型蛋白质的可能性是0.9734,说明Dpgc没有信号肽,可能是其它类型蛋白质。signal 6.0是最新版本,可预测各种类型的微生物,预测结果见图5(B),预测结果与signal 5.0预测结果一致。
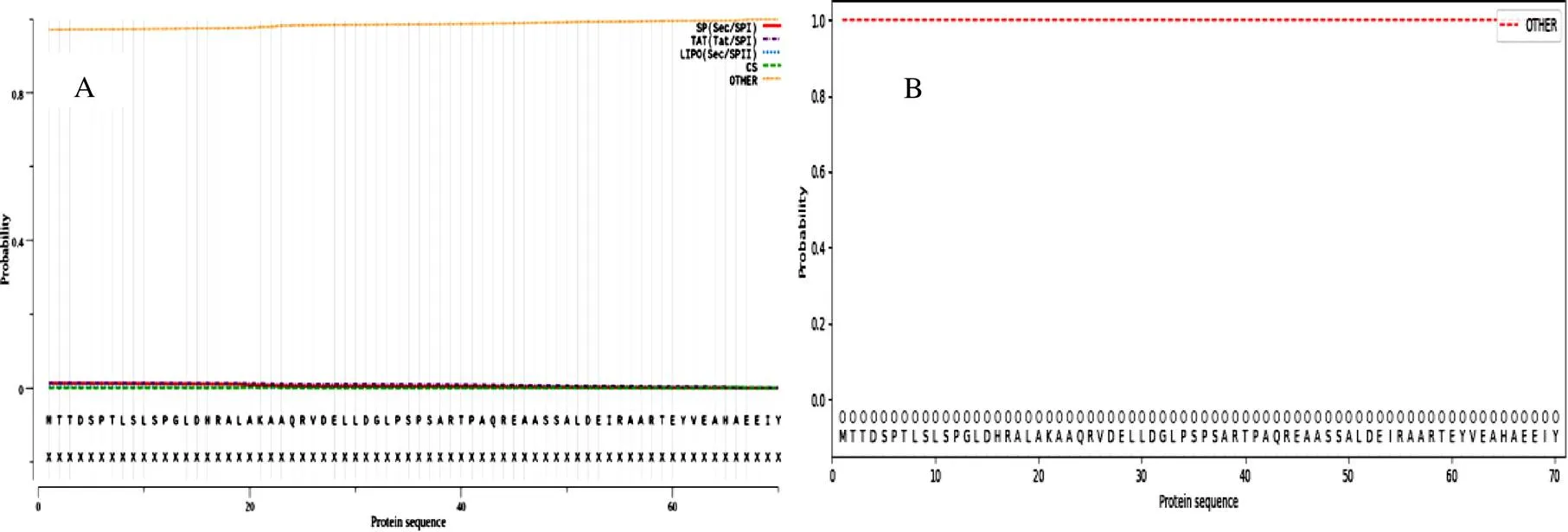
图 5 Dpgc信号肽预测结果
2.3.4 Dpgc跨膜分析膜蛋白是一类结构独特的蛋白质,广泛存在于各种细胞中,为神经信号分子、激素和受体等组成成分之一,是各种离子跨膜的通道,也是多种药物分子的靶点,可分外在膜蛋白、内在膜蛋白和脂锚定膜蛋白3种类型。由TMHMM-2.0预测结果见图6,结果显示:膜蛋白为跨膜螺旋数量为0,跨膜螺旋氨基酸期望值为0.13815,蛋白前60个氨基酸中跨膜螺旋的氨基酸量的期望值为0,由此知N端预测的跨膜螺旋区不存在信号肽,此结果与信号肽预测结果一致,也不存在跨膜区。
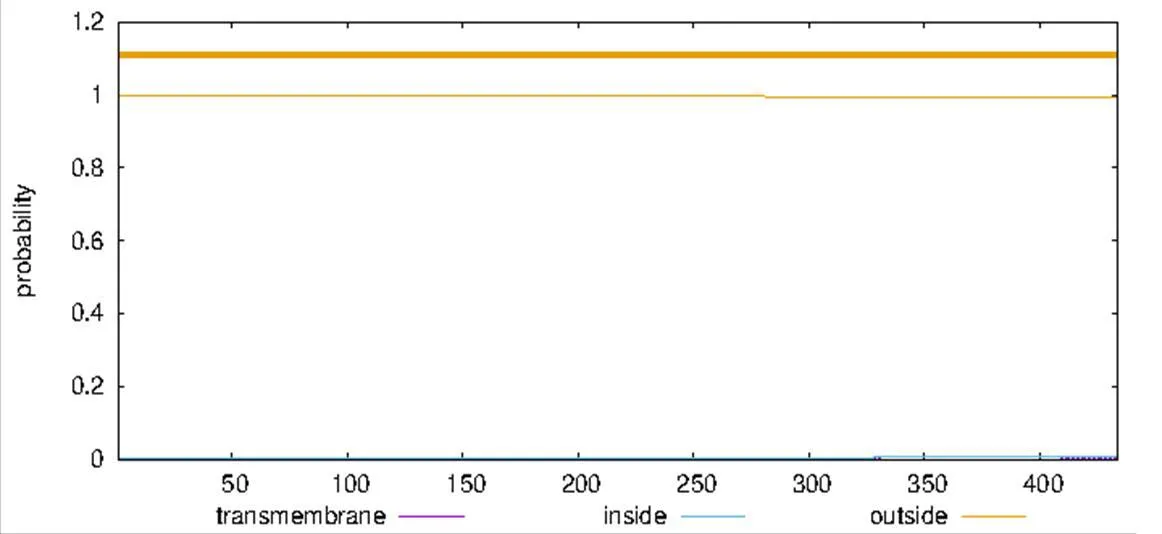
图 6 Dpgc跨膜区分析
2.3.5 亚细胞定位由Psortb 3.0 亚细胞定位计算结果显示,该酶在细胞膜中定位得分仅为1.15,在细胞壁中定位得分为0.62,在细胞质中定位得分为7.5分,据此判断该酶分布于细胞质中。
2.4 酶的结构分析
2.4.1 Dpgc结构域分析利用NCBI的CDD数据库对该酶结构域进行分析,结果见图7。其E-value为2.32e-40,小于阈值10-6,其中序列167-383氨基酸处有一结构域,属于巴豆酸酶/烯酰辅酶A (CoA)水合酶超家族。该家族包含多种酶:烯酰辅酶A水合酶、萘甲酸合酶、肉桂酸消旋酶、3-羟基丁酰辅酶A脱水酶和十二烷酰辅酶A δ异构酶。源自酰基辅酶A底物的烯醇阴离子中间体在氧阴离子空穴(OAH)中稳定,在大多数情况下由两个保守的主链NH基团形成。除了保守的结构核心外,该家庭成员的寡聚状态各不相同。有些形成三聚体或六聚体(三聚体的二聚体)或九聚体(三聚体的三聚体),底物结合位点位于三聚体中两个亚基之间的界面,每个三聚体包含三个底物结合位点。
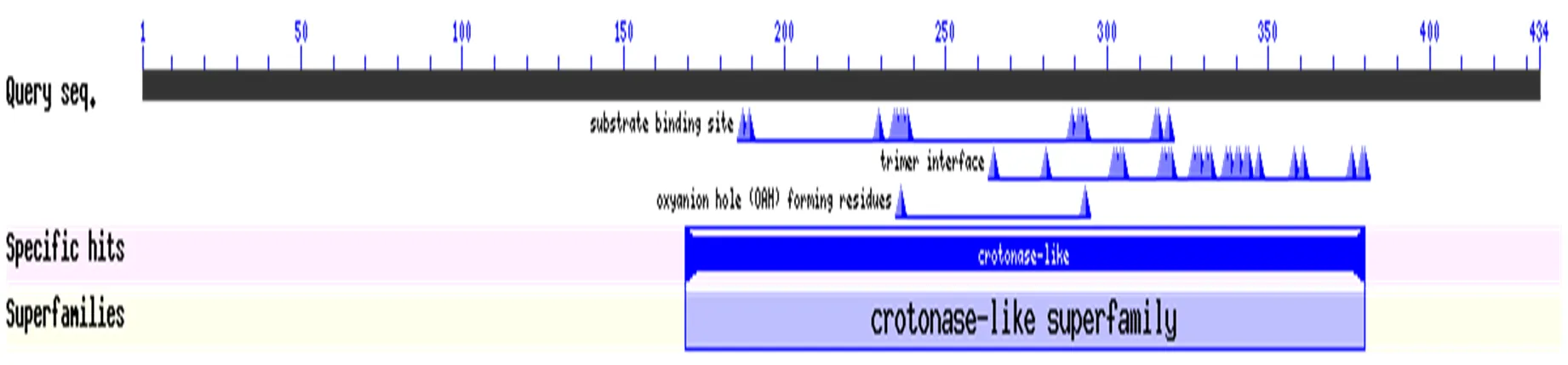
图7 Dpgc的结构域分析
2.4.2 Dpgc的二级结构分析蛋白质多肽链的二级结构主要形式有-螺旋、-折叠、-转角和无规卷曲。由SOPMA预测Dpgc结果见图8。结果显示:DPGC的二级结构主要由四个部分组成,其中螺旋占54.85%、转角占6.91%,无规则卷曲占29.26%,及延伸链(也叫片层)占8.99%,由此可知Dpgc主要由螺旋构成,其次为无规则卷曲,少数为片层和转角。

图 8 Dpgc的二级结构分析
注:H为螺旋,T为转角,E为片层,为无规则卷曲
2.4.3 Dpgc的三级结构预测及催化口袋分析采用RobeTTA对Dpgc结构进行在线预测,置信度为 0.87(满分为1),共获5个预测结构,每个结构中预测到1个结构域,结果见图9。从各模型的误差估算图可知,其中模型4具有相对较小的误差,可以此基础进行进一步结构解析。利用模型4对其催化口袋进行分析,建立了一个体积为171.52 ų的催化口袋,结果如图10,参数信息见表2。
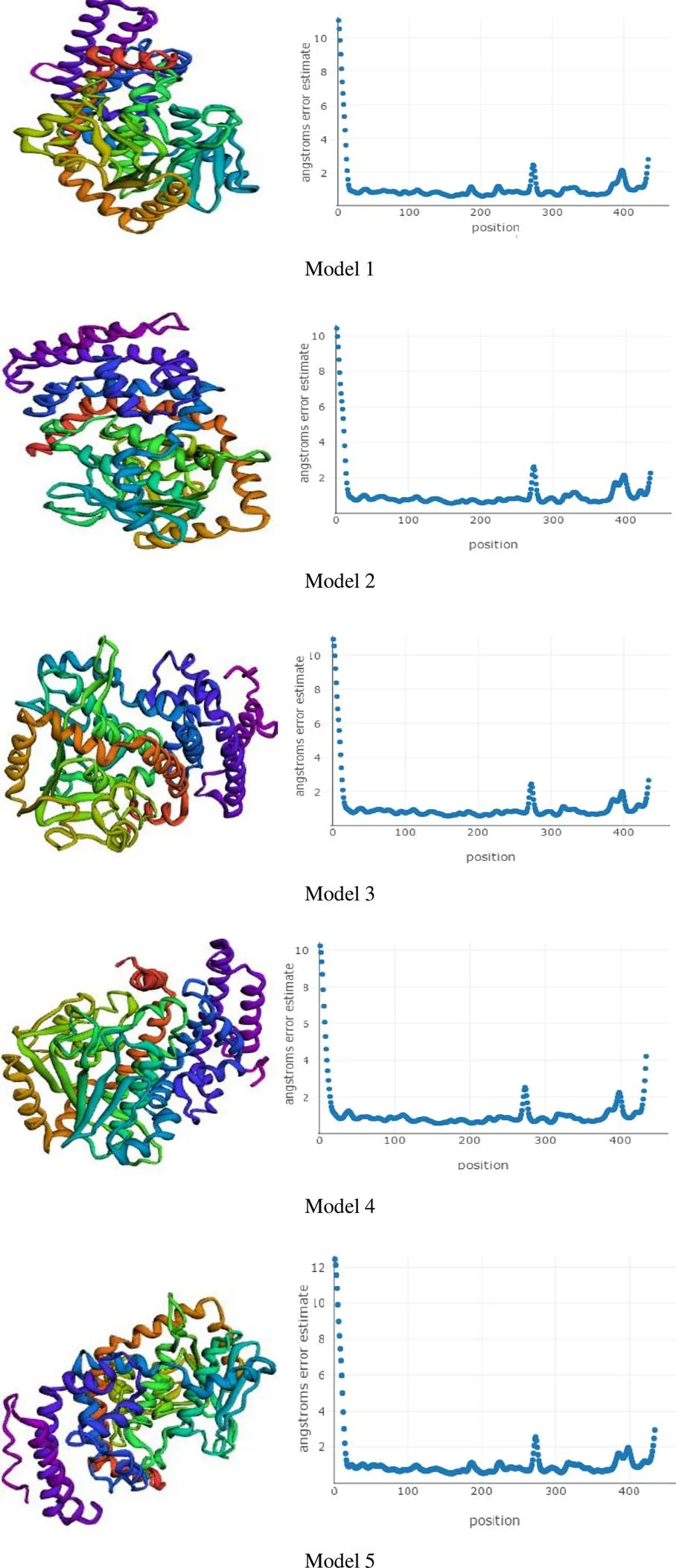
图 9 Dpgc的三级结构预测
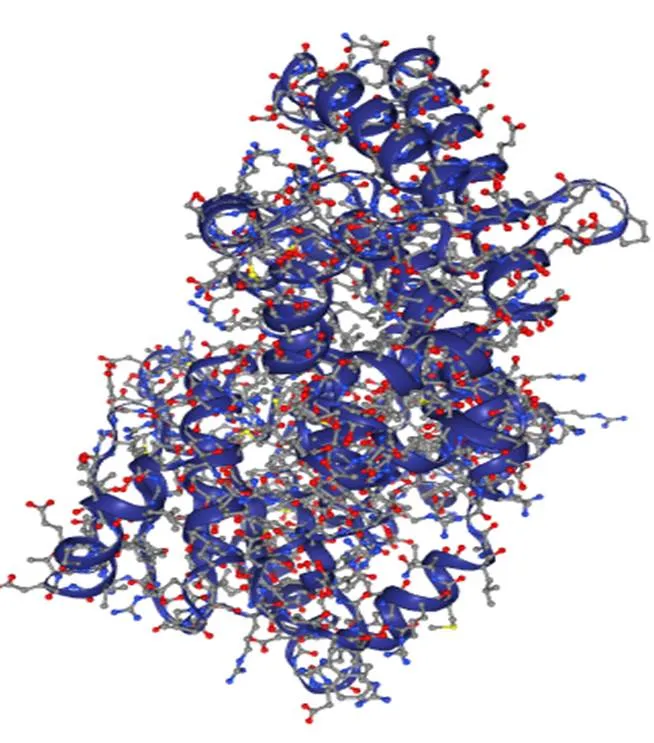
图 10 基于PDB结构模拟的Dpgc的催化口袋
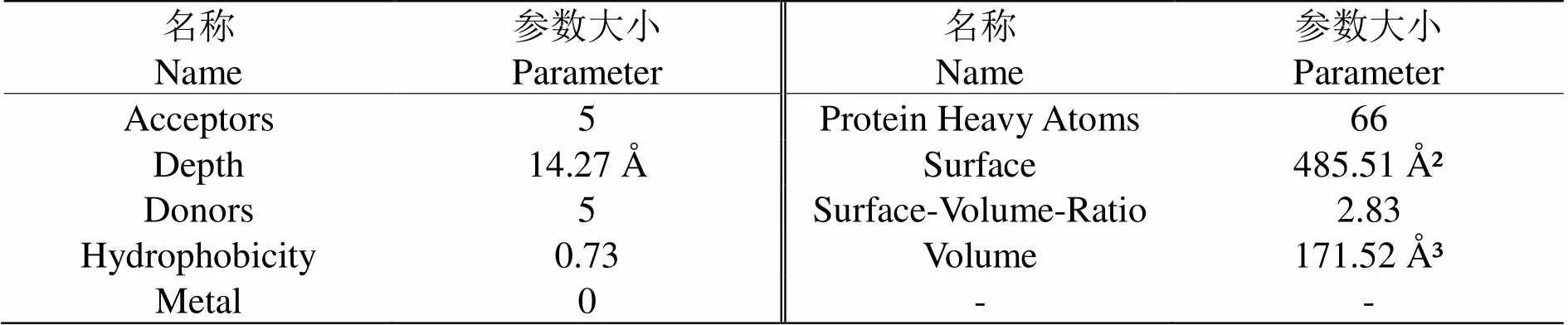
表 2 Dpgc催化口袋基本信息
2.4.4 Dpgc的三级结构预测及其同源建模分析采用SWISS-MODEL网站预测蛋白质结构,以P21, A319C突变巴豆酶超家族双加氧酶和2np9.1.A巴豆酸酶超家族双加氧酶的Dpgc晶体结构为模板进行建模,结果见图11。对其质量评估结果显示:相似度值比对后结果为69.72%,说明待预测蛋白与模板蛋白结构具有很高的同源性,该模板可用于预测该酶蛋白的三维结构,且预测模型的可信度高。建模结构的GMQE值为0.89,在0~1之间,比较接近1,说明建模质量很好;其QMEAN值为-0.36,在-4-0区间,接近0,说明模型匹配度高,由此可知,运用同源建模的方法预测得到的蛋白质结构准确可靠。并以此为模板采用拉式图评价预测模型质量,使用拉式图评价预测建立的Dpgc三级结构模型,结果见图12,有97.20%的氨基酸残基落在绿色最佳区域,说明模型结构可靠,可用于后续工作和研究。
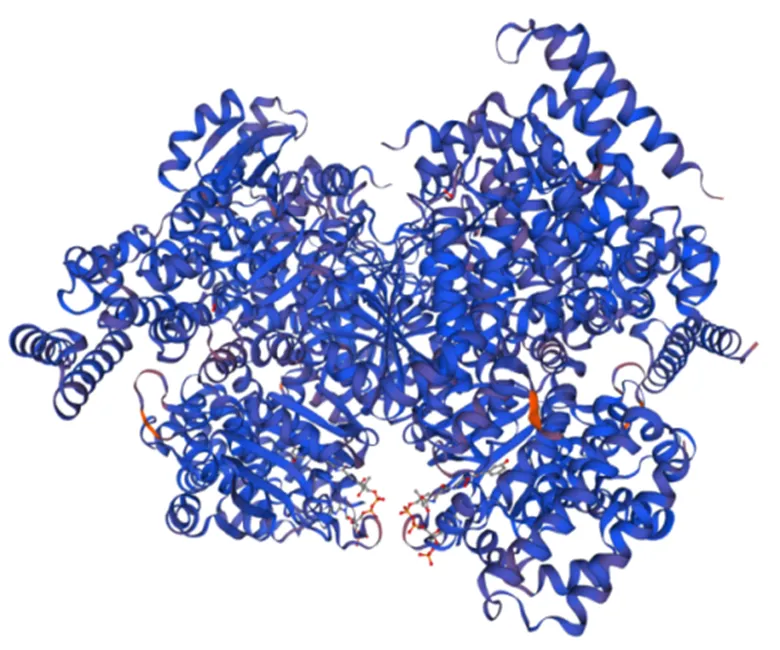
图 11 Dpgc的三级结构
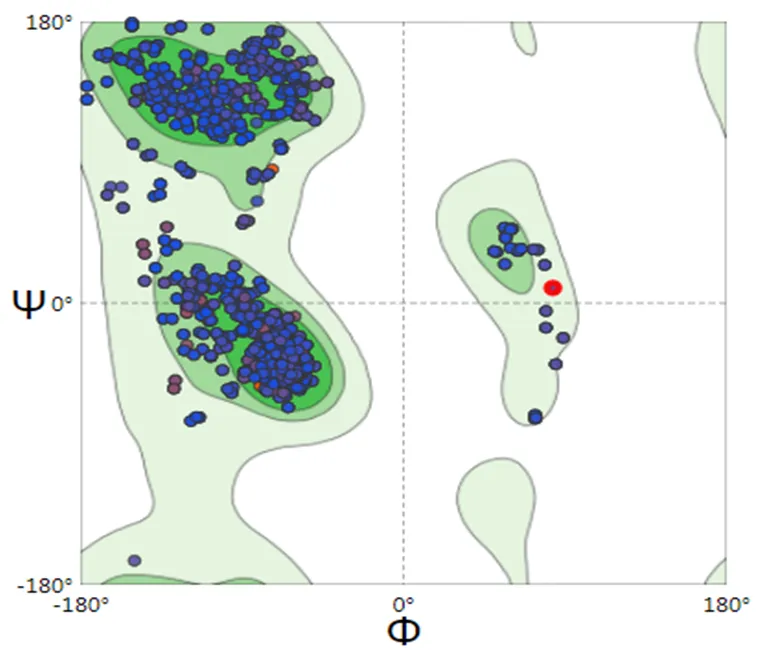
图 12 Dpgc的拉式构象
3 结论与讨论
抗生素是以放线菌为主要生产菌代谢合成的次级代谢产物,其调控关键酶都在合成阶段被合成,酶与氧结合和活化是参与分解代谢、合成代谢等多种细胞过程的基本部分[7]。多数加氧酶使用血红素或非血红素铁来结合和激活加氧,而Dpgc是一类为数不多的不依赖辅因子的双加氧酶,有关其与氧结合的机制研究较多。Kunhua L与Fielding EN等介绍了DpgC与双氧结合的机制[8,9]。随着万古霉素等抗生素的耐药性问题突显,对新糖肽类抗生素的开发及组合生物合成的研究越来越多,而基因分子水平上的操作是组合生物合成的重要手段,因而对酶的生信分析是基因水平操作的前提。
该酶属于亲水性蛋白,其中Arg和Glu的含量较高,极性氨基酸组成较非极性氨基酸含量高,这两种氨基酸在在底物识别和催化中具有重要的作用[9]。该酶虽预测到7个磷酸化位点,但目前还未有实验证实,可作为今后研究的一个方向。对其结构预测中,不同在线生信工具都预测到Dpgc有一个结构域,属于巴豆酸酶/烯酰辅酶A (CoA)水合酶超家族,其成员对酰基辅酶A底物进行各种各样的化学转化,有实验证据表明Dpgc的保守活性位点残基与其他巴豆激酶家族成员之间没有显著重叠[10,11]。预测的三级结构置信度高,误差较小,可作为后续研究的基础。
[1] 周良辅.万古霉素临床合理应用中国专家共识[J].中国新药与临床杂志,2011,8(30):561-573
[2] Nagarajan R. Antibacterial activities and modes of action of vancomycin and related glycopeptides [J]. Antimicrobial Agents and Chemotherapy, 1991,35(4):605-609
[3] Chen H, Tseng CC, Hubbard BK,. Glycopeptide antibiotic biosynthesis: Enzymatic assembly of the dedicated amino acid monomer ()-3,5-dihydroxyphenylglycine [J]. Proceedings of the National Academy of Sciences, 2001,98(26):14901-14906
[4] Widboom PF, Bruner SD. Complex oxidation chemistry in the biosynthetic path-ways to Vancomycin/Teicoplanin antibiotics [J]. Chem Bio Chem, 2009,10:1757-1764
[5] Tseng CC, Vaillancourt HF, Bruner DS,. DpgC is a metal- and cofactor-free 3,5-Dihydroxyphenylacetyl-CoA 1,2-Dioxygenase in the vancomycin biosynthetic pathway [J]. Chemistry & Biology, 2004,11(9):1195-1203
[6] Ortega P, Zanchet A, Sanz-Sanz C,. Dpgc-catalyzed peroxidation of DPA-CoA: insights into the spin-forbidden transition and charge transfer mechanisms [J]. Chemistry, 2021,27(5):1700-1712
[7] Laden BP, Tang Y, Porter TD. Cloning, heterologous expression, and enzymological characterization of human squalene monooxygenase [J]. Arch. Biochem. Biophys, 2000,374:381-388
[8] Kunhua L, Fielding EN, Condurso HL,. Probing the structural basis of oxygen binding in a cofactor-independent dioxygenase [J]. Acta Crystallographica Section D Structural Biology, 2017,73(7):573-580
[9] Fielding EN, Widboom PF, Bruner SD. Substrate recognition and catalysis by the cofactor-independent dioxygenase Dpgc [J]. Biochemistry, 2007,46(49):13994-14000
[10] Sleeman MC, Sorensen JL, Batchelar ET,. Structural and mechanistic studies on carboxymethylproline synthase (CarB), a unique member of the crotonase superfamily catalyzing the first step in carbapenem biosynthesis [J]. J. Biol. Chem. 2005,280:34956-34965
[11] Truglio JJ, Theis K, Feng Y,. Crystal structure ofMenB, a key enzyme in vitamin K2 biosynthesis [J]. J. Biol. Chem, 2003,278:42352-42360
Bioinformatics Analysis of the Cofactor-Independent Key Enzyme Dpgc for Antibiotic Synthesis inspp
MA Hong-mei1,2,3, YANG Dong-wei1, HUANG Hai1,2,3, SONG Yu1,2,3
1.5720222.572022,572022
To investigate the characteristics of key enzymes in antibiotic synthesis such as vancomycin, the sequence of Dpgc was analyzed and its physicochemical properties, structure, and functions were predicted using various biological information tools. The results showed that Dpgc was distributed in the cytoplasm without signal peptide, and was a hydrophilic soluble protein with 7 potential phosphorylation sites. It was predicted that a domain belongs to the crotonase /enoyl coenzyme A (CoA) hydratase superfamily, and its secondary structure was mainly composed of α Mainly spiral and irregular curl;five models had been predicted for the tertiary structure, one of which the error of model 4 was smaller. Based on it, a volume of 171.52 ų had been predicted Catalytic pocket. The results of homologous modeling using swiss model indicated that the structure of Dpgc modeling was reliable and can provide a theoretical reference for further analysis of the enzyme activity. Bioinformatics analysis of the key enzyme of Dpgc corroborated the results of some experimental studies on the enzyme structure, providing a reference for improving the production of antibiotics such as vancomycin.
spp.; antibiotic synthetase; bioinformatics
Q811.4
A
1000-2324(2023)02-0208-08
10.3969/j.issn.1000-2324.2023.02.008
2022-12-05
2023-02-04
海南省高等学校教育教学改革研究重点项目(Hnjg2020ZD-35);海南自然科学基金(421RC591)
马红梅(1976-),女,教授,研究方向为微生物代谢调控. E-mail:mahongmei612@163.com
