小麦G2-like转录因子家族基因鉴定与表达模式分析
2023-03-23贾玉库高宏欢冯健超郝紫瑞王晨阳谢迎新郭天财马冬云
贾玉库 高宏欢 冯健超 郝紫瑞 王晨阳 谢迎新 郭天财 马冬云
研究简报
小麦G2-like转录因子家族基因鉴定与表达模式分析
贾玉库**高宏欢**冯健超 郝紫瑞 王晨阳 谢迎新 郭天财 马冬云*
河南农业大学农学院 / 国家小麦工程技术研究中心/ 河南省小麦技术创新中心, 河南郑州 450046
()转录因子, 是属于MYB类转录因子中GARP超家族的成员, 在调节叶绿体发育中起重要作用。本研究利用生物信息学方法对小麦基因进行了全基因组鉴定, 并对其理化性质、亚细胞定位、启动子顺式作用元件及对非生物胁迫和激素的响应模式进行了分析。从小麦中共鉴定出87个基因, 不均匀的分布在小麦21条染色体上, 系统发育分析将这些基因分为14个亚族。蛋白质二级结构预测表明, 小麦基因的氨基酸序列均以α螺旋和随机卷曲为主要结构。启动子顺式作用元件分析表明其上游2 kb区域含有7种(P-box、SpI、LTR、ABRE、MBS、TGA-element和AE-box)与逆境胁迫诱导相关顺式作用元件。其中含有顺式调控元件结合位点最多, 一共有18个结合位点。qRT-PCR验证发现,、、和在PEG和盐胁迫下以及GA、IAA和ABA激素诱导下表达量显著上调, 这些基因可能介导了小麦对多种非生物逆境的响应。
小麦; G2-like转录因子; 生物信息学; 逆境胁迫; 表达分析
小麦是我国最主要的粮食作物之一, 其产量高低对国家粮食安全至关重要。近年来, 随着气候变化和农业生态环境污染, 各种环境胁迫包括低温[1-2]、高温[3]、干旱[4-5]和盐胁迫[6-7]等严重影响小麦生长。因此, 提升小麦在逆境条件下的防御水平, 对小麦增产增收意义重大。
植物中转录因子在逆境胁迫下可以激活或者抑制某些基因的转录, 影响其蛋白在植物体内的表达量及功能, 从而在植物逆境应答和生长发育中起重要作用[8-9]。(或)转录因子是属于MYB类转录因子中GARP超家族的成员, 广泛存在于陆地植物中。首先在玉米中被发现[10], 随后在拟南芥、水稻、辣椒和番茄中均有存在, 在叶绿体发育、果实品质、植物衰老及响应逆境胁迫等方面均起作用[11-13]。e家族成员的特征是在过渡和早期成熟阶段调节叶绿体的形成[14-15], 对叶绿体的发育是不可或缺的基因。玉米和这一对同源基因功能基本相同, 分别在叶肉细胞和维管束鞘细胞中表达[16]。过表达可以增强调控叶绿体的发育相关基因的表达, 进而影响成熟果实中的糖类和类胡萝卜素的含量[13]。
基因沉默后的番茄植株表现出对干旱和低温的抗性下降[17]。拟南芥在低温下基因通过抑制花青素合成基因和抑制花青素的积累并增强黄酮和香豆素苷的积累, 从而对低温产生抗性[18]。对玉米植株在低温(4℃)和干旱胁迫处理下的转录因子家族的20个基因表达量分析, 发现部分基因的表达与植株抗逆性有关[19]。同时转录因子影响控制气孔开闭和钾离子运动相关基因的表达, 从而对气孔起到调节作用[20]。植物激素如生长素和油菜素类固醇与果实叶绿体的发育有关, 且与的表达上调有关[21]。目前关于转录因子在小麦中的研究尚未有详细报道。因此, 通过对小麦基因组中转录因子进行了系统挖掘, 结合生物信息学分析和对部分小麦转录因子在干旱和盐胁迫下的表达模式分析, 筛选抗逆相关的家族成员, 为小麦抗逆基因的挖掘和在育种中的应用奠定基础。
1 材料与方法
1.1 小麦G2-like基因的全基因组鉴定、编码蛋白的理化性质、二级结构分析和亚细胞定位分析
从Ensembl Plants数据库(http://plants.ensembl.org/ index.html)获取小麦蛋白序列, 以玉米G2-like蛋白序列作为query序列进行BLASTP比对(-value<1E–5)获得同源序列。在Pfam数据库(http://pfam.xfam.org/)下载G2-like保守结构域PF00249.29, 利用HMM3.0进行保守结构域比对; 同时利用Pfam搜索(http://pfam.xfam.org/)、SMART (http://samrt.embl-heidelberg.de/)和HMMER (https://www. ebi.ac.uk/Tools/hmmer/search/hmscan)验证候选蛋白保守结构域。利用ExPASy-ProtParam (https://web.expasy.org/ protparam/)计算G2-like蛋白的氨基酸长度和分子量等基本理化性质。分别利用软件NPS-SOPMA (https://npsa- prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)和ProtComp 9.0 (https://linux1.Softbe-rry.com/berry.phtml)预测G2-like的二级结构和亚细胞定位。
1.2 小麦G2-like基因系统发育进化、基因结构和基序分析
利用Clustal_W工具对G2-like蛋白序列进行多重比对, 结果放入MEGA 7.0[22]软件, 采用邻接法(Neighbor- Jointing, NJ)构建系统发育进化树(bootstrap, 1000; 基序, 10)。利用TBtools软件[23]作图。
1.3 小麦G2-like基因系统发育进化、染色体定位和共线性分析
根据IWGSC (http://wheat-urgi.versailles.inra.fr/)小麦基因组数据BLASTN构建本地数据库, 通过比对获得基因在染色体上的物理位置。利用MG2C (http://mg2c.iask.in/mg2c_v2.0/)构建基因在染色体上的物理图谱。利用MCScanX[24]进行共线性分析(-value<1E–5), Circos软件可视化分析[25]。利用KaKs_Calculator软件计算非同义替代率(Ka)和同义替代率(Ks)[26], 预测基因CDS区的适应性进化。
1.4 小麦G2-like基因的顺式作用元件分析
利用TBtools软件截取基因CDS上游2.0 kb的DNA序列, 提交至PlantCARE数据库(http://bioinformatics. psb.ugent.be/webtools/plantcare/html)进行启动子顺式作用元件的预测。
1.5 小麦G2-like基因在不同组织部位、逆境胁迫下的表达分析
利用expVIP数据库(http://www.wheat-expression.com/)和Wheat Exp数据库(https://wheat.pw.usda.gov/WheatExp/)中小麦基因组和在干旱、热胁迫下的转录组数据, 分析基因在不同组织及逆境胁迫下表达模式。
1.6 小麦G2-like转录因子家族部分基因qRT-PCR验证
选用河南省主推小麦品种百农207为试验材料, 挑选籽粒饱满大小一致的种子经75%乙醇消毒处理后, 用蒸馏水冲洗5次, 然后置于光照培养箱中。在23℃/18℃ (白天/黑夜)、光/暗周期为16 h/8 h培养, 然后挑选发芽一致的种子采用Hoagland营养液培养, 每2 d更换一次培养液。待其长到二叶一心进行干旱(20% PEG-6000)、盐胁迫(200 mmol L–1NaCl)、以及生长素(100 μmol L–1IAA)、赤霉素(100 μmol L–1GA)和脱落酸(100 μmol L–1ABA)处理, 正常营养液培养作为对照, 所有处理重复3次。分别在处理0、1、6、12和24 h时取不同处理幼叶, 立即置于液氮中保存。使用TRIZOL (北京全式金生物技术股份有限公司)提取RNA, 进行反转录cDNA第一链合成, 用于Real-time PCR分析(表1)。在QuantStudio 5 Real-time PCR System (Themo Fisher, BIO INC, CA, 美国)上进行qRT-PCR分析, 反应体系与程序参考TB Green Premix ExII (TaKaRa, 大连)试剂盒说明书, 采用2–ΔΔCt法计算相对表达量[27]。

表1 部分G2-like基因荧光定量所用引物
2 结果与分析
2.1 小麦G2-like基因全基因组鉴定、编码蛋白的基本理化性质、二级结构和亚细胞定位分析
根据59个玉米基因的蛋白序列, 利用BLASTP和HMM比对搜索, 在小麦基因组数据库中得到110个候选基因。通过Pfam、SMART、HMMER验证是否有G2-like MYB DNA保守结构域, 共鉴定出87个基因, 据其在染色体上的位置进行命名(表2)。
蛋白序列一级结构理化性质分析显示, 87个G2-like蛋白序列长度为224~534氨基酸, 分子量为24.03~58.47 kD, 等电点范围为4.84~9.65, 其中43个蛋白理论等电点在酸性范围内, 其余均在碱性范围内, 表明G2-like蛋白质酸碱分布均匀; 疏水性指数范围为–0.945~ –0.319; 不稳定系数范围为28.00~73.87, 其中有8个G2-like蛋白不稳定系数小于40, 相对来说为稳定蛋白质。蛋白二级结构均含有α螺旋、延伸链、β转角和无规则卷曲, 但以α螺旋和无规则卷曲为主要结构。亚细胞定位预测表明, 大部分位于核膜, 3个位于线粒体膜, 9个位于质膜, 1个位于细胞质膜。
2.2 G2-like基因家族系统发育进化、基因结构和基序分析
基于进化关系、基序和结构分析,基因家族可分为14 (I~XIV)组(图1-A)。每组基因分布不均匀, 第9组数量最多(13个), 第3组最少只有2个。一般来说, 聚集在同一分组的基因表现出相似的基序结构和基因结构。利用MEME motif搜索工具, 鉴定出G2-like蛋白10个保守基序(图1-B)。几乎每个基因都有基序1和基序2结构域(没有)。此外, 每个分组都有独特的基序, 说明转录因子在进化过程中存在内部分化, 这可能是基因功能多样性的来源。依据基因组DNA和CDS序列, 获得了基因的外显子和内含子结构(图1-C)。所有的e基因都具有完整的基因结构, 但是有14个基因没有UTR结构。当缺失UTR时, 意味着基因的调控功能(mRNA的稳定性、折叠、核内运输相互作用, RNA加工、剪切和翻译等)会受到影响[28-29]。
2.3 小麦G2-like基因的染色体分布及共线性分析
基因在小麦A、B、D三种染色体上是不均匀分布, 且不同染色体上基因位置和数目明显不同(图2)。其中2A、2B和2D染色体上分布的最少(7个), 在7A、7B和7D染色体上分布的最多(22个)。有6个基因形成3对串联重复基因对:和、和和
通过BLASTP和MCscanX对进行重复模式分析, 表明小麦基因组内存在79,067个共线性区域(包括Un染色体), 共存在107,545个基因(图3)。有74个小麦基因定位在共线性区域内, 说明这些基因由片段复制模式产生。同时, 3个基因同时涉及到片段复制和串联重复事件, 表明这些基因由两种重复模式互作扩增而来。家族的同源基因对的Ka/Ks值均小于1, 表明这些同源基因对均受到负选择作用, 同时其进化过程中受到功能限制作用。
2.4 小麦G2-like基因启动子的顺式作用元件分析
对基因启动子序列分析表明, 其2 kb上游区域含有响应抗逆胁迫的7种潜在顺式作用调控元件(图4), 其中与激素、干旱、低温、盐胁迫诱导有关的MBS、P-box、Sp1、TGA-element、LTR、ABRE和AE-box顺式作用元件。本研究中62个基因至少含有5个抗逆胁迫顺式作用元件结合位点,含有顺式调控元件结合位点最多, 一共有18个结合位点。表明小麦基因家族可能参与多种非生物逆境的响应。
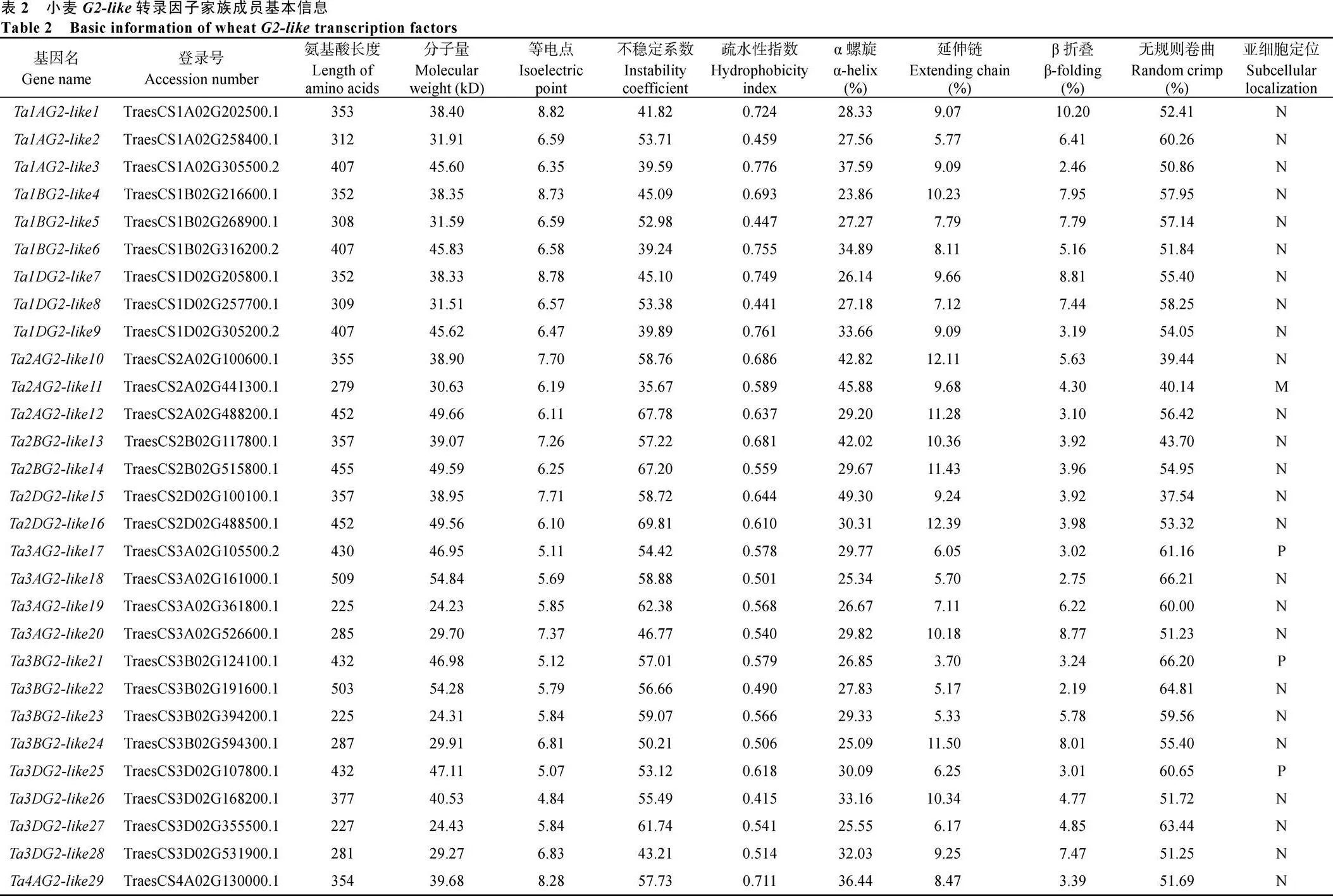

图1 小麦G2-like基因的系统发育关系、保守蛋白基序结构和基因结构
A: 利用MEGA7基于小麦G2-like蛋白全长序列构建系统发育树。B: 小麦G2-like蛋白的基序组成。不同颜色表示motif 1~motif 10; 蛋白质的长度可以用底部的刻度来估计。C: 小麦基因的外显子-内含子结构。绿色方框表示未翻译的5-和3-区域;黄色框表示外显子; 黑线表示内含子。
A: phylogenetic tree was constructed based on the full-length sequences of wheat G2-like proteins using MEGA 7. B: the motif composition of the wheat G2-like proteins. Different colors indicate motif 1–motif 10. The length of the protein can be estimated using the scale at the bottom. C: the exon-intron structures of wheatgenes. Green boxes indicate untranslated 5-regions and 3-regions; yellow boxes indicate exons; and black lines indicate introns.
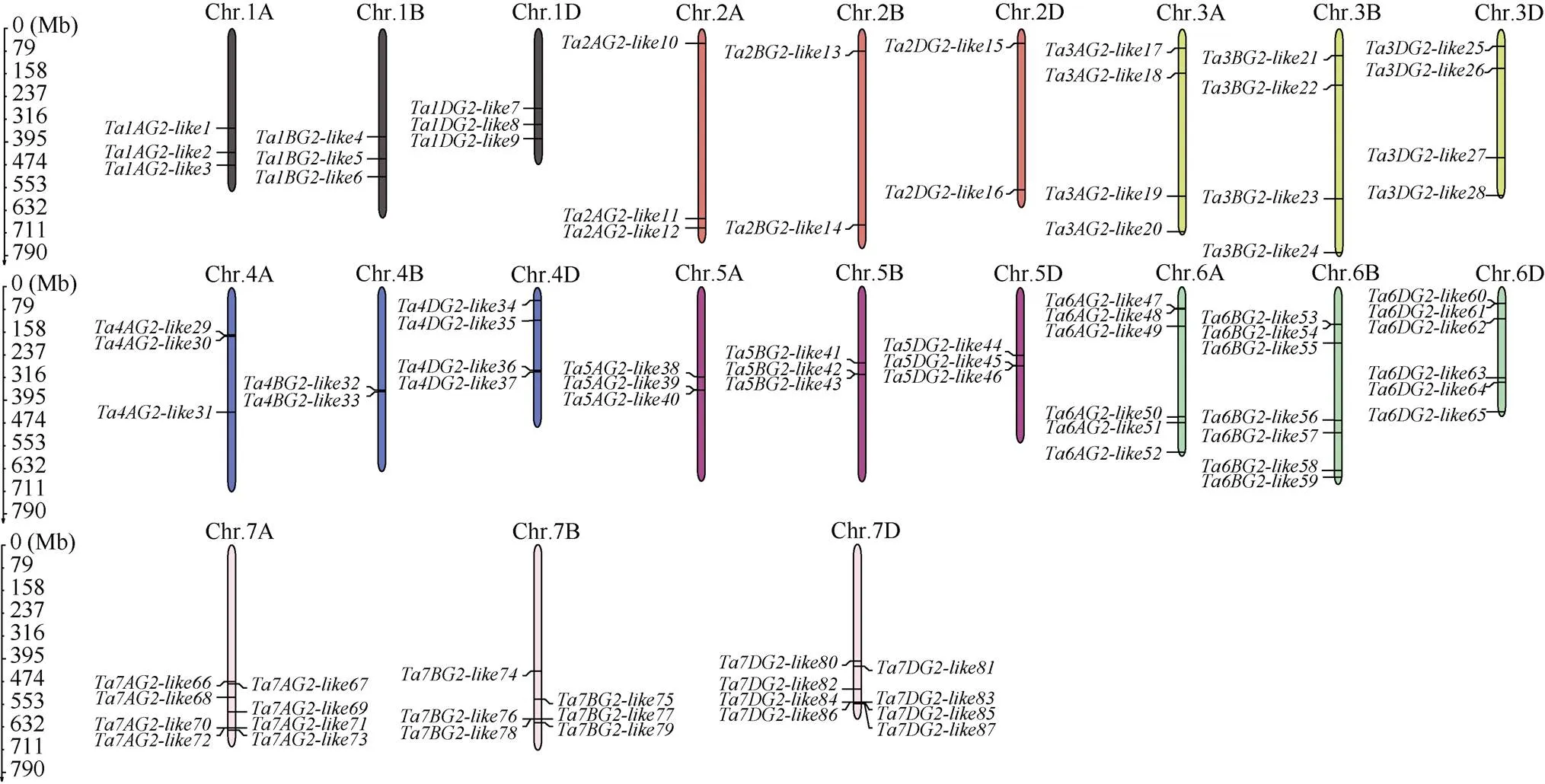
图2 小麦G2-like基因染色体定位
标度代表兆碱基(Mb)。The scale represents Mb.

图3 小麦G2-like基因的染色体位置及共线性关系
所有的共线区域和基因由灰线连接,基因的片段重复基因对用红线标出。
All syntenic blocks and genes are linked by the grey lines, and segmental duplication pairs ofgenes are highlighted by red lines.

图4 小麦G2-like基因启动子区域与抗逆相关的7种顺式作用元件
顺式作用元件用不同的彩色方框表示。
Different colored boxes indicate different cis-acting elements in the scale at the bottom.
2.5 小麦G2-like基因组织表达及逆境胁迫下表达分析
基因的表达模式分析表明(图5),基因家族可分为5类(A~E), 其中大部分基因在叶中均有表达; 模式A和E中的基因几乎在根、叶、穗和籽粒的所有时期都有较高水平表达; 模式B中的基因主要在根中表达; 模式D主要是在叶和穗中表达; 在模式C中有12个的基因在不同组织和不同时期的表达量都很低或没有表达。
对候选小麦基因在干旱、高温及干旱和高温双重胁迫下的表达情况分析(图6), 发现大部分基因在胁迫下均有较高的表达量, 表达模式基本相同。、、、、、和在3类胁迫下表达量均较高; 特别是, 其表达量在3种胁迫下最高, 表明这个基因在小麦遭受热、干旱胁迫时可能发挥重要作用。、、和在干旱胁迫下的表达量显著高于热胁迫, 表明这4个基因对干旱胁迫可能更加敏感。但是、、、和在不同的胁迫下的表达量均较低或者没有, 可能是这5个基因对逆境胁迫不敏感。

图5 不同时期籽粒、叶片、根和穗中87个G2-like基因表达谱热图
S: 幼苗期; V: 期; R: 生殖期。红色或绿色代表每个样品中每个转录本的较高或较低的相对丰度。
S: at seedling stage; V: at vegetative stage; R: at reproductive stage. The red or green colors stand for the higher or lower relative abundance of each transcript in each sample.

图6 小麦G2-like基因在热、干旱以及热和干旱共同胁迫下的表达模式
D1、D6:干旱处理1、6 h; H1、H6: 高温处理1、6 h; D+H1、D+H6: 在高温和干旱胁迫下处理时间为1、6 h。红色和绿色分别代表每个样品中每个转录本的较高和较低的相对丰度。
D1, D6: drought treatment of 1 h and 6 h; H1, H6: heat treatment of 1 h and 6 h; D+H1, D+H6: heat and drought treatments of 1h and 6 h. The red and green colors represent the higher or lower relative abundance, respectively.
2.6 小麦G2-like转录因子家族部分基因qRT-PCR验证分析
为探究基因对非生物胁迫及激素的响应, 根据基因在不同组织部位表达量, 以及启动子区域顺式调控元件结合位点数量等综合考虑, 挑选了14个小麦基因进行qRT-PCR表达分析(图7)。结果表明, 在盐胁迫下只有表达量显著降低, 在胁迫24 h后较CK下降了3倍; 而、、、、、、、和表达量显著升高, 其中、升高最为明显, 在胁迫24 h后其表达量相对于CK分别提高了8倍、15倍和11倍。
在干旱胁迫下,、、、和、、和表达量显著升高, 其中和上升最为显著, 在胁迫24 h后其表达量相对于CK分别升高了10倍、13倍和12倍; 而在胁迫下上调最不明显, 在胁迫24 h后相对于CK上升大致为1.3倍。同时发现在盐胁迫下显著上调, 但在干旱胁迫下其表达量并没有显著变化(胁迫12 h除外);在盐胁迫下显著下调, 而在干旱胁迫6 h之后其表达量较对照显著上调, 表明这2个基因在响应干旱和盐胁迫中可能存在差异。
在3种激素诱导下,、、、、、和表达量均显著升高(图8), 其中和升高最为明显, 其表达量相对CK上调10倍以上。、和表达量随GA和IAA诱导时间的延长呈现出先升高后降低的趋势, 且均在诱导1 h之后表达量显著上调。但其对ABA诱导的响应相对较弱,和分别在ABA诱导6 h和24 h、6 h时表达量较对照显著上调; 而在ABA处理12 h之后表达量显著上调。在IAA处理下表达量显著升高, 在诱导24 h后较CK上升3倍左右; 而在3种激素处理下表达量均表现出下降的趋势, 其中在IAA处理下降最明显, 在诱导24 h后较CK下调了0.3倍。
3 讨论
小麦在生产过程经常遭受各种逆境胁迫的影响, 挖掘小麦抗逆基因资源对于小麦抗逆育种有重要作用。植物在面临逆境胁迫时, 其体内转录因子迅速启动相应基因的表达和逆境信号的传递, 启动抗逆应答反应, 在植物逆境应答和生长发育过程中发挥重要作用[9]。转录因子, 是属于MYB类转录因子中GARP超家族的成员, 广泛存在于陆地植物中。本研究在小麦中共鉴定出87个基因, 不均匀的分布在21条染色体上; 通过共线性分析发现小麦基因家族主要由片段重复产生, 同时还有3个串联重复基因对; 植物基因家族扩张的主要途径是串联重复和片段重复[30], 片段复制在进化较慢的基因家族中是有规律出现的。计算Ka/Ks值后发现2种重复基因对比值均小于1, 表明基因在进化过程中可能是相对保守的, 进化较慢。
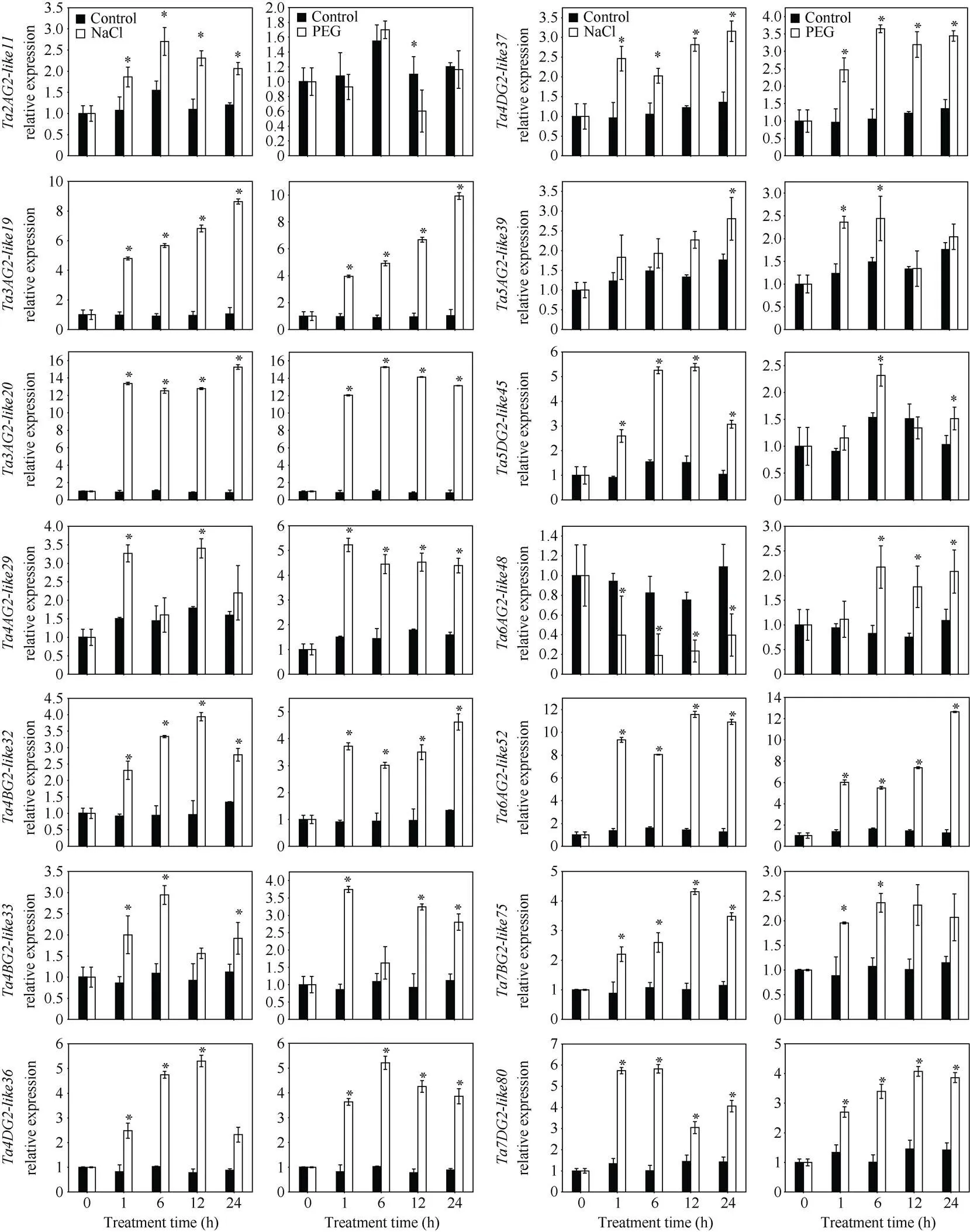
图7 定量实时PCR分析14个G2-like基因对NaCl、PEG处理的响应
*表示处理间存在显著差异(< 0.05)。
* indicates significant differences between the stress conditions and the control condition (< 0.05).
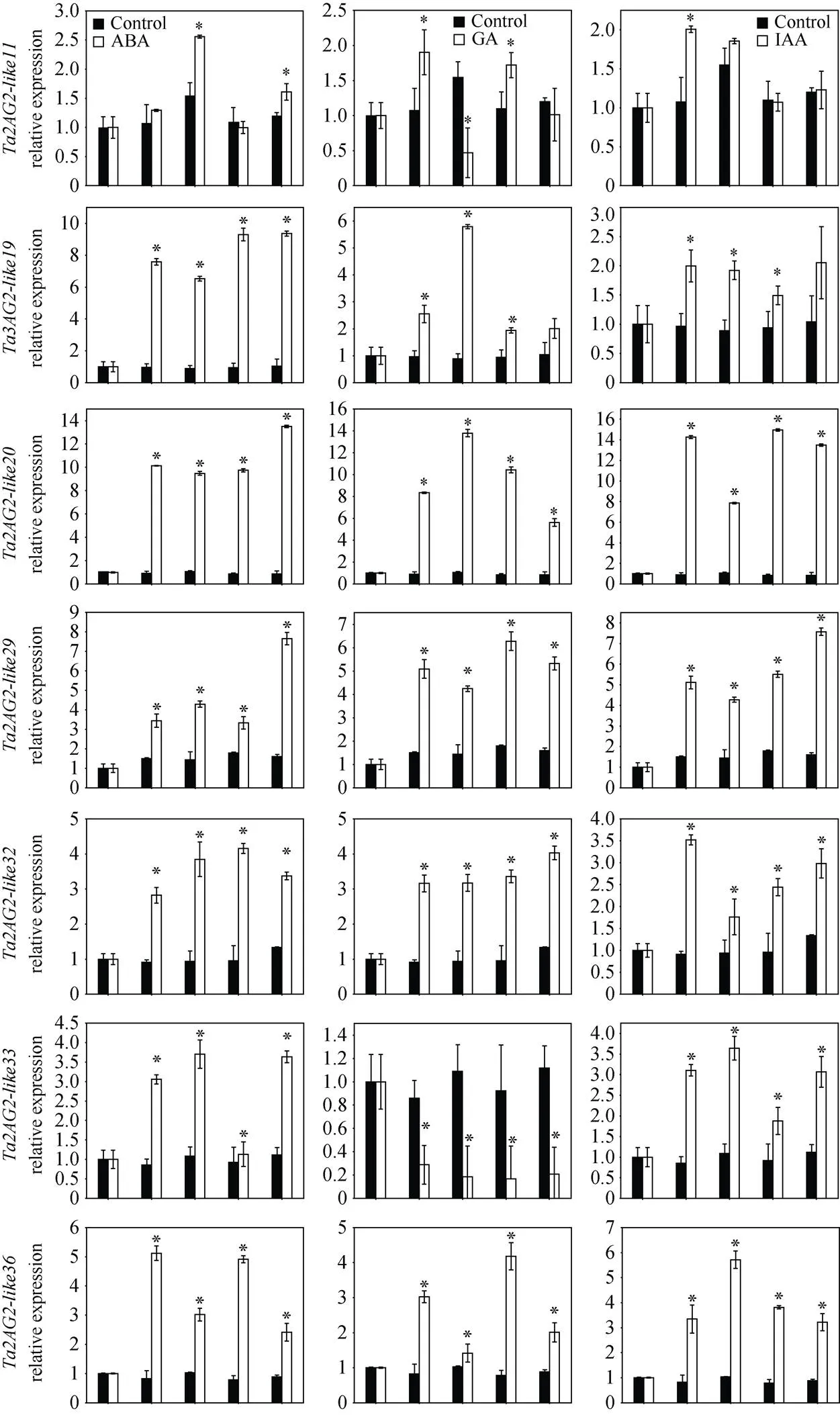
(图8)
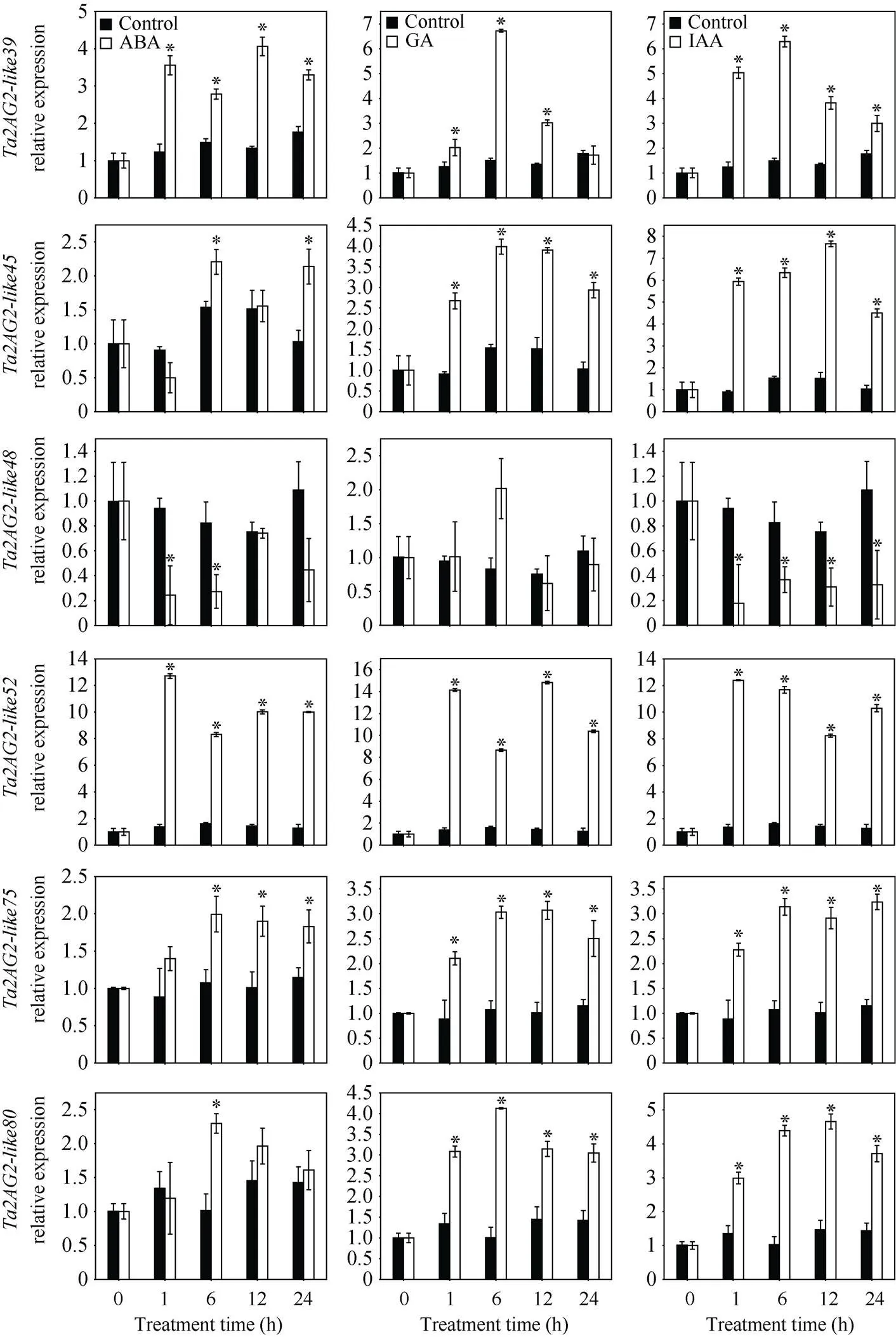
图8 定量实时PCR分析14个G2-like基因对ABA、GA和IAA处理的响应
*表示处理间存在显著差异(< 0.05)。
* indicates significant differences between the stress conditions and the control condition at< 0.05.
位于启动子区域的顺式作用元件通过对不同环境信号的响应来调控基因转录, 从而影响植物的生长发育[31]。在玉米中发现与激素、干旱和低温诱导响应有关的MBS、P-box、Sp1、TGA-element和AE-box顺式作用元件[32]; 在水稻中发现的ABRE顺式调控元件, 参与干旱诱导和脱落酸诱导[33-34]; LTR顺式调控元件, 在拟南芥盐胁迫中发挥重要作用[35]。本研究发现, 小麦基因启动子区域含有多种顺式作用元件, 包括MBS、P-box、ABRE等响应逆境胁迫元件, 表明小麦基因在参与逆境胁迫中可能发挥重要作用。已有研究结果表明基因在植物叶绿体的发育具有重要作用[36]。对不同组织部位基因表达模式分析表明, 不同基因在小麦叶片中均有表达, 且在干旱、高温以及干旱和高温共同胁迫下,基因都有较高的表达量, 表明基因可能在叶绿体发育和逆境胁迫响应中均发挥着重要作用, 但其具体的调控机制仍需进一步研究。
玉米中有10个基因在干旱和低温胁迫下表达量均上调, 其中基因在低温和干旱胁迫下表达量显著上调[19]。刘俊芳等[17]发现盐处理后有6个基因表达上调, 干旱处理后有3个基因表达上调, 其中基因在干旱和低温胁迫中应答明显, 沉默后番茄植株对干旱和低温的抗性降低。本研究中通过实时荧光定量分析发现, 14个基因中, 几乎所有的基因在盐胁迫和干旱胁迫下显著上调, 表明多数基因在盐和干旱胁迫下表现为正向调控; 同时发现基因在干旱胁迫下显著下调; 表明尽管转录因子的蛋白氨基酸序列相似, 但转录因子对干旱胁迫的调控机制不尽相同。其中、和在这两种胁迫下表达量上调幅度最高, 可能在小麦抗盐和抗旱中发挥重要作用, 有关其功能验证及作用机理有待进一步研究。
ABA信号转导途径是植物响应盐和干旱胁迫的关键途径之一;双突变体在拟南芥种子萌发和幼苗发育期间表现出对ABA高度敏感, 在幼苗发育期间也有抗渗透胁迫的作用;基因调控包括WRKY40在内的几种ABA响应基因, 在ABA反应中起负调控作用[37]。本研究中多数基因在ABA诱导下显著上调, 与干旱胁迫下表达模式相似, 表明这些转录因子参与干旱信号转导途径可能需借助ABA信号途径; 而表达量对ABA响应程度较小, 可能其不依赖ABA信号途径对抗旱能力进行调控。如在水稻的研究中表明参与介导植物对ABA和干旱的反应[38],而过表达植株对外源ABA没有响应[39]。在沉默了抑制生长素响应的基因株系中,的表达升高[40]。本研究14个基因中, 几乎所有的基因均在IAA和GA诱导下显著上调, 只有和分别在IAA和GA诱导下显著下调表达, 表明不同转录因子对激素的响应模式存在一定差异, 可能与其参与植株生长发育和逆境胁迫响应的调控模式有关。多数基因对胁迫和激素诱导均明显响应, 其中、、和在不同胁迫和激素处理下应答最为明显, 表明这4个基因可能在小麦抗逆应答过程中发挥着重要作用。同时必须指出的是,本研究在逆境胁迫和激素诱导基因表达分析中, 仅选用了1个品种, 且胁迫类型和激素种类较少; 尽管百农207为适应性好、综合抗逆能力强的品种, 但选择抗逆能力不同小麦品种、同时增加逆境胁迫和激素种类可能更有利于抗逆相关基因鉴定, 关于基因对不同逆境胁迫和激素诱导的响应需进一步验证。根据基因表达模式并结合生信分析,可能在小麦抗逆中发挥重要作用。通过载体构建和遗传转化, 已经成功获得了10株过表达的转基因株系, 有关过表达株系在逆境下的表型及相应生理机制有待进一步研究。
[1] Janmohannadi M, Zolla L, Rinalducci S M. Low temperature tolerance in plants: changes at the protein level., 2015, 117: 76–89.
[2] Rinalducci S, Egidi M G, Karimzadeh G, Jazii F R, Zolla L. Proteomic analysis of a spring wheat cultivar in response to prolonged cold stress., 2011, 32: 1807–1818.
[3] Gibson L R, Paulsen G. Yield components of wheat grown under high temperature stress during reproductive growth., 1999, 39: 1841–1846.
[4] Farooq M, Hussain M, Siddique K H M. Drought stress in wheat during flowering and grain-filling periods., 2014, 33: 331–349.
[5] Mohammadi R. Efficiency of yield-based drought tolerance indices to identify tolerant genotypes in durum wheat., 2016, 211: 71–89.
[6] Ding H, Ma D, Huang X, Hou J, Wang C, Xie Y, Wang Y, Qin H, Guo T. Exogenous hydrogen sulfide alleviates salt stress by improving antioxidant defenses and the salt overly sensitive pathway in wheat seedlings., 2019, 41: 123.
[7] Saqib M, Akhtar J, Qureshi R H. Pot study on wheat growth in saline and waterlogged compacted soil: I. Grain yield and yield components., 2004, 77: 169–177.
[8] Mizoi J, Shinozaki K, Yamaguchi-Shinozaki K. AP2/ERF family transcription factors in plant abiotic stress responses., 2012, 1819: 86–96.
[9] Chrispeels H E, Oettinger H, Janvier N, Tague B W., encodingC2H2zinc-finger protein 1, is expressed downstream of photomorphogenic activation., 2000, 42: 279–290.
[10] Jenkins M T. A second gene producing golden plant color in maize., 1926, 60: 484–488.
[11] Brand A, Borovsky Y, Hill T, Rahman K A A, Bellalou A, Van Deynze A, Paran I.regulates natural variation of chlorophyll content and fruit color in pepper fruit., 2014, 127: 2139–2148.
[12] Fitter D W, Martin D J, Copley M J, Scotland R W, Langdale J A.gene pairs regulate chloroplast development in diverse plant species., 2002, 31: 713–727.
[13] Powell A L T, Nguyen C V, Hill T, Cheng K L, Figueroa-Balderas R, Aktas H, Ashrafi H, Pons C, Fernández-Muñoz R, Vicente A, Lopez-Baltazar J, Barry C S, Liu Y S, Chetelat R, Granell A, Van Deynze A, Giovannoni J J, Bennett A B.encodes atranscription factor regulating tomato fruit chloroplast development., 2012, 336: 1711–1715.
[14] Jarvis P, López-Juez E. Biogenesis and homeostasis of chloroplasts and other plastids., 2013, 14: 787–802.
[15] Li P, Ponnala L, Gandotra N, Wang L, Si Y, Tausta S L, Kebrom T H, Provart N, Patel R, Myers C R, Reidel E J, Turgeon R, Liu P, Sun Q, Nelson T, Brutnell T P. The developmental dynamics of the maize leaf transcriptome., 2010, 42: 1060–1067.
[16] Chang Y M, Liu W Y, Shih A C-C, Shen M N, Lu C H, Lu M-Y J, Yang H W, Wang T Y, Chen S C-C, Chen S M, Li W H, Ku M S B. Characterizing regulatory and functional differentiation between maize mesophyll and bundle sheath cells by transcriptomic analysis., 2012, 160: 165–177.
[17] 刘俊芳. 番茄G2-like转录因子家族生物信息学分析及抗逆相关基因鉴定. 东北农业大学硕士学位论文, 黑龙江哈尔滨, 2018.
Liu J F. Bioinformatics Analysis of Tomato G2-like Transcription Factor Family and Identification of Resistance-related Genes. MS Thesis of Northeast Agricultural University, Harbin, Heilongjiang, China, 2018 (in Chinese with English abstract).
[18] Petridis A, Döll S, Nichelmann L, Bilger W, Mock H P.G2-LIKE FLAVONOID REGULATOR and BRASSINOSTEROID ENHANCED EXPRESSION1 are low-temperature regulators of flavonoid accumulation., 2016, 211: 912–925.
[19] Liu F, Xu Y, Han G, Zhou L, Ali A, Zhu S, Li X. Molecular evolution and genetic variation oftranscription factor genes in maize., 2016, 11: e0161763.
[20] Nagatoshi Y, Mitsuda N, Hayashi M, Inoue S, Okuma E, Kubo A, Murata Y, Seo M, Saji H, Kinoshita T, Oheme-Takagi M. GOLDEN 2-LIKE transcription factors for chloroplast development affect ozone tolerance through the regulation of stomatal movement., 2016, 113: 4218–4223.
[21] Chen M, Ji M, Wen B, Liu L, Li S, Chen X, Gao D, Li L. GOLDEN 2-LIKE transcription factors of plants., 2016, 7: 1509.
[22] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33: 1870–1874.
[23] Chen C, Xia R, Chen H, He Y. TBtools, a toolkit for biologists integrating various HTS-data handling tools with a user-friendly interface., 2018, 13: 1194–1202.
[24] Wang Y, Tang H, DeBarry J D, Tan X, Li J, Wang X, Lee T, Jin H, Marler B, Guo H, Kissinger J C, Paterson1 A H. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity., 2012, 40: e49.
[25] Krzywinski M, Schein J, Birol İ, Connors J, Gascoyne R, Horsman D, Jones S J, Marra M A. Circos: an information aesthetic for comparative genomics., 2009, 19: 1639–1645.
[26] Zhang Z, Li J, Zhao X Q, Wang J, Wong G K S, Yu J. KaKs_Calculator: calculating Ka and Ks through model selection and model averaging., 2006, 4: 259–263.
[27] Kang C H, Jung W Y, Kang Y H, Kim J Y, Kim D G, Jeong J C, Baek D W, Jin J B, Lee J Y, Kim M O, Chung W S, Mengiste T, Koiwa H, Kwak S S, Bahk J D, Lee S Y, Nam J S, Yun D J, Cho M J. AtBAG6, a novel calmodulin-binding protein, induces programmed cell death in yeast and plants., 2006, 13: 84–95.
[28] Koundrioukoff S, Polo S, Almouzni G. Interplay between chromatin and cell cycle checkpoints in the context of ATR/ATM- dependent checkpoints., 2004, 3: 969–978.
[29] Meshorer E. Chromatin in embryonic stem cell neuronal differentiation., 2007, 22: 311–319.
[30] Cannon S B, Mitra A, Baumgarten A, Young N D, May G. The roles of segmental and tandem gene duplication in the evolution of large gene families in., 2004, 4: 10.
[31] Kong F, Wang J, Cheng L, Liu S, Wu J, Peng Z, Lu G. Genome-wide analysis of the mitogen-activated protein kinase gene family in., 2012, 499: 108–120.
[32] Tao Y, Wang F, Jia D, Li J, Zhang Y, Jia C, Wang D, Pan H. Cloning and functional analysis of the promoter of a stress- inducible gene () in maize., 2014, 33: 200–208.
[33] Lee S C, Kim S H, Kim S R. Drought induciblepromoter is activated by OsDREB1A and OsDREB1D., 2013, 56: 115–121.
[34] Nakashima K, Jan A, Todaka D, Maruyama K, Goto S, Shinozaki K, Yamaguchi‑Shinozaki K. Comparative functional analysis of six drought-responsive promoters in transgenic rice., 2014, 239: 47–60.
[35] 张新宇, 赵兰杰, 李艳军. 盐胁迫对拟南芥基因的诱导表达及其启动子分析. 西北植物学报, 2014, 34(1): 54–59.
Zhang X Y, Zhao L J, Li Y J. Salt Stress induced expression and promoter analysis ofgene in, 2014, 34(1): 54–59 (in Chinese with English abstract).
[36] Yasumura Y, Moylan E C, Langdale J A. A conserved transcription factor mediates nuclear control of organelle biogenesis in anciently diverged land plants., 2005, 17: 1894–1907.
[37] Ahmad R, Liu Y, Wang T J, Meng Q, Yin H, Wang X, Wu Y, Nan N, Liu B, Zheng Y X. GOLDEN2-LIKE transcription factors regulateexpression in response to abscisic acid., 2019, 179: 1844–1860.
[38] Yin X, Cui Y, Wang M, Xia X. Overexpression of a novel MYB-related transcription factor,, confers improved drought tolerance and decreased ABA sensitivity in rice., 2017, 490: 1355–1361.
[39] Guo H, Wu T, Li S, He Q, Yang Z, Zhang W, Gan Y, Sun P, Xiang G, Zhang H, Deng H. The methylation patterns and transcriptional responses to chilling stress at the seedling stage in rice., 2019, 20: 5089–5106.
[40] Sagar M, Chervin C, Mila I, Hao Y, Roustan J, Benichou M, Gibon Y, Biais B, Maury P, Latche A, Pech J C, Bouzayen M, Zouine M. SlARF4, an auxin response factor involved in the control of sugar metabolism during tomato fruit development., 2013, 161: 1362–1374.
Genome-wide identification and expression analysis of G2-like transcription factors family genes in wheat
JIA Yu-Ku**, GAO Hong-Huan**, FENG Jian-Chao, HAO Zi-Rui, WANG Chen-Yang, XIE Ying-Xin, GUO Tian-Cai, and MA Dong-Yun*
College of Agronomy, Henan Agricultural University / National Engineering Research Center for Wheat / Technology Innovation Center of Henan Wheat, Zhengzhou 450046, Henan, China
() transcription factor, a member of the GARP superfamily of MYB transcription factors, plays an important role in regulating chloroplast development. In this study, genome-wide identification ofgenes in wheat was carried out by bioinformatics methods, and their physicochemical properties, subcellular localization, cis-acting elements of promoters, and response patterns to abiotic stresses and hormones were analyzed. A total of 87genes were identified from wheat, which distributed in evenly on 21 chromosomes in wheat. Phylogenetic analysis showed that these genes were divided into 14 subfamilies, and fragment replication was the main reason for the expansion of this gene family. The prediction of protein secondary structure revealed that α helix and random curl were the main amino acid sequences ofgene in wheat. Promoter-acting elements showed that there were seven-acting elements (P-box, SpI, LTR, ABRE, MBS, TGA-Element, and AE-box) in 2-kb region upstream of the promoter. Among them,contained the most-regulatory element binding sites with a total of 18 binding sites. The qRT-PCR revealed. that the relative expression levels of,,, andwere significantly up-regulated under PEG and salt stresses, and induced by GA, IAA, and ABA hormones. These genes may mediate the response of wheat plant to various abiotic stresses.
wheat;transcription factors; bioinformatics; adversitystress; the relative expression level
10.3724/SP.J.1006.2023.21036
本研究由国家科技支撑计划项目(2015BAD26B00)和河南省科技攻关项目(212102110281)资助。
This study was supported by the National Science and Technology Support Program of China (2015BAD26B00) and the Science and Technology Project of Henan Province (212102110281).
马冬云, E-mail: xmzxmdy@126.com
**同等贡献(Contributed equally to this work)
贾玉库, E-mail: 13633969649@163.com; 高宏欢, E-mail: 2972620298@qq.com
2022-05-13;
2022-07-22;
2022-08-18.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220817.1833.011.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
