盐胁迫下不同甘薯品种的转录组测序分析
2023-03-23张小红
张小红 彭 琼 鄢 铮
研究简报
盐胁迫下不同甘薯品种的转录组测序分析
张小红 彭 琼 鄢 铮*
福州市农业科学研究所, 福建福州 350018
为了获得甘薯耐盐转录组序列信息, 挖掘差异表达基因及其相关代谢途径, 本文以盐胁迫处理0 d、3 d和6 d的耐盐甘薯品种‘榕薯819’以及不耐盐甘薯品种‘榕薯910’的叶片为材料, 借助高通量测序技术进行转录组测序分析。结果表明, 2个品种共获得157,252条Unigenes, 平均组装长度为576 bp。其中有83,264条Unigenes在七大数据库中得到注释, 占总数的52.95%。NR注释分类结果显示, 在牵牛花()中比对到同源序列的Unigenes最多, 共43,620条, 占总数的57.05%。Unigenes在KOG数据库中的注释主要富集在普通功能预测(8752个)、信号转导机制(5067个)以及翻译后修饰、蛋白转换、分子伴侣(4471个)中。差异表达分析显示, 在‘榕薯819’中, 盐处理3 d和6 d的样品差异表达基因数分别为323个和3752个, 共参与了33个GO功能分类项和302条KEGG代谢通路。在‘榕薯910’中, 差异表达基因数则分别为5554个和7395个, 共参与了50个GO功能分类项, 涉及了329条KEGG代谢通路。以部分差异表达基因的转录组数据为基础, 进行了表达量热图绘制。结果显示, 注释到淀粉和蔗糖代谢途径的7个差异表达β-葡萄糖苷酶基因均表现为在耐盐品种中上调, 在不耐盐品种中下调; 注释到Ca2+信号途径的7个类钙调素基因中, 有2个在耐盐品种中特异上调, 5个在不耐盐品种中特异下调。差异表达转录因子表达量热图显示,仅在耐盐品种中特异表达,、、和仅在不耐盐品种中特异表达, 而在耐盐品种中下调表达, 在不耐盐品种中上调表达。综上分析, 甘薯耐盐转录组获得的Unigenes数量较大, 序列信息丰富, 盐胁迫下获得的差异表达基因及高丰度转录因子可能在甘薯抵御盐胁迫过程中发挥着重要作用。
甘薯; 盐胁迫; 转录组; 高通量测序
土壤作为农作物的直接载体, 盐渍化发展容易导致耕地土壤质量下降, 农作物减产减收, 严重影响着国家粮食安全生产与发展。合理种植耐盐植物被认为是最有效的改良土壤环境、保障作物生产水平的方法之一[1]。甘薯((L.) Lam.)作为重要的新型能源作物, 种植面积广泛, 具有高产稳产、适应性强、经济效益高等特点[2]。近年来, 甘薯更是被当作盐碱地改良的优势作物, 广泛种植于滨海盐碱土地, 但随着土壤盐渍化发展日趋严重, 过高的土壤盐分大大增加了甘薯的种植难度, 高盐会对甘薯产生毒害作用, 直接影响甘薯的生长发育, 进而导致甘薯减产减收[3-4]。因此, 研究甘薯耐盐相关代谢途径, 了解甘薯响应盐胁迫的分子调控机制, 对甘薯耐盐优良种质资源的筛选与利用具有重要的意义。
目前, 国内外关于甘薯耐盐性的相关研究主要集中在品种选育[5]、生理特性分析[6-7]和耐盐相关基因挖掘与鉴定[8-10]等方面, 在转录组水平上的研究仍落后于水稻、玉米、小麦等作物。而高通量测序技术的出现则大幅度丰富了甘薯在转录组学上的研究, 为甘薯的抗逆分子机理研究提供了重要手段[11-13]。转录组测序技术在甘薯转录组研究中已得到了广泛的应用。Wang等[14]于2010年对甘薯品种‘广薯87’块根进行了高通量转录组测序, 以产生大量转录物序列, 用于甘薯块根形成发育相关基因的挖掘和分子标记开发。Tao等[15]利用Illumina配对末端测序技术, 在甘薯中获得了128,052个转录本, 同时还鉴定出大量的差异表达和特异表达转录本。此外, 还对涉及病毒基因组、淀粉代谢以及潜在胁迫耐受性和抗虫性的基因进行了表达特征的鉴定。Tao等[16]利用RNA测序技术, 鉴定出了可能的甘薯花特异性基因和开花调控相关基因。Li等[17]通过对黄肉甘薯品种‘维多丽’及其突变体‘HBV-3’的转录组测序研究, 挖掘到58,277条转录本和35,909条Unigenes, 并鉴定出参与类胡萝卜素生物合成的22个差异表达基因和31个转录因子。Luo等[18]比较了盐胁迫和对照条件下的甘薯二倍体盐生野生近缘种假厚藤()的转录组信息, 发现有大量的基因参与了假厚藤直系根和叶的盐胁迫响应, 并涉及了4个不同的代谢过程。Zhu等[19]以耐旱型甘薯品种‘徐薯55-2’为研究对象, 通过Illumina高通量测序鉴定出11,359个差异表达基因, 涉及了脱落酸(abscisic acid, ABA)、乙烯(ethylene, ETH)和茉莉酸(jasmonic acid, JA)信号通路, 进一步揭示了甘薯抵御干旱胁迫的分子机制。Arisha等[20]从‘徐紫薯8号’的转录组研究数据库中挖掘到重要的功能基因, 获得了相对丰富的基因表达信息。高通量测序技术虽然已经在甘薯转录组研究中得到了广泛的应用, 但关于甘薯在盐胁迫条件下的转录组测序分析相关研究仍旧较少。本研究利用高通量技术手段, 将生物信息学分析与分子生物学技术相结合, 从转录组学水平研究不同甘薯品种对盐胁迫的应答, 通过对未知序列的挖掘和差异表达基因的分析, 全面了解甘薯响应盐胁迫过程中所涉及的代谢通路, 为揭示甘薯抵御盐胁迫的抗性机理提供理论保障。
1 材料与方法
1.1 试验材料
供试材料为耐盐甘薯品种‘榕薯819’和不耐盐甘薯品种‘榕薯910’, 2个品种均为福州市农业科学研究所自主选育品种。
1.2 方法
1.2.1 供试样品的盐胁迫处理 将甘薯扩繁苗种植于田间, 30 d后, 选取长势一致、含7片完全展开叶片的甘薯苗插入蛭石中, 并浸泡于200 mmol L-1NaCl溶液中, 分别于盐溶液浸泡处理后3 d、6 d对甘薯苗功能叶(顶叶向下数第3叶)进行取样。以处理0 d的样品作为对照组, 将叶片液氮冻存后, 置于-80℃冰箱保存, 用于总RNA提取。试验设置3组生物学重复, 共18份样品。样品名称标记为: 耐盐品种‘榕薯819’的盐胁迫对照组R 1-1~R 1-3; 3 d处理组R 2-1~R 2-3; 6 d处理组R 3-1~R 3-3; 不耐盐品种‘榕薯910’的盐胁迫对照组T 1-1~T 1-3; 3 d处理组T 2-1~T 2-3; 6 d处理组T 3-1~T 3-3。
1.2.2 总RNA提取、cDNA文库构建及高通量测序 总RNA 提取及质量检测、cDNA文库构建以及高通量测序均委托北京组学生物科技有限公司进行。RNA样品检测合格后, 进行mRNA的富集及cDNA文库的构建。文库构建完成后, 通过Qubit 2.0、Agilent 2100及Q-PCR的方法对文库质量进行检测。库检合格后, 进行Illumina高通量测序。
1.2.3 生物信息学分析 对原始数据(raw data)进行数据过滤, 去除其中的接头序列及低质量Reads, 获得高质量的Clean data。采用Trinity[21]组装软件对所有样品的Clean reads进行序列组装, 获得转录本信息, 随后选取转录本中最长的序列作为Unigene序列, 获得甘薯的Unigene库。
文库质量评估合格后, 使用BLAST[22]软件先将组装后的Unigene序列与NR (NCBI non-redundant protein sequences)、Swiss-Prot (A manually annotated and reviewed protein sequence database)、GO (Gene ontology)、COG (Clusters of orthologous groups of proteins)、KOG (Eukaryotic ortholog groups)和KEGG (Kyoto encyclopedia of genes and genomes)这六大公共数据库进行比对, 使用KOBAS 2.0[23]软件获得Unigene在KEGG中的KEGG Orthology结果, 预测完Unigene的氨基酸序列之后使用HMMER[24]软件与Pfam (Protein family)数据库进行比对, 获得Unigene的注释信息。
采用RPKM算法计算Unigene表达量, 其计算公式为:RPKM =×106/(×/103), 式中为比对到某一转录本上的片段数目,为比对到所有转录本上的片段总数,为转录本长度。利用DESeq[25]软件, 以差异倍数(Fold Change)≥2、错误发现率(False Discovery Rate, FDR) < 0.01作为筛选标准, 对18个样品进行差异表达基因筛选。最后对筛选出的差异表达基因进行GO富集分析和KEGG代谢通路分析。采用Bioinformatics在线工具(http://www. bioinformatics.com.cn/)绘制部分差异表达基因和差异表达转录因子的表达量热图。
2 结果与分析
2.1 测序结果评估及组装结果统计
通过Illumina测序, 18个样品共获得533,839,471的Clean reads数。各样品中Clean reads的碱基质量值Q20均高于97%, Q30均高于93%, 表明其碱基识别准确性较高, 测序结果质量良好。由此可见, 该转录组测序结果满足后续组装的质量要求。
为提高测序深度和转录本组装的完整性, 采用Trinity组装软件对所有样品的Clean reads进行序列组装。共获得211,688条转录本, 平均组装长度为759 bp。其中, 长度超过500 bp的转录本有71,388条, 占转录本总数的33.72%; 长度超过1.0 kb的有44,790条, 占转录本总数的21.16%。随后选取转录本中最长的序列作为Unigene序列, 总共获得157,252条Unigene, 平均组装长度为576 bp, N50达到了1064 bp (表1)。其中Unigene长度在500 bp以上的有34,641条, 占Unigene总数的22.03%; 长度在1.0 kb以上的有19,856条, 占总数的12.63%。转录本和Unigenes的长度频数分布见图1。

表1 样品拼装结果统计
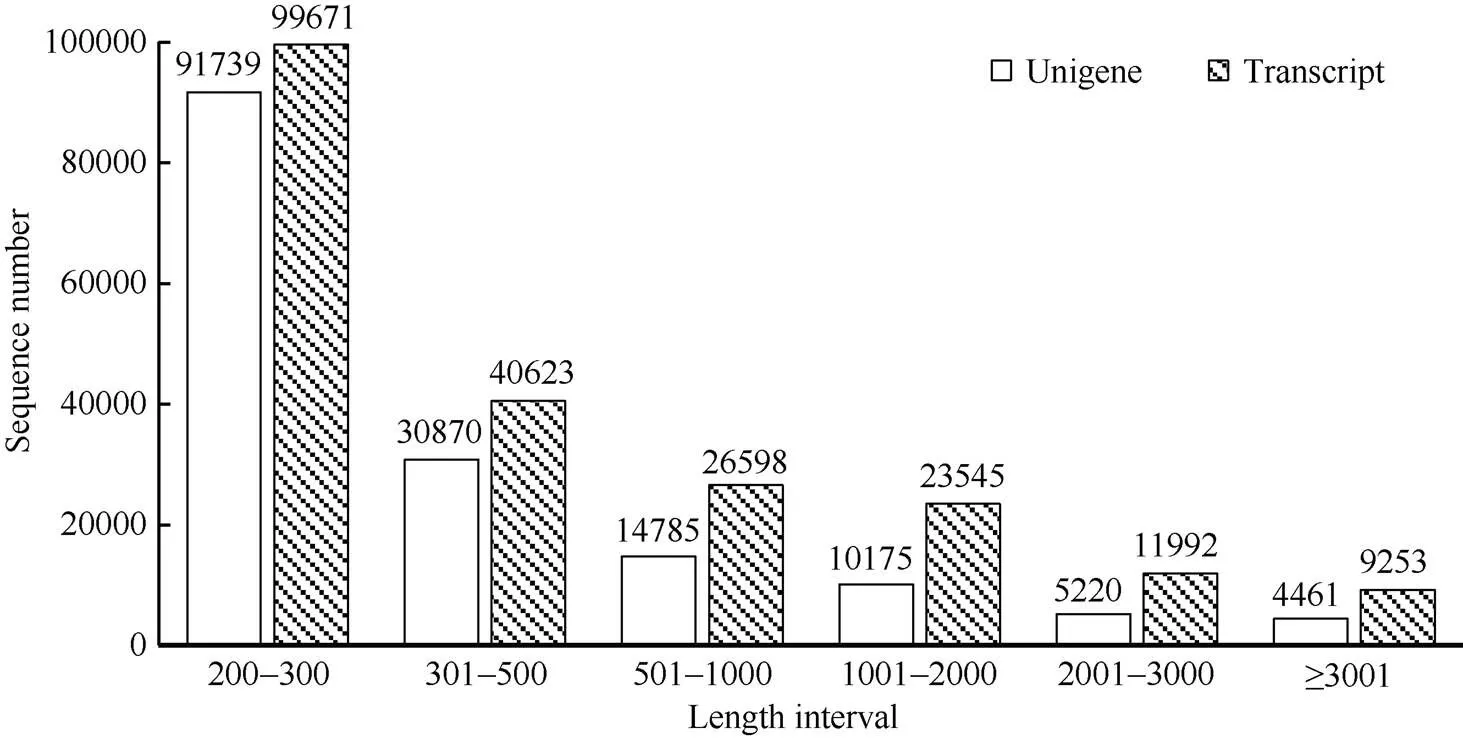
图1 组装所得转录本和Unigenes长度分布图
2.2 Unigene的功能注释
将组装后的Unigenes序列与七大数据库(NR、Swiss-Prot、GO、COG、KOG、KEGG和Pfam)进行比对后(表2)发现, 共有83,264条Unigenes注释成功, 占总数的52.95%, 剩下的73,988条Unigenes均未获得注释, 占总数的47.05%。其中, 注释到NR数据库的Unigenes数量最多, 有76,583条, 占注释成功Unigenes总数的91.98%。
通过与NR数据库的比对(图2)发现, 在牵牛花()中比对到同源序列的Unigenes最多, 共43,620条, 占总数的57.05%。其次为拟除虫菊, 共2786条, 占总数的3.64%, 而与其他物种的相似性均较低(0.82%~2.05%)。
为了进一步分析Unigene的功能, 将甘薯Unigene库与KOG数据库进行比对。结果显示, 42,285个Unigenes共获得47,213个KOG注释, 主要富集在普通功能预测(general functional prediction only, 8752个)、信号转导机制(signal transduction mechanisms, 5067个)以及翻译后修饰、蛋白转换、分子伴侣(posttranslational modification, protein turnover, chaperones)共计4471个(图3)。
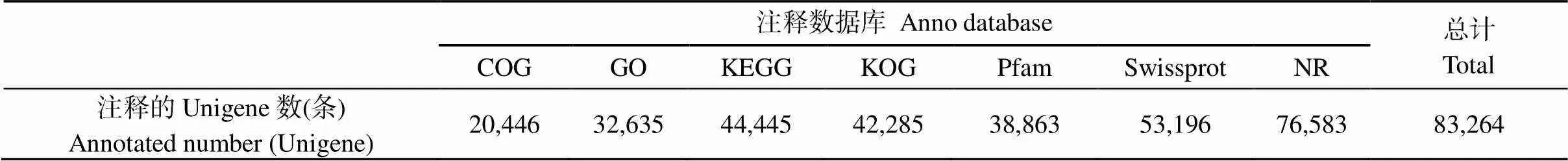
表2 Unigene注释统计表
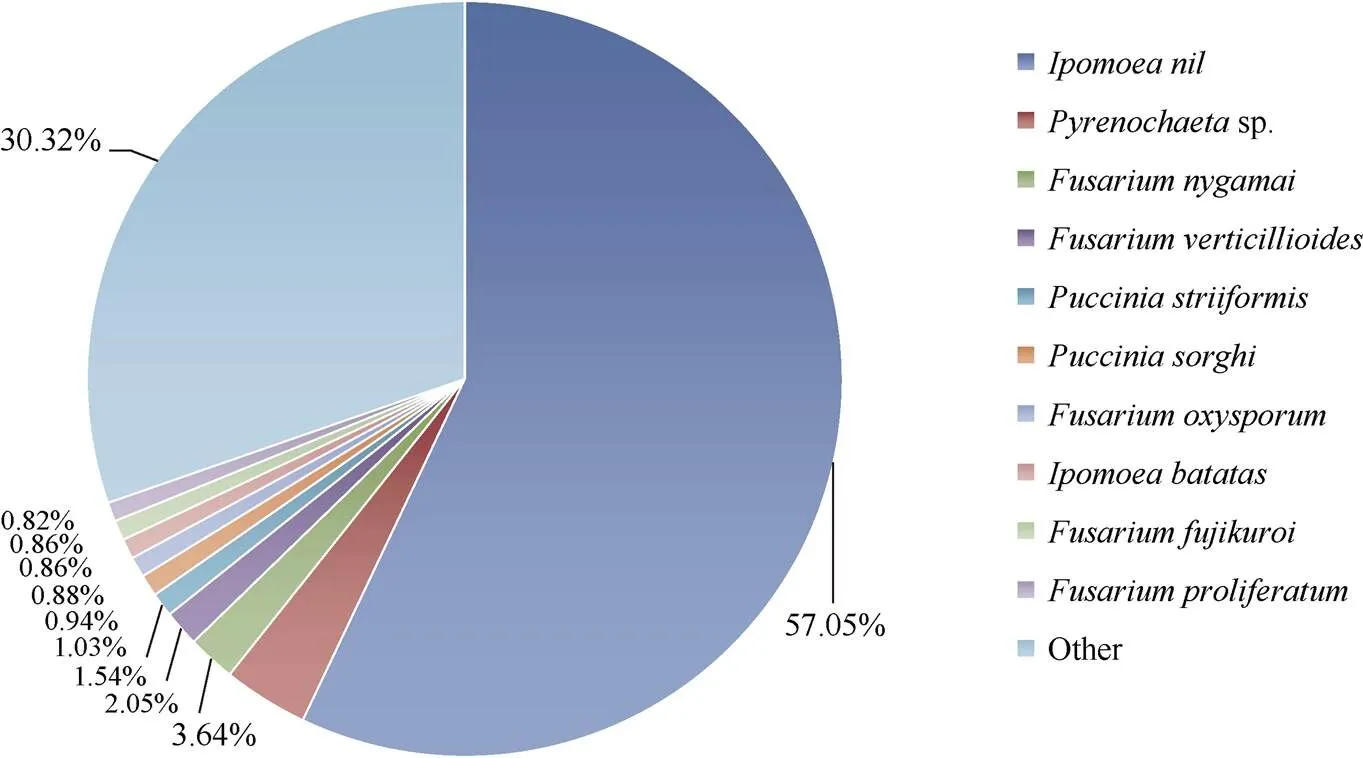
图2 NR注释物种分类图
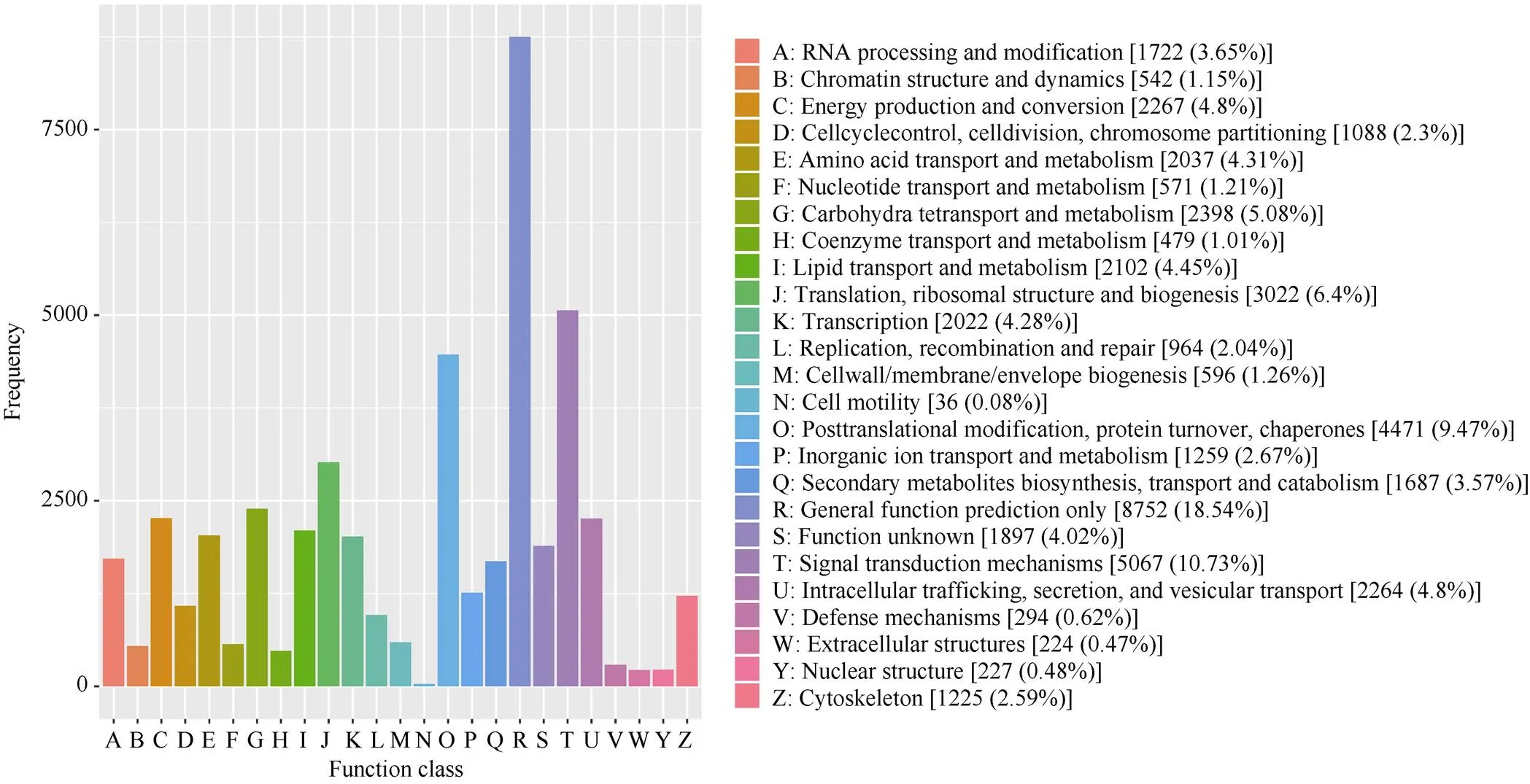
图3 KOG功能注释分类图
2.3 差异表达基因的筛选
利用DESeq进行差异表达基因的分析(表3)发现, 在耐盐品种‘榕薯819’中, 与对照相比, 盐处理3 d和6 d的样品差异表达基因数分别为个323个和3752个, 且差异表达基因的上调数量均略高于下调数量。在不耐盐品种‘榕薯910’中, 盐处理3 d、6 d后的样品与对照相比, 差异表达基因分别有5554个和7395个, 上调表达基因数量明显低于下调表达基因数量。说明, 在盐胁迫条件下, 甘薯不耐盐品种中的差异表达基因远多于甘薯耐盐品种, 且多数为下调表达差异基因。在品种间差异表达分析结果中可以看出, 盐处理6 d后的样品差异表达基因数量要高于对照及盐处理3 d后的样品, 且除对照组外, 不同基因型甘薯在相同的盐处理条件下, 下调表达的差异基因明显多于上调表达差异基因。
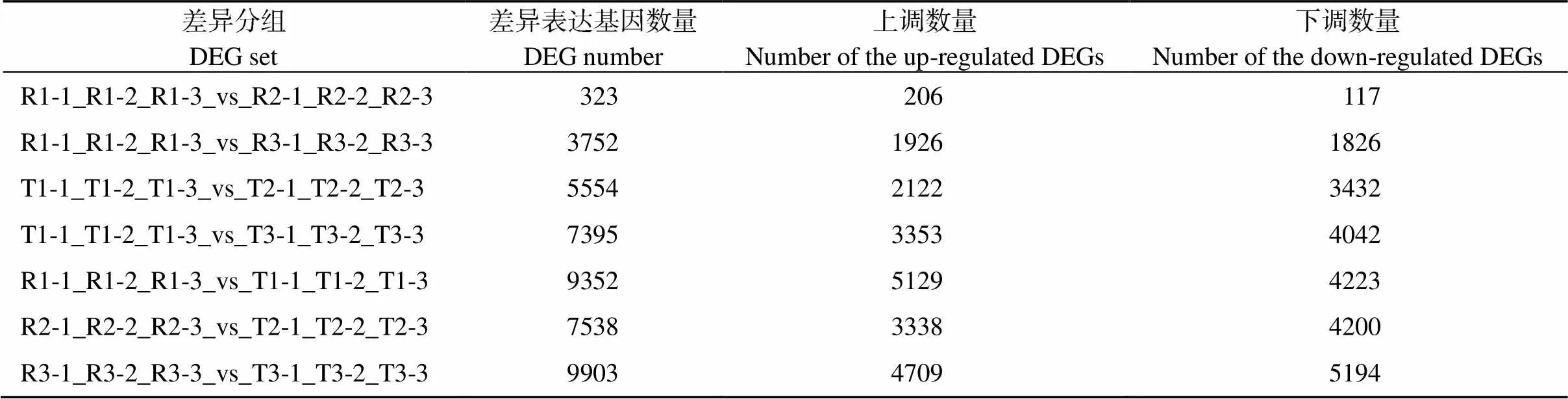
表3 差异表达基因数目统计表
R1-1_R1-2_R1-3_vs_R2-1_R2-2_R2-3和R1-1_R1-2_R1-3_vs_R3-1_R3-2_R3-3分别表示‘榕薯819’对照组与盐胁迫3 d、6 d处理组之间转录组数据的比对组合; T1-1_T1-2_T1-3_vs_T2-1_T2-2_T2-3和T1-1_T1-2_T1-3_vs_T3-1_T3-2_T3-3分别表示‘榕薯910’对照组与盐胁迫3 d、6 d处理组之间转录组数据的比对组合; R1-1_R1-2_R1-3_vs_T1-1_T1-2_T1-3、R2-1_R2-2_R2-3_vs_T2-1_T2-2_T2-3和R3-1_R3-2_R3-3_vs_T3-1_T3-2_T3-3分别表示‘榕薯819’与‘榕薯910’对照组之间、盐胁迫3 d处理组之间和盐胁迫6 d处理组之间转录组数据的比对组合。
R1-1_R1-2_R1-3_vs_R2-1_R2-2_R2-3 and R1-1_R1-2_R1-3_vs_R3-1_R3-2_R3-3 represent the combination between ‘Rongshu 819’ control group and treated group under salt stress for 3 days and 6 days, respectively. T1-1_T1-2_T1-3_vs_T2-1_T2-2_T2-3 and T1-1_T1-2_ T1-3_vs_T3-1_T3-2_T3-3 represent the combination between ‘Rongshu 910’ control group and treated group under salt stress for 3 days and 6 days, respectively. R1-1_R1-2_R1-3_vs_T1-1_T1-2_T1-3 represent the combination between ‘Rongshu 819’ control group and ‘Rongshu 910’ control group. R2-1_R2-2_R2-3_vs_T2-1_T2-2_T2-3 represent the combination between ‘Rongshu 819’ treated group and ‘Rongshu 910’ treated group under salt stress for 3 days. R3-1_R3-2_R3-3_vs_T3-1_T3-2_T3-33 represent the combination between ‘Rongshu 819’ treated group and ‘Rongshu 910’ treated group under salt stress for 6 days.
2.4 差异表达基因功能注释和富集分析
2.4.1 差异表达基因GO分类 对耐盐品种‘榕薯819’不同处理组间的差异表达基因进行GO富集分析发现, 盐胁迫3 d的处理组有154个差异表达基因参与了33个GO功能分类项, 主要富集在分子功能(molecular function)大类中的催化活性(catalytic activity, 92个)和结合(binding, 73个), 以及生物过程(biological process)大类中的代谢过程(metabolic process, 84个) (图4-A)。盐胁迫6 d的处理组有1807个差异表达基因参与了33个GO功能分类项, 其中有949个同时参与了催化活性和代谢过程(图4-B)。
对不耐盐品种‘榕薯910’不同处理组间差异表达基因进行GO富集分析发现, 盐胁迫3 d的处理组有2797个差异表达基因参与了49个GO功能分类项(图4-C), 盐胁迫6 d的处理组则有3601个差异表达基因参与了50个GO功能分类项(图4-D)。2个处理组中, 富集程度最高的3个分类项均为催化活性、代谢过程以及结合, 差异表达基因数分别为催化活性(1520个, 1897个)、代谢过程(1423个, 1846个)以及结合(1293个, 1631个)。
2.4.2 差异表达基因KEGG注释 KEGG分析结果表明, 盐胁迫后‘榕薯819’的差异表达基因共涉及了302条通路, 其中盐胁迫3 d处理组涉及了82条通路, 6 d处理组涉及了302条通路。将富集程度最显著的20条通路进行散点图绘制。结果显示, 当Corrected-value < 0.05时, ‘榕薯819’盐胁迫3 d处理组仅分析到4条代谢通路, 主要有: 单菌素生物合成(ko00261)、光合作用天线蛋白(ko00196)、硫代谢(ko00920)以及硒化合物代谢(ko00450), 均属于代谢途径大类(图5-A); 盐胁迫6 d处理组则分析到22条, 主要有光合作用天线蛋白、单菌素生物合成、糖胺聚糖生物合成-硫酸乙酰肝素/肝素(ko00534)、维生素消化吸收(ko04977)和类黄酮生物合成(ko00941)等, 分属于代谢、有机体系统、环境信息处理和人类疾病四大类(图5-B)。
在‘榕薯910’中, 差异表达基因共涉及了329条通路, 其中盐胁迫3 d和6 d处理组分别涉及了314和327条通路。当Corrected-value < 0.05时, 盐胁迫3 d处理组共分析到29条代谢通路, 主要有: 光合作用天线蛋白、光合作用(ko00195)、苯乙烯降解(ko00643)、二萜生物合成(ko00904)、单萜生物合成(ko00902)、植物激素信号转导ko04075)、NF-kappa B信号通路(ko04064)、Toll样受体信号通路(ko04620)等, 分属于代谢、有机体系统、环境信息处理和人类疾病四大类(图5-C); 盐胁迫6 d处理组则分析到28条通路, 主要有: 光合作用天线蛋白、光合作用、苯乙烯降解、类胡萝卜素生物合成(ko00906)、异喹啉生物碱生物合成(ko00950)、植物激素信号转导、NF-kappa B信号通路、Toll样受体信号通路等, 分属于代谢、细胞过程、有机体系统、环境信息处理和人类疾病五大类(图5-D)。
2.5 部分差异表达基因的表达量分析
在对差异表达基因KEGG的分析中, 选取了2条代谢通路中的关键基因进行表达量热图的绘制(图6)。结果显示, 注释到淀粉和蔗糖代谢途径的7个差异表达β-葡萄糖苷酶基因(TRINITY_DN28654_c0_g1、TRINITY_ DN12057_c2_g1、TRINITY_DN3985_c0_g1、TRINITY_ DN35527_c0_g1、TRINITY_DN9914_c0_g1、TRINITY_ DN9535_c0_g1、TRINITY_DN1683_c0_g1)均表现为在耐盐品种中上调, 在不耐盐品种中下调; 注释到Ca2+信号途径的7个类钙调素基因在2个品种中均发生特异性表达,其中2个(TRINITY_DN2515_c0_g1、TRINITY_DN6673_ c0_g1)在耐盐品种中特异上调, 5个(TRINITY_DN30953_ c0_g1、TRINITY_DN21585_c0_g1、TRINITY_DN23699_ c0_g1、TRINITY_DN2168_c0_g1、TRINITY_DN18268_ c0_g1)在不耐盐品种中特异下调。根据以上结果推测, 这几个基因能够响应盐胁迫并且参与了甘薯抵御盐胁迫的代谢过程。
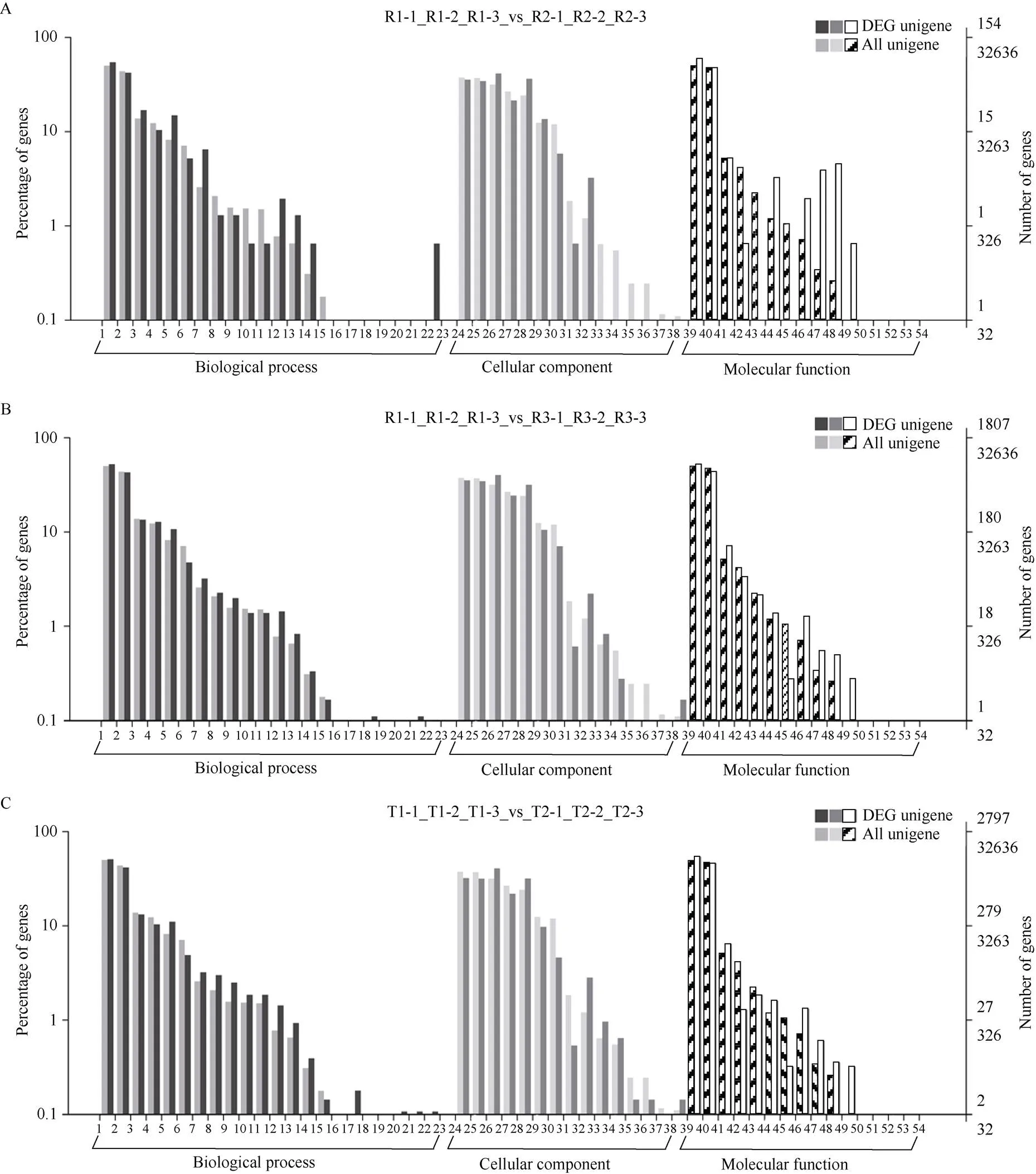
(图4)
样品名称缩写同表3。1: 代谢过程; 2: 细胞过程; 3: 生物调节: 4: 定位; 5: 响应刺激; 6: 细胞成分组织或生物发生; 7; 信号; 8: 发展过程; 9: 多细胞生物过程; 10: 繁殖; 11: 生殖过程; 12: 解毒; 13: 多生物过程; 14: 生长; 15: 免疫系统过程; 16: 移动; 17: 细胞杀伤; 18: 节律过程; 19: 氮肥利用; 20: 细胞群增殖; 21: 色素沉着; 22: 碳利用; 23: 生物粘附; 24: 细胞; 25: 细胞部分; 26: 膜; 27: 细胞器; 28: 膜部分; 29: 细胞器部分; 30: 含蛋白质复合物; 31: 膜腔; 32: 胞外区; 33: 细胞连接; 34: 超分子络合物; 35: 病毒粒子; 36: 病毒粒子部分; 37: 细胞核; 38: 胞外区部分; 39: 催化活性; 40: 结合; 41: 转运活性; 42: 结构分子活性; 43: 转录调节活性; 44: 分子功能调节; 45: 翻译调节活性; 46: 抗氧化活性; 47: 分子转导活性; 48: 小分子传感器活性; 49: 营养贮存活性; 50: 分子载体活性; 51: 蛋白质标签; 52: 货物受体活性; 53: 蛋白质折叠伴侣; 54: 货物适配器活性。
Abbreviations of sample names are the same as those given in Table 3. 1: metabolic process; 2: cellular process; 3: biological regulation; 4: localization; 5: response to stimulus; 6: cellular component organization or biogenesis; 7: signaling; 8: developmental process; 9: multicellular organismal process; 10: reproduction; 11: reproductive process; 12: detoxification; 13: multi-organism process; 14: growth; 15: immune system process; 16: locomotion; 17: cell killing; 18: rhythmic process; 19: nitrogen utilization; 20: cell population proliferation; 21: pigmentation; 22: carbon utilization; 23: biological adhesion; 24: cell; 25: cell part; 26: membrane; 27: organelle; 28: membrane part; 29: organelle part; 30: protein-containing complex; 31: membrane-enclosed lumen; 32: extracellular region; 33: cell junction; 34: supramolecular complex; 35: virion; 36: virion part; 37: Nucleoid; 38: extracellular region part; 39: catalytic activity; 40: binding; 41: transporter activity; 42: structural molecule activity; 43: transcription regulator activity; 44: molecular function regulator; 45: translation regulator activity; 46: antioxidant activity; 47: molecular transducer activity; 48: small molecule sensor activity; 49: nutrient reservoir activity; 50: molecular carrier activity; 51: protein tag; 52: cargo receptor activity; 53: protein folding chaperone; 54: cargo adaptor activity.
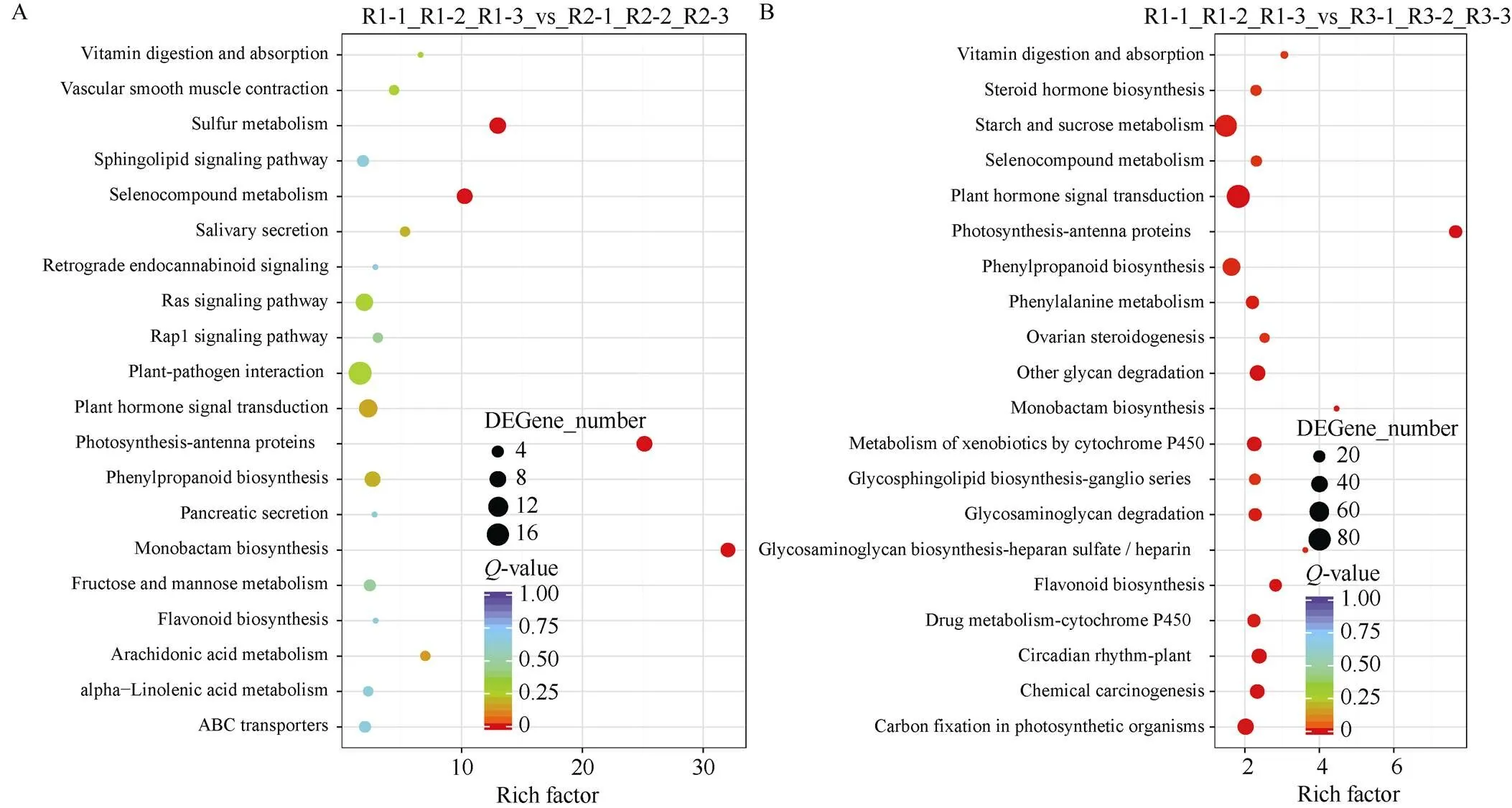
(图5)
样品名称缩写同表3。Abbreviations of sample names are the same as those given in Table 3.
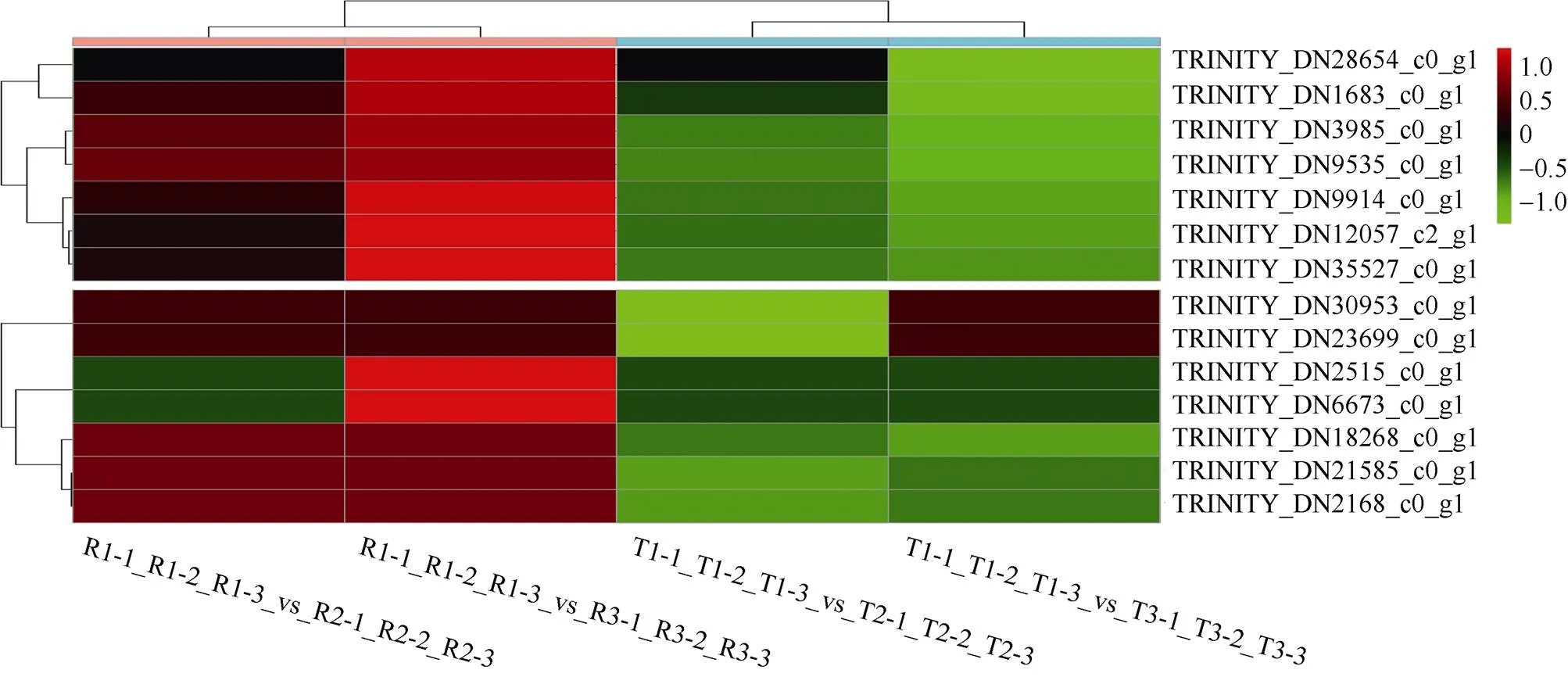
图6 甘薯部分差异表达基因表达量热图
样品名称缩写同表3。Abbreviations of sample names are the same as those given in Table 3.
2.6 差异表达转录因子的分析
盐胁迫处理后有很多响应盐胁迫的转录因子家族基因发生了特异性表达(表4)。‘榕薯819’盐胁迫3 d处理组有166个差异表达基因涉及了45类转录因子家族, 其中C3H 14个、NAC 12个、mTERF 12个、MYB 9个、WRKY 8个、GRAS 6个、C2H2 6个、GNAT 5个、SNF2 5个、AP2/ERF 3个。盐胁迫6 d处理组则有1630个差异表达基因涉及了66类转录因子家族, 其中MYB 131个、C3H 97个、AP2/ERF 88个、C2H2 75个、NAC 69个、B3 63个、GNAT 62个、HB 60个、C2C2 49个、WRKY 38个。
‘榕薯910’盐胁迫3 d、6 d处理组分别鉴定到转录因子家族67个和69个, 包括了2677个和3303个差异表达基因, 其中数量最多的10个转录因子家族分别为MYB (177个, 244个)、AP2/ERF (152个, 182个)、C3H (128个, 166个)、WRKY (113个, 149个)、NAC (117个, 133个)、B3 (114个, 131个)、bHLH (111个, 138个)、HB (98个, 108个)、GNAT (97个, 144个)、C2H2 (96个, 126个)。
2.7 部分差异表达转录因子的表达量分析
本研究从69类转录因子家族中筛选出2类家族(NAC和AP2/ERF)的6个家族成员进行表达量热图绘制(图7)。从图中可以看出, 在NAC家族中,(TRINITY_ DN5042_c0_g1)和(TRINITY_DN6922_c0_g1)仅在不耐盐品种中特异表达, 而(TRINITY_ DN82578_c0_g1)在耐盐品种中下调表达, 在不耐盐品种中上调表达, 且仅在盐处理6 d时有表达, 表明在盐处理前期并不受诱导表达, 随着盐处理时间的延长, 基因得到表达。
在AP2/ERF家族中,(TRINITY_DN113_ c1_g1)仅在耐盐品种中特异下调表达, 而(TRINITY_DN7097_c0_g2)和(TRINITY_DN11829_ c0_g1)则仅在不耐盐品种中特异下调表达, 推测这几个特异表达的转录因子有可能在甘薯抵御盐胁迫过程中发挥了作用。
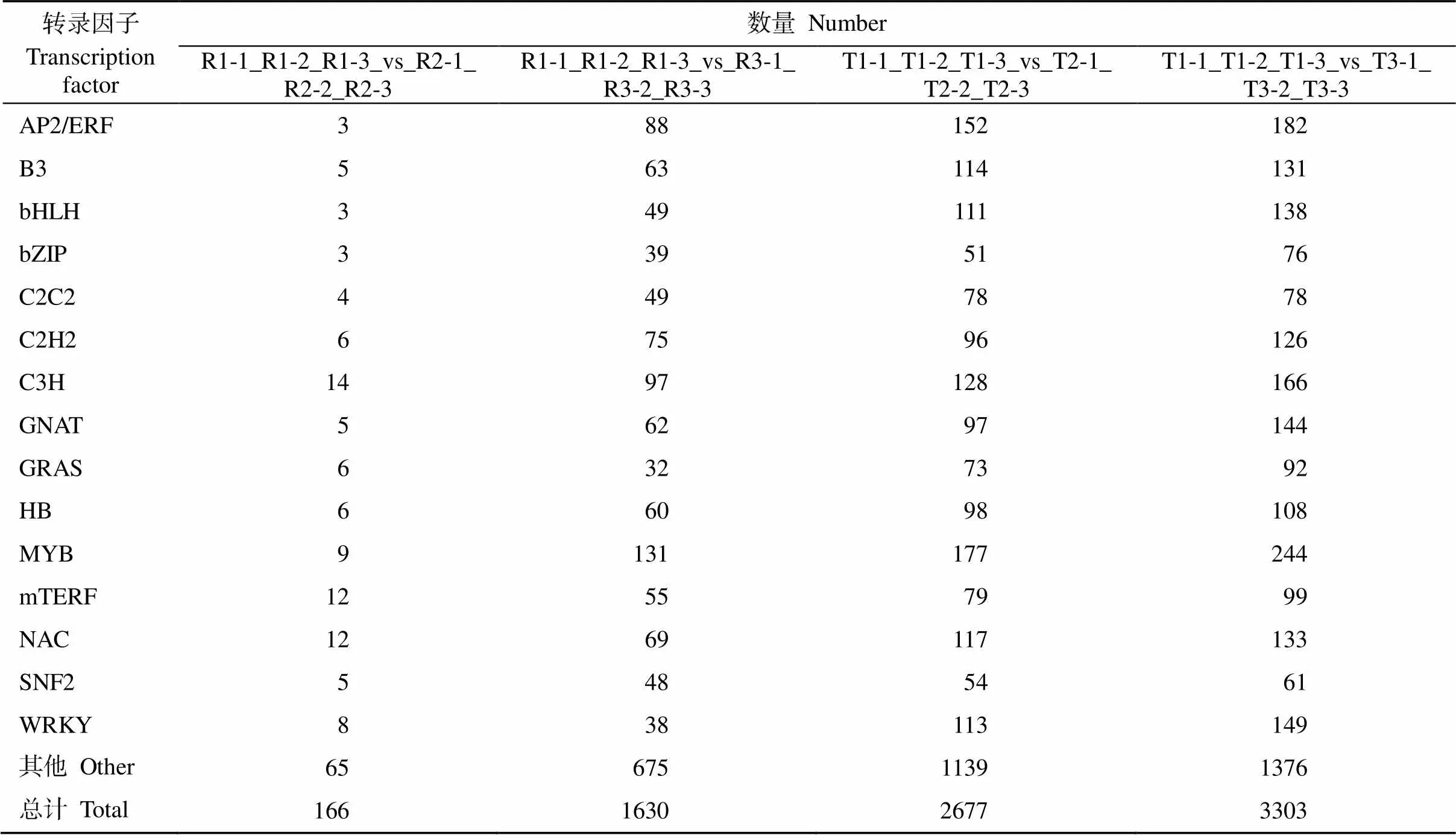
表4 差异表达转录因子数目统计表
样品名称缩写同表3。Abbreviations of sample names are the same as those given in Table 3.

图7 甘薯部分差异表达转录因子表达量热图
样品名称缩写同表3。Abbreviations of sample names are the same as those given in Table 3.
3 讨论
利用高通量测序手段对不同耐盐基因型甘薯品种进行转录组测序分析的研究结果发现, 2个甘薯品种共获得211,688条转录本和157,252条Unigenes, 平均组装长度分别为759 bp和576 bp, 较前人对甘薯转录组的研究而言[15,18,20,26], 获得的Unigenes数量更大, 信息数据更加丰富, 表明本研究测序质量更高、组装效果更好。有83,264条Unigenes在七大数据库中得到注释, 占总数的52.95%, 且注释到NR数据库的Unigenes数量最多, 此结果也超出Arisha等[20]研究中的NR注释数据。在成功注释到NR数据库的Unigenes中, 有57.05%的Unigenes与牵牛花的同源性最高, 与物种间的亲缘关系以及传统的生物学分类结果完全相符[22]。但研究仍有47.05%的Unigenes未能获得注释信息, 表明仍有大量新的转录本有待进一步挖掘与鉴定。
差异表达基因的GO富集分析结果显示, 富集程度最高的3个GO功能分类项为催化活性、代谢过程和结合, 此结果与Wang等[14]和Luo等[18]的研究一致, 而差异表达基因在催化活性和结合中的高富集能力也证明了植物分子功能是造成植物耐盐性差异的主要原因之一[28]。通过KEGG注释分析可知, 在所有的代谢通路中, 参与代谢大类的基因占比最多。其中, 淀粉和蔗糖代谢途径是代谢大类中注释基因最多的一条途径。
在糖代谢过程中, β-葡萄糖苷酶作为参与代谢过程最关键的糖苷水解酶, 对纤维素糖化水解具有重要作用。纤维素在降解过程中经由内切葡聚糖酶和外切葡聚糖酶水解会产生纤维二糖, 纤维二糖在β-葡萄糖苷酶的作用下则会水解形成葡萄糖, 以此释放能量, 用于抵御外界胁迫[29-30]。本研究中, 注释到该途径的7个差异表达β-葡萄糖苷酶基因均表现为在耐盐品种中上调, 在不耐盐品种中下调, 可以推测这几个β-葡萄糖苷酶基因在盐胁迫后的上调作用促进了耐盐品种中葡萄糖的形成, 可能为甘薯抵御盐胁迫过程提供了能量。
此外, 还在Ca2+信号途径中挖掘到7个类钙调素蛋白基因。类钙调素蛋白作为植物中普遍存在的一类Ca2+结合蛋白, 能够介导Ca2+信号与下游基因间的相互作用, 从而响应植物逆境胁迫[31]。Xu等[32]将水稻类钙调素蛋白基因转入拟南芥后, 发现拟南芥的耐盐性明显增加。本研究中, 挖掘到的7个类钙调素蛋白基因在2个品种中均发生了特异性表达, 其中2个在耐盐品种中特异上调, 5个在不耐盐品种中特异下调。因此, 推测这7个类钙调素蛋白基因的特异性表达很可能涉及了甘薯的耐盐生物过程, 在甘薯对盐胁迫的耐受性方面起了重要作用。
多项研究表明, 转录因子MYB[33]、AP2/ERF[34]、C3H[35]、WRKY[36]、NAC[37]和C2H2[38]能够通过对下游基因的调控作用响应盐胁迫, 在植物抵御盐胁迫过程中发挥了重要作用。
NAC家族是一类重要的转录因子家族, 广泛参与了植物的生长发育以及生物和非生物胁迫应答[37]。有研究发现, 在盐胁迫条件下, 转日本结缕草的酵母菌株长势明显弱于对照, 推测在盐胁迫下起负调控作用。随后, 该研究对过表达的拟南芥植株进行盐处理, 发现其生长情况明显弱于野生型植株, 表明降低了拟南芥的耐盐性, 进一步证明了是一个负调控基因[39]。本研究中,仅在不耐盐品种中上调表达, 推测其有可能在盐胁迫条件下通过诱导表达抑制了甘薯的耐盐性, 使得该品种呈现出盐敏感特性。Wang等[40]从番茄中分离出一个基因, 并利用RNAi干扰技术对其进行沉默, 发现相较于野生型植株,突变株的耐盐性明显降低, 表明该基因在盐胁迫下起到正调控作用。本研究中,仅在不耐盐品种中下调表达, 推测其有可能与起到了类似的功能, 调控了该品种耐盐性。是水稻中纤维素合成酶基因(cellulose synthase,)的调节因子, 能够直接结合并上调下游的, 从而激活的表达[41]。是纤维素合成的关键基因, 植物中纤维素含量的变化则会引起植物对非生物和生物胁迫的响应[42]。此外, Han等[43]研究发现, 转拟南芥植株在高盐下的存活率明显高于野生型植株, 且转基因植株呈现出更高的抗氧化酶活性, 推测可能通过增强活性氧的清除能力在抗盐胁迫中发挥了重要作用。本研究中,在不同耐盐品种中呈现差异表达, 且仅在盐处理6 d时有表达, 表明在盐处理前期并不受诱导表达, 随着盐处理时间的延长, 基因得到表达, 推测其在甘薯抵御盐胁迫后期发挥了作用。
AP2/ERF家族是植物中特有的一类转录因子家族, 可以通过结合启动子中的特定基序来调节下游基因的表达[44]。在盐胁迫下, 拟南芥可以与启动子中的DRE元件特异结合从而调控下游基因的表达, 使转基因拟南芥获得更高的抗性, 表明在植物的抗逆性上具有积极正向的调控作用[45]。本研究中,仅在耐盐品种中特异表达, 推测其有可能发挥了自身调节功能, 增强甘薯的耐盐性。尽管大多数AP2/ERF家族成员被当作转录激活因子发挥作用, 但仍有一部分ERF-B1亚族的成员由于包含EAR基序, 被作为转录抑制因子, 通过下调下游基因或其他转录因子的活性发挥作用[46-47]。和都被证实能够受到高盐诱导, 转和转的拟南芥植株相较野生型都呈现出更高的盐敏感性, 表明其在植物抵御逆境过程中起到了抑制作用[46-49]。本研究中,和仅在不耐盐品种中特异表达, 推测这2个基因的特异表达有可能抑制了下游目标基因的表达, 削弱了该甘薯品种的耐盐性。
[1] 牛东玲, 王启基. 盐碱地治理研究进展. 土壤通报, 2002, 33: 449–455.
Niu D L, Wang Q J. Research progress on saline-alkali field control., 2002, 33: 449–455 (in Chinese with English abstract).
[2] 王福琴. 甘薯对盐胁迫的生理响应及甜菜碱合成相关基因的克隆与表达. 山东大学硕士学位论文, 山东济南, 2015.
Wang F Q. Physiological Response to Salt Stress and Cloning, Expression of Betaine Biosynthesis Related Genes in Sweet Potato. MS Thesis of Shandong University, Jinan, Shandong, China, 2015 (in Chinese with English abstract).
[3] 马箐, 于立峰, 孙宏丽, 唐琪, 王宝山, 杜希华. NaCl胁迫对不同甘薯品种体内离子分配的影响. 山东农业科学, 2012, 44(1): 43–46.
Ma J, Yu L F, Sun H L, Tang Q, Wang B S, Du X H. Effect of NaCl stress on ion distribution in plants of different sweet potato varieties., 2012, 44(1): 43–46 (in Chinese with English abstract).
[4] 洪立洲, 邢锦城, 魏福友, 刘冲, 董静, 吴春. 滩涂地区不同种植密度下甘薯生长对盐渍土壤理化特性的影响. 湖南农业科学, 2018, (7): 52–55.
Hong L Z, Xing J C, Wei F Y, Liu C, Dong J, Wu C. Effects of sweet potato growth on saline soil physicochemical properties under different planting densities in coastal beach., 2018, (7): 52–55 (in Chinese with English abstract).
[5] 王文婷, 侯夫云, 王庆美, 李爱贤, 郭钢, 张立明. 耐盐性甘薯品种的初步筛选. 山东农业科学, 2012, 44(11): 35–37.
Wang W T, Hou F Y, Wang Q M, Li A X, Guo G, Zhang L M. Initial screening of sweet potato varieties with salt tolerance., 2012, 44(11): 35–37 (in Chinese with English abstract).
[6] 过晓明, 李强, 王欣, 马代夫. 盐胁迫对甘薯幼苗生理特性的影响. 江苏农业科学, 2011, 39(3): 107–109.
Guo X M, Li Q, Wang X, Ma D F. Effects of salt stress on physiological characteristics of sweet potato seedlings., 2011, 39(3): 107–109 (in Chinese).
[7] 霍恺森, 成小威, 曹清河, 唐君, 朱国鹏, 陈艳丽. 甘薯近缘野生种马鞍藤对盐胁迫的生理生化响应特征. 江苏农业科学, 2019, 47(3): 94–98.
Huo K S, Cheng X W, Cao Q H, Tang J, Zhu G P, Chen Y L. Physiological and biochemical responses of wild species of sweet potato [(L.) R. Brown] to salt stress., 2019, 47(3): 94–98 (in Chinese).
[8] Wang L J, He S Z, Zhai H, Liu D G, Wang Y N, Liu Q C. Molecular cloning and functional characterization of a salt tolerance-associated genefrom sweet potato., 2013, 12: 27–35.
[9] 余静, 孟小庆, 娜菲莎·艾买提江, 李格, 张颖, 李淑清, 朱明库. 甘薯盐胁迫响应基因的克隆、生物信息学及表达模式分析. 四川农业大学学报, 2019, 37: 828–835.
Yu J, Meng X Q, Amatjian N, Li G, Li G, Zhang Y, Li S Q, Zhu M K. Cloning, bioinformatics and expression analysis of salt-responsivegene from sweet potato ()., 2019, 37: 828–835 (in Chinese with English abstract).
[10] 李格, 孟小庆, 李宗芸, 朱明库. 甘薯盐胁迫响应基因的表达特征及生物信息学分析. 植物学报, 2020, 55: 41–51.
Li G, Meng X Q, Li Z Y, Zhu M K. Expression patterns and bioinformatic analyses of salt stress responsive genein., 2020, 55: 41–51 (in Chinese with English abstract).
[11] 于聘飞, 王英, 葛芹玉. 高通量DNA测序技术及其应用进展. 南京晓庄学院学报, 2010, (3): 1–5.
Yu P F, Wang Y, Ge Q Y. High-fluxed DNA sequencing technology and its application development., 2010, (3): 1–5 (in Chinese with English abstract).
[12] Martin J A, Wang Z. Next-generation transcriptome assembly., 2011, 12: 671–682.
[13] 颜朗, 魏昌赫, 张义正. 甘薯‘徐薯18’转录组分析. 植物生理学报, 2017, 53: 772–780.
Yan L, Wei C H, Zhang Y Z. Transcriptomic analyses in sweetpotato [(L.) Lam. cv. ‘Xushu 18’]., 2017, 53: 772–780 (in Chinese with English abstract).
[14] Wang Z Y, Fang B P, Chen J Y, Zhang X J, Luo Z X, Huang L F, Chen X L, Li Y J. Deassembly and characterization of root transcriptome using Illumina paired-end sequencing and development of cSSR markers in sweet potato ()., 2010, 11: 726.
[15] Tao X, Gu Y H, Wang H Y, Zheng W, Xiao L, Zhao C W, Zhang Y Z. Digital gene expression analysis based on integratedtranscriptome assembly of sweet potato [(L.) Lam.]., 2012, 7: 36234.
[16] Tao X, Gu Y H, Jiang Y S, Zhang Y Z, Wang H Y. Transcriptome analysis to identify putative floral-specific genes and flowering regulatory-related genes of sweet potato., 2013, 77: 2169–2174.
[17] Li R J, Zhai H, Kang C, Liu D G, He S Z, Liu Q C.transcriptome sequencing of the orange-fleshed sweet potato and analysis of differentially expressed genes related to carotenoid biosynthesis., 2015, 2015: 843802.
[18] Luo Y, Reid R, Freese D, Li C B, Watkins J, Shi H Z, Zhang H Y, Loraine A, Song B H. Salt tolerance response revealed by RNA-Seq in a diploid halophytic wild relative of sweet potato., 2017, 7: 9624.
[19] Zhu H, Zhou Y Y, Zhai H, He S Z, Zhao N, Liu Q C. Transcriptome profiling reveals insights into the molecular mechanism of drought tolerance in sweet potato., 2019, 18: 9–23.
[20] Arisha M H, Aboelnasr H, Ahmad M Q, Liu Y J, Tang W, Gao R F, Yan H, Kou M, Wang X, Zhang Y G, Li Q. Transcriptome sequencing and whole genome expression profiling of hexaploid sweet potato under salt stress., 2020, 21: 197.
[21] Grabherr M G, Haas B J, Yassour M, Levin J Z, Thompson D A, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q D, Chen Z H, Mauceli E, Hacohen N, Gnirke A, Rhind N, Palma F D, Birren B W, Nusbaum C, Lindblad-Toh K, Friedman N, Regev A. Trinity: reconstructing a full-length transcriptome without a genome from RNA-Seq data., 2011, 29: 644–652.
[22] Altschul S F, Madden T L, Zhang J, Zhang Z, Miller W, Lipman D J. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs., 1997, 25: 3389–3402.
[23] Xie C, Mao X, Huang J, Wu J, Dong S, Lei K, Ge G, Li C Y, Wei L. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases., 2011, 39: W316–W322.
[24] Eddy S R. HMMER: profile HMMs for protein sequence analysis., 1998, 14: 755–763.
[25] Anders S, Huber W. Differential expression analysis for sequence count data., 2010, 11: R106.
[26] Firon N, Labonte D, Villordon A, Kfir Y, Solis J, Lapis E, Perlman T S, Doron-Faigenboim A, Hetzroni A, Althan L. Transcriptional profiling of sweetpotato () roots indicates down-regulation of lignin biosynthesis and up-regulation of starch biosynthesis at an early stage of storage root formation., 2013, 14: 460–460.
[27] 勾晓婉, 侯文倩, 平艳飞, 韩永华, 李宗芸. 甘薯及其野生近缘种的基因组测序研究进展. 江苏师范大学学报(自然科学版), 2020, 38(2): 52–54.
Gou X W, Hou W Q, Ping Y F, Han Y H, Li Z Y. Research advance in genomic sequencing of sweetpotato and its wild relatives.(Nat Sci Edn), 2020, 38(2): 52–54 (in Chinese with English abstract).
[28] 张飞, 王艳秋, 朱凯, 张志鹏, 朱振兴, 卢峰, 邹剑秋. 不同耐盐性高粱在盐逆境下的比较转录组分析. 中国农业科学, 2019, 52: 4002–4015.
Zhang F, Wang Y Q, Zhu K, Zhang Z P, Zhu Z X, Lu F, Zou J Q. Comparative transcriptome analysis of different salt tolerance sorghum (L. Moench) under salt stress., 2019, 52: 4002–4015 (in Chinese with English abstract).
[29] 来亚鹏, 刘刚, 王娟. 嗜热真菌β-葡萄糖苷酶基因克隆表达与调控的研究进展. 纤维素科学与技术, 2017 , 25(2): 69–76.
Lai Y P, Liu G, Wang J. Advances on gene cloning, expression and regulation of β-glucosidase from., 2017, 25(2): 69–76 (in Chinese with English abstract).
[30] 谢欠影, 曹晟阳, 赵晨阳, 伊凯, 戴峰, 秦会发, 秦玉雪, 方蕾.翅碱蓬响应高盐胁迫的分子机制研究. 大连海洋大学学报, 2019, 34(2): 160–167.
Xie Q Y, Cao S Y, Zhao C Y, Yi K, Dai F, Qin H F, Qin Y X, Fang L. Study on the molecular mechanisms ofin response to high salt stress., 2019, 34(2): 160–167 (in Chinese with English abstract).
[31] 杨秀, 许艳超, 杨芳芳, 蔡小彦, 侯宇清, 王玉红, 王星星, 王坤波, 刘方, 周忠丽. 棉花基因家族成员鉴定与功能分析. 棉花学报, 2019, 31: 307–318.
Yang X, Xu Y C, Yang F F, Cai X Y, Hou Y Q, Wang Y H, Wang X X, Wang K B, Liu F, Zhou Z L. Identification and functional analysis ofgene family in cotton., 2019, 31: 307–318 (in Chinese with English abstract).
[32] Xu G Y, Rocha P, Wang M L, Xu M L, Cui Y C, Li L Y, Zhu Y X, Xia X J. A novel rice calmodulin-like gene,, enhances drought and salt tolerance and increases ABA sensitivity in., 2011, 234: 47–59.
[33] Liao Y, Zou H F, Wang H W, Zhang W K, Ma B, Zhang J S, Chen S Y. Soybean,, andgenes confer stress tolerance in transgenicplants., 2008, 18: 1047–1060.
[34] Zhang G, Ming C, Li L, Xu Z S, Chen X P, Guo J M, Ma Y J. Overexpression of the soybeangene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco., 2009, 60: 3781–3796.
[35] Seok H Y, Nguyen L V, Park H Y, Tarte V N, Ha J, Lee S Y, Moon Y H.non-TZF genefunctions as a positive regulator in salt stress response., 2018, 498: 954–959.
[36] 蔡荣号, 李尉, 陈浩伟, 罗国伟, 戴浩然, 宋文雷, 伯晨. 异源表达玉米基因增强拟南芥对盐胁迫的敏感性. 安徽农业大学学报, 2019, 46(6): 145–152.
Cai R H, Li W, Chen H W, Luo G W, Dai H R, Song W L, Bo C. The maizegene negatively regulates salt stress tolerance in transgenic., 2019, 46(6): 145–152 (in Chinese with English abstract).
[37] Liu Q L, Xu K D, Zhao L J, Pan Y Z, Jiang B B, Zhang H Q, Liu G L. Overexpression of a novel chrysanthemum NAC transcription factor gene enhances salt tolerance in tobacco., 2011, 33: 2073–2082.
[38] 苏莹, 甄军波, 张曦, 王玉美, 华金平. 陆地棉转录因子基因的克隆与功能分析. 棉花学报, 2016, 28: 555–564.
Su Y, Zhen J B, Zhang X, Wang Y M, Hua J P. Cloning and functional analysis of a transcription factor gene, G, in upland cotton (L.)., 2016, 28: 555–564 (in Chinese with English abstract).
[39] Jiang H Y, Fan X F, Wen H F, Han C, Teng W J, Teng K, Yin S X. Functional characterization ofgene in response to salt stress., 2021, 38: 1706–1714.
[40] Wang L L, Hu Z L, Zhu M Q, Zhu Z G, Hu J T, Qanmber G, Chen G P. The abiotic stress-responsive nac transcription factoris involved in drought and salt response in tomato (L.)., 2017, 129: 161–174.
[41] Huang D B, Wang S G, Zhang B C, Shang-Guan K K, Shi Y Y, Zhang D M, Liu X L, Wu K, Xu Z P, Fu X D, Zhou Y H. A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice., 2015, 27: 1681–1696.
[42] 康忱, 赵雪芳, 王鹏, 李亚栋, 田哲娟, 吴志明. 黄瓜纤维素合成酶CESA家族基因鉴定及非生物胁迫响应分析. 中国蔬菜, 2022, (3): 29–41.
Kang C, Zhao X F, Wang P, Li Y D, Tian Z J, Wu Z M. Identification and abiotic stress response analysis of CESA family genes in cucumber (L.)., 2022, (3): 29–41 (in Chinese with English abstract).
[43] Han D G, Du M, Zhou Z Y, Wang S, Li T M, Han J X, Xu T L, Yang G H. An NAC transcription factor gene from,, increases cold and high salinity tolerance in., 2020, 56: 588–599.
[44] 覃利萍. 刚毛柽柳AP2ERF转录因子ThCRF1响应盐胁迫的调控机理研究. 新疆大学博士学位论文, 新疆乌鲁木齐, 2018.
Qin L P. Study on the Regulatory Mechanism of an AP2/ERF Transcription Factor, ThCRF1, in Response to Salt Stress in. PhD Dissertation of Xinjiang University, Urumqi, Xinjiang, China, 2018 (in Chinese with English abstract).
[45] Cheng M C, Liao P M, Kuo W W, Lin T P. Theregulates abiotic stress- responsive gene expression by binding to different cis-acting elements in response to different stress signals., 2013, 162: 1566–1582.
[46] Ohta M, Matsui K, Hiratsu K, Shinshi H, Ohme-Takagi M. Repression domains of class II ERF transcriptional repressors share an essential motif for active repression., 2001, 13: 1959–1968.
[47] Dong W, Ai X H, Xu F, Quan T Y, Liu S W, Xia G M. Isolation and characterization of a bread wheat salinity responsive ERF transcription factor., 2012, 511: 38–45.
[48] Fujimoto S Y, Ohta M, Usui A, Shinshi H, Ohme-Takagi M.ethylene-responsive element binding factors act as transcriptional activators or repressors of GCC box-mediated gene expression., 2000, 12: 393–404.
[49] Yang Z, Tian L N, Latoszek-Green M, Brown D, Wu K Q.ERF4 is a transcriptional repressor capable of modulating ethylene and abscisic acid responses., 2005, 58: 585–596.
Transcriptome sequencing analysis of different sweet potato varieties under salt stress
ZHANG Xiao-Hong, PENG Qiong, and YAN Zheng*
Fuzhou Institute of Agricultural Sciences, Fuzhou 350018, Fujian, China
The objective of this study is to obtain the salt tolerant transcriptome sequence information of sweet potato, mine and identify the differentially expressed genes and their related metabolic pathways. Rongshu 819 (salt-tolerant variety) and Rongshu 910 (salt-sensitive variety) under salt treatment for 0, 3, and 6 days were conducted for the transcriptome by high-throughput sequencing. The results showed that a total of 157,252 Unigenes with an average length of 576 bp were obtained from the two varieties. The 83,264 Unigenes, accounting for 52.95% of the total Unigenes, were annotated in the 7 functional databases. NR annotation revealed that sweet potato Unigenes had the most homologous sequences inwith a total of 43,620 accounting for 57.05%. The annotation of Unigenes in the KOG database is mainly concentrated in general functional prediction only (8752), signal transformation mechanisms (5067), and post translation modification, protein transformation, molecular chaperones (4471). The qRT-PCR indicated that, in Rongshu 819, the number of differentially expressed genes in 3 days and 6 days of salt treatment was 3752 and 807, respectively, which classified into 33 GO functional categories and 302 KEGG metabolic pathways. In Rongshu 910, the number of differentially expressed genes in 3 days and 6 days of salt treatment was 5554 and 7395, respectively, which classified into 50 GO functional categories and 329 KEGG metabolic pathways. The heat map was drawn based on the transcriptome data of partial differentially expressed genes. The result showed that 7 differentially expressed β-Glucosidase genes annotated into starch and sucrose metabolic pathway were up-regulated in salt-tolerant variety while down-regulated in salt-sensitive variety. Seven calmodulin-like genes annotated into Ca2+signaling pathway were specifically expressed in two varieties, among which 2 were specifically up-regulated in salt-tolerant variety and 5 were specifically down-regulated in salt-sensitive variety. The expression heat map of differentially expressed transcription factors indicated thatwas only specifically expressed in salt-tolerant varieties,,,, andwere only specifically expressed in salt-sensitive varieties, whilewas down-regulated in salt-tolerant varieties and up-regulated in salt-sensitive varieties. In conclusion, the number of Unigenes obtained from transcriptome of sweet potato under salt stress was large and the sequence information was rich. The differentially expressed genes and high abundance transcription factors may play an important role in sweet potato response to salt stress.
sweet potato; salt stress; transcriptome; high-throughput sequencing
10.3724/SP.J.1006.2023.24143
本研究由福建省科技厅引导性项目(2019N0037)资助。
This study was supported by the Guiding Project of Science and Technology Department of Fujian Province (2019N0037).
鄢铮, E-mail: moosey@163.com
E-mail: ahjane@163.com
2022-06-16;
2022-10-11;
2022-10-17.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20221017.1428.008.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
