昆明裂腹鱼CYP1A1基因结构与进化分析*
2023-03-13毕映慧苗贵东杨申才吕彤彤
毕映慧,苗贵东,杨申才,吕彤彤,杨 进
(兴义民族师范学院生物与化学学院,贵州 兴义 562400)
昆明裂腹鱼已成为我国重要的经济养殖鱼类之一[1],属鲤科(Cyprinidae)裂腹鱼属(Schizothorax Hecke),地方名常称之为细鳞鱼[2],主要生活在我国的西南部分地区,贵州省境内主要生活在南盘江、北盘江上游的支流中,此外金沙江下游、乌江上游等都有分布。昆明裂腹鱼的最适生长水温范围在14~18℃之间,为冷水性鱼类,由于具有较强的耐低温能力以及肉味鲜美的优点,因此具有开发养殖前景,已被列入贵州省“十四五”乡村振兴生态渔业产业重要的经济鱼种。然而,昆明裂腹鱼存在性成熟时期较晚(一般雌性鱼4龄、雄性鱼3龄的个体性腺才开始逐渐发育成熟)、繁殖能力较低、生长畸形率较高[3]等困扰产业发展的问题。
细胞色素P450(Cytochrome P450,CYP450)由一群基因超家族(super family)编码的酶蛋白所形成。CYP450在生物体内广泛分布,是微粒体混合功能氧化酶中不可或缺的一族氧化酶。CYP450与雌激素在内的外源性以及内源性化学物质在生物体内的相互转化是依赖于酶来实现的。在雌激素致癌链上,雌激素代谢酶的活性以及功能会对雌激素与靶细胞的作用产生直接影响[2]。研究表明CYP450就包括了CYPlAl、CYP1B1以及CYPl9等。细胞色素P4501A1(Cytochrome P4501A1,CYP1A1)是一种芳香烃羟化酶[4],是一种重要的体内I相代谢解毒酶,能对多种环境致癌物以及突变物进行失活[5]。许多研究表明,CYP1A1是一种重要的激活环境污染物(多环芳香碳氢化合物)的酶[6-7]。通过开展发育调控及性别控制相关基因研究,进一步研究其功能,为认识昆明裂腹鱼的发育特点及性别控制相关育种研究奠定理论基础。
1 实验材料与方法
本研究材料昆明裂腹鱼个体取自中国科学院昆明动物研究所云南濒危物种保育中心,通过解剖取雌雄成熟个体性腺,构建了雌雄个体的全长转录组文库,并进行高通量测序。取鱼鳍,用95%酒精脱水,-20℃保存。通过对昆明裂腹鱼全长转录组文库数据分析与挖掘,获得昆明裂腹鱼CYP1A1基因的全长,设计引物,然后通过PCR扩增产物直接测序方法,验证CYP1A1基因的全长。
2 实验结果
2.1 昆明裂腹鱼基因组DNA提取
取适量鱼鳍组织,采用天根生物海洋动物组织基因组提取试剂盒,按照试剂盒方法提取高质量基因组DNA,-20℃保存备用。
基因组DNA经检测,DNA琼脂糖凝胶电泳检测结果图显示主条带清晰,点样孔干净无荧光,表明DNA质量良好,见图1。图1中,M为D2000 Marker。

图1 DNA琼脂糖凝胶电泳检测结果图
2.2 昆明裂腹鱼CYP1A1基因的全长测序验证
对获得昆明裂腹鱼CYP1A1基因的全长序列进行基因组扩增,PCR扩增产物直接测序验证,获得序列与高通量测序所获基因序列进行比较,高通量测序所获基因序列的碱基准确性大于99%,高通量测序所获基因序列结果较为可靠(见图2、图3)。图2中,M为D2000 Marker。

图2 部分引物PCR扩增产物结果
图3 CYP1A1基因部分引物PCR扩增产物测序结果
2.3 昆明裂腹鱼CYP1A1基因结构分析
根据整理的基因序列,采用美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)网站上的ORF Finder程序,对测序获得的mRNA序列进行开放阅读框定位;确定其起始密码子以及终止密码子位点;进而获得其CDS序列全长。昆明裂腹鱼CYP1A1基因的CDS序列全长为1 498 pb。采用DNASTAR软件,把CDS序列碱基翻译成氨基酸序列,得到499个氨基酸。采用SMART软件,对昆明裂腹鱼CYP1A1基因的CDS序列进行预测,发现其氨基酸的45~368区域和382~499区域为Pfam(见图4),跨膜区可能在9~31区域(见表1)。

图4 SMART软件预测的CDS序列蛋白质结构
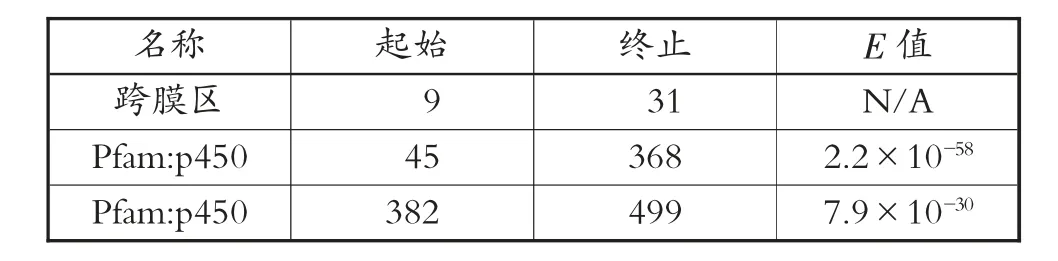
表1 SMART软件预测的昆明裂腹鱼蛋白结构数据(%)
运用NCBI网站上的Blast程序,对扩增获得的昆明裂腹鱼CYP1A1基因与近源物种的同源性进行比对。昆明裂腹鱼CYP1A1基因与鲤鱼、安水金线鲃、鲫鱼、犀角金线鲃、金线鲃、南亚野鲮、草鱼、卡特拉鱼、黑头呆鱼和斑马鱼等物种的CYP1A1基因的CDS序列区的同源性比对结果,见表2。
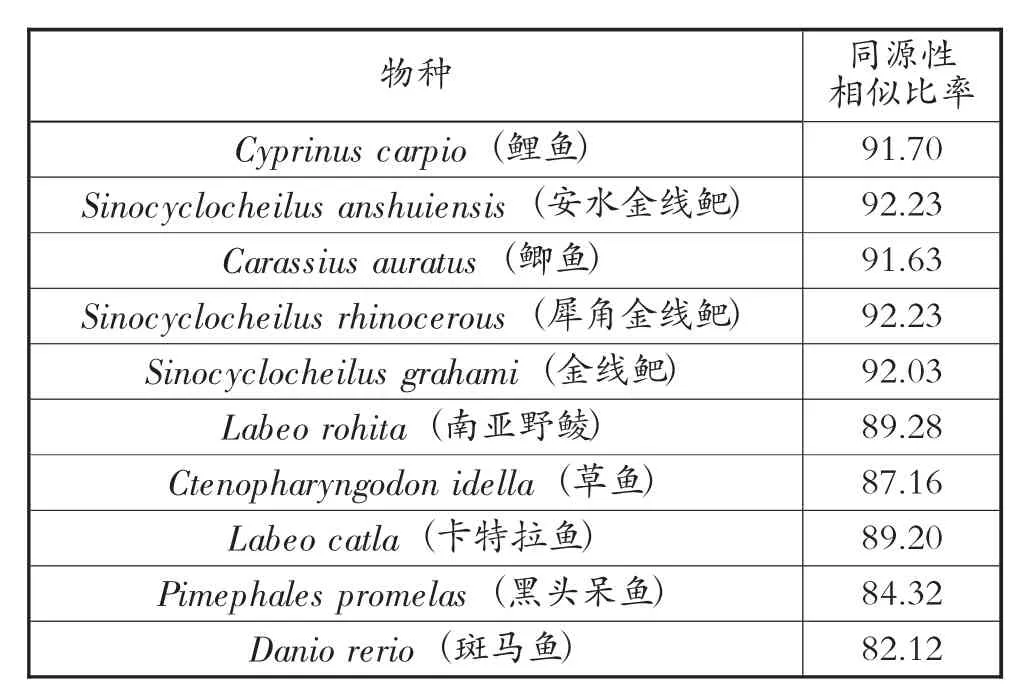
表2 CYP 1A1基因的CDS序列区的同源性比对结果(%)
由表2分析可知,昆明裂腹鱼CYP1A1基因与安水金线鲃和犀角金线鲃的同源性相似比率较高,均为92.23%;其次与鲤鱼、鲫鱼、南亚野鲮、卡特拉鱼、草鱼和黑头呆鱼的同源性相似比率依次为91.70%、91.63%、89.28%、89.20%、87.16%、84.32%;与斑马鱼的同源性相似比率较低,为82.12%。
CYP1A1基因的氨基酸序列的同源性比对结果见表3。
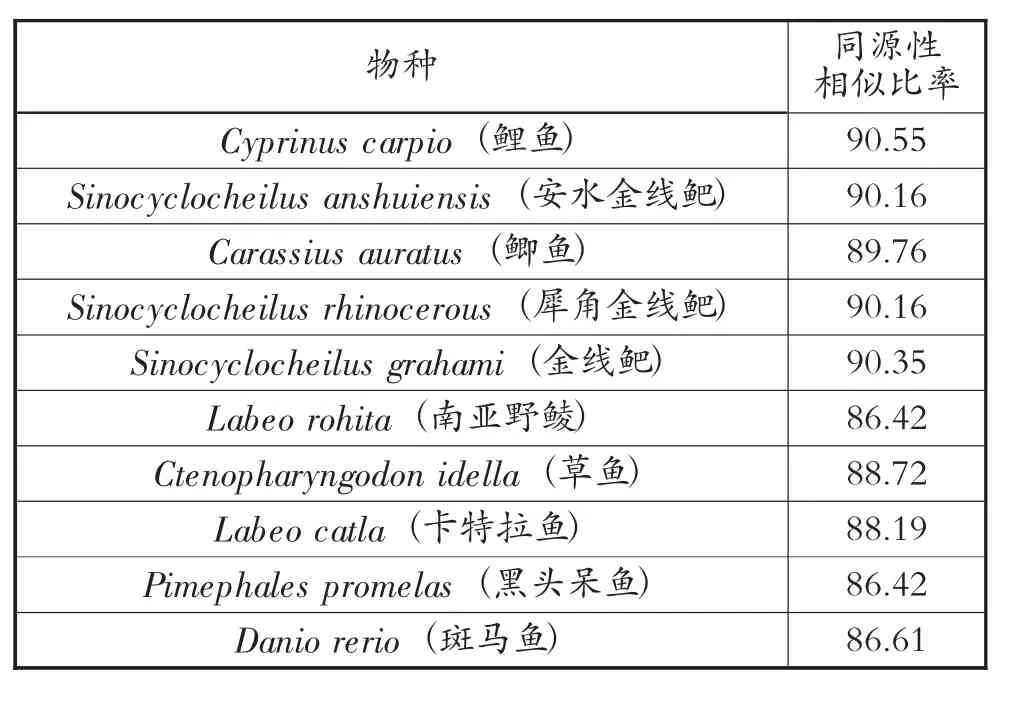
表3 CYP1A1基因的氨基酸序列的同源性比对结果(%)
由表3分析可知,昆明裂腹鱼CYP1A1基因的氨基酸序列与鲤鱼的氨基酸序列同源性相似比率最高,为90.55%;与安水金线鲃和犀角金线鲃的氨基酸序列同源性相似比率均为90.16%;与南亚野鲮、和黑头呆鱼的氨基酸序列同源性相似比率为86.42%。
可以推测,CDS序列区和氨基酸序列的同源性比对结果显示,扩增得到的基因即为昆明裂腹鱼的CYP1A1基因。
运用BioEdit软件,对本次试验扩增获得的CYP1A1基因的CDS序列进行碱基组成成分分析。CYP1A1基因的4种碱基组成含量见表4。
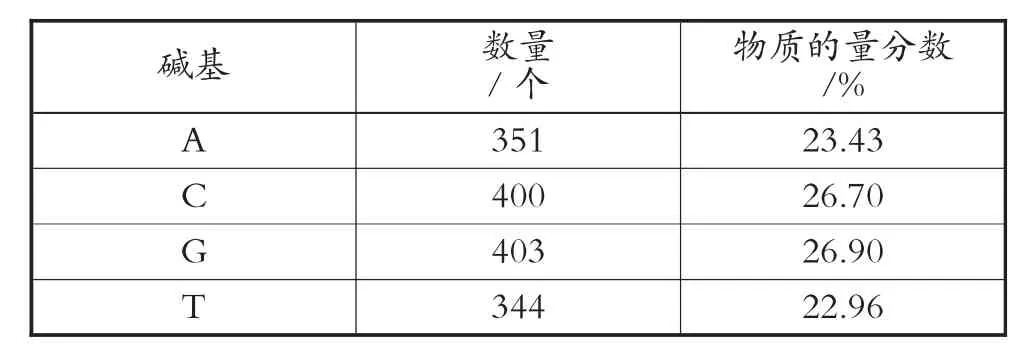
表4 昆明裂腹鱼CYP1A1基因的CDS序列4种碱基含量
由数据分析可知,CDS序列的单链蛋白质的相对分子质量为455 822.00 KD,双链蛋白质的相对分子质量为911 589.00 KD;GC含量为53.60%,AT含量为46.40%。
2.4 昆明裂腹鱼CYP1A1基因进化分析
从NCBI网站的Ge Ba nk数据库里下载鲤鱼、安水金线鲃、鲫鱼、犀角金线鲃、金线鲃、卡特拉鱼、南亚野鲮、黑头呆鱼、草鱼和斑马鱼等同源性物种的CYP1A1基因氨基酸序列。采用MEG A7.0软件里面P hyrogeny的N eighbor-Joining Tree程序,对该序列构建分子系统进化树(见图5)。
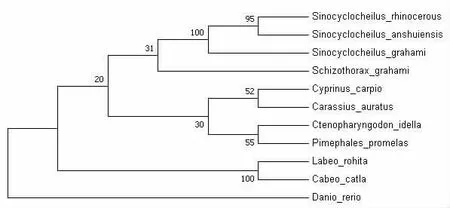
图5 CYP1A1基因氨基酸序列的分子系统进化树
2.5 昆明裂腹鱼CYP1A1基因蛋白质预测与分析
2.5.1 昆明裂腹鱼CYP1A1基因蛋白质疏水性分析
运用ExPASy在线平台上的ProtScale程序,对扩增得到的CYP1A1基因的蛋白质进行疏水性分析,将蛋白质序列提交到该程序,由程序给出CYP1A1基因的疏水性图谱(见第29页图6),从而对其进行分析。
分析图6疏水性图谱可知,最小值为-3.144,位于序列214位置上;最大值为3.478,位于序列472位置;昆明裂腹鱼CYP1A1基因序列的N-端的5~32位置区域、192~208位置区域、322~341位置区域和465~479位置区域表现为明显的疏水性,在其序列N-端的209~218位置区域表现为存在明显的亲水性,由此预测其信号肽可能存在于疏水性区域内。
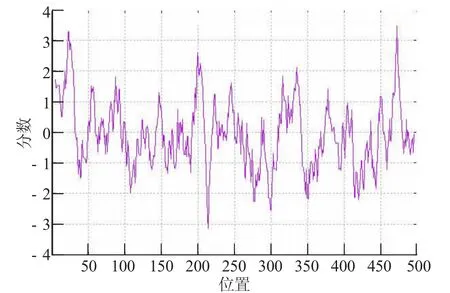
图6 CYP 1A1基因的疏水性图谱
通过ExPASy在线平台上的ProtParam程序可知,昆明裂腹鱼CYP1A1基因的氨基酸序列的平均亲水性为-0.078;理论等电点(pI)为6.44,氨基酸的相对分子质量为55 867.48 Da,原子组成为C(2505)、H(3965)、N(671)、O(728)、S(23);分子式为C2505H3965N671O728S23。该序列的N-端是甲硫氨酸(Met)。由于其不稳定指数(II)为35.37,因此将其蛋白质归类为稳定蛋白质。
2.5.2 昆明裂腹鱼CYP1A1基因信号肽分析
基于对昆明裂腹鱼CYP1A1基因蛋白质的疏水性分析,其蛋白质序列可能存在信号肽,将CYP1A1基因的氨基酸序列上传到SignaIP 3.0在线服务器,通过隐马可夫模型(HMM),预测该序列中是否含有信号肽,见图7。运用神经网络法(NN)程序,预测该序列中的信号肽位点(见图8)。

图7 预测CYP1A1基因的氨基酸序列中是否含有信号肽
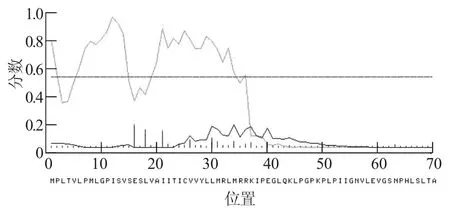
图8 预测CYP 1A1基因的氨基酸序列中的信号肽位点
信号肽分析主要涉及3个参数值,分别为C、S和Y。程序会给出一个相对应的数据,并能够对是否含有信号肽做出判断。根据SignaIP3.0服务器呈现的数据(见表5),可对其进行分析。
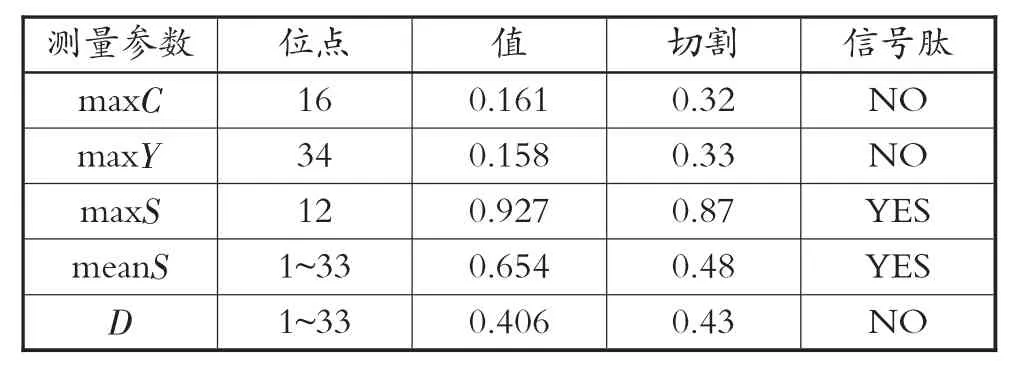
表5 昆明裂腹鱼CYP1A1基因信号肽预测结果测量参数
由表5的预测结果表明,最可能的裂解位点在33和34之间MRL-MR,可能存在信号肽的概率为0.081;信号锚定概率为0.869;该序列最大切割位点概率为0.050;位点在15和16之间。根据数据并结合图形分析可知,昆明裂腹鱼CYP1A1基因的蛋白质的N-端存在1个信号肽,位点在33和34之间MRL-MR。
2.5.3 昆明裂腹鱼CYP1A1基因跨膜区分析
将昆明裂腹鱼CYP1A1基因的氨基酸序列提交到ExPASy在线平台上的TMPRED程序,让其对该序列进行跨膜区预测,见图9。
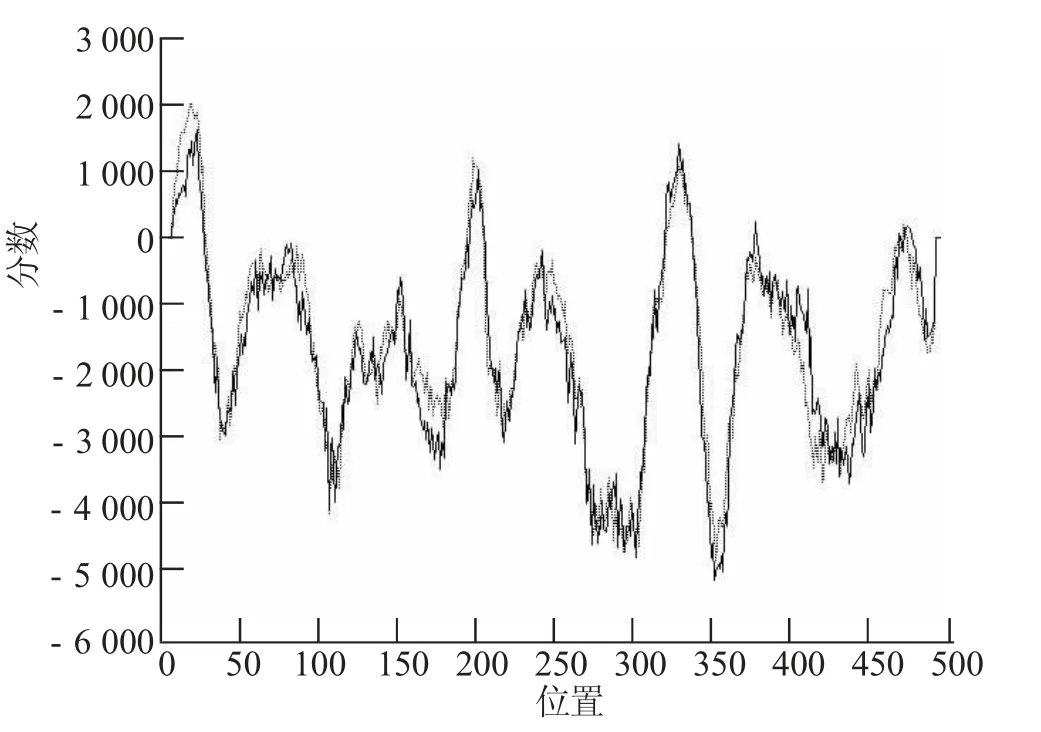
图9 预测CYP1A1基因的跨膜区
由图9分析表明,该序列的N-端有2个强跨膜螺旋,由内到外的螺旋有4个,分别位于14~34位置区域、194~210位置区域、319~339位置区域以及368~388位置区域;由外到内的螺旋有2个,见第30页表6。因此,该基因有2个跨膜区,并且能够预测跨膜区的长度为17到33之间。
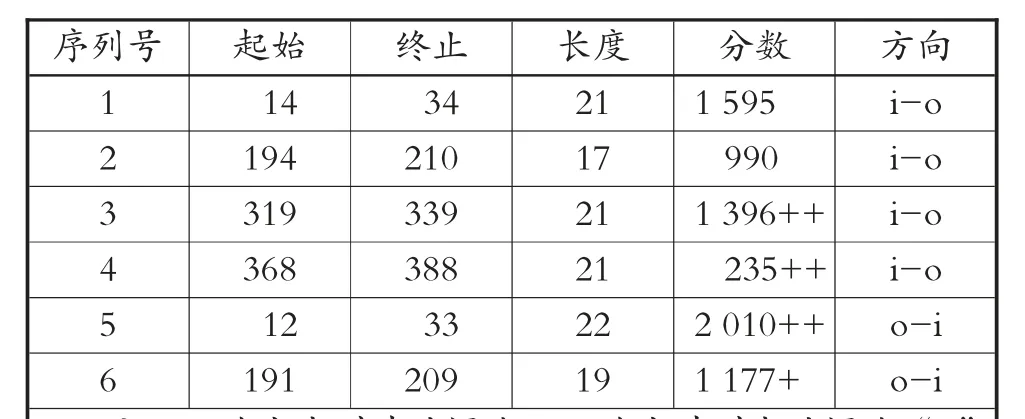
表6 CYP1A1基因的氨基酸序列的强跨膜螺旋区
2.5.4 昆明裂腹鱼CYP1A1基因磷酸化位点和糖基化位点
采用NetPhos3.1服务器,在线对扩增的昆明裂腹鱼CYP1A1基因进行磷酸化位点预测,见第30页图10。
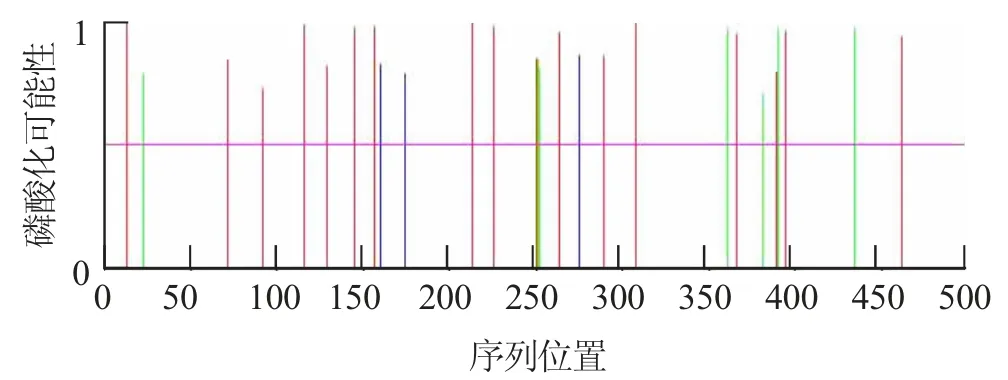
图10 预测CYP 1A1基因的磷酸化位点
蛋白质的磷酸化主要是指氨基酸残基被磷酸化后能够导致蛋白质结构发生改变,进而导致蛋白质活性发生改变的过程。由图10分析结果显示,昆明裂腹鱼CYP1A1基因中,共发现27个磷酸化位点,其中,苏氨酸(Thr)为7个,丝氨酸(Ser)为17个,酪氨酸(Tyr)为3个。
利用NetNGlyc1.0及NetOGlyc3.1服务器,分别对扩增的昆明裂腹鱼CYP1A1蛋白质进行N-糖基化位点预测和O-糖基化位点预测。N-糖基化位点预测见图11。
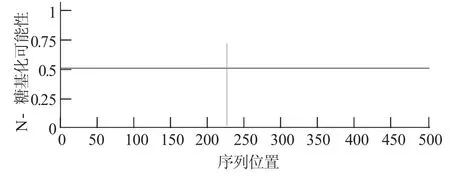
图11 预测CYP 1A1基因的N-糖基化位点
由图11分析结果显示,昆明裂腹鱼CYP1A1蛋白质序列有1个N-糖基化位点,位于224位置上,该蛋白质序列暴露N-糖基化机制,因此能被糖基化。O-糖基化位点预测见图12。由图12分析结果可知,昆明裂腹鱼CYP1A1基因的O-糖基化位点有3个,见表7。分别位于15位置、198位置和391位置。O-糖基化发生在丝氨酸(Ser)上。
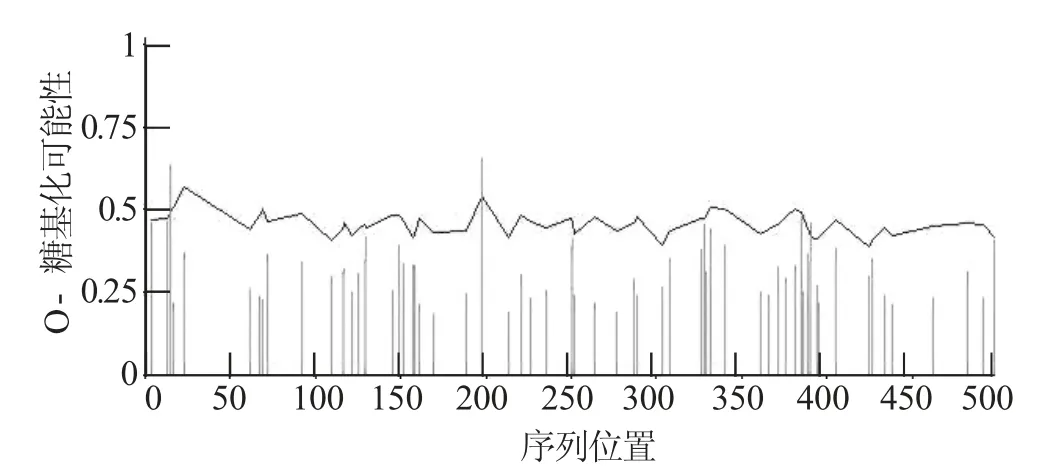
图12 预测CYP 1A1基因的O-糖基化位点
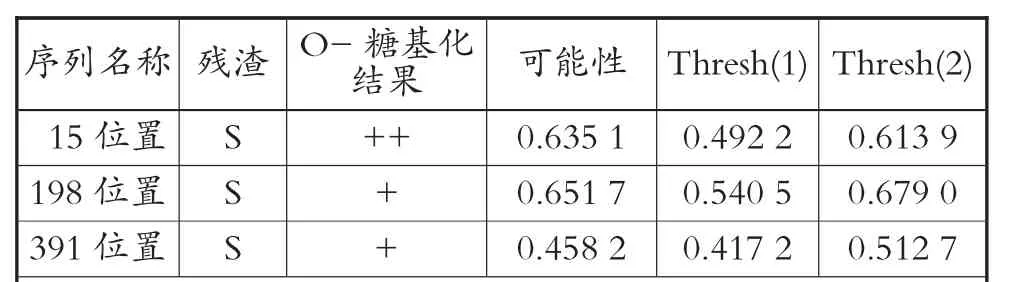
表7 昆明裂腹鱼CYP1A1基因O-糖基化位点数据统计
2.5.5 CYP1A1基因二级结构分析
运用SOPMA在线服务器,对扩增的昆明裂腹鱼CYP1A1基因进行预测,见图13。
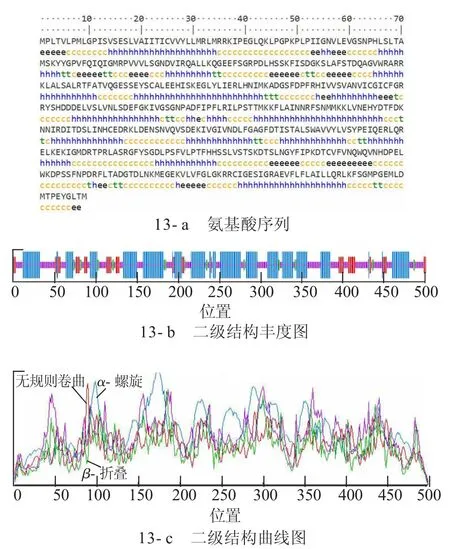
图13 CYP1A1基因的二级结构
由图13分析显示,昆明裂腹鱼CYP1A1基因主要以α-螺旋(Alpha helix)为主;无规则卷曲(Random coil)次之;β-折叠(Beta bridge)最少。昆明裂腹鱼CYP1A1基因的无规则卷曲区域、α-螺旋区域、β-折叠区域分别占36.87%、45.89%、5.01%。
2.5.6 CYP1A1基因三级结构分析
运用同源建模的方法,将昆明裂腹鱼CYP1A1基因的氨基酸序列提交到SWISS-MODEL在线服务器,对该序列进行建模,见第31页图14。分析可知,建模以4i8v.1.A序列作为模板,与目标序列之间的一致度为56.99%,远大于30%,满足同源建模的基本条件。
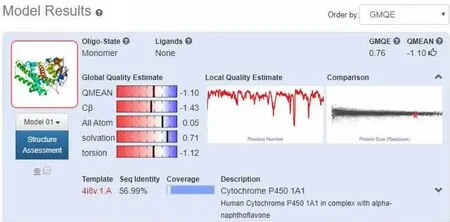
图14 昆明裂腹鱼CYP1A1基因的氨基酸序列的同源建模
基于可信度(GMQE)范围为0~1,QMEAN区间为0~1,由数据分析可知,待测蛋白质的GMQE值为0.76,QMEAN值为-1.10。因此,可根据模板蛋白质的三维结构,预测待测蛋白质的三维结构,见图15。在图15中,图像由彩虹着色N→C端;尺寸如下:x=64.745Å,y=47.352Å,z=72.452Å。
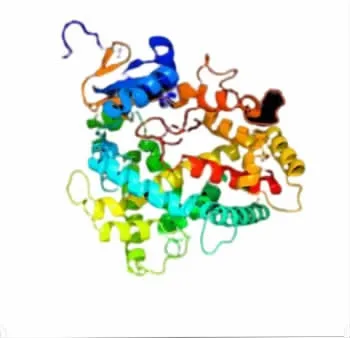
图15 昆明裂腹鱼CYP 1A1基因的蛋白质的三维结构预测图
分析可知,旋转异构体离群值(Rotamer Outliers)为2.72%;C-Beta偏差(C-Beta Deviations)有5个,分别位于A366 SER,A299 LEU,A336 VAL,A122 ASP,A324 PHE;非脯氨酸顺式肽键(Cis Non-Proline)有1个,位于A438 THR-A439 ASP。昆明裂腹鱼CYP1A1基因的各氨基酸残渣质量见图16。分析显示,昆明裂腹鱼CYP1A1基因的N-端是甘氨酸(Gly),C-端是甲硫氨酸(Met);缺失突变位点有1个,位于SER 386;半胱氨酸(Cys)位点有6个,分别位于序列162位置、204位置、207位置、294位置、406位置和458位置上。
图16 昆明裂腹鱼CYP1A1基因的各氨基酸残渣质量
3 分析与讨论
采用NCBI网站上的ORF Finder程序,对测序获得的mR NA序列进行开放阅读框位置定位;确定其序列的起始密码子以及终止密码子的位置;从而确定试验扩增CYP1A1基因的CDS序列全长为1 498 bp,通过DNAS T A R软件,对该序列进行翻译,可得出该序列共编码299个氨基酸。从NCBI网站的Ge Ba nk数据库中,获取鲤鱼、安水金线鲃、鲫鱼、犀角金线鲃、金线鲃和黑头呆鱼等物种的CYP1A1基因的CDS序列。利用Blast程序,对这些物种的CYP1A1基因进行全CDS序列比对,通过比对发现昆明裂腹鱼与安水金线鲃和犀角金线鲃的碱基同源性相似比率达到92.23%,与鲤鱼的碱基同源性相似比率达到91.70%。比对结果表明,昆明裂腹鱼与安水金线鲃以及犀角金线鲃的氨基酸同源性相似比率达到90.16%,与鲤鱼的氨基酸同源性相似比率达到90.55%,根据CDS序列和氨基酸序列同源性比对结果分析,进而可以确定扩增获得的基因序列即为昆明裂腹鱼的CYP1A1基因序列。利用SMART软件,对昆明裂腹鱼CYP1A1基因的CDS序列进行预测,发现其氨基酸的45~368位置区域和382~499位置区域为Pfam(P450),进一步证实了扩增获得的基因序列就是昆明裂腹鱼的CYP1A1基因序列。同源性比对结果分析表明,昆明裂腹鱼与鲤科鱼类和高原冷水鱼类的CYP1A1基因同源性相似比率高,这与其均属于冷水鱼类和具有相似的栖息环境是一致的。
昆明裂腹鱼的CYP1A1基因的氨基酸序列与其他多种鱼类的分子系统进化树表明,昆明裂腹鱼与金线鲃、犀角金线鲃以及安水金线鲃的亲缘关系较为相近;与斑马鱼的亲缘关系较远。通过分析可知,安水金线鲃是金线鲃属的一个新种[8];金线鲃属于鲤形目鲤科[9];犀角金线鲃是云南鲤科鱼类的一个新种[10]。这些物种与昆明裂腹鱼均属于鲤科冷水性鱼类,主要分布于我国西南地区高原河流与湖泊内。斑马鱼是鲤形目鲤科,是一种常见的热带鱼,主要分布于印度、孟加拉国以及巴基斯坦等南亚国家[11]。不同的地理分布以及生存环境可能是导致其亲缘关系之间远近不同的一个原因。
运用ExPASy在线平台上的相应程序,对扩增获得的昆明裂腹鱼CYP1A1基因的蛋白质序列进行疏水性预测,发现其序列有4个明显的疏水性区域,进而推测在疏水性区域内可能存在信号肽。运用ExPASy在线平台上的ProtParam程序分析表明,该序列的氨基酸平均亲水性为-0.078,理论等电点为6.44。基于疏水性预测可能存在信号肽,通过SignaIP3.0在线服务器,运用隐马可夫模型(HMM)预测其序列中是否含有信号肽;运用神经网络法(NN)程序预测信号肽位点研究发现,昆明裂腹鱼CYP1A1基因的蛋白质序列的N-端存在1个信号肽,信号肽位点为33和34之间MRL-MR。在跨膜区预测的分析中,发现序列的N-端有2个强跨膜螺旋,因此,预测昆明裂腹鱼CYP1A1基因有2个跨膜区。
蛋白质翻译后修饰过程主要有糖基化以及磷酸化等,蛋白质的磷酸化是生物界最普遍同时也是比较重要的一种蛋白质翻译后修饰。在磷酸化调节的过程中,细胞的形态和功能都会随着磷酸化状态发生改变[12]。本次试验扩增获得的昆明裂腹鱼CYP1A1基因蛋白质翻译后修饰主要是磷酸化位点和O-糖基化位点。有27个磷酸化位点,其中,苏氨酸(Thr)有7个,丝氨酸(Ser)有17个,酪氨酸(Tyr)有3个。有3个O-糖基化位点,分别位于15位置、198位置和391位置上。昆明裂腹鱼CYP1A1基因的蛋白质序列只有1个N-糖基化位点,位于该序列的224位置上。
本次试验扩增获取的昆明裂腹鱼CYP1A1基因蛋白质二级结构以α-螺旋为主,占45.89%;无规则卷曲次之,占36.87%。采用同源建模的方法,将昆明裂腹鱼CYP1A1基因的蛋白质序列提交到SIWSS-MODEL程序,得到与模板蛋白质序列较高的一致度,为56.99%,远大于30%,可用于预测待测蛋白质序列的三维结构。由SIWSS-MODEL程序计算的数据分析显示,扩增获得的昆明裂腹鱼CYP1A1基因在保守区域内存在6个半光氨酸(Cys);在保守区域内有3个二硫键,其二硫键主要是以相邻的2个Cys依次构成的亚域。昆明裂腹鱼CYP1A1基因的蛋白质氨基酸序列的稳定CYP1A1球状结构是通过二硫键折叠而形成的,维持了CYP1A1的高级结构。蛋白质结构特征具备典型的鱼类CYP1A1基因的高级结构特点[13]。
4 结论
通过构建昆明裂腹鱼全长转录组文库获得全长基因的结果是可靠的,通过引物设计、PCR扩增产物直接测序获得序列分析,昆明裂腹鱼CYP1A1基因的碱基和氨基酸水平分析结果表明,昆明裂腹鱼与高原区抗寒鱼类和多倍体鲤科鱼类的同源性相似比率较高,与鲤科鱼类斑马鱼同源性相似比率较低,表明基因与低温鱼类及多倍体高原鱼类有相似的进化方向;其蛋白质预测结果表明,该基因能够形成稳定蛋白,与鲤科鱼类CYP1A1同源性相似比率较高,具备维持其生物学特性的完整的蛋白质空间构型,该研究为进一步认识CYP1A1基因功能及在昆明裂腹鱼性别发育及控制及发育中的作用研究奠定了一定基础。
