芥子酸和米糠谷蛋白非共价相互作用的分子机制研究
2023-03-12梁富强段姗姗濮欣然郭锐林石嘉怿
梁富强,段姗姗,濮欣然,郭锐林,石嘉怿
(南京财经大学食品科学与工程学院,江苏省现代粮食流通与安全协同创新中心,江苏南京 210023)
作为稻米加工过程的重要副产物,米糠不仅富含优良蛋白质,还含有具有多种生物活性的小分子物质,如酚酸类化合物[1]。谷蛋白是米糠中主要的贮藏蛋白之一(含量可达22%[2]),不仅具有较高的必需氨基酸含量,还是多种生物活性肽的重要来源,如抗氧化肽[3]、降血压肽[2]和降血糖肽[4]等,因此具有较高的营养健康价值。根据分子结构的不同,酚酸类化合物可以分为羟基苯甲酸和羟基肉桂酸两大类,芥子酸(sinapic acid,SA)是米糠中一种常见的含量较高的羟基肉桂酸(分子结构如图1A所示)[5]。酚酸类物质不仅具有良好的抗氧化活性,也能够通过作用于不同的靶点发挥多种生物活性。因此,酚酸单体及其协同效应被认为是米糠及全谷物糙米具有多种健康效应的重要原因[6]。冉玲等[7]的研究表明SA和阿魏酸的ABTS+自由基清除能力不仅强于咖啡酸,而且高于常见的内源抗氧化剂谷胱甘肽。而与阿魏酸的结构不同,SA分子结构的苯环上有两个甲氧基(图1),研究发现这对于SA的生物活性具有重要影响。Yang等[8]发现SA的DPPH自由基清除能力明显高于阿魏酸。Jeon等[9]的研究也表明SA对于紫外损伤人皮肤成纤维细胞的保护效果显著优于阿魏酸。因此,SA作为一种代表性的酚酸化合物近年来受到越来越多的关注。
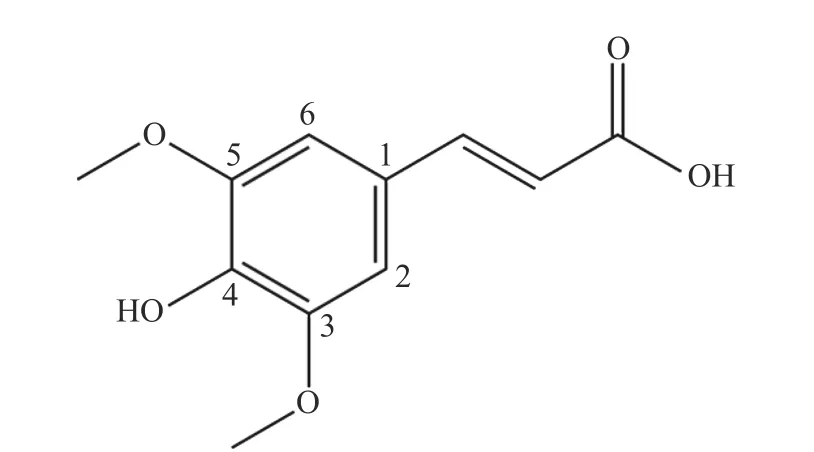
图1 SA的分子结构Fig.1 Molecular structure of SA
作为共存于食品体系中的小分子和生物大分子,酚酸和蛋白在加工、膳食和消化过程中非常容易发生共价结合。Dai等[10]的研究表明大米谷蛋白与原花青素在疏水相互作用的驱动下发生非共价结合。研究表明这不仅能改变蛋白质的营养功能特性,也会对小分子多酚的化学稳定性、生物利用率及生物活性产生显著影响[11−12]。小分子化合物与蛋白的结合位点及作用机制是决定其结合亲和力及生物效应的基础[13]。然而,作为共存于米糠中的小分子和生物大分子,SA与米糠谷蛋白(rice bran glutelin,RBG)非共价相互结合的动态行为及其分子机制仍不清楚。因此,研究拟采用光谱学方法结合同源模建、分子结合和分子动力学模拟探究RGB和SA的结合行为及分子机制,以期为RGB-SA非共价复合物在食品营养中的应用提供分子和结构基础。
1 材料与方法
1.1 材料与仪器
米糠 由南京远望富硒农产品有限责任公司提供;芥子酸(纯度>95%) 上海阿拉丁生化科技股份有限公司;二甲亚砜(纯度≥96.0%) 北京索莱宝科技有限公司;氢氧化钠(纯度≥96.0%) 上海麦克林生化科技股份有限公司;盐酸(分析纯) 中国医药集团有限公司。
磁力搅拌器 常州国华电器有限公司;FL-7000荧光分光光度计、U3900型紫外-可见光分光光度计 日本日立公司。
1.2 实验方法
1.2.1 RBG制备 参考实验室提取大米谷蛋白的方法[14]提取RBG:将脱脂米糠依次分离清蛋白、球蛋白和醇溶蛋白后的残渣,加入0.05 mol/L NaOH提取谷蛋白,离心取上清(8000 r/min,15 min),调节pH至4.8,静置沉淀。将沉淀物水洗至pH中性,冻干得到RBG提取物。采用凯氏定氮法测得RBG提取物中蛋白质含量约为85.3%±2.6%。
1.2.2 荧光光谱测定 将0.2 g RBG溶解于50 mL磷酸盐缓冲液中离心制备蛋白母液,与不同浓度的SA混合制备SA终浓度为0、2.5、5.0、7.5、10.0、12.5、15.0、17.5、20.0 μmol/L的RBG-SA复合物体系。设定激发波长为280 nm,狭缝为5 nm,荧光分光光度计发射波长为300~500 nm范围的荧光光谱。此外,将Δλ分别设定为15和60 nm,测定SA对RBG同步荧光光谱的影响。采用式(1)~式(4)计算SA猝灭RBG荧光的速率常数(Ksv)、猝灭常数(Kq)、结合常数(Ka)、结合位点数(n)及热力学参数,包括焓变(∆H),熵变(∆S)和吉布斯自由能(∆G)[15]。
式中:F0和F分别是SA加入前后RBG的荧光强度; τ0表示生物分子的荧光平均寿命(10−8s);[Q]表示SA浓度;T表示开尔文温度;R表示气体常数(8.314 J/mol/K)。
1.2.3 RBG三维结构同源模建 基于多模板同源模建的方法构建RBG的三维结构模型[16]。从Uniprot数据库(https://www.uniprot.org/)下载谷蛋白的氨基酸序列(P07728),利用BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastp&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)在线网站进行序列比对以寻找适合的模板结构。根据比对结果选择同源模建模板,采用Modeller 10.0软件构建RBG的三维结构模型,通过PROCHECK程序(https://saves.mbi.ucla.edu/)评估模型质量。
1.2.4 分子对接 为寻找小分子配体SA在RBG上所有可能的结合位点,将同源模建获得的RBG三维结构文件和小分子配体SA的三维结构文件上传到CB-dock2(https://cadd.labshare.cn/cb-dock2/php/index.php)进行口袋搜索和分子对接[17]。通过Discovery Studio客户端和Pymol可视化对接结果。
1.2.5 分子动力学(Molecular dynamic,MD)模拟以分子对接获得的5种结合构象为初始结构,采用Gromacs 2019.6软件包参考实验室的方法[18]进行MD模拟。6个模拟体系(单独的RBG和5个RBGSA复合物)均采用立方体盒子,TIP3P水模型,力场为amber99sb-ildn。添加Na+和Cl−离子做为抗衡离子,以中和模拟体系电荷。首先采用最陡下降法对模拟体系进行能量最小化,然后依次进行100 ps等温系综和等温等压系综下的限制性模拟以平衡体系,使体系的温度和压力分别达到298 K和1 bar。最后对平衡后的体系进行100 ns的无限制动力学模拟,时间步长为2 fs。对模拟轨迹通过Gromacs 2019.6结合视觉分子动力学软件进行分析。采用分子力学泊松-玻尔兹曼比表面积(Molecular mechanics Poisson-Boltzmann surface area,MM-PBSA)方法计算SA与RBG的结合自由能。
1.3 数据处理
所有试验重复三次或以上,数据以平均数±平均偏差表示。采用Origin 2018软件绘图,SPSS22.0进行统计分析,P<0.05表示差异显著。使用chemoffice软件绘制SA分子结构。
2 结果与分析
2.1 荧光猝灭及机制分析
图2A~图2C显示了不同浓度SA存在下RBG荧光光谱在三个温度下的变化。可以看出RBG在345 nm处有最大发生峰。随着SA浓度的不断增加,RBG的内源荧光强度明显下降。研究认为这种荧光猝灭效应可能是由于非共价结合形成了复合物,改变了蛋白中色氨酸和酪氨酸残基的微环境[19]。
动态猝灭和静态猝灭是两种常见的荧光猝灭机制,可以通过Stern-Volmer方程(图2D)和结合参数(表1)判断。三个温度下Ksv随着温度的增大而增大,说明存在动态猝灭。然而,最小的猝灭速率(Kq=1.55×1012L/mol/s)远大于最大动态猝灭常数(2.0×1010L/mol/s),表明SA对RBG的荧光淬灭主要是由于基态复合物的形成而不是动态碰撞。因此,可以推断SA对于RBG荧光淬灭主要是静态猝灭。前人研究表明包括芥子酸与玉米醇溶蛋白结合的Kq也远大于最大动态猝灭常数,判断猝灭类型为静态猝灭[20]。Li等[21]的研究认为矢车菊素-3-O-葡萄糖苷对于大米谷蛋白的荧光猝灭也属于静态猝灭。对于静态猝灭,可以进一步采用双对数方程(图2E)计算SA与RBG之间的结合常数与结合位点数。由表1可以看出,SA与RBG的结合常数随着温度的升高而增大,表明形成复合物的稳定性与温度正相关。与前人研究相比,SA与RBG的结合常数(298 K)大于SA与白菇鱼肌球蛋白[22]及大米谷蛋白与没食子酸[23]的结合常数。岳一珂[20]的研究也显示大多数羟基肉桂酸与玉米醇溶蛋白的结合能力强于羟基苯甲酸。可以推测SA与RBG能够发生非共价结合且表现出较强的结合能力。
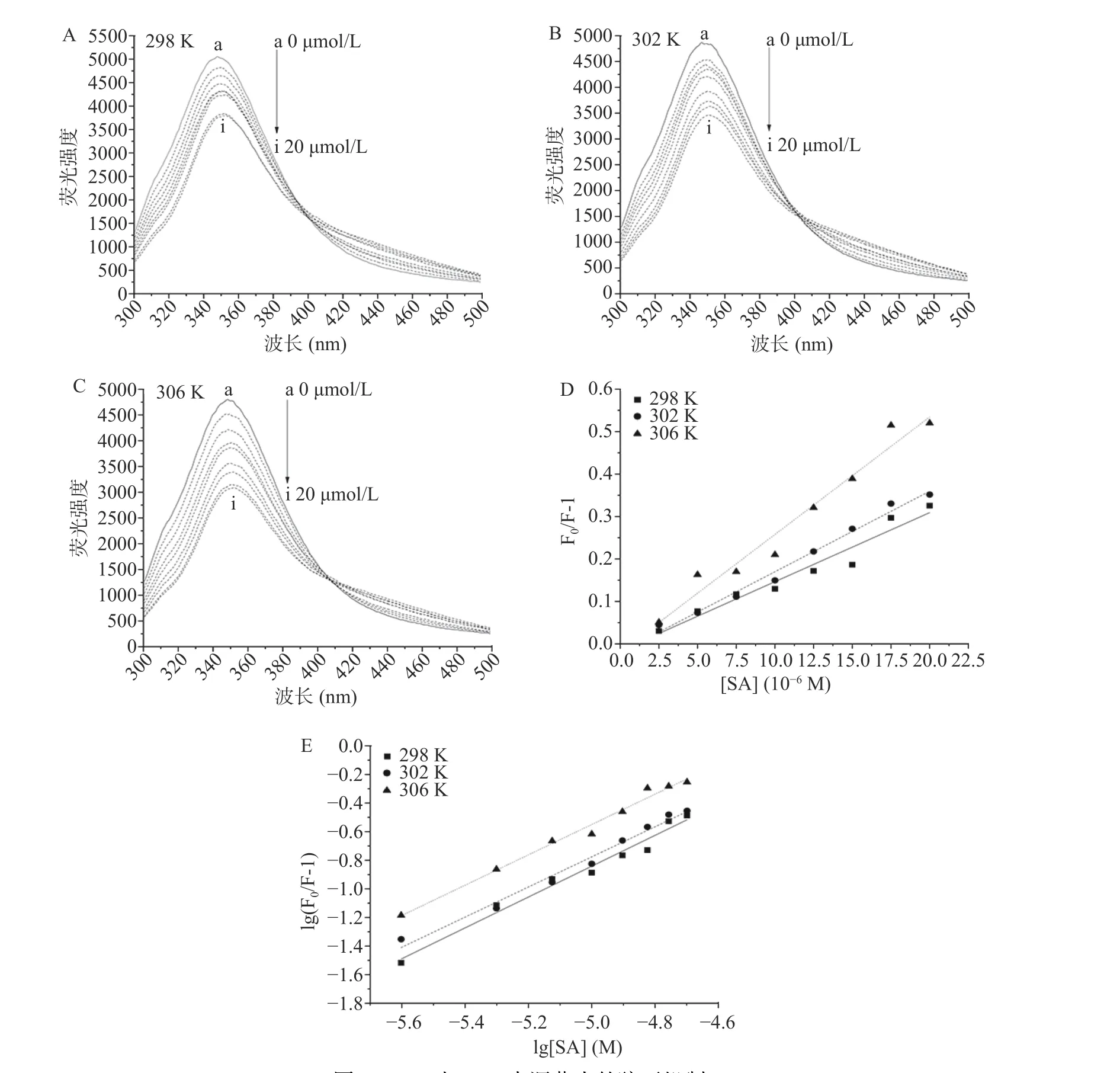
图2 SA对RBG内源荧光的猝灭机制Fig.2 Quenching mechanism of SA on RBG endogenous fluorescence
表1 SA与RBG相互作用的猝灭常数及热力学参数Table 1 Quenching parameters and thermodynamic parameters for SA-RBG interaction
氢键、范德华力、静电和疏水相互作用是蛋白与多酚非共价结合的主要相互作用力。这些作用力类型可以通过热力学参数来确定。根据前人理论,一般认为,当ΔS<0,ΔH<0时为氢键相互作用;ΔS>0,ΔH>0时为疏水相互作用;当ΔS>0,ΔH<0时为静电相互作用[24]。由表1可知,ΔG为负值,说明SA与RBG的结合是自发行为;而ΔS和ΔH均大于0,表明RBG-SA非共价复合物的形成主要由疏水相互作用驱动。Jia等[25]的研究表明SA与杏仁蛋白的非共价结合也主要是由疏水相互作用力驱动的。
2.2 同步荧光
同步荧光光谱可以表征蛋白中色氨酸和酪氨酸残基微环境极性变化,可以进一步反映SA对RBG构象的影响。图3A和图3B分别显示的是不同浓度SA存在下RBG中酪氨酸(Δλ=15 nm)和色氨酸(Δλ=60 nm)的同步荧光光谱变化。可以发现色氨酸(5069)的荧光强度明显高于酪氨酸(1011),说明RBG的内源荧光主要由色氨酸贡献,这与戴涛涛[23]的结果一致。随着SA浓度的增加,RBG中的酪氨酸和色氨酸的荧光强度均呈现规律性下降的趋势,可以发现最大发射峰的位置发生了轻微蓝移,说明SA的结合导致酪氨酸和色氨酸残基所处微环境的疏水性有所增加。
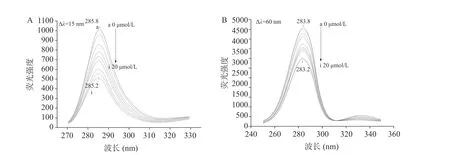
图3 SA对RBG同步荧光光谱的影响Fig.3 Effect of SA on the synchronous fluorescence spectra of RBG
2.3 分子对接结果
实验结果说明SA与RBG发生了非共价结合,然而仅通过实验的手段难以阐述受体蛋白识别小分子配体的过程、结合稳定性及分子机制。近年来分子对接已经成为研究受体和配体相互作用分子机制的重要手段。由于目前尚无RBG的晶体结构,因此首先通过多模板同源模建的方法建立了其三维结构。根据序列比对结果中的相似性(>30%)选择5WPW(51.36%)、5XTY(50.66%)、2E9Q(45.05%)、3QAC(44.52%)和6B4S(45.15%)五个蛋白结构作为模板进行多模板同源模建,获得RBG的三维结构模型(图4A)。拉氏图分析(图4B)显示,没有氨基酸残基(0.0%)位于不允许区域(disallowed regions),90%以上的氨基酸残基位于允许区域(allowed regions)和最佳区域(most favoured regions)。上述说明得到的RBG三维结构构象合理,质量较高,可用于后续研究[26]。
通过基于口袋搜索的分子对接可以发现RBG存在5个潜在的配体结合位点(图4C),根据位点的三维坐标依次命名为C1~C5位点。SA在这5个位点的分子对接打分均为负值(表2)表明SA能够自发与RBG结合。很多研究一般根据分子对接打分选择多酚与蛋白的结合构象[27]。然而,这5个位点的对接打分非常接近,因此,仅依据分子对接结果无法确定SA在RBG上最可能的结合位点。近年来越来越多的研究认识到分子对接只能提供蛋白和小分子配体的静态结合模式,而无法分析两者相互作用的动态过程和结合稳定性[28]。MD模拟能够从时间尺度和分子水平探究受体蛋白和小分子配体结合识别的稳定性及动态行为。因此,需要进一步结合MD模拟以探究SA与RBG最可能的结合位点。
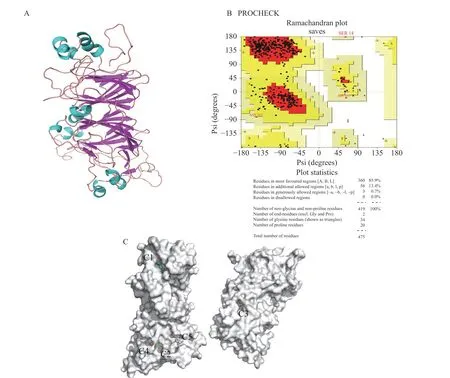
图4 RBG三维结构及其与SA的分子对接结果Fig.4 Three-dimensional structure of RBG and the molecular docking result between it and SA
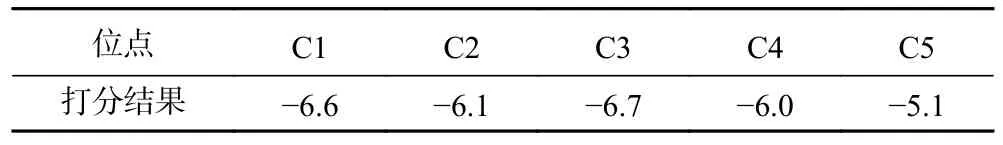
表2 SA在RBG 5个潜在结合位点的分子对接打分Table 2 Docking scores for SA at five potential binding sites of RBG
2.4 SA与RBG最可能结合位点的确定
为了确定SA的RBG最可能的结合位点,以分子对接得到的5种结合构象为起始结构进行了MD模拟。均方根位移(root mean square deviation,RMSD)是衡量模拟体系是否达到稳定状态的重要指标,小分子配体的RMSD能够反映其在结合口袋中运动的稳定性[29]。MD过程中RBG蛋白骨架的RMSD的变化如图5A所示,可以发现在配体SA不存在(RBG)和存在(pro_C1-pro_C5)的情况下,蛋白骨架的RMSD值10 ns后均能达到平衡状态(0.2~0.3 nm之间),说明MD过程中蛋白RBG能够很快达到稳定状态。五个结合位点MD过程中配体SA的RMSD变化情况和运动轨迹如图5B~图5F所示。由图5F可以发现,60 ns后配体SA的RMSD发生了急剧升高,进一步结合其MD过程中的运动轨迹可以发现SA在60 ns后脱离了蛋白结合位点,说明SA不能稳定结合在位点C5。而结合在位点C1~C4时,根据图5B~图5E可以看出,虽然在MD过程中配体SA的RMSD也都产生了一定的波动,但是结合SA整个运动轨迹可以看出其均未脱离,这表明SA能够稳定结合在RBG上除C5外的4个位点。
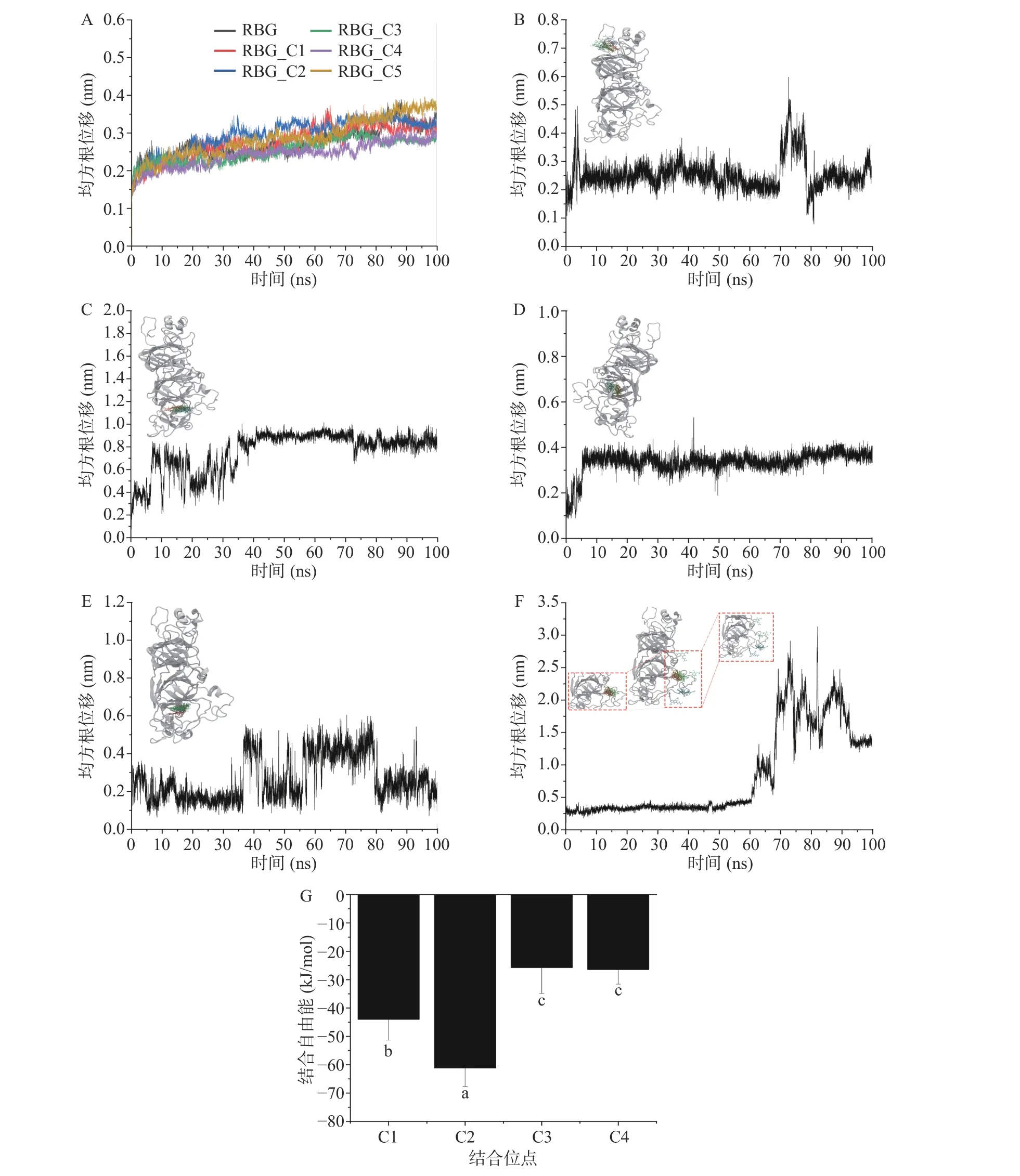
图5 RMSD曲线及结合自由能Fig.5 RMSD plots and binding free energy
实验结果(表1)表明,SA与RBG的结合位点数n接近1,因此,为了确定小分子配体SA在RBG上这个最可能的结合位点,进一步采用MM-PBSA的方法计算了SA在C1~C4四个结合位点的结合自由能。如图5G所示,SA结合在剩余四个位点的结合自由能值分别为−43.99、−61.15、−25.75和−26.44 kJ/mol。结合自由能均为负值说明SA与RBG的结合是自发行为,这与实验获得的热力学参数和分子对接结果一致[30]。SA在C2位点的结合自由能显著低于其他位点(P<0.05),表明在这一位点的亲和力最强。同时,图5C显示在结合位点C2,35 ns后小分子配体SA的RMSD值稳定在约0.8 nm。因此,根据结合自由能和RMSD变化情况可以推测位点C2是SA与RBG最可能的结合位点。与前人的研究比较可以发现,SA在RBG上的结合位点不同于原花青素[10]、没食子酸[31]和表没食子儿茶素没食子酸酯[32]的结合位点。这一方面可能是由于前人研究采用静态的分子对接分析三种多酚与RBG的相互作用,没有考虑结合的动态过程;另一方面配体SA的分子结构与这三种多酚类物质不同,也可能导致结合位点存在明显差异。因此,多酚类化合物与RBG非共价相互作用的构效关系仍需深入研究。
2.5 SA与RBG的结合稳定性及分子机制分析
蛋白质回旋半径(radius of gyrate,Rg)可以表征蛋白质的密实度,值越大表示蛋白溶胀和结构松弛[29]。图6A可以看出,MD过程中单独RBG和RBG-SA复合物的Rg平均值分别稳定在约2.59和2.58 nm,说明SA的结合对于RBG的密实度没有明显影响。均方根涨落(root mean square fluctuation,RMSF)能够反映MD过程中单个氨基酸残基的波动情况。图6B表明,配体SA存在的情况下RBG结构中多数氨基酸残基的波动程度明显降低至低于0.2 nm,说明SA的结合增强了结构稳定性。这也进一步证实SA能够稳定结合在RBG的C2位点。
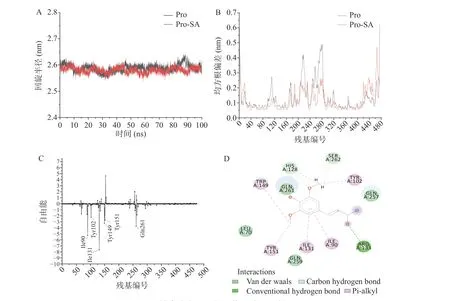
图6 结合稳定性及相互作用分子机制Fig.6 Binding stability analysis and the interaction molecular mechanism
为深入揭示SA与RBG相互作用的分子机制,进一步将结合自由能分解到蛋白的每个氨基酸残基。如图6C所示,可以发现其中Ile131、Ile90、Gln261、Trp149、Tyr151和Tyr102 6个氨基酸残基(<-2 kJ/mol)对两者的结合自由能有重要贡献,提示它们可能对于SA和RBG的结合稳定性和亲和力起着重要作用。SA与Trp149、Tyr151和Tyr102残基的相互作用可能是实验中导致RBG内源荧光猝灭的重要原因。对MD最后一帧的结合构象分析(图6D)可以发现,SA的苯环和甲氧基可以通过π-烷基相互作用与Ile131、Ile90、Trp149、Tyr151及Tyr102形成疏水相互作用;还可以通过范德华力与Gln261接触。有研究显示SA的苯环和甲氧基可以通过π-σ、π-烷基和氢键与靶点蛋白的关键氨基酸残基相互作用,从而发生特异性结合[33]。前人研究表明酚酸分子结构中甲氧基的取代位置,如3位和5位,对提高其与蛋白的亲和力强弱有重要影响[34]。可以推测SA的两个甲氧基基团对其与RBG的结合具有重要作用。本文的结果能够为后续探究酚酸与RBG相互作用的结构-亲和力关系及其复合物的结构改造提供依据。
3 结论
本文通过荧光光谱结合同源模建、分子对接和分子动力学模拟研究了SA与RBG非共价相互作用及其动态过程,解析了SA与RBG最可能的结合位点及相互作用的分子机制。结果表明SA与RBG通过疏水相互作用以约1:1的比例自发形成复合物,其结合导致RBG微环境和构象的改变。分子模拟揭示了RBG上5个潜在的结合位点中,C2位点是最可能的结合位点,并进一步揭示了SA在RBG的C2位点亲和力高的关键氨基酸残基和结构基础。本文深入探究了SA与RBG非共价结合的动态过程和相互作用的分子机制,为进一步研究酚酸类化合物与RBG非共价相互作用的构效关系提供了新思路,为其复合物作为功能性食品配料的应用开发提供了理论基础。
