油莎豆 5-烯醇式丙酮酰莽草酸-3-磷酸合酶基因 CeEPSPS的克隆与分析
2023-02-27肖艳华赵永国
邹 智,肖艳华,张 丽,赵永国
1.中国热带农业科学院三亚研究院/中国热带农业科学院热带生物技术研究所/海南省南繁生物安全与分子育种重点实验室,海南海口 571101;2.中南民族大学生命科学学院/武陵山区特色资源植物种质保护与利用湖北省重点实验室,湖北武汉 430074;3.广东石油化工学院生物与食品工程学院,广东茂名 525000
草甘膦是现代农业生产上应用最广的一类广谱型除草剂,其作用靶标为莽草酸途径的5-烯醇式丙酮酰莽草酸-3-磷酸合酶(5-enolpyruvylshikimate-3-phosphate synthase,EPSPS,EC 2.5.1.19)。草甘膦可竞争性地抑制EPSPS而使芳香氨基酸合成受阻,最终导致植物黄化死亡[1]。EPSPS基因于1983年首次在鼠伤寒沙门氏菌中得到克隆,后发现其广泛存在于各类微生物和植物中[2]。根据进化关系及其对草甘膦的敏感性,微生物源EPSPS可分为两大类,Ⅰ型对草甘膦敏感,Ⅱ型则具有较高的耐受能力,其典型代表是根癌农杆菌CP4-EPSPS,已被广泛用于作物除草剂抗性育种[1,3]。植物源EPSPS按敏感性可归为I型,然而,草甘膦的长期施用致使越来越多的杂草(>30种)出现抗性变异,其抗性机制主要表现为EPSPS基因扩张和单/多位点突变,主要对应于拟南芥EPSPS成熟蛋白的Thr102、Ala103和Pro106[2,4]。
油莎豆(Cyperus esculentusL.)隶属于禾本目莎草科,是一种起源于非洲和地中海沿岸的多年生草本,但在生产上作为一年生作物栽培[5]。油莎豆地下结豆(即块茎)、地上长草,其地上部分全为叶片,叶长80~150 cm,可产鲜草3×104kg/hm2以上,是牛、羊、兔等食草动物的优质饲料;匍匐茎起源的块茎富含淀粉(25%~45%)、油脂(24%~35%)和糖(15%~30%),以及丰富的蛋白质(5%~10%)、膳食纤维(8%~10%)、维生素C/E(8~14 mg/100 g)和矿物质(如钾、磷、钠、钙、镁),综合利用价值高,可开发成可口食品、饮料、保健食用油、糖、酱油、醋、酒和饲料等。作为一种新型的油料作物,油莎豆具有产量高(产鲜豆1.2万~3万kg/hm2,折合油脂>100 kg)、适应性广、抗逆性强、适合机械化等特点,这便于其在不挤占现有耕地的情况下利用沙化边际土地增加我国的食用油和饲料原料供给,提高人民的生活质量,并减少对国外大豆的依赖程度,进而服务国家的战略需求[6]。然而,随着劳动力成本的日益攀升,草害成为制约油莎豆大面积推广的重要因素,因此培育耐除草剂的新品种具有积极的现实意义。本研究对油莎豆的EPSPS基因进行克隆,并在此基础上重点比较分析其序列特征、遗传变异、进化及表达特性,以期为今后的开发与利用奠定基础。
1 材料与方法
1.1 材料
1.1.1 植物材料 供试材料为‘热研1号’及另外55份油莎豆种质[7]。用于DNA提取的幼嫩叶片直接采集于中国热带农业科学院热带生物技术研究所文昌试验基地,而用于RNA提取的‘热研1号’参照邹智等[8]的方法,在采样前10 d移栽至海口温室,然后单独采集幼嫩叶片、成熟叶片、衰老叶片、芽、芽茎(即分化产生芽的匍匐茎)和块茎等组织样本。
1.1.2 菌株及载体 感受态大肠杆菌DH5α和植物表达载体pCAMBIA1301由本实验室制备和保存。1.1.3 主要试剂 引物合成和常规 DNA测序委托生工生物工程(上海)股份有限公司完成;Omega快速质粒小提试剂盒、Omega DNA纯化回收试剂盒、诺唯赞 ClonExpress II One Step Cloning Kit(C112-02)试剂盒、宝生物高保真DNA聚合酶、普洛麦格DNase I及各类分析纯生化试剂和实验耗材均购自相应的试剂公司。
1.2 方法
1.2.1EPSPS基因的鉴定 从Araport11(表1)下载拟南芥的AtEPSPS(AT2G45300)和AtEPSPS2(AT1G48860)基因,并以其蛋白序列作为种子tBLASTn搜索代表性植物的基因组(表1)和油莎豆的全长转录组[8]。

表1 生物信息学分析所用数据库、软件及其网址Tab.1 Databases, software and related websites used for bioinformatics analysis
1.2.2 总 RNA提取与 cDNA第一链的合成 不同组织的总RNA采用天根植物多糖多酚RNA提取试剂盒单独提取,经纯度、浓度和完整性检测合格后,用普洛麦格DNase I清除残存的DNA,并用赛默飞反转录试剂盒合成cDNA第一链。
1.2.3 基因克隆 基于转录组获得的CeEPSPS转录本序列,采用Primer Premier 5.0设计引物对CeEPSPSF/R(GTCACCTCCACTGCATTTCTT/ACT TTGATACAAGCAATATGC)和 CeEPSPSHF/R(C ACGGGGGACTCTTGACC ATGGCGCAAGCGAC CATGGCC/CTGGTCACCTGTAATTCACACCTAA TATTTAGCAAACTTCTG ,下划线为可与pCAMBIA1301重组的同源臂),分别用于基因克隆和植物表达载体的构建。基因克隆参照邹智等[9]的方法,PCR产物用1%琼脂糖凝胶电泳检测;为进一步完成植物过表达载体的构建,以稀释100倍的第一轮PCR产物作为模板,利用CeEPSPSHF/R进行第二轮PCR,产物电泳检测后切胶回收目的条带;同时,用NcoⅠ和PmlⅠ于37℃酶切pCAMBIA1301 12 h,产物经0.8%琼脂糖凝胶电泳检测后回收目的条带;然后,将上述线性化载体和PCR胶回收产物按1∶2比例混合,并用ExnaseⅡ于37℃催化2 h,构建载体pCAMBIA1301-CeEPSPS。重组质粒转化 DH5α感受态细胞后,参照邹智等[9]的方法进行后续的菌落PCR、测序及质粒的提取。
1.2.4 生物信息学分析 参照肖艳华等[7]的方法,分析EPSPS成熟蛋白的理论分子量(Mw)、等电点(pI)、总平均疏水指数(GRAVY)、脂肪族指数(AI)、不稳定系数(II)等,所用软件及其网址详见表1。
1.2.5 基因表达分析 荧光定量分析具体参照邹智等[10]的方法,以18S rRNA作为内参,引物18SF/R(TGTGATGCCCTTAGATGTTCTGG/GAC GTAGTCAACGCGAGCTGA)和 CeEPSPSFq/Rq(CGAGTAGGGGTGCTGTTGTCA/TGCGATTG GAGAGGGATTTAG),每样品3次生物学重复;用2ΔΔCT法和SPSS软件分别进行基因相对表达分析和方差分析。
2 结果与分析
2.1 CeEPSPS基因的克隆
通过搜索团队前期获得的油莎豆全长转录组文库[8]共得到2条同源转录本,其长度为1896、2144 bp,分别包含1545、1344 bp的开放读码框(ORF)。前者的序列如图1所示,其编码的蛋白序列和长度与其他植物类似,将该基因命名为CeEPSPS;后者则缺失了正常的C端。将序列比对到本地的油莎豆基因组,结果发现CeEPSPS含有7个内含子,而上述较长的转录本为第6内含子滞留的可变剪接形式。

图1 CeEPSPS的全长cDNA及其推测编码的氨基酸Fig.1 Full-length cDNA sequence of CeEPSPS and its deduced coding amino acids
为克隆CeEPSPS基因,在基因的转录非翻译区(UTR)设计引物对CeEPSPSF/R,以‘热研1号’反转录的cDNA作为模板,首轮PCR成功扩增得到一条约1853 bp的特异条带;然后以上述PCR产物作为模板,CeEPSPSHF/R作为引物,第二轮PCR扩增得到一条约1584 bp的目标条带,将该条带切胶回收,再通过同源重组法克隆到pCAMBIA1301上。测序结果显示,分离到的CDS与全长转录本的相应区域完全一致,并与 NCBI中释放的 4条序列(登录号为 KT757586.1、KT757587.1、KM052384.1和 KM052385.1)的一致性分别为100.00%、98.30%、98.50%和98.60%。
2.2 代表性单子叶植物中EPSPS基因的鉴定
为揭示基因在单子叶植物中的进化特征,研究基于康藏嵩草(Carex littledalei)等植物的全基因组序列对EPSPS基因进行系统鉴定。结果显示,EPSPS在研究的 13种代表性单子叶植物中多以单拷贝的形式存在,约占69.23%;含有2个拷贝的分别为椰枣(Phoenix dactylifera)、油棕(Elaeis guineensis)、菠萝(Ananas comosus)和香蕉(Musa acuminata),其在核酸和蛋白水平的一致性分别为99.40%/99.40%、90.20%/89.70%、100.00%/100.00%、88.40%/90.90%,接近或略高于拟南芥的 89.60%/89.30%;拟南芥和 15种单子叶植物中的EPSPS基因均含有 7个内含子(表2)。此外,从 NCBI中还搜索到 4条香附子(C.rotundus)的EPSPS完整或部分编码区序列,即KT757585.1(1545 bp)、KM052381.1(1530 bp)、KM052382.1(1474 bp)和KM052383.1(1477 bp),其与CeEPSPS的一致性分别为97.20%、97.50%、97.40%和97.10%。
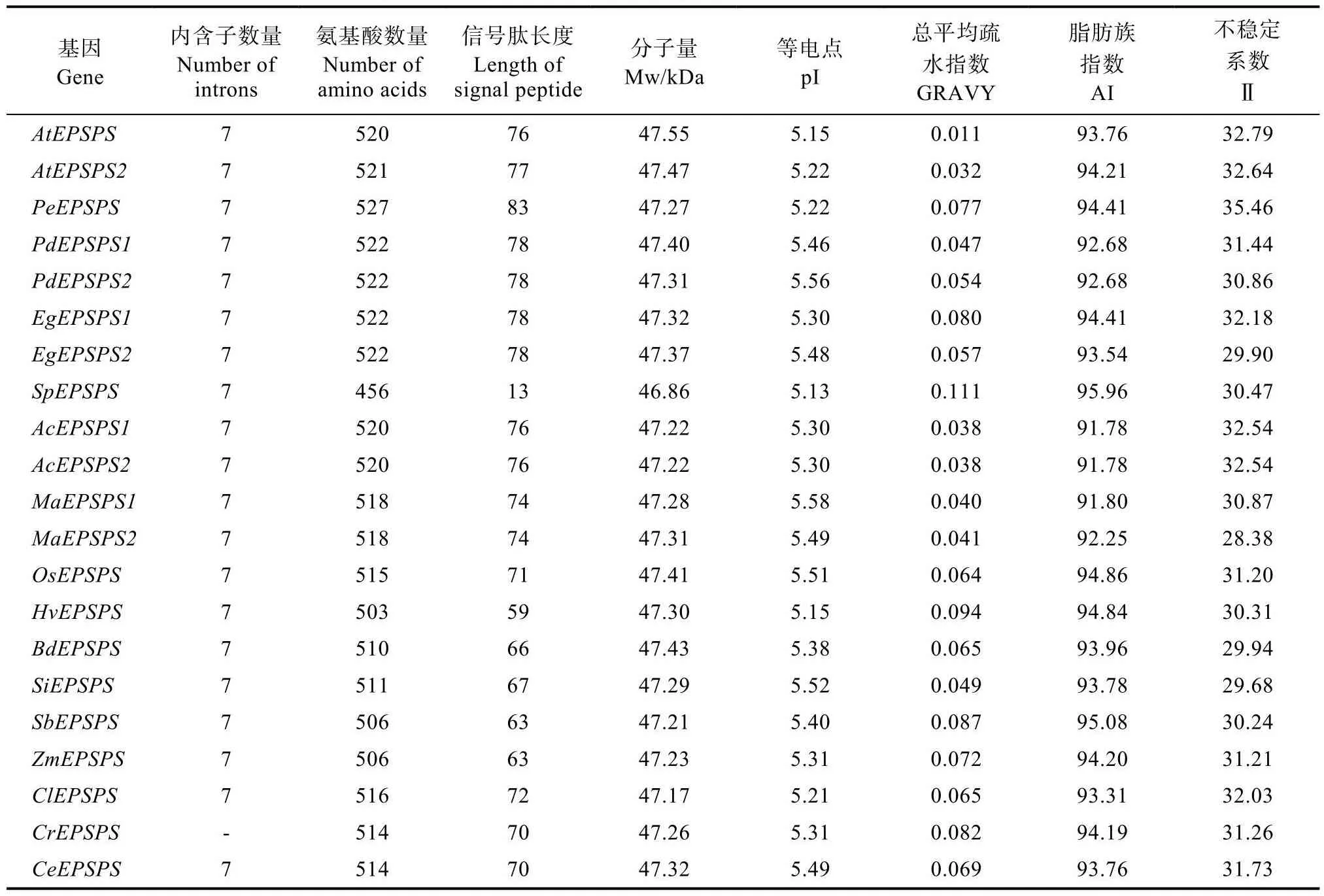
表2 拟南芥和15种单子叶植物中EPSPS基因的结构及其编码蛋白的理化特性Tab.2 Gene structures and protein physicochemical properties of EPSPS genes in arabidopsis and 15 monocots
2.3 CeEPSPS基因的生物信息学分析
2.3.1CeEPSPS基因编码蛋白的理化特性、亚细胞定位及保守结构域分析 如图1所示,CeEPSPS基因编码区的GC含量为49.64%,预测编码 514个氨基酸,其中含量较高的为 Ala(10.30%)、Gly(9.70%)、Val(9.10%)、Leu(8.90%)和Ser(8.20%);强酸性、强碱性、极性和疏水氨基酸分别占10.70%、11.09%、22.18%和37.16%。亚细胞定位及与拟南芥中的AtEPSPS蛋白比对分析显示,CeEPSPS属于叶绿体定位蛋白,其N端的前70个残基为信号肽,长度与所分析的多数物种相近;值得注意的是,紫萍(Spirodela polyrhiza)中的SpEPSPS蛋白的信号肽仅有13个残基,由碱基插入与缺失造成,其是否足以介导叶绿体定位还有待研究。EPSPS成熟蛋白的理论分子量、等电点、总平均疏水指数、脂肪族指数、不稳定系数在所分析的物种中均非常接近,暗示其均为稳定的疏水型酸性蛋白(表3)。TMHMM和SMART分析显示,CeEPSPS无跨膜螺旋,其 77~508位为 EPSP合酶结构域(PF00275,E值 2.7e-150)(图2);InterPro分析显示,其主要参与芳香族氨基酸的生物合成(GO:0009073),具有 3-磷酸莽草酸-1-羧基乙烯基转移酶活性(GO:0003866)、甲基以外的芳烃基转移酶活性(GO:0016765)以及催化活性(GO:0003824)等分子功能。
2.3.2 SNP分析 如图2所示,可引起草甘膦抗性改变的Thr102、Ala103和Pro106在所比对的蛋白间高度保守,暗示‘热研1号’的CeEPSPS蛋白可能对该除草剂敏感。为摸清团队所收集的其他种质是否存在抗性突变,我们利用前期获得的基因组重测序数据(即将56份材料的叶片DNA等量混合后测序)[7]进行多态性位点鉴定,共计获得20个单核苷酸多态性(SNP)位点(表3);经深入分析显示,它们仅造成3个氨基酸的变异,即对应于成熟蛋白的His116Asn、Leu139Val和Asn153Asp。

表3 SNP分布情况Tab.3 Distribution of SNPs identified in this study
2.3.3 进化分析 为揭示 EPSPS的进化特征及油莎豆的分类学地位,研究构建了包括拟南芥EPSPS在内21个蛋白的无根进化树。如图3所示,AtEPSPS和AtEPSPS2作为唯一的双子叶植物来源蛋白位于进化树的基部,而单子叶植物来源蛋白较好地聚为7支,其中,同为莎草属的CeEPSPS和 CrEPSPS聚在一起,并与同为莎草科的ClEPSPS形成一个分支;同为禾本目但隶属于禾本科的 OsEPSPS、HvEPSPS、BdEPSPS、SiEPSPS、SbEPSPS和 ZmEPSPS聚为莎草科的姊妹支;棕榈目棕榈科的 PdEPSPS1/2、EgEPSPS1/2聚为一支;禾本目凤梨科的AcEPSPS1/2、芭蕉目芭蕉科的MaEPSPS1/2、微子目兰科的PeEPSPS和天南星目浮萍科的SpEPSPS则形成另外的5个分支。由于来自同一物种的旁系同源蛋白都聚在一起,并具有很高的序列相似性(89.3%~100.0%),暗示其可能通过物种特异性的基因重复产生。
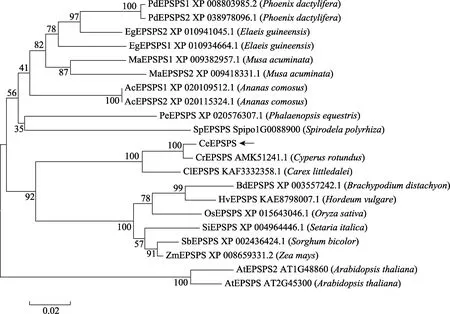
图3 EPSPS蛋白的进化分析Fig.3 Phylogenetic analysis of EPSPS proteins
2.4 CeEPSPS基因的表达特性分析
为揭示CeEPSPS的表达特性,并为草甘膦的适时施用提供参考,研究采用qRT-PCR技术检测基因在叶片(分幼嫩、成熟和衰老3个时期)、芽、芽茎和块茎等主要组织中的表达水平。如图4所示,CeEPSPS在成熟叶片和块茎中的表达丰度最高,显著高于芽、幼嫩叶片、衰老叶片和芽茎;从叶片的发育过程来看,基因呈现先增后减的趋势,其在成熟叶片中的表达水平显著高于幼嫩和衰老叶片,而幼嫩和衰老叶片之间差异不显著。

图4 CeEPSPS在不同组织中的表达模式Fig.4 Expression patterns of CeEPSPS in various tissues
3 讨论
作为莽草酸途径的关键酶,EPSPS催化烯醇式丙酮酸和莽草酸-3-磷酸形成 EPSP,继而参与酪氨酸、苯丙氨酸、色氨酸等芳香族氨基酸的生物合成,以及香豆素、绿原酸、类黄酮等次生代谢物的合成。同时,EPSPS又是广谱型除草剂草甘膦的作用靶标[2]。虽然绝大多数植物的 EPSPS对草甘膦高度敏感,但高选择压引起的基因扩张及关键位点的突变赋予了部分群体或生态型的抗药性,这些位点包括 Thr102、Ala103和 Pro106[2,4,11-12]。Pro106突变是抗性获得的常见形式,至今已在 10种以上的杂草中发现,如Pro106Ser/Thr/Ala/Leu,虽然此类突变多赋予低水平的抗性(<10倍),但Pro106Ser突变在黑麦草、长芒苋和Leptochloa virgata等植物中也发现了高水平的抗性(>10倍);Thr102Ile突变一般赋予高水平的抗性,但Thr102Ser突变仅赋予低水平的抗性;与单突变相比,多位点突变如 Thr102Ile+Pro106Ser/Ala/Thr(TIPS/A/T)、Thr102Ile+Ala103Val+Pro106Ser可赋予更高水平的草甘膦抗性[4,13-16]。其中,编码TIPS突变的EPSPS基因已成功用于玉米(Zea mays)除草剂抗性育种[17]。在油莎豆中,至今还未见有关于草甘膦抗性变异的报道。
本研究利用基因组和转录组数据对油莎豆及另外13种代表性单子叶植物的EPSPS基因进行了鉴定,并在此基础上完成了CeEPSPS编码区序列的分离及植物过表达载体的构建。这些单子叶植物包括莎草科的康藏嵩草,禾本科的水稻(Oryza sativa)、大麦(Hordeum vulgare)、二穗短柄草(Brachypodium distachyon)、谷子(Setaria italica)、高粱(Sorghum bicolor)和玉米,凤梨科的菠萝,棕榈科的椰枣和油棕,芭蕉科的香蕉,兰科的小兰屿蝴蝶兰,以及浮萍科的紫萍。结果显示,与拟南芥一样,这些植物的EPSPS基因均含有7个内含子;多数植物以单拷贝的形式存在,而椰枣、油棕、菠萝和香蕉中各自存在1对重复基因,主要通过物种特异性的基因重复产生(由于菠萝AcEPSPS1和AcEPSPS12的编码区序列完全一样,不排除是基因组错误组装所致);进化树依科将所分析的蛋白分成9支,其中,属于双子叶的拟南芥位于基部,CeEPSPS与CrEPSPS的亲缘关系最近,并与ClEPSPS形成莎草科分支;莎草科分支与禾本科分子形成姊妹支,支持油莎豆划归为禾本目[7,18-19]。
研究克隆到的CeEPSPS编码514个氨基酸,其序列长度与其他物种相近,并含有叶绿体定位的信号肽和高度保守的 EPSPS合酶结构域。CeEPSPS成熟蛋白的分子量为47.32 kDa、pI为5.49、GRAVY 为 0.069、AI为 93.76、II为 31.73,与其他物种中同源蛋白的理化特性基本一致,即分别为 46.86~47.55 kDa、5.13~5.58、0.011~0.111、91.78~95.96、28.38~35.46,暗示其为稳定的疏水型酸性蛋白。进一步的序列比对证实CeEPSPS不存在已知的草甘膦抗性突变。育种实践中也发现团队收集的 56份油莎豆种质均对草甘膦高度敏感,研究利用前期获得的基因组重测序数据[8]进一步对相关SNP进行了鉴定,虽然获得了20个SNP,但它们均与已知的抗药性变异无关,这表明现有群体不存在抗药性突变。从表达模式来看,CeEPSPS倾向于在成熟叶片和块茎中表达,这与其活跃的次生代谢一致。
综上,研究完成了包括油莎豆在内的代表性单子叶植物EPSPS基因的鉴定、CeEPSPS编码区的分离与植物过表达载体的构建及序列特征、遗传变异、进化和表达特性分析,较好地揭示了油莎豆的分类学地位,初步证实团队前期收集的56份油莎豆种质不存在草甘膦抗性变异,这为油莎豆的开发与合理栽培提供了理论支撑,并为通过利用过表达或基因编辑技术创制油莎豆新种质奠定了坚实的基础。
