玉米生物钟基因ZmPRR1-2的克隆及表达分析
2023-02-21董柯清王雷立刘青青张严玲王翠玲
董柯清,王雷立,刘青青,张严玲,王翠玲
(河南科技大学 农学院,河南洛阳 471023)
生物对昼夜交替中日照时间长短的周期性变化的响应被称为光周期现象,普遍存在于动植物中。光周期途径是植物开花的主要途径之一,生物钟是光周期途径传递信号的重要一环[1-3]。中央振荡器是生物钟的核心部分,由LHY(late elongated hypocoty1)、CCA1(circadian clock associated 1)和伪应答调节蛋白家族(pseudo-response regulators,PRRs) 构成的负反馈调节环组成[4-5],不仅能对从输入途径中传递的不同信号进行加工处理,还能经过一系列的反馈调节机制,在保持自身的生物节律的同时,将节律输出给下游的节律调节基因GI等[6-7]。PRR基因家族作为生物钟中央振荡器的主要组成部分,在生物钟系统中起着重要作用,目前拟南芥中共发现了5个PRRs成员,分别命名为PRR1、PRR3、PRR5、PRR7和PRR9,其中PRR1又被称为TOC1(timing of cab expression 1)[8-9]。
TOC1在结构上有PRR家族均含有的REC和CCT结构域[10],但是没有PRR5、PRR7和PRR9的EAR结构域,因此其发挥转录抑制功能的机制与其他PRR家族成员不同[11]。研究发现TOC1与LHY和CCA1构成了中央振荡器的核心负反馈调控环,在这个负反馈调控环中TOC1的表达量达到最高时CCA1/LHY能抑制TOC1的表达,而当CCA1/LHY表达量最高时TOC1又可以抑制CCA1/LHY的表达[12-15]。PRR家族成员在相互调控下呈现节律性表达,TOC1在黄昏后1~3 h达到表达高峰[8]。其作为生物钟重要的晚间表达基因,随着夜间蛋白质含量增加,还能够抑制从早上到晚上表达的生物钟基因[16-17]。目前在模式植物拟南芥(Arabidopsisthaliana)中研究发现TOC1参与生物钟循环并调控其下游基因表达[18-20],能抑制PIFs介导的下胚轴伸长[20-22],还与响应冷胁迫有关[22]。除此之外,水稻(Oryzasativa)中的TOC1也有相应冷胁迫的功能[23],还发现OsTOC1有抗旱相关作用[24]。大豆(Glycinemax)中PRR1参与调控ABA信号及气孔孔径和非生物逆境反应等[25]。目前关于TOC1的研究主要集中在拟南芥和水稻中,其在玉米中的功能及调控关系还不清晰。通过全基因组鉴定发现玉米中存在2个PRR1的同源基因,将其命名为ZmPRR1-1和ZmPRR1-2[26],其中前人克隆研究了ZmTOC1[27],经鉴定该基因为ZmPRR1-1,关于ZmPRR1-2至今还无相关研究。
本研究以玉米骨干自交系‘黄早4’(HZ4)为材料,克隆了ZmPRR1-2基因,并对其进行生物信息学分析和表达规律分析,以期为揭示玉米生物钟基因ZmPRR1-2在玉米光周期反应途径中的调控机制奠定基础。
1 材料和方法
1.1 植物材料与处理
本研究所用试验材料为玉米自交系‘黄早4’,种植于河南科技大学人工气候室中,在玉米苗期选取长势良好的幼嫩叶片组织提取RNA进行基因克隆。在玉米吐丝期,选取3株长势一致的玉米分别取其根、茎、叶、花丝、未授粉果穗、叶耳、叶鞘及雄穗等组织,经过液氮速冻后保存于-80 ℃冰箱中,用于进行组织特异性表达分析。昼夜节律表达分析所用的玉米材料分别种植于人工气候室的长、短日照处理室中,短日照条件为9 h光照/15 h黑暗,长日照条件为15 h光照/9 h黑暗,温度控制为(28±2)℃,相对湿度为40%~60%。前期试验显示‘黄早4’光周期诱导期是6~9叶期,本试验选择在第8片叶完全展开时选取长势一致的3株玉米植株,从光照开始时采集叶片,每隔3 h取1次,每次约0.1 g,连续取样48 h,各个时间点的叶片经过液氮速冻后保存于-80 ℃冰箱中备用。
1.2 方 法
1.2.1 总RNA的提取及cDNA合成采用TRIzol法提取玉米‘黄早4’的叶片及其他不同组织的总RNA,反转录用PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)(TaKaRa,中国大连)进行,具体步骤参照说明书,用于qRT-PCR试验的样品所反转录的RNA量为1 μg。
1.2.2ZmPRR1-2基因的克隆使用Primer Premier 6.0软件以已公布的玉米参考基因组B73序列(Zm00001d017241_T001)为模板设计获得特异性引物(表1)。用反转录得到的cDNA作为模板进行ZmPRR1-2基因的cDNA克隆。PCR反应体系为:KOD OneTMPCR Master Mix 25 μL,DNA模板2 μL,上下游引物各1.5 μL,ddH2O 20 μL。反应条件:94 ℃预变性2 min;98 ℃变性10 s,56 ℃退火5 s,68 ℃延伸11 s,37个循环;最后4 ℃保存。扩增产物用1%的琼脂糖凝胶电泳检测,将符合要求的产物送至生工生物工程(上海)股份有限公司进行测序。

表1 特异性引物信息表Table 1 Specific primer sequences
1.2.3 生物信息学分析运用DNAMAN6.0软件进行序列比对分析,利用ExPASy (http://web.expasy.org/protparam/)分析氨基酸的理化性质,利用MEME(https://meme-suite.org/meme/)对ZmPRR1-2基因编码氨基酸序列进行保守motif分析,利用NetPhos3.1(https://services.healthtech.dtu.dk/service.php?NetPhos-3.1)在线软件进行磷酸化位点预测,利用NPS@:HNN (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_server.html) 预测蛋白质的二级结构,利用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)和Plant-mPLocserver(http://www.csbio.sjtu.edu.cn/cgi-bin/PlantmPLoc.cgi)进行跨膜结构域和亚细胞定位预测,利用SignalP(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析,利用NCBI的保守结构域数据库CDD-search(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)分析保守结构域。应用MEGA7.0软件邻近法构建系统发生树。
1.2.4 组织特异性及节律表达分析分别提取各个样本的总RNA并反转录成cDNA第一链,用qRT-PCR技术进行ZmPRR1-2基因的组织特异性和节律表达分析,内参基因为Ubiquitin,qRT-PCR试验所用特异引物具体信息见表1。用荧光定量 PCR 试剂盒(全式金,北京)进行试验,反应总体积为20 μL,上下游引物(10 μmol/L)各 0.5 μL,cDNA模板1 μL,无核酸酶水8 μL,2×PerfectStart Green qPCR Super Mix 10 μL。q RT-PCR反应程序为:94 ℃预变性30 s,94 ℃变性5 s,57 ℃退火15 s,72 ℃延伸10 s,45个循环。每个处理均进行3次技术重复及3个生物学重复,得出的数据采用2-ΔΔCt法进行分析。
2 结果与分析
2.1 ZmPRR1-2基因的克隆及序列比对
cDNA的扩增结果与预测片段大小一致(图1),克隆得到的cDNA含有一个完整的开放阅读框(ORF),ZmPRR1-2基因CDS序列长度为1 554 bp,含有6个外显子和5个内含子,共编码517个氨基酸。比对发现ZmPRR1-2基因在‘黄早4’和B73中CDS区域大小相同,但是存在14处单碱基的差异,最终由于密码子的简并性造成2个玉米材料的ZmPRR1-2蛋白共有7个氨基酸不同。保守结构域预测发现,ZmPRR1-2基因所编码的氨基酸序列含有1个REC结构域和1个CCT结构域,分别在氨基酸序列的第30~133个氨基酸区域及第442~483个氨基酸区域,REC结构域与CCT结构域之间的部分为IR(internal region)区域。结合氨基酸序列比对结果发现‘黄早4’和B73的ZmPRR1-2氨基酸序列在REC结构域中序列完全一致,高度保守,但在IR区域中有5个氨基酸的差异,在CCT结构域中有1个氨基酸的差异(图2)。

M.Marker;H4.Huangzao 4图1 ZmPRR1-2基因PCR扩增结果Fig.1 PCR amplification results of ZmPRR1-2 gene

图2 玉米自交系‘黄早4’和B73中ZmPRR1-2氨基酸序列比对Fig.2 Sequence alignment of ZmPRR1-2 proteins in ‘Huangzao 4’ and B73
2.2 ZmPRR1-2蛋白的理化性质分析及结构预测
使用在线软件ExPAsy对ZmPRR1-2基因所编码的氨基酸进行理化性质分析,结果表明,‘黄早4’中ZmPRR1-2基因所编码517个氨基酸,分子式为C2473H3910N734O796S29,总平均亲水性系数为-0.603,属于亲水性蛋白,不稳定系数为49.45,属于不稳定蛋白,其相对分子质量为57.59 kD,理论等电点为6.41。ZmPRR1-2蛋白中相对较多的氨基酸是丝氨酸(Ser,S),占比11.6%,色氨酸(Trp,W)含量最少仅占0.8%(表2)。二级结构分析表明该蛋白主要以无规则卷曲和α-螺旋为主体结构,分别含有23.21%的α-螺旋、59.96%的无规则卷曲、13.35%的延伸链以及3.48%的β折叠。亚细胞定位预测结果显示其定位于细胞核内,信号肽与跨膜结构域预测结果表明ZmPRR1-2蛋白不包含跨膜结构域和信号肽。磷酸化位点预测结果显示ZmPRR1-2蛋白共有55处磷酸化位点,其中丝氨酸(Ser,S)为最多(38个),占比69.09%,苏氨酸(Thr,T)次之(14个),酪氨酸(Tyr,Y)最少(3个),该结果也符合在真核生物中磷酸化主要发生在丝氨酸上这一结论[28]。
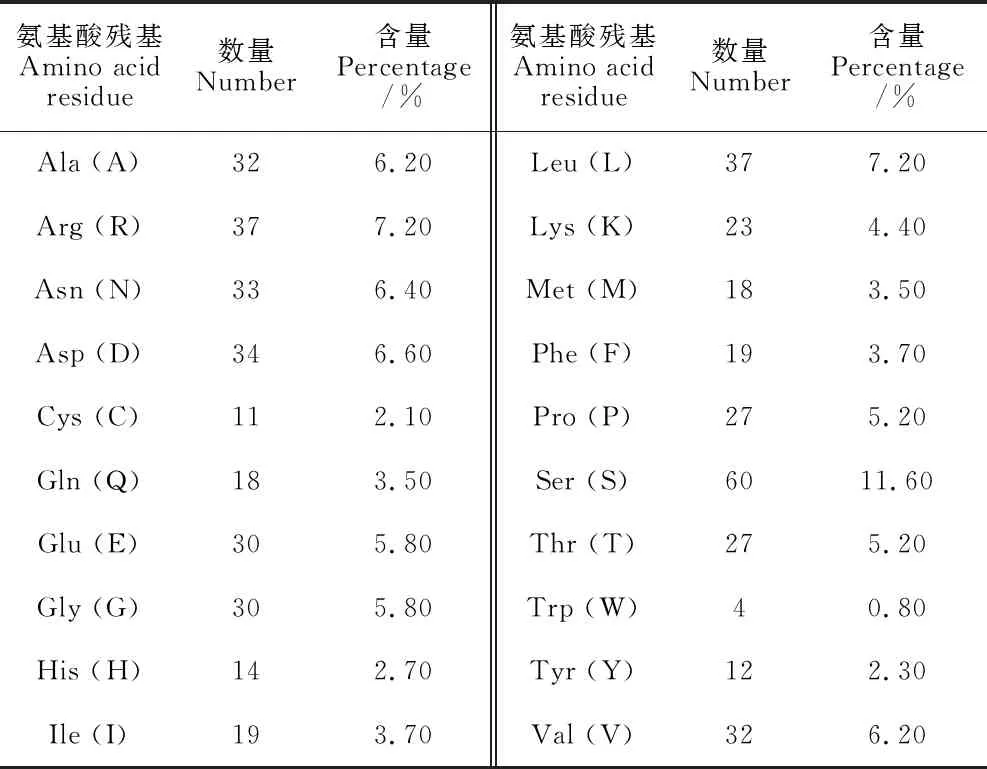
表2 ZmPRR1-2蛋白中的20种氨基酸含量表Table 2 The list of 20 amino acids in ZmPRR1-2 protein
2.3 多序列比对及系统进化分析
以本试验克隆所得的氨基酸序列为模板,在NCBI数据库进行Blast同源比对,将所得结果筛选后进行序列比对,PRR1蛋白在多个物种中的2个重要保守结构域REC和CCT的氨基酸序列比对结果(图3)显示,PRR1蛋白的REC和CCT结构域在不同物种中相对保守,特别是禾本科物种的PRR1蛋白保守性非常高。进一步系统进化关系分析表明,玉米ZmPRR1-2基因与同属于禾本科黍亚科的高梁PRR1(Sorghumbicolor,XP_002452462.1)亲缘关系最近(89.89%),并且与谷子(Setariaitalica,XP_004953120.1)、水稻(Oryzasativa,BAD38854.1)、大麦(Hordeumvulgare,AEW48242.1)等禾本科作物处于同一进化分支中,表明PRR1基因进化过程中在禾本科内相对保守,而与双子叶植物拟南芥(Arabidopsisthaliana,Q9LKL2.1),及油棕(Elaeisguineensis,XP_010943377.1)、椰子(Cocosnucifera,EHA8587847.1)等棕榈科植物亲缘关系较远(图4)。

A.REC结构域的保守序列;B.CCT结构域的保守序列;Zm.玉米;Td.野生二粒小麦;Sb.高粱;Ph.黍;Si.谷子;La.高羊茅;Bt.马甲竹;Tu.乌拉尔图小麦;Hv.大麦;At.拟南芥;Zo.姜;Ma.小果野蕉;Ao.石刁柏;Ac.凤梨;Pd.海枣;Eg.油棕;Cn.椰子;Os.水稻;Pm.糜子;Bd.二穗短柄草图3 ZmPRR1-2蛋白的保守结构域的多序列比对A.Conserved sequence of REC domain;B.Conserved sequence of CCT domain;Zm.Zea mays;Td.Triticum dicoccoides;Sb.Sorghum bicolor;Ph.Panicum hallii;Si.Setaria italica;La.Lolium arundinaceum;Bt.Bambusa tulda;Tu.Triticum urartu;Hv.Hordeum vulgare subsp.vulgare;At.Arabidopsis thaliana;Zo.Zingiber officinale;Ma.Musa acuminata subsp.malaccensis;Ao.Asparagus officinalis;Ac.Ananas comosus;Pd.Phoenix dactylifera;Eg.Elaeis guineensis;Cn.Cocos nucifera;Os.Oryza sativa;Pm.Panicum miliaceum;Bd.Brachypodium distachyonFig.3 Multiple sequence alignment of conserved domain of ZmPRR1-2 and other PRR1-2

图4 ZmPRR1-2蛋白的系统进化分析Fig.4 Phylogenetic analysis of protein sequences of ZmPRR1-2 and other PRR1-2
2.4 不同组织表达分析
不同组织的表达分析结果(图5)显示:在玉米‘黄早4’植株的根、茎、叶、花丝、果穗、叶耳、叶鞘及雄穗这8个组织部位中,ZmPRR1-2基因的相对表达水平在叶片中最高,并显著高于其他7个组织,在果穗和花丝相对较低,且显著低于雄穗,推测玉米中ZmPRR1-2基因主要在叶片中进行表达并发挥功能,参与玉米光周期调控,并且该基因在玉米开花转化过程中对雄穗发育和雌穗发育的调控功能存在差异。
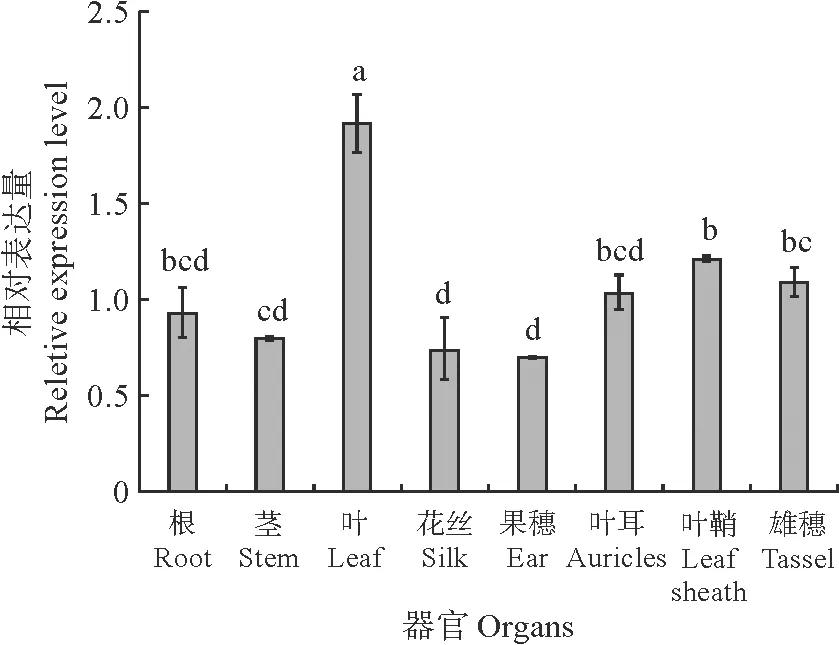
不同小写字母表示在0.05水平差异显著(P<0.05)图5 玉米 ZmPRR1-2基因的组织特异性表达分析Different normal letters indicate significant difference at 0.05 level (P<0.05)Fig.5 Tissue-specific expression analysis of ZmPRR1-2 gene
2.5 昼夜节律表达分析
对长/短日照条件下玉米‘黄早4’的ZmPRR1-2基因进行连续48 h的表达分析,结果(图6)表明,ZmPRR1-2基因在两种日照条件下均具有以24 h为一个周期的节律性表达规律。且在两种光照条件下,在光照起始时分ZmPRR1-2的相对表达水平较低。短日照条件下于光照后3 h起始ZmPRR1-2基因的表达,在光照后12 h或黑暗后3 h达到表达高峰;长日照条件下在光照后6 h起始该基因的表达,于光照后15 h或光照刚刚结束时达到表达高峰,这也使得ZmPRR1-2基因的表达高峰在长/短日照处理下相差3 h(图6)。该结果与前人对ZmPRR1-1的研究结论相一致[27]。

横坐标的实心方框显示黑暗处理时长,空心方框为光照处理时长图6 玉米ZmPRR1-2基因的昼夜节律表达分析The solid box portion of the abscissa means the time that are dark,and the hollow box portion is lightedFig.6 Circadian rhythm expression of ZmPRR1-2 gene
3 讨 论
植物生长发育受到生物钟的严密调控,PRRs家族基因是生物钟系统的重要组成部分中央振荡器的核心组成部分,PRR1(TOC1)作为PRRs基因家族最重要的成员,与MYB家族成员LHY和CCA1组成中央振荡器的核心负反馈环,是中心反馈环的关键组成要件,在植物光周期调控开花途径中发挥着关键作用[29]。在拟南芥中,CCA1和LHY在清晨时由光激活表达,在黎明时表达量最高,LHY和CCA1蛋白与TOC1启动子的黑夜元件(AAATATCT)相结合从而抑制TOC1在白天表达。随后,早晨环中PRR9的表达由于TOC1被抑制而得到解除,PRR9/PRR7/PRR5依次积累,极大地抑制了CCA1的表达,随着CCA1/LHY表达水平的下降,从而可解除对TOC1基因表达的抑制作用,TOC1表达量逐渐升高,傍晚表达量达到峰值[4,6]。TOC1基因表达量过高或过低时,会造成生物钟周期变短或延长,TOC1过量表达的突变体中生物钟紊乱,表明TOC1的节律表达和蛋白丰度对于维持生物钟的稳定发挥重要作用,TOC1基因表达节律失衡会造成植物细胞周期紊乱、开花时间异常、甚至影响植物正常的生长发育[30-31]。本研究中玉米ZmPRR1-2基因的昼夜节律表达规律与拟南芥TOC1基因的表达高度一致,在光照开始时,由于CCA1和LHY蛋白对ZmPRR1-2基因的抑制作用,ZmPRR1-2基因的表达水平非常低。ZmPRR1-2蛋白的减少解除了其对PRR9基因的抑制作用,随着PRR9蛋白的增加,反过来抑制了ZmCCA1基因的表达,因而逐渐解除了CCA1对ZmPRR1-2基因的抑制作用,ZmPRR1-2基因的表达量逐渐增加。本研究中在短日照条件下于光照开始3 h后ZmPRR1-2基因的表达量开始逐渐上升,而长日照条件下在光照开始6 h后ZmPRR1-2基因的表达量才开始逐渐上升,表明光周期的长短对解除CCA1对ZmPRR1-2基因的抑制作用有重要影响,至于是光照时间还是黑暗时间发挥更为重要的作用,需要进一步探索。韦小敏试验结果表明暗间断能提高ZmCCA1、ZmGI基因的表达量,而降低ZmCN8和hd6的表达水平[32]。因此可以推测长日照条件下相对较长的光照时间有利于ZmCCA1的表达,故而延长了其对ZmPRR1-2基因的抑制作用,以致于ZmPRR1-2基因的表达起始时间比短日照下推迟了3 h,进一步试验验证这一假说将是一个非常有意义的研究课题。
同源比对和系统进化结果显示,玉米中ZmPRR1-2基因与高粱、水稻、乌拉尔图小麦等禾本科植物亲缘关系较近,序列一致性非常高,但是与这些作物不同的是,在玉米中PRR1基因的同源基因有2个,分别是ZmPRR1-1和ZmPRR1-2,分别位于第4和第5染色体,推测原因可能与玉米基因组在与高粱分化后,大约5~1 200万年前经历的一次四倍体化事件有关,玉米的四倍体化的历史导致玉米基因组中直系同源基因的数量明显高于其近缘系高粱和蜀黍族的其他核心作物[8,26]。生物钟系统的调控网络在生物钟相关基因节律表达的动态变化过程中维持平衡,从而保持其功能的稳定性,如果这2个基因均在昼夜节律网络中发挥作用,则玉米生物钟调控网络可能会变得更加复杂。那么ZmPRR1-1和ZmPRR1-2是否会出现功能分化或者功能冗余?它们如何参与玉米生物钟系统的调控网络,仍有待阐明。
LHY和CCA1蛋白对TOC1基因的抑制作用的分子机制已经阐述的非常清楚[13]。然而关于TOC1对CCA1和LHY的抑制作用的分子机制,至今尚不明晰。与其他PRR家族成员不同的是,PRR9、PRR7和PRR5蛋白能通过其EAR结构域与TOPLESS和HDA6(histone deacetylase 6)形成一个复合体,结合到CCA1和LHY的启动子上抑制其转录,而TOC1没有EAR结构域,不能直接与CCA1的启动子结合从而抑制其转录[11]。本研究结果表明ZmPRR1-2蛋白含有REC和CCT两个重要结构域,符合其作为PRR家族成员的基本结构特征,而其IR区间与拟南芥TOC1一样不含有其他家族成员所共有的重要基序EAR-motif[20]。Pruneda-Paz等[15]的研究表明TOC1可以与TCP转录因子CHE(CCA1 hiking expedition)互作,而CHE能结合到CCA1的启动子的TBS位点(GGTCCCAC),从而抑制CCA1的表达。又有研究表明TOC1可通过其CCT结构域的T1ME元件(TGTG)直接结合到CCA1和LHY的启动子上从而抑制二者的转录[20]。由此可见,TOC1对CCA1和LHY的抑制作用的分子机制非常复杂,到底ZmPRR1-2是否能通过其CCT结构域直接结合到ZmCCA1和ZmLHY的启动子上,抑或是通过与CHE蛋白的互作间接地结合到ZmCCA1和ZmLHY的启动子上,从而抑制其表达,需要进一步深入研究。而且,玉米中存在ZmPRR1-1和ZmPRR1-2两个TOC1的同源基因,使得问题更为复杂,这两个基因如何与CCA1和LHY相互抑制是玉米生物钟调控机制研究中需要解决的关键问题。
综上所述,本研究以玉米自交系‘黄早4’为试验材料,扩增获得玉米PRR家族转录因子ZmPRR1-2,并分析了其组织性特异表达特征和不同光周期环境下的昼夜表达规律,初步验证了其昼夜节律表达节律与拟南芥TOC1高度一致,推测其在玉米光周期调控开花途径中发挥重要作用,为揭示其在玉米生物钟中的分子功能奠定了基础,但是ZmPRR1-2与CCA1和LHY的相互反馈抑制调控的分子机制需要进一步深入研究。
