ARMS-PCR技术在玫瑰果鉴别中的应用
2023-02-08邵婕杨正修邹强陆峥飞李凡
邵婕,杨正修,邹强,陆峥飞,李凡*
1. 康宝莱蕾硕(湖南)天然产物有限公司(长沙 410100);2. 康宝莱全球测试中心HP1实验室(托伦斯 90502)
玫瑰果(Rosa canina)在世界上有着悠久的药食两用历史。玫瑰果中多酚、黄酮、维生素C等活性物质含量丰富,具有抗氧化、抗癌、抗辐射和抑菌等多种药理作用[1]*。但玫瑰果原料品种繁多,蔷薇属果实的化学成分、抗氧化能力和个体酚类物质含量存在显著差异[2]*。刺玫果(Rosa duvarica)和玫瑰果属于同一植物属,两者不仅在外观上非常相似,活性物质也非常相似,主要包括黄酮、多糖、三萜等[3]*。由于化学成分的相似性,常见的化学鉴定方法难以对二者进行区分,使得玫瑰果掺假特别是混合掺假的检测具有挑战性。扩增阻滞突变系统-聚合酶链式反应(amplification refractory mutation system-PCR,ARMSPCR)技术[4]*常用于遗传疾病[5]*、基因分型[6]*和微生物学[7]*等许多快速检测研究领域,在植物鉴定和分析化学中的应用鲜有报道。
试验通过对玫瑰果和刺玫果基因组序列的比较分析,建立一种基于特征SNP位点的ARMS-PCR方法,可用于鉴定玫瑰果和刺玫果。
1 材料与方法
1.1 材料与设备
化学标准品Isoquercetin(ChromaDex ASB-00009505-025)。
植物对照药材(Botanical Reference Material,BRM):玫瑰果果实BRM(Lot 00030792-473,ASB-00030792-005,ChromaDex. Inc);刺玫果果实BRM(Lot 121255-201203,121255,购自National Institute of Food and Drug Control,NIFDC)。
植物材料:从中国原料市场采购11份玫瑰果用于验证。
Automatic TLC Sampler 4(Chromacim,Moirans,法国);硅胶板(60 F254,20×10 cm,Merck,德国);CAMAG TLC可视化仪(Chromacim,Moirans,法国);DNeasy mericon Food Kit;Qubit 3.0荧光计(Thermofisher,广州,中国);C1000 Touch(BIORAD,上海,中国);Integrated DNA Technologies(Thermofisher,广州,中国);AmpliTaq GoldTM*360 Master Mix(Thermo Fisher,立陶宛)。
1.2 高效薄层色谱(high-performance thin layer chromatography,HPTLC)
样品处理:称取500 mg玫瑰果或刺玫果果实BRM到15 mL试管中,加入10 mL甲醇,涡旋30 s。超声处理15 min。将一部分样品通过0.45 μm滤膜过滤到HPLC进样瓶中。
使用Automatic TLC Sampler 4将样品喷涂到预制硅胶板上,Isoquercetin作为化学标准品喷涂到预制硅胶板上。用流动相(乙酸乙酯/乙酸/甲酸/超纯水100/11/11/26)展开,使用10%硫酸进行显色。用CAMAG TLC可视化仪和Vision cats软件拍摄照片。
1.3 DNA提取
DNA提取过程均根据制造商的说明使用DNeasy mericon Food Kit进行。过程:将50~100 mg玫瑰果或刺玫果果实BRM,1 mL Food Lysis Buffer和2.5 μL Proteinase K加入离心管中,在60 ℃下孵育30 min,并以2 500×g离心5 min;取700 μL上清液于新的离心管中,加入500 μL氯仿,涡旋15 s,并将混合物以14 000×g离心15 min;将350 μL上清液转移到新的离心管中,加入350 μL PB Buffer,将混合液转移到吸附柱中,以17 000×g离心1 min;弃去收集管中溶液;向吸附柱中加入500 μL AW2 Buffer,并以17 000×g离心1 min;将收集管中的溶液再次倒出,并将吸附柱以17 000×g离心2 min;将吸附柱放入新的离心管中,加入100 μL TE缓冲液,并将其以17 900×g离心2 min;使用Qubit 3.0荧光计进行定量。
1.4 DNA Barcode序列采集及植物鉴定
使用2对引物对玫瑰果和刺玫果gDNA(genomic DNA)进行扩增,扩增体系包含10 μL AmpliTaq GoldTM*360 Master Mix、2 μL引物混合物(每种引物5 μmol/L)、5 μL gDNA和3 μL Free-Nuclease Water。在C1000 Touch上进行PCR扩增,反应条件为95 ℃预变性5 min,95 ℃变性30 s(58 ℃退火30 s,72 ℃延伸30 s)×35,72 ℃终末延伸3 min。RbcL-F:5’-TGTAAAACGACGGCCAGTATGTCACCACAAACAGAAAC-3’;RbcL-R:5’-CAGGAAACAGCTATGACCGTAAAA TCAAGKCCACCRCG-3’;ITS2-F:5’-TGTAAAACGACGGCCAGTATCCGATACTTGGTGTGAAT-3’;ITS2-R:5’-CAGGAAACAGCTATGACCGACGCTTCTCCAGACTACAAT-3’。获得RbcL与ITS2基因片段,通过sanger测序获取序列信息。引物由Integrated DNA Technologies(Thermofisher,广州,中国)合成。
双盲试验:将11份玫瑰果市场样品与14份刺玫果果实BRM由试验人员1随机编排顺序并编号,通过DNA barcode技术对25个样品进行鉴定。提取25个样品gDNA后,通过RbcL和ITS2这2对引物对gDNA进行PCR扩增,扩增体系包含10 μL AmpliTaq GoldTM*360 Master Mix、2 μL引物混合物(每种引物5 μmol/L)、5 μL gDNA和3 μL Free-Nuclease Water。在C1000 Touch上进行PCR扩增,反应条件为95 ℃预变性5分钟,95 ℃变性30 s(58 ℃ 退火30 s,72 ℃延伸30 s)×35,72 ℃终末延伸3 min。琼脂糖凝胶电泳检测扩增结果后进行Sanger测序,确认样品种属信息。试验人员2通过ARMS-PCR对25个样品进行鉴别。
1.6 ARMS-PCR
使用AmpliTaq GoldTM*360 Master Mix对玫瑰果与刺玫果的gDNA进行扩增,扩增体系包含10 μL AmpliTaq GoldTM*360 Master Mix、2 μL引物混合物(每种引物5 μmol/L)、5 μL gDNA和3 μL Free-Nuclease Water。在C1000 Touch上进行PCR扩增,反应条件为95 ℃预变性5 min,95 ℃变性30 s(58 ℃退火30 s,72 ℃延伸30 s)×35,72 ℃终末延伸3 min。原始DNA洗脱用于PCR反应,无需调整浓度。每次运行都包括阳性对照和阴性对照,PCR产品在带有DNA染料的2%琼脂糖凝胶中可视化。各基因及其引物的扩增引物序列详见表1。引物由Integrated DNA Technologies合成。

表1 ARMS-PCR引物信息表
1.7 生物信息学分析
从NCBI(https://www.ncbi.nlm.nih.gov/)下载所有序列,从NCBI下载的所有序列在MEGA X中使用ClustalW默认设置进行比对,引物设计使用Primer 3.0 plus(https://www.primer3plus.com/)。DNA条形码序列使用CodonCode Aligner和APE软件进行分析。Sanger测序结果在NCBI上进行比对。
1.8 统计学分析
双盲试验所获得的试验数据通过POI(probability of identification)进行分析[8]*。
2 结果与分析
2.1 HPTLC鉴别玫瑰果和刺玫果
通过HPTLC方法检测玫瑰果和刺玫果,玫瑰果果实含有Isoquercetin[9]*,以Isoquercetin作为化学标准品,其位置和可见度如图1所示。Rf值为0.59。玫瑰果和刺玫果HPTLC指纹图谱非常相似,两者之间的主要区别是检测到的条带的相对密度。因此,HPTLC难以区分玫瑰果和刺玫果。
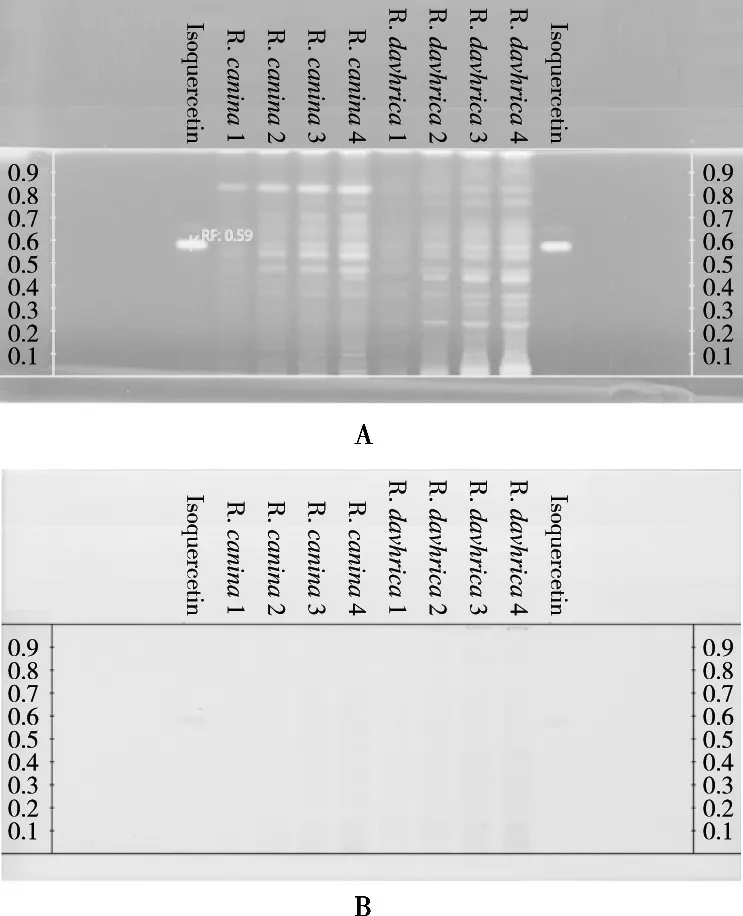
图1 玫瑰果和刺玫果HPTLC指纹图谱
2.2 玫瑰果与刺玫果DNA barcode序列采集及引物设计
由于植物中ITS和rbcL区域的选择压力小、变异大,因此选择该区域进行ARMS-PCR分析可更有效地区分遗传关系密切的物种。通过DNA barcode方法获取玫瑰果和刺玫果的ITS2和RbcL基因片段,Sanger测序得到序列信息(表2)。

表2 玫瑰果及刺玫果ITS2和RbcL序列信息
为验证ARMS-PCR区分玫瑰果和刺玫果的可行性,根据3个不同的SNP位点分别设计P1、P2和P3这3种方案。P1方案的设计基于RbcL区域序列,而P2和P3方案的设计基于ITS2区域序列(图2)。
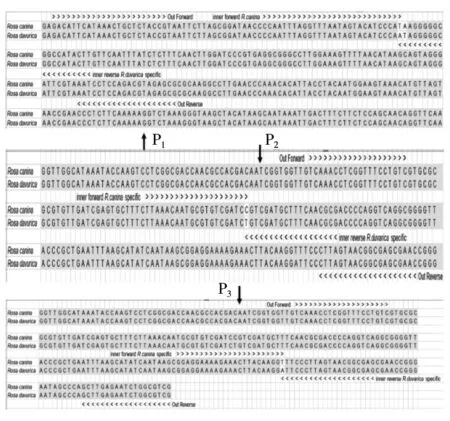
图2 候选ARMS-PCR引物在RbcL(P1)和ITS2区域(P2和P3)中的位置
以P2方案为例,分析ARMS-PCR的工作原理(图3)。基于SNP位置设计4条引物(表1)Outer F,Outer R,Inner F和Inner R,为玫瑰果和刺玫果提供不同长度的PCR片段。Outer F和Outer R配对从基因组DNA中扩增出1个182 bp的片段作为内参,Inner F和Inner R分别作为玫瑰果和刺玫果的特异性引物。玫瑰果DNA存在时,Inner F与玫瑰果的DNA模板之间形成完美匹配,Inner F与Outer R配对扩增出1段135 bp的片段。只有当Inner R与刺玫果的DNA模板之间形成完美匹配时,Inner R才能与Outer F配对扩增出1段86 bp片段。DNA模板中只存在玫瑰果DNA时,可以得到1条182 bp的内参条带和1条135 bp的特异性条带。DNA模板中只存在刺玫果DNA时,可以得到1条182 bp的内参条带和1条86 bp的特异性条带。当DNA模板中同时存在玫瑰果和刺玫果DNA时,可以得到1条182 bp的内参条带,1条135 bp的特异性条带和1条86 bp的特异性条带。

图3 ARMS-PCR的P2设计示意图

接表2
2.3 ARMS-PCR条件优化
为从P1、P2和P3中筛选最佳的试验方案,使用玫瑰果和刺玫果果实BRM,通过DNA提取和PCR扩增,2%琼脂糖凝胶电泳结果显示,P1方案针对玫瑰果和刺玫果进行扩增,其扩增产物一致,均为1条100 bp左右的条带,P3方案针对玫瑰果和刺玫果均得到两条200 bp左右的条带,P1和P3方案均无法区分玫瑰果和刺玫果。P2方案针对玫瑰果可得到1条182 bp的内参条带和1条135 bp的特异性条带,针对刺玫果可得到1条182 bp的内参条带和1条86 bp的特异性条带,可明显区分玫瑰果和刺玫果。结果表明P2方案优于P1和P3方案,可以达到预期结果(图4)。

图4 3种设计的ARMS-PCR的验证结果
由于设计的4条引物的退火温度不同,设计梯度PCR试验。从图5(A)可以看出,最佳退火温度为58℃。PCR的循环数决定扩增产物的产量,循环数太多会易出现非特异条带。为避免非特异性条带干扰,分别测试25 cycles和28 cycles的扩增效率,从图5(B)可以看出,28 cycles和25 cycles均无非特异性条带,但28 cycles电泳信号强度优于25 cycles,表明28 cycles优于25 cycles。

图5 ARMS-PCR条件优化
2.4 ARMS-PCR验证
为验证ARMS-PCR玫瑰果鉴定的准确性,通过双盲试验测试玫瑰果市场样品和刺玫果果实BRM,25个样品经DNA barcode技术进行鉴定。ARMS-PCR结果表明(图6),S-1,S-3,S-6,S-8,S-9,S-10,S-12,S-15,S-16,S-17,S-21,S-22,S-24,S-25这14个样品被鉴定为刺玫果,S-2,S-4,S-5,S-7,S-11,S-13,S-14,S-18,S-19,S-20,S-23这11个样品被鉴定为玫瑰果。该结果与DNA barcode方法结果完全一致。基于POI统计分析,该测定对于鉴别玫瑰果和刺玫果果实原材料具有大于0.9(95%CI)的识别概率。
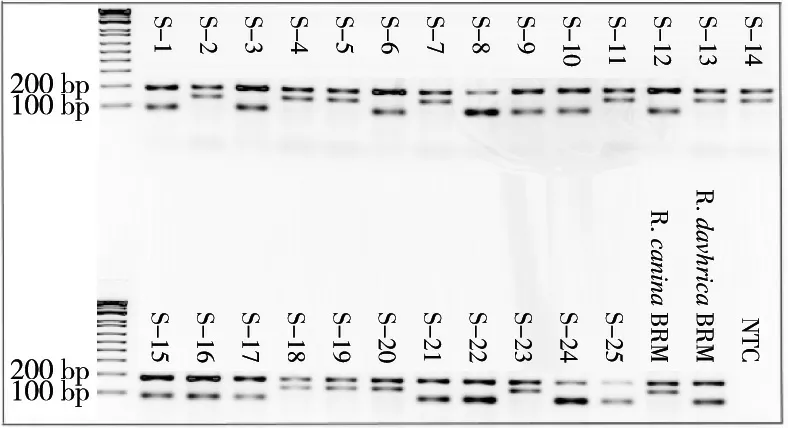
图6 ARMS-PCR验证市场25批样品
2.5 玫瑰果中刺玫果的最低检测限确认
为进一步探索ARMS-PCR的检出限,将玫瑰果和刺玫果BRM粉末按质量比10/90,20/80,30/70,40/60,50/50,60/40,70/30,80/20和90/10进行混合。从这些混合材料中提取DNA用于扩增。结果显示(图7A),100%玫瑰果电泳结果仅有1条182 bp的内参条带和1条135 bp的特异性条带,添加10%~90%刺玫果后,电泳结果显示多1条86 bp的刺玫果特异性条带,且随着刺玫果添加量的升高,135 bp的玫瑰果特异性条带强度降低。为确认刺玫果的最低检测限,对10%的刺玫果进行重复验证,结果如图7(B)所示。结果显示,10次重复结果一致,均可检出86 bp的刺玫果特异性条带。综上所述,该方法对玫瑰果中刺玫果的最低检测限为10%。

图7 ARMS-PCR最低检测限测定
3 结论与讨论
建立一种基于特征SNP位点的ARMS-PCR方法,并应用于玫瑰果及其近缘种刺玫果的鉴定。该方法对玫瑰果和刺玫果的最低检测限为10%。试验方法客观、分辨率高。与DNA Barcode方法相比,检测周期短,无需测序,结果以图位差异的形式呈现,易于进行判断。
为确保目标序列的扩增和鉴定,高质量DNA的获取极为重要。植物材料中蛋白质、脂肪、多糖、多酚等物质含量较高,这些物质可能抑制PCR扩增。因此,在DNA提取过程中尽可能地去除这些物质非常重要。此外,植物的加工过程也会影响DNA的最终产量、纯度以及完整性。
基于DNA技术的植物鉴定方法通常只能得到种属、基因型等信息,而无法获得植物中活性物质的鉴定及定量信息。这些信息通常需要化学方法提供,如LC、LC-MS、GC-MS、NMR。在组学时代,基因组学、蛋白质组学和代谢组学相辅相成,从而能够对各种复杂的植物原料进行全面鉴定。
