基于Super-GBS技术的高粱籽粒酿造相关性状QTL定位
2023-01-16丁延庆徐建霞周棱波张国兵邵明波张立异
丁延庆 徐建霞 汪 灿 周棱波 张国兵 赵 强 邵明波 张立异
(贵州省农业科学院旱粮研究所,贵州贵阳 550006)
高粱(Sorghum bicolorL.Moench)是世界第五大粮食作物,在食品、饲料、纤维、酿造和生物能源原料等方面被广泛应用。在我国,高粱主要种植在西南、华北和东北地区,每年有超过80%的高粱籽粒作为白酒酿造原料[1-3]。高粱籽粒酿造性状包括总淀粉含量、支链淀粉含量、单宁含量、硬度和颜色等。淀粉是产生酒精的主要物质,籽粒总淀粉含量与出酒率成正比,而支/直链淀粉含量与糊化率相关,影响酿造工艺。籽粒硬度由角质(玻璃质)率决定,而角质率与蛋白质含量呈正比,对白酒品质有较大影响。单宁又称原花青素,在白酒酿造过程中不仅具有抑制有害微生物作用,而且能产生丁香酸、丁香醛、香草醛、阿魏酸等化合物,从而赋予白酒独特的香味[4]。此外,籽粒颜色受单宁含量影响,也是酒用高粱育种的一个重要指标[5]。
高粱籽粒酿造性状是受多基因控制的数量性状。结合限制性片段长度多态性(restriction fragment length polymorphism,RFLP)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)和简单重复序列(simple sequence repeat,SSR)等分子标记,利用遗传群体开展高粱数量性状位点(quantitative trait locus,QTL)定位是研究数量性状的重要方法[6]。目前,位于10号染色体上编码颗粒结合淀粉合成酶的Wax基因已被克隆[7]。Murray等[8]利用85个AFLP和68个SSR标记对176个家系的重组自交系(recombinant inbred lines,RIL)群体(BTx623×Rio)开展基因分型,在1、3和4号染色体上确定了4个与籽粒总淀粉含量有关的QTL。基于115个RFLP和85个AFLP标记的基因分型,前人构建了包含125个家系的F5群体(RTx430×Sureno)和379个家系(IS2807×249)的RIL的遗传图谱,并在2、3和7号染色体上定位了4个影响籽粒硬度的QTL[9-10]。在籽粒颜色和单宁含量方面,目前科研人员已经克隆了位于1号染色体上影响籽粒颜色的Y基因[11],以及分别位于4号和2号染色体上控制籽粒单宁含量的Tan1和Tan2基因[12-13]。利用118个SSR和8个Indel标记,白春明等[14]构建了包含325个家系RIL群体(BTx623×Rio)的遗传图谱,在1号、2号、4号和6号染色体上检测到3个与单宁含量和6个与籽粒颜色相关的QTL位点。
随着高粱基因组测序的完成,基于高通量测序的简化基因组测序技术(genotyping by sequencing,GBS)越来越多地应用于高粱QTL定位。GBS是在二代测序的基础上发展起来的一种基于酶切试验对特定区域进行测序从而降低基因组复杂度的方法,具有快速、简便、低成本发现单核苷酸多态性标记(single nucleotide polymorphism,SNP)标记的优点[15]。通过GBS技术,Habyarimana等[16]利 用S.bicolor×S.halepense构 建 的RIL,准确定位了控制高粱籽粒中多酚、单宁和类黄酮含量的QTL。Sukumaran等[17]对包含248个家系的RIL群体(Tx436×MN7645),使用GBS进行基因分型获得了7 144个多态性SNP位点,准确定位了与高粱产量、开花时间和持绿性状相关的QTL。同样利用该技术,Kong等[18]对来源于BTx623×IS3620C的399个RILs构建了包含616个Bin标记的高粱遗传图谱,检测到36个调控株型和开花性状的QTL[18]。Super-GBS是基于GBS进一步发展的简化基因组测序技术,在SNP标记的重现性、稳定性、分型准确性等特点上优于GBS,已应用于遗传图谱构建、QTL定位以及全基因组关联分析等研究[19-21]。但目前利用Super-GBS技术在高粱中开展遗传图谱构建和QTL定位的研究仍鲜见报道。
鉴于此,本研究通过美国品种BTx623与贵州茅台酒专用品种红缨子杂交构建包含205个家系的RIL群体,利用Supper-GBS技术开展全基因组基因分型,以期确定影响籽粒总淀粉含量、支链淀粉含量、单宁含量、硬度和颜色的QTL,并对重要遗传区段内的基因进行注释,确定影响重要QTL的候选基因,旨在为关键基因的进一步克隆和酒用高粱遗传改良提供理论依据。
1 材料与方法
1.1 试验材料
本研究利用贵州省旱粮研究所收集的美国高粱籽粒品种BTx623为母本,贵州酒用高粱品种红缨子为父本配置杂交组合,得到F1植株后,采用单粒传法在贵州贵阳和海南三亚两地进行轮流加代,构建了包含205个家系的RIL群体。BTx623是非糯高粱,总淀粉含量较高,籽粒较硬,颜色为白色,不含单宁;红缨子是糯高粱,籽粒较软,颜色为红色,单宁含量较高。
1.2 表型调查
2020—2021年连续两年分别在贵州贵阳、贵州安顺和海南三亚进行RIL群体及双亲材料种植。小区长2 m,行距60 cm,株距20 cm,人工点播,适时进行常规大田管理工作。在抽穗期,每个株系中选择5个高粱穗用硫酸钠纸袋套袋自交,在整穗完成授粉时,将纸袋换成网袋,直至种子成熟。收获后进行晒干脱粒,选择饱满无损伤的籽粒200 g,利用XDS谷物品质分析仪(美国福斯公司)进行籽粒总淀粉含量(total starch content,TSC)、支链淀粉含量(amylopectin content,AC)和单宁含量(tannin content,TC)的测定:(1)采用改进最小二乘法(modified PLS)回归技术,通过不同散射处理和导数处理建立预测模型;(2)将100份不同类型高粱资源的化学测定值作为验证集,对预测模型进行交叉验证和非参数检验,从而获得最优预测模型;(3)测定时将样品放入分析盘的样品杯中,在波长400~2 489 nm范围内进行3次光谱扫描,每个性状以3株的平均值为最终的表型值。使用GWJ-2谷物硬度计(杭州金科利达公司)测定高粱籽粒硬度(grain hardness,GH),每个株系随机选取30粒种子进行测量,重复3次,取平均值作为表型数据。籽粒颜色(grain color,GC)参照《高粱种质资源描述规范和数据标准》[22]进行分级赋值,1~5级依次对应白色、灰色、黄色、黄褐色和红褐色。
1.3 基因分型
在2020年贵州贵阳环境,采集四叶期RIL家系的新鲜嫩叶,采用十六烷基三甲基溴化铵(cetyltrimethylammonium bromide,CTAB)方法提取DNA,将质检合格的DNA样品送至上海欧易生物医学科技有限公司进行Super-GBS文库的构建,并通过Illumina Nova PE150平台测序。以高粱品种BTx623的基因组序列作为参考(https://phytozome-next.jgi.doe.gov/info/Sbicolor_v3_1_1),使 用bwa和GATK软 件进 行SNP筛 选,利用vcftools软件对获得的SNP分型结果进行过滤,过滤条件为:缺失率小于20%,次等位基因频率大于5%的二等位位点。利用亲本的基因型数据进行多态性标记开发,其中BTx623基因组为参考基因组,红缨子已完成基于深度重测序的全基因组测序[23]。完成亲本间标记开发后,对RIL群体的基因型数据进行标记位点筛选,只保留亲本基因型为纯合且有差异的标记,共获得16 474个多态性标记。
1.4 遗传图谱构建与QTL分析
使用JoinMap 5.0软件对RIL群体的基因型数据进行标记过滤和遗传图谱构建,过滤条件为:(1)删除卡方检验显著性P<0.05的位点;(2)删除RIL群体中基因型缺失值>5%的个体;(3)相似度等于1的位点只保留1个。采用Kosambi函数构建遗传图谱,根据5个籽粒性状的表型数据,使用QTL IciMapping 4.2软件的完备区间作图法(inclusive composite interval mapping,ICIM)进行QTL鉴定,染色体步移长度设定为0.1 cM,逐步回归概率为P<0.001,筛选似然率(likelihood of odds,LOD)大于2.5的QTL位点,最后计算每个QTL的贡献率和加性效应,QTL参照苏代群等[24]的方法进行命名。
1.5 数据分析
利用SPSS 21.0软件对各个环境的表型数据进行平均值、标准差、频率分布等描述性统计分析和相关性分析。在Sorghum QTL(http://aussorgm.org.au/sorghumqtl-atlas/)网站查找已被报道的QTL信息,从Phytozome(https://phytozome-next.jgi.doe.gov/)网站上获得高粱品种BTx623的参考基因组信息。
2 结果与分析
2.1 RIL群体的性状表型变异与相关性分析
在贵州贵阳(2020—2021年)、贵州安顺(2021年)和海南三亚(2020年)的4个环境下开展亲本和RIL群体的表型数据调查。结果表明,除籽粒颜色外,RIL群体的总淀粉含量、支链淀粉含量、单宁含量和籽粒硬度均表现为连续变异,表型平均值在2个亲本表型值范围内,变异系数为1.72%~64.97%。RIL群体各性状的偏度和峰度绝对值均小于1,说明各性状指标均呈正态分布(表1),是受多基因控制且易受环境影响的数量性状,适合进行QTL定位。为了解不同环境下各性状之间的关系,进行了Pearson相关性分析,结果如表2所示。籽粒颜色与单宁含量在4个环境下均呈极显著正相关(P<0.01),R值平均为0.55,变异范围为0.53~0.56;支链淀粉含量与总淀粉含量在所有环境下均呈极显著正相关(P<0.01),R值平均为0.28,变异范围为0.23~0.33。
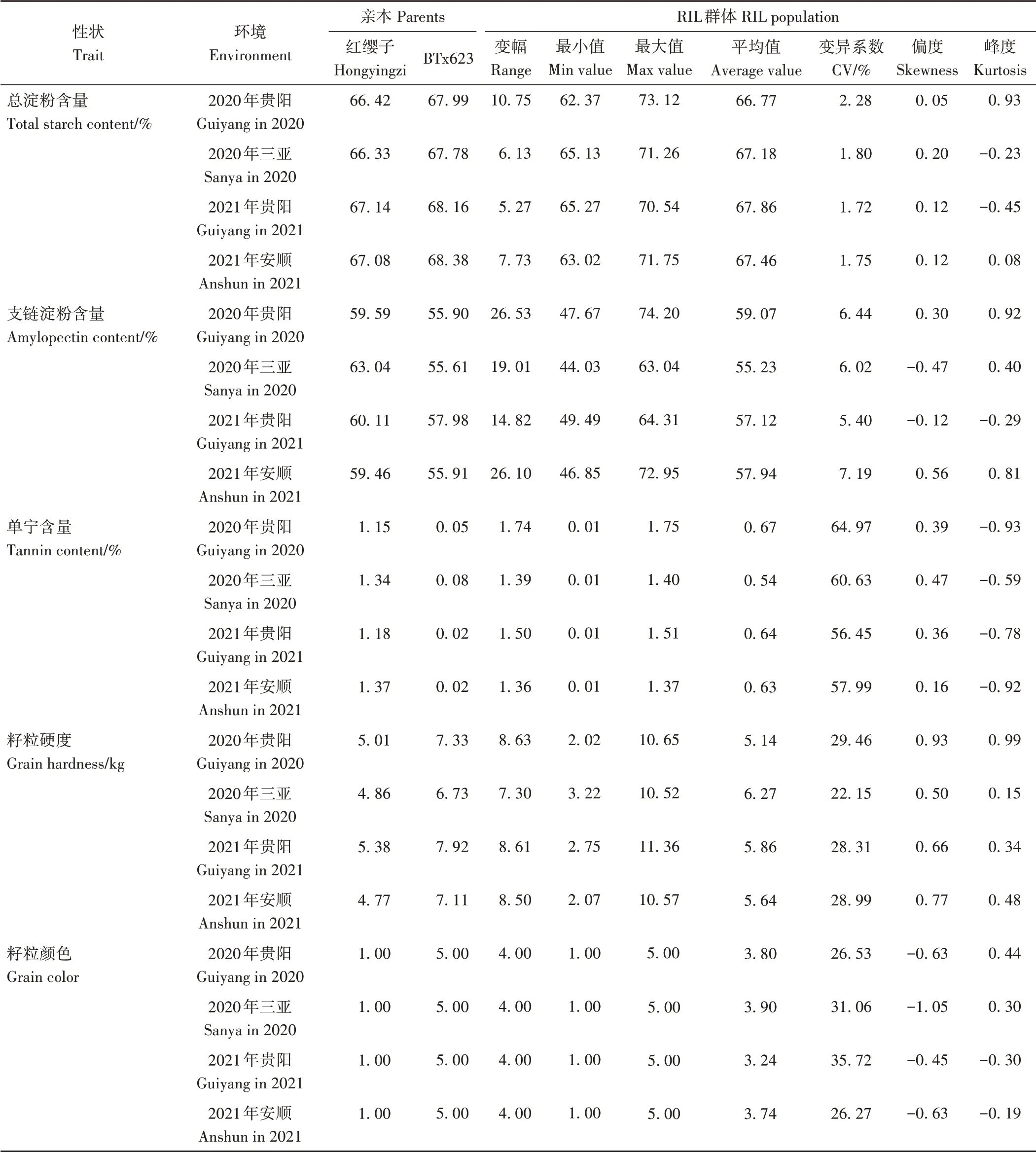
表1 4个环境下亲本及RIL群体籽粒酿造相关性状的表型统计Table 1 Phenotypic statistics of grain brewing related traits in parents and RIL population in four environments
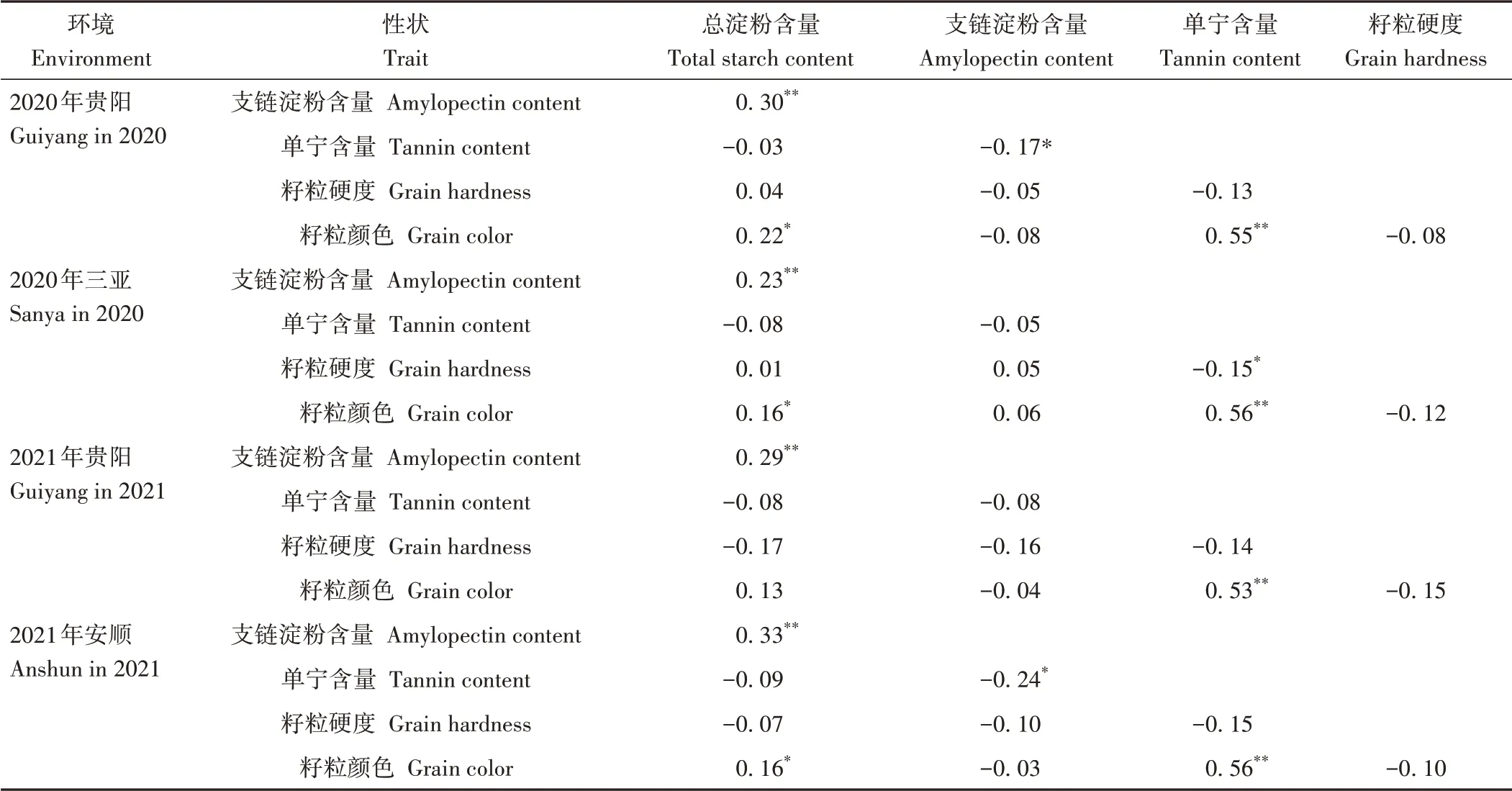
表2 高粱籽粒酿造相关性状的相关性分析Table 2 Correlation analysis of grain brewing related traits in sorghum
2.2 遗传图谱的构建
利用Super-GBS技术对205个家系的RIL群体进行全基因组基因分型,共获得16 474个多态性SNP标记。采用Kosambi函数构建了覆盖了高粱10条染色体的遗传连锁图谱。除去冗余SNP后,最终图谱的标记数为1 910个,图谱长度为905.10 cM,标记间平均距离为0.47 cM(表3)。不同染色体的图谱距离和标记密度变化范围较大,其中遗传图距最长和标记密度最高的是2号染色体,其遗传长度和标记间平均距离分别为128.45和0.38 cM;图距最短的是5号染色体(61.48 cM)和10号染色体(61.94 cM);密度最低的是7号染色体,其标记间平均距离为0.72 cM。
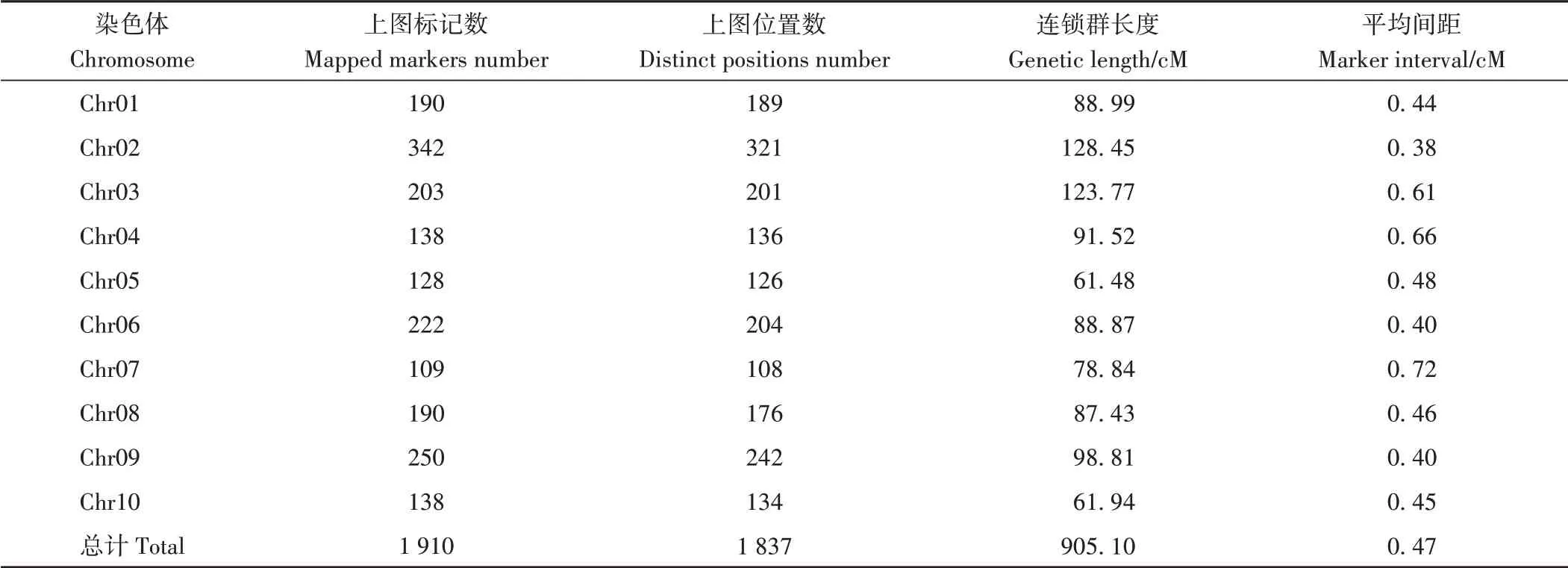
表3 RIL群体中10条染色体遗传图距长度及SNP标记密度Table 3 Genetics distance length and SNP marker density of 10 chromosomes in the RIL population
2.3 籽粒酿造相关性状QTL定位
利用ICIM方法,在4个环境下共定位到35个QTL与籽粒的5个酿造性状相关,分别位于1~9号染色体上(表4、图1)。与总淀粉含量、支链淀粉含量、单宁含量、籽粒硬度和籽粒颜色相关的QTL分别有9、7、11、5和3个,一共涉及到28个染色体区段。
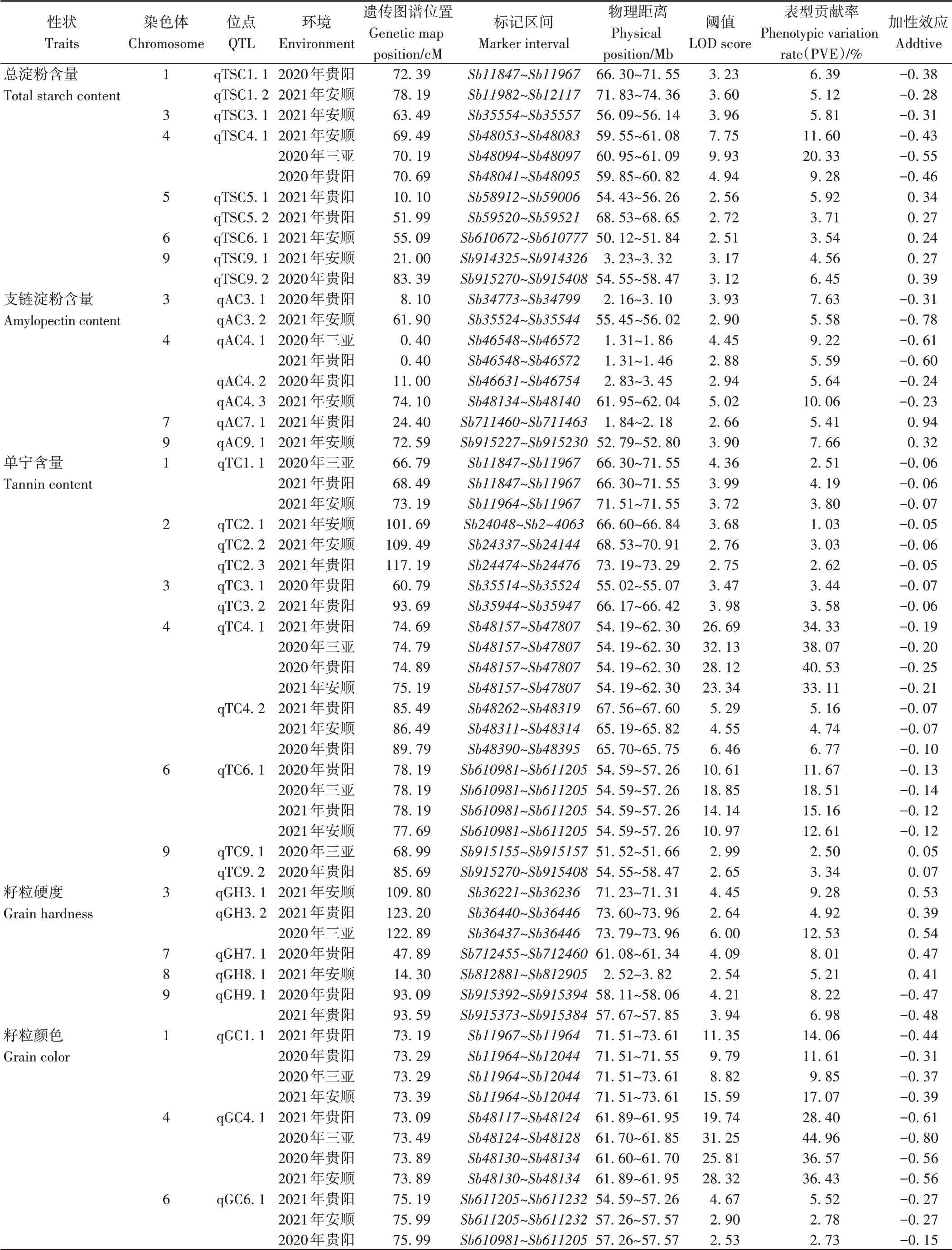
表4 4个环境下高粱籽粒酿造相关性状的QTL定位Table 4 QTL mapping of sorghum grain brewing related traits in in four environments
影响总淀粉含量的9个QTL分别位于1号(2个)、3号(1个)、4号(1个)、5号(2个)、6号(1个)和9号(2个)染色体上。在3个环境下均能检测到位于4号染色体的qTSC4.1,其最大LOD值和表型贡献率分别为9.93和20.33%。在2020年贵阳环境下检测到2个QTL(qTSC1.1和qTSC9.2),LOD值变化范围为3.12~3.23,可解释的表型变异率为6.39%~6.45%。除qTSC1.1、qTSC1.2、qTSC3.1和qTSC4.1外,其他5个QTL的增效等位基因均来源于亲本BTx623。
影响支链淀粉含量的7个QTL分别位于3(2个)、4(3个)、7(1个)和9(1个)号染色体上。在2个环境下都定位到1个位于4号染色体上的重要QTL(qAC4.1),其最大LOD值和表型贡献率分别为4.45和9.22%。5个QTL(qAC3.1、qAC3.2、qAC4.1、qAC4.2、qAC4.3)的增效等位基因来源于亲本红缨子,其他2个QTL(qAC7.1和qAC9.1)的增效等位基因来源于亲本BTx623。
与单宁含量相关的11个QTL分别位于1号(1个)、2号(3个)、3号(2个)、4号(2个)、6号(1个)和9号(2个)染色体上。2个重要的QTL在4个环境中均能够被检测到,位于4号的qTC4.1的平均LOD值为27.57,变化范围为23.34~32.13,可解释平均表型贡献率为36.51%,变化范围为33.11%~40.53%。位于6号染色体的qTC6.1的平均LOD值为13.62,变化范围为10.61~18.85,可解释平均表型贡献率为14.49%,变化范围为11.67%~18.51%。在3个环境下检测到2个QTL(qTC1.1和qTC4.2),最大LOD值分别为4.36和6.46,表型贡献率分别为4.19%和6.77%。除qTC9.1和TC9.2以外,其他9个QTL增效的等位基因均来源于亲本红缨子。
影响籽粒硬度的5个QTL分别位于3号(2个)、7号(1个)、8号(1个)和9号(1个)染色体上。其中,位于3号和9号染色体上的2个重要QTL(qGH3.2和qGH9.1)能够在2个环境下被检测到,最大的LOD值分别为6.00和4.21,表型贡献率分别为12.53%和8.22%。在5个QTL中,除了qGH9.1以外,其他4个QTL的增效等位基因均来源于亲本BTx623。
与籽粒颜色相关的3个QTL分别定位于1号(1个)、4号(1个)和6号(1个)染色体上。在4个环境下均检测到的2个重要QTL(qGC1.1和qGC4.1)定位于1号和4号染色体上,LOD值分别为8.82~15.59和19.74~31.25,表型贡献率范围为9.85%~17.07%和28.40%~44.96%。在3个环境下检测到的qTC6.1位于6号染色体,其LOD值和表型贡献率分别为2.53~4.67和2.73%~5.52%。上述3个QTL的增效等位基因均来源于亲本红缨子。
综合比较,与5个性状相关的QTL涉及到28个遗传区段,其中3个重要区段在多个性状中被同时定位(图1)。与总淀粉含量、单宁含量和籽粒颜色相关的QTL在1号染色体的66.30~71.55 Mb区段上重叠,与总淀粉含量、支链淀粉含量、单宁含量和籽粒颜色相关的QTL在4号染色体的54.00~62.3 Mb区段上重叠,与单宁含量和籽粒颜色相关的QTL在6号染色体的54.59~57.57 Mb区段上重叠。
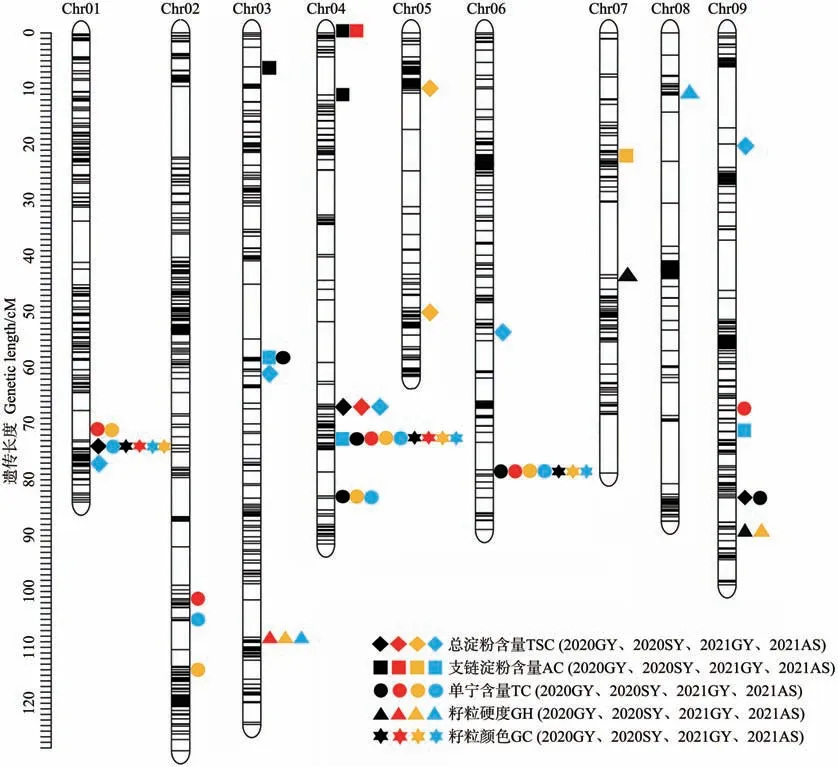
图1 高粱籽粒酿造相关性状QTL在遗传图谱上的分布Fig.1 Distribution of QTLs for sorghum grain brewing related traits on genetic map
2.4 候选基因预测
根据上述QTL定位结果,对1号染色体(66.30~71.55 Mb)、4号染色体(54.00~62.3 Mb)以及6号染色体(54.59~57.57Mb)的三个重要区段进行了候选基因分析,利用植物基因组数据库Phytozome(https://phytozome-next.jgi.doe.gov/),识别QTL区间内高粱相关基因ID、蛋白质注释信息及相应的其他物种(水稻和拟南芥)的同源基因,结合前人研究文献[11,13,26,34-35],确定了8个候选基因(表5)。除已经被克隆的Y基因和Tan1基因以外,本研究还注释到了6个新的候选基因,包括与类黄酮合成途径相关的bHLH转录因子Sobic.006G175700以及编码黄烷酮3-羟化酶的基因Sobic.006G253900。其他4个基因则与植物淀粉和蔗糖代谢途径相关,包括编码β-淀粉酶的基因Sobic.001G372100,是催化植物淀粉转化为麦芽糖的重要基因,以及编码淀粉合酶Ⅱ的Sobic.004G238600基因,是淀粉合成途径的4种关键基因之一。
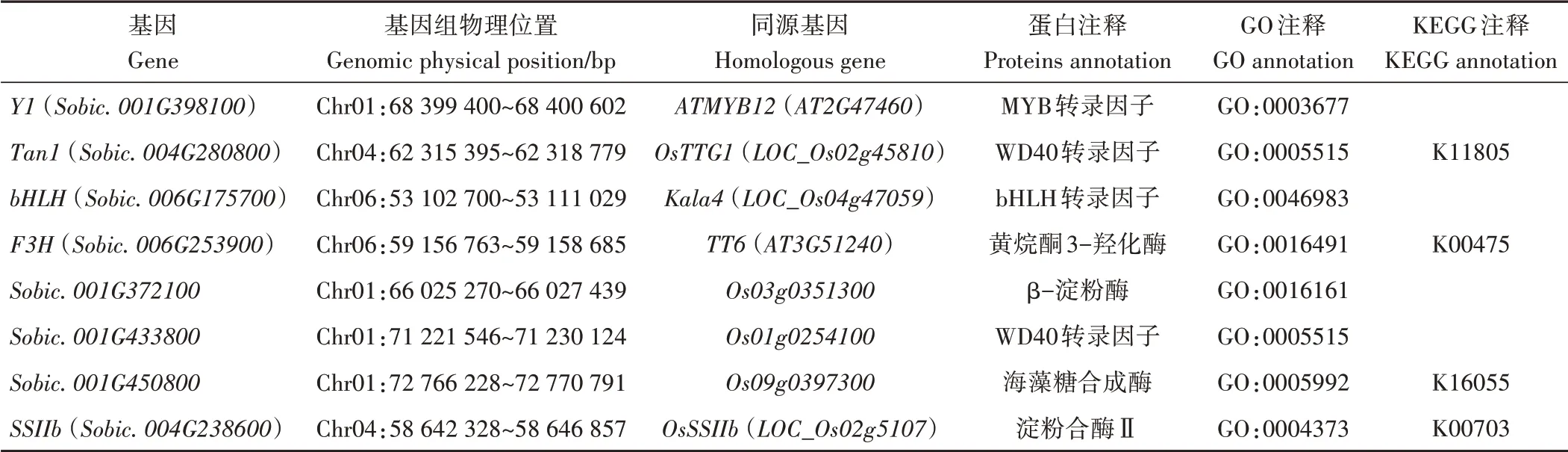
表5 QTL区间候选基因功能注释Table 5 Functional annotation of candidate genes in QTL mapping
3 讨论
自元朝(公元前749-652年)以来,我国高粱一直就是酿造著名白酒的重要原料[25],如贵州茅台、泸州老窖、五粮液、汾酒等都是利用高粱作为主要的酿酒原料。因此,开展酿造相关性状的遗传学研究,发展紧密连锁的分子标记,利用标记辅助育种技术加快酒用高粱新品种选育具有重要意义。
籽粒颜色是选育高粱单宁含量的指标,即随着籽粒颜色的加深,单宁含量增加[14]。但是本研究的相关性分析结果显示,单宁含量和籽粒颜色之间呈正相关关系,Pearson系数约等于0.5。这说明籽粒颜色并不是高粱单宁含量的可靠指标,在酒用高粱新品种选育中,仅根据籽粒颜色的深浅来判断单宁含量的可能会导致较大的偏差。
目前,在水稻和玉米中涉及淀粉合成途径的相关基因研究较为透彻[26],而在高粱中,虽然对籽粒总淀粉含量、直(支)链淀粉含量以及胚乳蜡质/角质性状进行了一些QTL定位[6],但是目前仅有Wax基因被克隆[7]。本研究中,影响支链淀粉含量的2个QTL(qAC3.2和qAC9.1)与前人的研究结果接近[27],而总淀粉含量的4个QTL(qTSC1.2、qTSC3.1、qTSC4.1和qTSC9.1)则与已报道的QTL区间重叠[28]。通过同源基因比对,在qTSC1.2和qTSC4.1的区间上发现了4个候选基因,与其他作物中已报道的淀粉和蔗糖代谢相关基因同源[26],推测是参与高粱籽粒淀粉合成的候选基因。然而,由于本试验所构建的遗传图谱在10号染色体顶端存在一个缺口(Gap),导致Wax基因所在染色体区域缺少SNP标记,因此,在本研究中Wax基因未能被检测到。
在粮食作物籽粒硬度的遗传基础研究方面,控制小麦籽粒硬度的基因Pina和Pinb已经被克隆[29]。但关于高粱籽粒硬度的QTL定位报道仍较少[9-10,30],目前已定位的8个QTL位于2号、3号、4号、7号和10号染色体上,其中位于3号和7号染色体的QTL与本研究发现的qGH3.2和qGH7.1定位区间一致。推测本研究中发现的其他3个遗传位点可能是新的QTL,但尚未找到与小麦等其他作物籽粒硬度相关的同源基因,需要进一步验证。
单宁和花青素由类黄酮途径的一个特定分支产生,涉及数十个结构基因和多个调控基因,这些基因已在水稻[31]和玉米[32]和拟南芥[33]中得到较为全面的研究。迄今为止,在高粱中只克隆了2个调控单宁合成的基因,分别是编码WD40蛋白的Tan1(Sobic.004G280800)基因[13]和编码bHLH结构域蛋白的Tan2(Sobic.002G0 76600)基因[12]。在本研究中,位于4号染色体的qTC4.1和qGC4.1与Tan1基因位点重叠。而位于2号染色体的Tan2基因则没有被检测到,其原因是2个亲本BTx623和红缨子在这个基因上没有多态性,而基因测序结果也证实这两个亲本的Tan2基因序列完全相同(结果未报道)。而与单宁含量相关的另外3个QTL(qTC1.1、qTC3.2和qTC9.1)也与前人定位结果重叠[25]。本研究确定的控制籽粒颜色的qGC1.1位于1号染色体66.30~71.55 Mb区间,与控制高粱籽粒颜色的Y1基因位点一致[11]。同时,在4个环境中还检测发现,与单宁含量和籽粒颜色相关的8个QTL均位于6号染色体上的同一区段(54.59~57.26 Mb),目前在高粱研究中还未见报道。通过同源基因比对,在其附近确定了2个重要的候选基因:一个候选基因为Sobic.006G175700,与水稻Kala4基因同源,属于bHLH转录因子,通过激活类黄酮合成相关基因产生特异色素,导致水稻籽粒变黑[34];另一个候选基因为Sobic.006G253900,是拟南芥中黄烷酮3-羟化酶(F3H)同源物[35],属于类黄酮合成途径中的关键酶,转基因试验也证实了高粱品种Tx430中转入F3H基因后植株中类黄酮化合物含量较野生型显著上升[36]。
此外,本研究还确定了几个潜在的控制高粱单宁和淀粉生物合成的候选基因。为了进一步精确鉴定等位变异位点,后续将利用基于转录组测序的集群分离法(bulked segregation analysis,BSA),加密作图群体的目标区段;随后,将对候选基因进行序列分析和表达分析,以确定基因多态性,为单宁和淀粉遗传变异在作物改良中的应用奠定基础。
4 结论
本研究利用Super-GBS技术,构建了包含205个RILs(Bx623×红缨子)的SNP高密度遗传连锁图谱。在四个环境下共定位到9个总淀粉含量、7个支链淀粉含量、11个单宁含量、5个籽粒硬度和3个籽粒颜色相关的QTLs。在多个环境和性状中确定了3个重要遗传区段,分别位于1号、4号和6号染色体,包含了已经克隆的Y1和Tan1基因位点,同时筛选出6个与籽粒淀粉含量、支链淀粉含量、单宁含量以及颜色相关的候选基因。
