宏基因组测序技术在入海排污口污水监测中的研究进展
2023-01-14傅松哲
傅松哲
(1.设施渔业教育部重点实验室(大连海洋大学),辽宁 大连 116023;2.大连海洋大学 海洋科技与环境学院,辽宁 大连 116023)
宏基因组学(Metagenome)是由美国Wisconsin大学的Handelsman等于1998年提出的新概念[5]。目前,该方法已经广泛应用于描述和研究环境中的微生物群落,以及它们与环境中其他生物、非生物之间的相互作用。宏基因组学研究的是特定环境下所有生物遗传物质的总和,包括传统方法不能培养的微生物的遗传信息。得益于宏基因组测序技术的飞速发展,研究者已经有能力深入研究之前不可培养微生物的特性,从而全面地刻画不同微生物群落对于整个生态系统的重要意义[6]。
与传统方法相比,宏基因组测序技术可以检测特定环境下所有生物的遗传物质,是污水生物污染物监测较为理想的解决方案。近年来,宏基因组测序技术在未知病原的临床诊断[7]、环境保护与污染修复[8]及污水监测[9]中得到了广泛应用。本文从污水中致病性细菌、病毒、有毒藻类及ARGs的丰度与动态变化的角度,综述了宏基因组测序技术在入海排污口污水生物污染物监测中的应用进展,并结合团队前期研究和工作实践,提出了污水监测预警模型的构建思路,以期为保障中国海洋生态环境安全提供理论参考和技术支撑。
1 宏基因组测序技术的发展历程
1.1 第一代宏基因组测序技术
1996年,毛细管电泳技术的发明,第一次使宏基因组测序技术得以实现。2001年,Cai等在提取环境中的总DNA后,将纯化后的DNA进行16S rRNA基因扩增,再连接到适当的质粒载体上,并通过转化技术将质粒转入宿主菌,从而形成重组DNA文库,进一步对获得的克隆子进行筛选,并用毛细管测序仪测序,得到了宏基因组产物片段[10]。这套方法也称为鸟枪法(Short-gun method)(图1)。由于整个试验过程涉及从环境样品提取总DNA及其纯化、宿主菌的转化和从文库中筛选克隆子等多个步骤,每一步操作都可能会对分析结果产生不同程度的影响,近年来已不再使用。
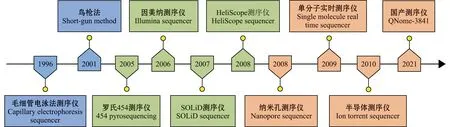
图1 宏基因组测序技术的发展历程Fig.1 Development roadmap of metagenomic sequencing
1.2 第二代宏基因组测序技术
高通量测序技术(high-throughput sequencing)也称为第二代宏基因组测序技术(metagenomic next-generation sequencing)。利用这一新方法,在成功制备基因组文库样品后,可以直接进行测序,样品制备的操作时间也极大地缩短。同时,因为采用了大规模平行测序的方式,测序的通量范围也得以呈指数级扩大。目前,主要有3种测序平台应用于宏基因组测序:罗氏公司的454焦磷酸测序仪、Illumina测序仪和SOLiD测序仪[11]。
1)罗氏454焦磷酸测序仪。2005年,454 Life Science公司首先建立了第二代高通量测序技术,并在随后推出了454 FLX Pyrosequencer测序仪(罗氏454焦磷酸测序仪)。其测序过程:首先,在具有超过200万个寡核苷酸的玻璃板上进行大规模的并行焦磷酸测序[12];随后,DNA在荧光素酶催化下发出荧光,被高分辨率电荷耦合器件(CCD)相机捕获,从而测定扩增的DNA序列。罗氏454测序仪的局限性在于其高错误率。与第一代测序序列的错误率(10-4~10-5)相比,罗氏454焦磷酸测序仪平均错误率为10-3~10-4[13]。由于聚合酶和荧光素酶效率的限制,罗氏454焦磷酸测序仪的平均读取长度约为400 bp。
刺绣最需要的就是耐心,金际珍经常在阳台边的“专座”上一坐就是一下午。小狮子、柳树叶、长命锁……黑色的线,白色的布,在老人的针下,变得异常精美。“走针时,三针一挑,要数线,只要有耐心,人人都能绣好。”她说。
2)因美纳Illumina测序仪。2006年,因美纳公司推出了基于单分子簇的Illumina测序仪。与Sanger测序中使用的合成测序法类似,Illumina测序仪也是基于可逆终止的化学反应[14],但具有边合成边测序(sequencing by synthesis)的特点。 其测序过程:首先,制备基因组文库,类似于罗氏454测序仪,文库的制备涉及将基因组DNA片段化成特定长度,进行末端修饰,并添加接头(adapter);随后,DNA随机附着到光学透明的玻璃表面(flow cell),通过聚合酶掺入荧光标记并实现DNA扩增,这些DNA片段经过扩增后,在flow cell上形成数以百万计的序列簇(cluster);最后,通过相机读取每个核苷酸扩增时产生的荧光标记,实现序列测定。Illumina 基因组分析仪的局限性在于其体外扩增步骤容易导致高背景错误率(错误率为10-2~10-3)[15]。此外,Illumina图像分析时可能将化学晶体、灰尘和棉绒颗粒识别为簇,从而误读为DNA序列。尽管Illumina测序的错误率稍高于罗氏454焦磷酸测序仪,但由于其测序成本较低且测序通量较高,故Illumina测序平台已成为最受欢迎的测序选择之一。
3)SOLiD测序仪。2007年,美国ABI公司推出了一款名为SOLiD系统的基因测序仪。该测序仪由于测序读长较短、运行较慢,随后退出了市场。2010年,ABI公司推出的Ion torrent测序仪采用的是半导体芯片测序原理[16],将高密度半导体小孔芯片置于测序仪中的离子感受器之上。其测序过程:首先,制备DNA文库,其过程与Illumina建库相似,但其DNA的接头不带有突出的T碱基黏性末端,而是平头;随后,进行乳液PCR,其成分包括水相和油相,其中水相是核心,DNA文库的PCR扩增在水相中完成,而油相起到分隔作用;最后,由半导体芯片捕捉测序信号,每当扩增出来的DNA链掺入芯片时就会释放出质子,离子感受器随即感受到这种信号,半导体芯片将化学信号直接转换为数字信号,从而读出相应的核苷酸序列。Ion torrent测序仪由于采用的是半导体芯片测序,不需要昂贵的物理成像和光学检测设备,故测序成本相对较低。此外,由于其测序操作简单,无修饰核苷酸的添加,可以在2 h内完成测序工作,因而近年来得到了迅速普及。Ion torrent测序仪的缺点在于其测序读长仍然较短(100~200 bp),对于重复序列和移动遗传元件的测序效果依然不佳[17]。
1.3 第三代宏基因组测序技术
第二代宏基因组测序技术普遍采取PCR扩增技术对初始DNA样品进行扩增,这会引入一定的测序偏倚。为解决这一问题,不依赖PCR扩增的第三代宏基因组测序技术应运而生。目前,Helicos基因分析系统(Helicos’ genetic analysis system)、牛津大学的纳米孔测序技术(Oxford nanopore sequencing)和Pacific Bioscience公司开发的单分子实时测序技术(single molecule real time, SMRT)经过不断地发展完善,已成为主流的第三代宏基因组测序平台。
1)HeliScope测序仪。2008年4月,Helico BioScience公司利用单分子测序技术对M13病毒的基因组进行了重测序[18],开启了第三代测序技术的研发浪潮。一方面,由于其制备过程中不需要PCR步骤,大大减少了由PCR引起的偏倚和误差;同时,由于其读长可达1 300 bp,比任何第二代测序技术都要长,这使得测序的质量又一次得到大幅度地提高。HeliScope测序仪是第一个能够对单个分子进行测序的测序平台,与二代测序中的Illumina测序仪类似,也采用边合成边测序的技术。但该技术无需对样本进行PCR扩增,简化了测序文库的构建过程,也避免了DNA扩增中出现的错误。HeliScope的测序过程相对简单:首先,将待测DNA随机打断成小片段,然后在3′末端加上50 bp带有荧光标记的poly(A) 尾,并与芯片上固定的Oligo dT探针结合,利用poly(A)上的荧光标记进行精确定位;随后,依次加入4种Cy5荧光染料标记的单核苷酸,在DNA聚合酶的作用下,与模板互补配对并延伸一个碱基;最后,用CCD相机采集荧光信号,获得测序序列。
2)单分子实时测序仪(SMRT)。2009年,Pacific Bioscience公司在HeliScope测序仪的基础上做了进一步改进,发展出单分子实时测序仪,其测序过程与HeliScope类似。SMRT测序过程:首先,DNA聚合酶和模板结合,分别将四色荧光基团标记在4种脱氧核苷酸的磷酸基团的末端;随后,随着不同碱基的加入,激发波会激发基团发出不同荧光;最后,根据光的波长与峰值判断进入的碱基类型。由于使用了活性持久且高保真的DNA聚合酶,与HeliScope测序仪20~50 bp的读长相比,SMRT测序技术平均测序读长可达3 000~5 000 bp,最长读长能达到20 000 bp,实现了超长读长测序[19]。
3)纳米孔测序仪。2008年,牛津纳米孔公司开发了依靠纳米孔捕获单分子实时电信号的纳米孔测序仪。其测序过程:首先,通过纳米孔通道蛋白结合并解螺旋DNA/RNA链,在电压差的作用下,DNA/RNA链以稳定的速率通过纳米孔通道蛋白,由于DNA/RNA链上的不同碱基将引起不同电学信号的变化,通过对这些信号进行捕获,即可得知相应碱基的类型,并完成序列的实时测定[20]。纳米孔测序技术有3个突出的优点:(1)测序读长较长,为10 kbp~100 kbp,对于复杂的基因组结构具有较好的效果;(2)数据可实时读取,这使得纳米孔测序可以边测序边检测,便于实现现场检测;(3)由于核酸不会在测序过程中被破坏,故可以直接对RNA进行测序。这些特点使其成功应用于新型冠状病毒(SARS-CoV-2)的实时在线监测中,并显示出显著高于荧光定量PCR检出限的检测效果[21]。2021年,中国成都奇碳科技公司推出了第一台基于纳米孔测序技术的国产基因测序仪 QNome-3841,相对于纳米孔测序仪,其读长更长,而测序成本更低。
综上,3种三代测序技术各有千秋。HeliScope测序仪测序成本最低,但由于其读长过短,因此,难以对含有复杂基因结构的序列进行测序。SMRT测序技术准确率和敏感性最高,准确率最高可达99.999%,并能检测到甲基化等碱基修饰,但其测序成本过高,不适用于大规模样本测序。纳米孔测序技术的优点在于其读长较长,最高可达100 kbp,但其错误率目前仍然较高,且错误位置较为随机[22]。随着测序技术的进一步发展,可以预见未来宏基因组测序技术将会朝着更高通量、更准确和更低成本的方向发展。一些当前只能依靠传统手段监测的病原生物,包括以血吸虫为代表的寄生虫和以贾第鞭毛虫为代表的原生动物也将有可能通过宏基因组测序手段进行监测。
2 宏基因组测序技术在入海排污口微生物监测中的应用
2.1 宏基因组测序技术在致病性细菌监测中的应用
致病性细菌是能在人体内寄生、增殖并引起疾病的一类细菌的统称。在中国沿海地区,每天有成千上万吨的养殖废水、生活污水、工业废水和医疗废水经入海排污口排入海洋,排污口排放的污水极易在沿海人口稠密地区造成如霍乱等致病性细菌病大规模流行[23]。因此,加强入海排污口污水中致病性细菌的监测至关重要。
1) 养殖污水中致病性细菌的监测。近几年来的宏基因组测序结果表明,排污口污水的溶解氧、盐度和温度的季节性变化,潮汐及排污口类型、地理位置上的空间分布差异等,均会对入海排污口污水中细菌群落结构、时空分布和致病性细菌的丰度产生不同程度的影响[23-25](表1)。如Suttner等[26]采用Illumina测序技术对美国加利福尼亚州沿岸的农业养殖污水进行了9个月的监测,结果表明,农业养殖污水主要致病性细菌为产志贺毒素的大肠埃希氏菌Escherichiacoli,并指出用宏基因组测序技术对农业养殖污水进行微生物风险评估具有可行性。Moore等[27]采用454焦磷酸测序技术对美国新罕布什尔州大湾区河口细菌群落进行了高通量测序,发现了多种贝类病原。
2)城市污水中致病性细菌的监测。近年来,中国也在各个沿海省份通过宏基因组测序开展了入海排污口的污水监测工作,发现排污口类型和排污季节是细菌群落变化的两个关键因素。刘霜等[28]采用454焦磷酸测序仪对青岛海域近海港口区、水产养殖区、工业区和休闲度假区4种类型排污口邻近海域致病性细菌的组成与分布进行了分析,结果显示,青岛排污口邻近海域致病性细菌类群多样性非常高,且不同类型排污口细菌种类存在较大差异,在工业区和养殖区均检测到溶藻弧菌Vibrioalginolyticus和副溶血弧菌V.parahaemolyticus等致病菌。郭明月[29]采用Illumina测序技术对青岛市麦岛海域和海泊河的污水监测发现,工业污水中细菌的群落结构比起生活污水更易受到季节演替的影响,其夏季致病菌的种类远远高于冬季,且夏季致病菌主要是与肺炎相关的军团菌Legionella及与腹泻相关的弧菌属Vibrio、弓形杆菌属Arcobacter等。姜南等[30]采用罗氏454焦磷酸测序技术,对宁波市沿海的5个陆源入海排污口在3—10月进行了采样和宏基因组测序,并对不同排污口细菌群落的时空变化进行了分析,结果显示,宁波市沿海5个入海排污口均含有大量的致病细菌,主要病原菌包括肠杆菌属Enterobacter、弧菌属和假单胞菌属Pseudomonas等,不同排污口在不同季节的细菌群落结构差异较大。
3)污水致病性细菌的风险评估。通过宏基因组测序也可对污水致病性细菌的风险进行评估。杨文新等[31]采用Illumina测序技术对大连市入海排污口及邻近海域的微生物种群演替进行了动态分析,结果显示,市政排污口排放的污水中致病性细菌非常丰富,包括希瓦氏菌属Shewanella、弧菌属、肠杆菌属和假单胞菌属等,其中,肠杆菌科为主要优势类群, 在各排污口均有检出,提示该菌风险较大。郎秀璐等[32]用宏基因组测序表明,相对于排入河流后进行生态处理的方式,直接排海的排放方式使得致病性细菌更易持久性存在于近岸海域,并使某些致病性细菌如假单胞菌属和弓形杆菌属,在扩散后仍然保持较高的相对丰度,因此,应对这几种致病性细菌进行重点监测。2017—2020年,本研究团队采用Illumina测序技术在辽宁省东港市的2个入海排污口进行污水监测,在东港市宏基因组数据中共检测到76种致病性细菌,其中,夏季污水中气单胞菌属Aeromonas、不动杆菌属Acinetobacter、霍乱弧菌Vibriocholerae和副溶血性弧菌最为丰富,而弓形杆菌属则在冬季达到峰值[33]。此外,本研究团队进一步分析了宏基因组数据中的毒力基因,包括不动杆菌、铜绿假单胞菌等机会性致病菌的毒力,从而更为准确地对微生物风险进行了评估[33]。
2.2 宏基因组测序技术在病毒监测中的应用
病毒是污水中的另一大类生物污染物。最早应用宏基因组学技术分析病毒群落的科学家是Breitbart Mya教授,他于2002年首次应用宏基因组监测技术对美国加州两处海岸中的病毒进行研究,分别发现了374、7 114个潜在的新病毒[34]。随后,包括诺如病毒和冠状病毒等在内的许多人类相关病毒也通过宏基因组测序技术在水环境中被检测到[35-36]。
1)提高病毒鉴定的分辨率。传统细胞培养法只能在科水平上鉴定病毒,而宏基因组工具提高了许多与人类相关病毒多样性的分辨率,为改进水质和更科学地防治病毒侵染提供了重要的管理工具。Adriaenssens等[37]通过RNA和DNA病毒学分析,对污水处理厂及下游河流、河口的病毒群落进行研究,在种水平上发现了8 400余种不同的病毒群落,表明来自污水的病毒群落也会沉积在河口水体中,同时这种现象也会受当地地理、天气和潮汐的影响。Xu等[38]通过对珠江口表层水的病毒群落深度进行宏基因组测序,发现其病毒群落主要由感染细菌、无脊椎动物和人类的病毒组成。
2)实现新冠疫情的早期预警。新冠疫情暴发以来,污水病毒宏基因组测序也在新冠疫情早期预警方面发挥着关键作用。自2020年6月首篇用污水监测新冠病毒SARS-CoV-2的研究报告发表以来[39],已有大量研究采用病毒宏基因组测序技术,在污水中检测到了该病毒的不同变异株[40-41]。最近研究发现,对污水的病毒宏基因组测序不仅有助于跟踪新冠病毒不同变异株的进化趋势,还能为病毒传播提供早期预警信号[42]。此外,采用病毒宏基因组学方法还可以通过测定污水中SARS-CoV-2的浓度,依据相关模型预测社区感染人数[42]。如本研究团队通过测定污水中SARS-CoV-2的浓度,采用蒙特卡洛模拟法构建了定量污水预测模型[43],并实时预测了陕西省西安市和江西省南昌市等地新冠疫情规模和感染人数,为相关部门决策提供了科学依据。
3)发现未知病毒。通过对污水样本进行宏基因组测序,还可以直接获得环境中不可培养的病毒信息,并阐明某些中间宿主的病毒携带情况[44]。这不仅增加了对环境病毒遗传多样性的认识,而且实时监测到一些潜在致病性病毒的进化情况,有助于对一些新发病原体进行早期预警。中国采用病毒宏基因组测序技术进行污水监测起步较晚,近3年来,应用逐步增多。Wang等[45]对中国香港地区2家污水处理厂排污口污水进行了跨度为9年的采样,用宏基因组测序技术共获得8 478 种病毒片段,并发现了一些之前未有的新病毒。
综上,有相当多的病毒类群稳定出现在污水中,这为揭示病毒种群的时空演替规律奠定了基础。这些工作显示了病毒宏基因组测序技术在污水病毒监测中的应用潜力。
2.3 宏基因组测序技术在有害藻华监测中的应用
有害藻华(harmful algal blooms,HAB)是指伴随着浮游藻类的骤然大量增殖而直接或间接导致水面发生变色的现象,是威胁、危害海洋生态环境和人类健康的另一种海洋灾害。宏基因组测序技术在对有害藻华的监测中发挥着日益重要的作用。2016年7月,美国佛罗里达州奥基乔比湖及整个圣露西河口发生了一次大型有毒蓝藻引起的水华,导致佛罗里达州宣布进入紧急状态。随后Kramer等[47]通过宏基因组测序对这一河口地区进行了污水监测,宏基因组测序分析表明,与产生微囊藻毒素和藻类神经毒素相关的基因来自微囊藻属,在所有调查位点中,总氮、微囊藻毒素与微囊藻毒素合成基因丰度之间存在显著相关性,这表明含高浓度氮的污水排放导致了水华期间微囊藻毒素的产生。这些结果表明,奥基乔比湖、圣露西河及河口的监测与监管策略应考虑管理氮负荷,以控制未来藻类和微囊藻毒素产生的蓝藻大量繁殖。有害藻类也可能通过河口污水排放迅速传播并造成大规模水华。
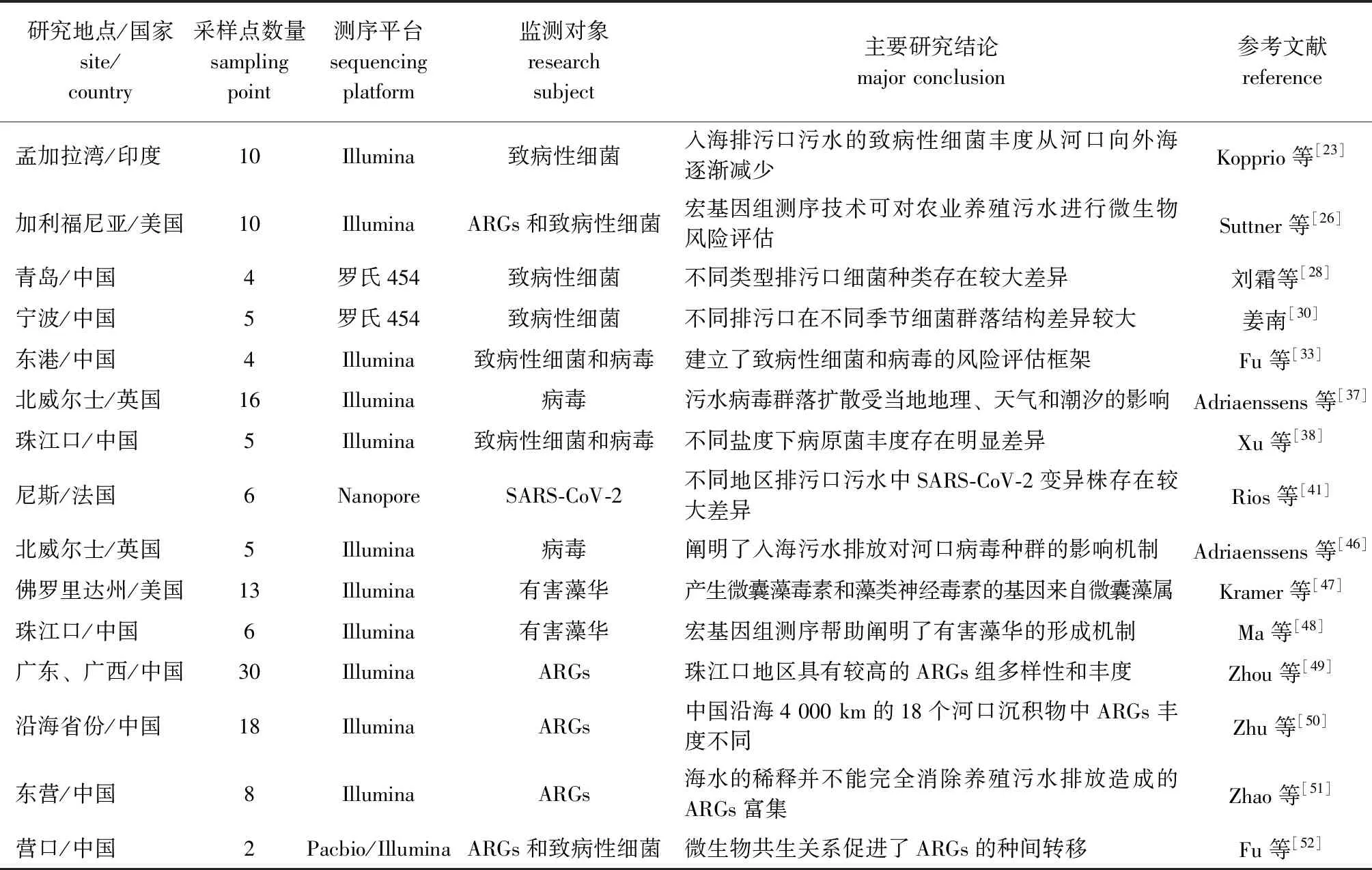
表1 宏基因组测序技术在入海排污口污水监测中的典型应用案例Tab.1 Application of metagenomic sequencing technology in the monitoring of marine sewage outfalls
宏基因组测序也帮助阐明了有害藻华的形成机制。Ma等[48]通过宏基因组测序,阐明了原核生物群落在有害藻华的建立和生长中的作用,宏基因组测序表明,硅藻产生的不稳定碳水化合物和有机氮促成了某些原核生物的激增。值得注意的是,Planctomycetaceae科细菌和硅藻可能形成了共生关系,硅藻为Planctomycetaceae提供了有机物质,而细菌则向硅藻提供维生素和过氧化氢作为交换,并最终促进了硅藻水华的形成。此外,微囊藻还可以与固氮微生物形成共生关系,依靠固氮微生物提供的氮源,微囊藻在氮缺乏状态下也能形成水华。
宏基因组测序也进一步揭示了促进有害藻华发生的环境因素。王靖淇等[53]对盘锦辽河口水体进行宏基因组测序发现,硝态氮、氨氮和磷酸盐等营养盐是影响真核浮游藻类种群结构特征最重要的环境因子。综上,宏基因组测序为认识和监测污水中有害浮游藻类的演替规律提供了借鉴。
3 宏基因组监测技术在入海排污口ARGs监测中的应用
抗生素自发现以来为人类治疗细菌性感染做出了重大贡献,然而,随着抗生素的长期滥用,ARGs不断出现,已对细菌性感染的治疗产生了严重影响。入海排污口是抗生素、耐药性细菌及ARGs的重要储存库,又是耐药性细菌驯化繁殖的“温床”,同时它还是向环境中传播扩散抗生素、耐药性细菌和ARGs的重要源头[54]。生活污水、制药废水及医院污水中均含有大量的ARGs,这些污水经污水处理系统处理后,仍含有相当数量的抗生素、耐药性细菌和ARGs,其对人类健康及生态环境构成了潜在威胁。一项全球入海排污口ARGs的监测表明,根据样本采样位置的人口密度,所有研究样本的ARGs丰度差异明显,高人口密度环境具有更高的ARGs丰度,其中23.78%的ARGs对人类健康构成严重威胁[54]。Suttner等[26]对加利福尼亚州河口水体、沉积物进行9个月的样品采集,通过宏基因组测序分析发现,所有样本中携带ARGs的宏基因组读数明显高于其他淡水和土壤宏基因组中的ARGs读数,这表明农业养殖污水可能是ARGs的天然储存库。
中国学者很早就对入海排污口的ARGs问题给予了高度关注。Zhou等[49]采用宏基因组测序技术测定了广东、广西地区30个主要河口的ARGs污染水平,结果发现,珠江口地区具有较高的抗性组多样性和丰度,特别是在珠江的主要河口,水体pH是塑造成广东、广西地区河口ARGs时空演变的重要因素。Zhu等[50]从中国东部海岸18个河口排污口沉积物样本中监测到近200个不同类型的ARGs,其中,四环素胺类基因及其他磺胺类药物ARGs检出率相对较高。Zhao等[51]从山东省东营市黄河口保护区和海水养殖区排污口分别采集水样,采用高通量测序技术和实时定量PCR芯片技术,测定了87种ARGs在沉积物中的组成和丰度,结果表明,养殖区排污口样品中ARGs的检出数目(7~15种)显著高于保护区ARGs的检出数目(5种),海水养殖污水样品中,ARGs的相对丰度也比黄河口保护区提高了10倍左右。此外,尽管大部分ARGs的丰度在入海后有所下降,但是个别ARGs的丰度依然较高,这说明海水的稀释并不能完全消除养殖污水排放所造成的ARGs富集。
致病性细菌间的共生关系是ARGs转移的另一个关键驱动因素。本研究团队通过解析2018—2020年辽河口养殖区域水体样品中微生物的种群演替,发现了与致病性弧菌存在共生关系的物种类型,随后有针对性地分析了这些物种的移动遗传元件,发现致病性弧菌与其存在共生关系的物种间存在广泛的ARGs水平转移[52]。该研究对于探究ARGs的传播途径及阐明抗生素耐药机制具有重要意义。
4 存在问题及展望
4.1 宏基因组测序面临的主要技术问题
目前,宏基因组测序技术已经在污水监测领域得到了广泛应用,然而,未来研究还需要攻克以下几个技术难题,以进一步拓宽其应用范围。
1)当前宏基因组测序技术只能表征微生物种群的相对丰度。由于细菌总数的季节性变化,通过相对丰度的变化来评估污水病原和ARGs的健康风险可能会产生偏差,这些潜在的偏差可能高估或低估微生物和ARGs风险。尽管定量聚合酶链式反应(qPCR)可以定量测定某个致病性细菌或病毒的绝对丰度,但为每一种微生物开发一套qPCR检测方案是不现实的。可能的解决方案是开发基于绝对定量的宏基因组测序技术,即通过细菌总量乘以每一种细菌的相对丰度来推断每一种细菌的绝对丰度。目前,测定污水样品细菌总量的方法主要包括流式细胞术、16S qPCR和Spike-in内参序列法等[55]。对于病毒宏基因组测序,未来也可通过荧光显微计数法测定样品中病毒的绝对丰度,从而推算出每种病毒的数量。
2)对于宏基因组测序得到的海量数据,如何构建更加快速的分析流程,并自动生成更加清晰易懂的分析报告,仍然是需要克服的技术难题。
3)如何通过宏基因组测序技术构建污水监测的健康风险评估模型,也是未来研究的重点。本研究团队提出了一个由病原菌筛选、病原菌基因分型和风险评估3个步骤组成的微生物风险评估流程,利用这一流程,笔者初步评估了辽宁省东港市入海排污口致病性细菌和病毒的健康风险。目前,已有初步研究可通过测定污水中新冠病毒的浓度,从而推测出区域内的感染人数。未来如何将这一方法拓展到对SARS-CoV-2及其他病原的健康风险评估中值得深入探索。这些技术的应用将极大地提高宏基因组测序技术对城市和养殖污水进行微生物风险评估的可行性(图2)。
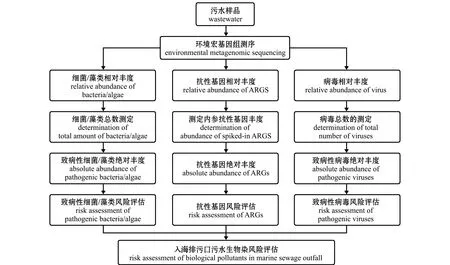
图2 绝对定量宏基因组测序技术在入海排污口污水风险评估中的流程Fig.2 Workflow of absolute quantification of metagenomic sequencing technology in the risk assessment of wastewater
4.2 构建基于绝对定量宏基因组测序技术的污水监测预警体系
随着宏基因组测序技术的不断发展,对污水样品进行现场测序已经具备了可行性。特别是便携式纳米孔测序仪的出现,更是让研究人员能够在野外读取污水样本中的生物遗传信息,从而为防止生物污染物的进一步扩散提供及时有效的信息。污水监测预警体系的构建主要包括以下两个方面。
1)提供所有病原微生物和ARGs的绝对丰度。相对于一次只能对1种或少数几种病原进行定量检测的传统方法,在理论上可以覆盖成千上万种所有可能的生物污染物,并对每一种生物污染物的丰度进行推测,从而实时提供包括SARS-CoV-2等高致病性病毒、有毒藻类和抗生素ARGs丰度数量等信息。这将给未来传染性疾病和生物污染物的监测预警带来革命性的变革。绝对定量宏基因组测序技术,可以在数小时内快速测定污水样品中的生物遗传信息,计算所有病原微生物和ARGs的丰度,实现在线风险评估。未来研究人员将可以通过一次宏基因组测序,全面掌握监测水体中生物污染物的数量,大大提升污水监测的效率。
2)实现定量风险评估。结合本研究团队开发的定量污水预测模型和其他定量风险评估工具,人们可以进一步通过绝对定量宏基因组测序获取生物污染物的种类和数量,并预测出包括新冠病毒肺炎和感染性腹泻等多种疾病的感染人数。同时,给上述检测指标设置不同阈值后,可以通过污水监测,实现对诸如SARS-CoV-2疫情、感染性腹泻疫情和有害藻华暴发等重大公共安全事件的早期预警。
可以预见,在不远的将来,随着现场测序技术、绝对定量宏基因组测序技术和风险评估模型的不断完善,人们将实现对所有生物污染物指标的定量在线监测和风险评估,并构建多层级污水监测预警体系。这些技术的研发将有助于实现海洋污染物的早期预警和及时处置,从而有力地保障公共卫生和海洋生态环境安全。
