冷胁迫下甘蓝型冬油菜表达蛋白及BnGSTs基因家族的鉴定与分析
2023-01-12赵玉红孙柏林侯献飞王旺田蒲媛媛刘丽君陶肖蕾孙万仓武军艳
马 骊 白 静 赵玉红 孙柏林 侯献飞 方 彦 王旺田 蒲媛媛 刘丽君 徐 佳 陶肖蕾 孙万仓,* 武军艳,*
冷胁迫下甘蓝型冬油菜表达蛋白及基因家族的鉴定与分析
马 骊1白 静2赵玉红3孙柏林3侯献飞4方 彦1王旺田1蒲媛媛1刘丽君1徐 佳1陶肖蕾1孙万仓1,*武军艳1,*
1省部共建干旱生境作物学国家重点实验室 / 甘肃省油菜工程技术研究中心 / 甘肃农业大学农学院, 甘肃兰州 730070;2张掖市农业科学院, 甘肃张掖 734000;3甘肃亚盛农业研究院有限公司, 甘肃兰州 730030;4新疆农业科学院经济作物研究所, 新疆乌鲁木齐 830091
谷胱甘肽S-转移酶(glutathione S-transferases, GST)参与调节植物生长、发育和逆境胁迫反应的许多方面。本研究利用双向电泳和质谱技术分析‘16VHNTS309’在冷胁迫下差异表达的蛋白质, 基于GO和KEGG分析鉴定出BE、APX、SOD、GST等参与冷胁迫反应的蛋白质。利用qRT-PCR和生理指标鉴定到响应冷胁迫的关键蛋白质GST, 采用同源克隆法克隆到‘16VHNTS309’的基因。该基因CDS长度为642 bp, 编码213个氨基酸, 是一个不稳定蛋白, 属于GST_N_3谷胱甘肽S-转移酶家族, 与甘蓝型油菜‘ZS11’序列相似性为99.22%, 与‘ZS11’和‘Vision’两者的氨基酸序列相比较发现第127位亮氨酸(L)突变为脯氨酸(P)。基因家族分析表明, 在甘蓝型油菜中共鉴定到153个BnGSTs成员, 按其功能主要分为Zeta、Phi、Theta、CHQ、DHAR、Lambda和Tau这七大类型, 大部分的BnGSTs属于Phi和Tau这2种类型。系统进化将BnGSTs分为12个亚家族, 亚家族I和VIII包含了较多的成员,不均匀的分布在18条染色体上, C06染色体上分布基因数量最多, 含有10个保守的蛋白质基序。BnGSTs基因家族中有99对基因存在共线性关系, 131个基因来自基因复制事件, 片段重复事件在基因的进化中起着重要作用。冷胁迫下、、、和在强抗寒品种中的显著高表达, 是弱抗寒品种的7~12倍, 并且强抗寒品种具有较高的生理酶活性。另外, 在冷冻胁迫下鉴定到一些瞬时和持续表达的关键候选基因。这些结果为进一步研究基因在强抗寒甘蓝型冬油菜抗寒分子调控中的作用奠定基础。
甘蓝型冬油菜; 冷胁迫; 蛋白质谱; GST基因家族; 表达模式
在中国北纬35°以北的大部分地区, 极端冬季气温低于-20℃, 负积温低于-500℃, 冬季降雨量低于30 mm, 这种寒旱的生态条件使北方冬油菜生产环境十分严酷[1-2]。随着育种进程的推进, 一批强冬性甘蓝型冬油菜品种已被成功选育, 这些新品种(系)能够在甘肃(庆阳、平凉、白银、定西等)、河北(衡水、保定等)安全越冬, 越冬率达70%以上, 这极大的提升了北方强冬性冬油菜区的冬油菜产量、品质和营养价值, 对中国北方甘蓝型冬油菜产业发展具有重要意义[3-4]。因此, 研究并解析强冬性甘蓝型冬油菜抗寒机制, 对我国北方强抗寒甘蓝型冬油菜品种选育和种质改良至关重要。
植物遇到冷害后在分子水平上响应主要是基因表达、蛋白质水平和代谢产物变化[5]。蛋白质直接响应植物逆境应激反应, 并且在植物响应逆境胁迫期间可以观察到其变化[6]。基于质谱(MS)的蛋白质组学已经成为揭示蛋白质丰度和植物蛋白质对胁迫响应之间关系的重要工具[7]。谷胱甘肽S-转移酶是一类在植物受到胁迫时可以保护植物细胞及蛋白活性, 并提高植物抗逆性的多功能蛋白酶[8]。植物GST分为Phi (GSTF)、Tau (GSTU)、Lambda (GSTL)、Theta (GSTT)、Zeta (GSTZ)、EF1Bγ、脱氢抗坏血酸还原酶(DHAR)和四氯代氢醌脱卤素酶(TCHDQ) 8类, Phi、Tau、Lambda和DHAR为植物所特有, Phi和Tau在植物中含量最为丰富[9-10]。众多报道显示, GSTs在植物应激反应中发挥着重要作用。亚麻()的GSTs和UGTs在ROS的解毒过程和细胞壁修饰中具有重要作用[11]。研究表明核桃(L.)和香蕉(L.)中在渗透胁迫和非生物胁迫反应中都起着关键作用, 并且可能受到多个上游调节因子的调控[12-13]。烟草基因在盐、冷、干旱、ABA和水杨酸胁迫下都会上调表达, 并且在过表达Tau类基因的烟草表现出较好的非生物胁迫耐受性[14]。
前人研究已经证实基因调控植物的冷应答途径, 因此, 研究强冬性甘蓝型冬油菜的GST蛋白和基因功能有助于解析强冬性甘蓝型冬油菜抗寒机制。本研究采用双向电泳技术, 分离强抗寒甘蓝型冬油菜‘16VHNTS309’中响应冷胁迫的蛋白质, 对响应冷胁迫的关键蛋白质进行功能注释和通路分析,鉴定到差异表达的GST蛋白, 并对基因进行生物信息学和表达模式分析, 旨在为甘蓝型冬油菜抗寒性研究提供理论依据。
1 材料与方法
1.1 材料种植与冷胁迫处理
强抗寒甘蓝型冬油菜‘16VHNTS309’由甘肃农业大学油菜育种课题组选育, 是以‘Vision’为母本、超强抗寒白菜型冬油菜‘陇油7号’为父本杂交, 通过集团选择结合系谱法选育的强抗寒甘蓝型冬油菜品种, 适应甘肃天水周边、陇东南部、陕西渭北旱源、山西上党盆地、河北冀中南地区秋播种植。弱抗寒甘蓝型冬油菜‘天油2238’是由天水市农业科学研究所选育, 以‘SW03-500056’为母本, ‘华双3号’为父本杂交, 后代经过系统选择、自交纯化选育出的甘蓝型优质冬油菜常规品种, 适宜在甘肃省天水、陇南干旱、半干旱、二阴山区, 海拔1800 m以下地区秋播种植。不抗寒甘蓝型冬油菜‘Vision’由甘肃农业大学油菜育种课题组从欧洲引进。将种子置于装有湿滤纸的玻璃培养皿中进行发芽, 在植物培养箱中(22/18℃, 16 h光照/8 h黑暗周期, 光照强度(46.8mmol m–2s–1)放置2 d。选取生长状况良好、生长阶段一致的幼苗移植到直径15 cm的育苗盆中, 盆中填充基质和蛭石(3∶1)。六叶期时将幼苗移入人工气候室, 在4℃低温下分别处理1、3、6、12和24 h, 22℃为对照处理[3]。处理结束后收集每株植物的生长锥, 用蒸馏水冲洗, 在滤纸上吸干, 剖成约5 mm厚的切片, 立即液氮冷冻, 随后储存在–80℃冰箱备用。参照杨宁宁等[15]方法测定丙二醛(MDA)、可溶性糖(SS)、可溶性蛋白质(SP)和游离脯氨酸(Pro)含量以及过氧化氢酶(CAT)、过氧化物酶(POD)、超氧化物歧化酶(SOD)、过氧化物酶(APX)活性。
1.2 双向电泳(2-DE)与蛋白质谱
采用Ettan IPGphor III和Ettan DALTsix进行双向电泳(General Electric Company, GE, 美国), 根据操作指南进行试验。采用Epson Expression 12000XL扫描仪(Seiko Epson Corporation, Epson, 日本)对脱色后的凝胶进行图像获取(600 dpi), 将2-DE凝胶保存于含有少量冰乙酸的MilliQ水中, 置于4℃冰箱备用。利用PDQuest 8.0.1软件(Bio-Rad Laboratories, 美国)对凝胶图谱进行图像裁剪与降噪、点和图像匹配与矫正、蛋白点表达定量, 对蛋白表达量进行-test分析, 选择表达丰度2倍以上且可重复的点送生工生物工程(中国上海)股份有限公司进行质谱鉴定[16]。
1.3 GST基因家族鉴定与进化关系分析
从十字花科数据库(http://brassicadb.cn/)中下载甘蓝型油菜基因组、蛋白质和CDS数据, 拟南芥基因组信息由拟南芥数据库(https://www.arabidopsis. org/)下载, 从Pfam数据库(http://pfam.sanger.ac.uk/)下载GST_N_3结构域(PF13417)的隐马尔可夫模型文件。利用HMMER 3.1软件(http://hmmer.org/down load.html)构建HMM模型, 在甘蓝型油菜和拟南芥基因组数据库中搜索GST成员, 采用BLASTP软件再次比对(-value≤1e–5), 删除冗余序列, 将候选成员提交至在线网站SMART (http://smart.embl-heidelberg.de/)和CD-Search (https://www.ncbi.nlm.nih. gov/Structure/cdd/wrpsb.cgi)中进行手动筛选, 最终获得甘蓝型油菜153个GST成员, 拟南芥59个GST成员[17]。利用ClustalW对甘蓝型油菜和拟南芥GST蛋白序列进行多序列比对, 利用MEGA7.0软件中最大似然法构建系统发育树[18]。
1.4 GST基因家族成员的序列结构、染色体定位和共线性分析
利用MEME软件(http://alternate.meme-suite. org/tools/meme)分析BnGSTs蛋白序列中的基序(motif)类型, Gene Structure Display Server软件(GSDS2.0, http://gsds.cbi.pku.edu.cn)分析基因的外显子和内含子的结构[19]。利用基因在染色体上的位置信息, 通过MapChart软件绘制基因的染色体位置图。采用MCScanX分析甘蓝型油菜BnGSTs基因间的重复事件和共线性关系, 并用Tbtools软件绘图[20]。
1.5 基因克隆与qRT-PCR
使用TRIzol Regent (DP419, 天根生化科技(北京)有限公司)试剂盒按照说明书提取总RNA。根据PrimeScript RT试剂盒(RR036A, 宝生物工程有限公司, TaKaRa, 中国大连)说明书合成cDNA第一条链。根据蛋白质谱鉴定结果得到谷胱甘肽S-转移酶DHAR1蛋白质序列, 并在NCBI中查找其对应的mRNA序列(XM_013786426.2)。采用Primer Premier 5.0软件设计引物, GST-F: 5¢-ATGGCCACTATCACC GTCGTTAAG-3¢, GST-R: 5¢-CTAGTACGGCTCCAC AGGCTTGCG-3¢。以‘16VHNTS309’和‘Vision’生长锥cDNA为模板, 按程序扩增: 94.0℃ 4 min; 94.0℃ 30 s, 60.0℃ 30 s, 72.0℃ 60 s, 35个循环; 72℃ 10 min。PCR结束后对扩增产物进行电泳检测(1%)并回收目的片段(DP209, 天根生化科技(北京)有限公司); 与pMD-19T载体连接成功后转化至DH5α感受态细胞, 培养12 h后进行蓝白斑筛选, 挑选单独白斑开始菌液培养, PCR检测后各选取5个阳性菌落进行测序(生工生物工程(上海)股份有限公司)。使用SYBR Premix ExII (RR820A, 宝生物工程有限公司, TaKaRa, 大连)在LightCycler 96 (罗氏集团, Roche, 瑞士)上进行qRT-PCR分析(表1)。以甘蓝型油菜基因为内参对照以标准化模板cDNA的表达量, 采用ΔCt方法计算基因的相对表达量[21]。冷胁迫和冷冻胁迫下的甘蓝型冬油菜叶片的转录组信息来自甘肃农业大学油菜课题组(SRP540905)[22]。

表1 qRT-PCR引物序列
2 结果与分析
2.1 冷胁迫下甘蓝型冬油菜差异表达蛋白质鉴定
利用PDQuest软件分析‘16VHNTS309’中的蛋白质表达差异, 有351个和396个蛋白点分别在对照组和冷应激下被匹配, 匹配率为88.6%。有169个蛋白点在冷胁迫后发生了明显的变化(Fold Change≥2)。对其表达丰度进行分析后筛选出29个差异变化的蛋白点(图1、附图1和附表1), 经测序质谱分析后匹配成功且得分(Protein scorn C.I.%)大于95%共25个。对这些蛋白质的表达分析得到18个上调表达蛋白、1个下调表达蛋白以及6个诱导表达蛋白。PSORT在线软件预测这些蛋白质定位于细胞质、微体、线粒体、内质网、叶绿体、细胞核和细胞外。
2.2 冷胁迫下差异蛋白质的功能与通路分析
利用Blast 2 go软件对‘16VHNTS309’差异蛋白进行GO功能富集分析, 并依据差异蛋白在发育过程中所行使的功能将这些蛋白质归为3类: 生物进程(GO:0008150)占33.64%、分子功能(GO:0003674)占36.36%、细胞组份(GO:0005575)占30%。生物进程中细胞过程、单生物过程、代谢过程、刺激反应分别为19.89%、17.52%、16.97%、16.97%。分子功能中结合作用占40.89%, 催化活性占35.56%。细胞组成中细胞组分、细胞、细胞器所占比分较大, 分别为16.28%、16.28%、16.28% (图2-A~C)。对差异蛋白质进一步分析发现, RNA结合蛋白CP29B (spot2)、双功能烯醇酶2/转录激活剂(spot6)、kunitz型丝氨酸蛋白酶抑制剂(spot24)、L-抗坏血酸过氧化物酶-1 (spot15)参与对寒冷响应途径, 超氧化物歧化酶(spot18)参与超氧化物歧化酶的代谢过程途径, 谷胱甘肽S-转移酶DHAR1 (spot27)参与L-抗坏血酸代谢过程。利用KEGG数据库注释鉴定的29种蛋白质, 共参与18个代谢通路(图2-D)。主要包括碳代谢(map01200)、乙醛酸和二羧酸代谢(map00630)、光合生物中的碳固定(map00710)、谷胱甘肽代谢(map00480)、抗坏血酸和醛酸代谢(map00053)、光合作用(map00195)、RNA降解(map03018)。

图1 冷胁迫下差异表达蛋白的鉴定
A: CK; B: 4℃处理24 h。箭头表示25个蛋白点被鉴定(≤ 0.05)。
A: CK; B: 4℃ for 24 hours. Arrows indicate 25 protein spots that are positively identified at≤ 0.05.
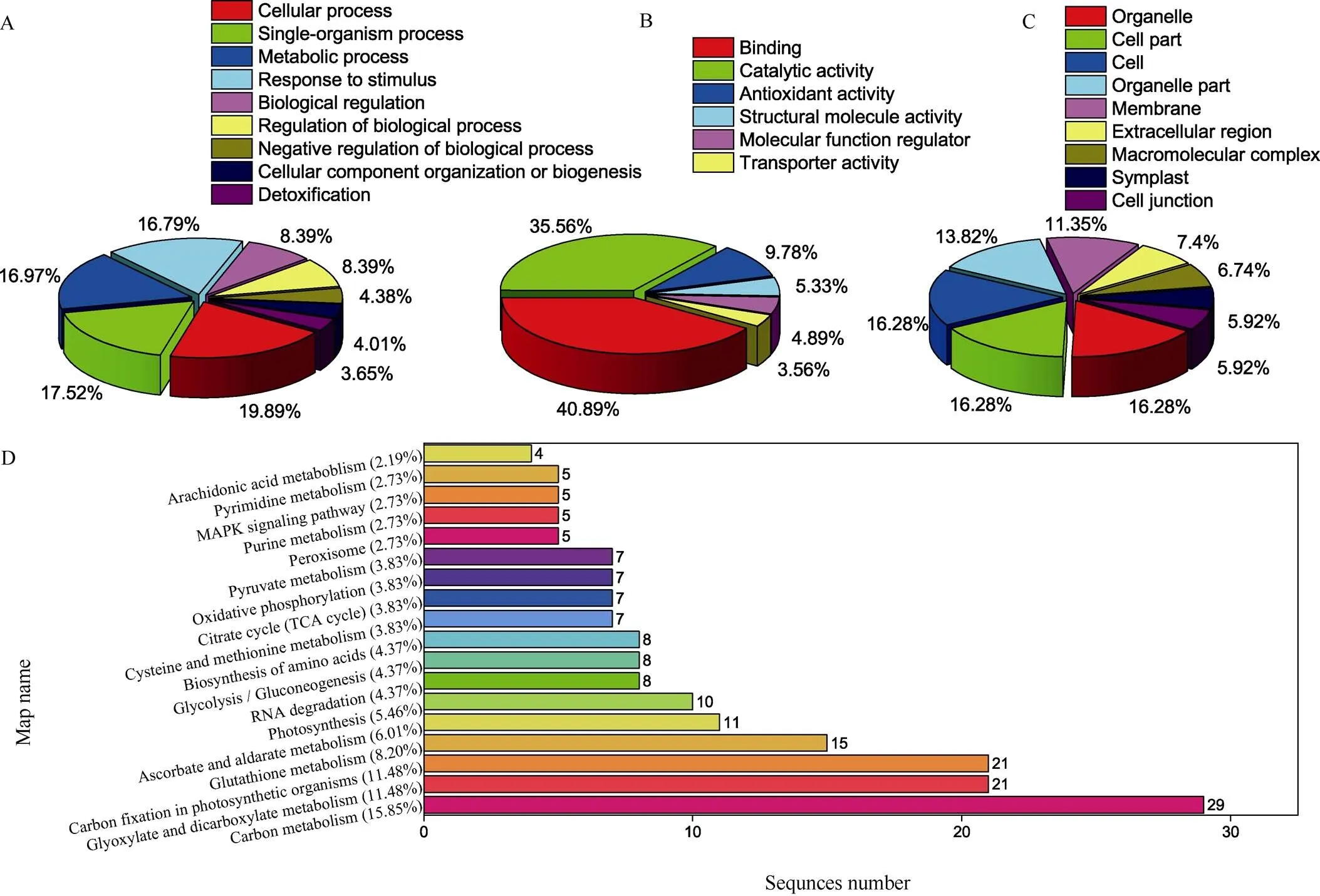
图2 冷胁迫下‘16VHNTS309’差异蛋白的功能及通路注释
A: 细胞组分; B: 分子功能; C: 生物学过程; D: KEGG注释。
A: cellular component; B: molecular function; C: biological process; D: KEGG annotations.
2.3 蛋白质、基因及生理表达模式变化的比较
根据差异蛋白点的表达丰度, 采用基于丰度的聚类方法25个鉴定的蛋白质进行聚类, 获得2个主要类群(图3-A)。第1类包含冷胁迫下17个上调表达的蛋白质, 包括Bifunctional enolase 2/transcriptional activator (BE, spot 6)、L-ascorbate peroxidase 1 (APX, spot 15, 25)、Superoxide dismutase [Cu-Zn] 2 (SOD, spot 18)、Glutathione S-transferase DHAR1 (GST, spot 27)等。第2类包含冷胁迫下下调表达、不表达及常温处理下不表达的8个蛋白质, 包括RNA-binding protein CP29B (RBP, spot 2)和Kunitz-type serine protease inhibitor (KTI, spot 24, 30)等。冷胁迫下观察到在‘16VHNTS309’中BE、APX、SOD、GST为表达丰度较高的蛋白质斑点, 较对照分别上调0.99、1.39、1.46、1.07倍(log2水平)。其mRNA表达水平随着冷胁迫时间增加出现不同幅度的上调, ‘16VHNTS309’中的和基因在冷胁迫6 h时表达量最大, 分别为对照的2.89倍和2.67倍。‘16VHNTS309’的基因在冷胁迫3 h时显著表达, 为对照的2.85倍, 并且显著高于‘Tianyou2238’和‘Vision’冷胁迫处理下的表达量。‘16VHNTS309’的基因在冷胁迫12 h时表达最高, 为对照的4.86倍, 是不抗寒品种‘Vision’的1.61倍。这4种与冷胁迫应答相关的蛋白和基因在冷胁迫下具有一致表达模式, 但基因的表达时间要比蛋白更早。而在冷胁迫处理下蛋白未检测成功, 其mRNA表达水平在冷胁迫下上调表达,的蛋白和其mRNA表达模式相反。冷胁迫后、、、、在不同抗寒性甘蓝型冬油菜中的表达趋势均表现强抗寒品种>弱抗寒>不抗寒品种,基因则呈现相反趋势, 并且其生理指标也反映出相似的变化模式(图3-C)。

图3 冷胁迫下差异蛋白质和基因表达模式及生理指标的分析
A: 蛋白质的表达谱与聚类分析; B: 6个具有急剧变化的DEPs采用qRT-PCR揭示其基因表达水平; C: 冷胁迫下的生理变化模式。小写字母表示所有品种和处理间的显著性(≤ 0.05)。
A: protein expression profiling and clustering analysis; B: six DEPs with drastic changes in gene expression levels revealed by qRT-PCR; C: the patterns of physiological changes under cold stress. Lowercase letters indicate significance difference at≤ 0.05 among all varieties and treatments.
2.4 甘蓝型油菜GST克隆及蛋白质理化性质分析
筛选出蛋白和mRNA水平均显著高表达的谷胱甘肽S-转移酶DHAR1, 其参与冷胁迫和过氧化物酶体等途径。以‘16VHNTS309’和‘Vision’为模板进行克隆及生物信息学分析发现, ‘16VHNTS309’基因() CDS长度为642 bp (图4), 与甘蓝型油菜‘ZS11’基因组序列相似性达99.22%, 共有5个碱基发生变化; 与“Vision”序列相似性为98.75%。该基因编码213个氨基酸, ‘ZS11’和“Vision”的氨基酸序列是完全相同的, 与‘16VHNTS309’相比较发现第127位亮氨酸(L)突变为脯氨酸(P) (附图2)。‘16VHNTS309’的氨基酸组成中Leu、Lys、Ser 3种氨基酸所占比例最高, 分别为11.3%、10.3%、8.9%, 相对分子质量约23.53 kD, 等电点为5.76, 有3333个原子, 分子式为C1076H1671N267O314S5, 含有30个负电荷残基, 24个正电荷残基。GST含有1个跨膜区域, 91至110位氨基酸之间的由外到内螺旋, 平均亲水指数为-0.490, 为亲水性蛋白, 无信号肽。预测不稳定指数为45.17, 是一个不稳定蛋白。预测翻译后磷酸化修饰的主要是Ser、Thr和Tyr的残基位点。搜索到其包含5个保守的domain, 包括GST_N_3等谷胱甘肽S-转移酶家族和细胞内氯离子通道蛋白家族。GST结构包含44.13%α螺旋、36.62%不规则卷曲、14.08%延伸链、5.16%β转角(附图2)。
2.5 BnGSTs基因家族染色体分布与进化分析
在甘蓝型油菜基因组中共检索到153个基因, 这些基因不均匀的分布在18条染色体上(图5), 除A01染色体上没有基因, C06染色体上分布基因数量最多, C07染色体上只有一个基因, 表明甘蓝型油菜GST基因的分布广泛。为了进一步研究GST的进化关系, 利用甘蓝型油菜(153个家族成员)和拟南芥(59个家族成员)的GST蛋白序列构建了系统发育树(图6), 根据序列的相似性和拓扑结构将GST被归类为17个亚家族(I-XVII), 亚家族IV和XVII包含最多的家族成员, 分别为53个和69个家族成员, 亚家族XIV和XVI分别只有一个家族成员。基于序列结构和功能, 将这些GST分为七大类, 分别为Zeta、Phi、Theta、CHQ、DHAR、Lambda和Tau, 而大部分的GST属于Phi和Tau类。亚家族VII没有被注释在这些类群中。此外, 本研究对153个BnGSTs成员进行了无根发育树的构建, 同样将这些成员分为12个亚类, 其中亚簇IV、VII、IX和XI分别包含2、2、1和1个成员(图7-A)。

图4 ‘16VHNTS309’GST基因序列比对
黑色: 3个品种氨基酸序列一致; 红色: 2个品种氨基酸序列一致; 白色: 氨基酸序列与另2个品种不一致。
Black: the amino acid sequences of 3 varieties are consistent; red: the amino acid sequences of 2 varieties are consistent; white: the amino acid sequences are not consistent with the other 2 varieties.

图5 甘蓝型油菜GST基因的染色体分布图
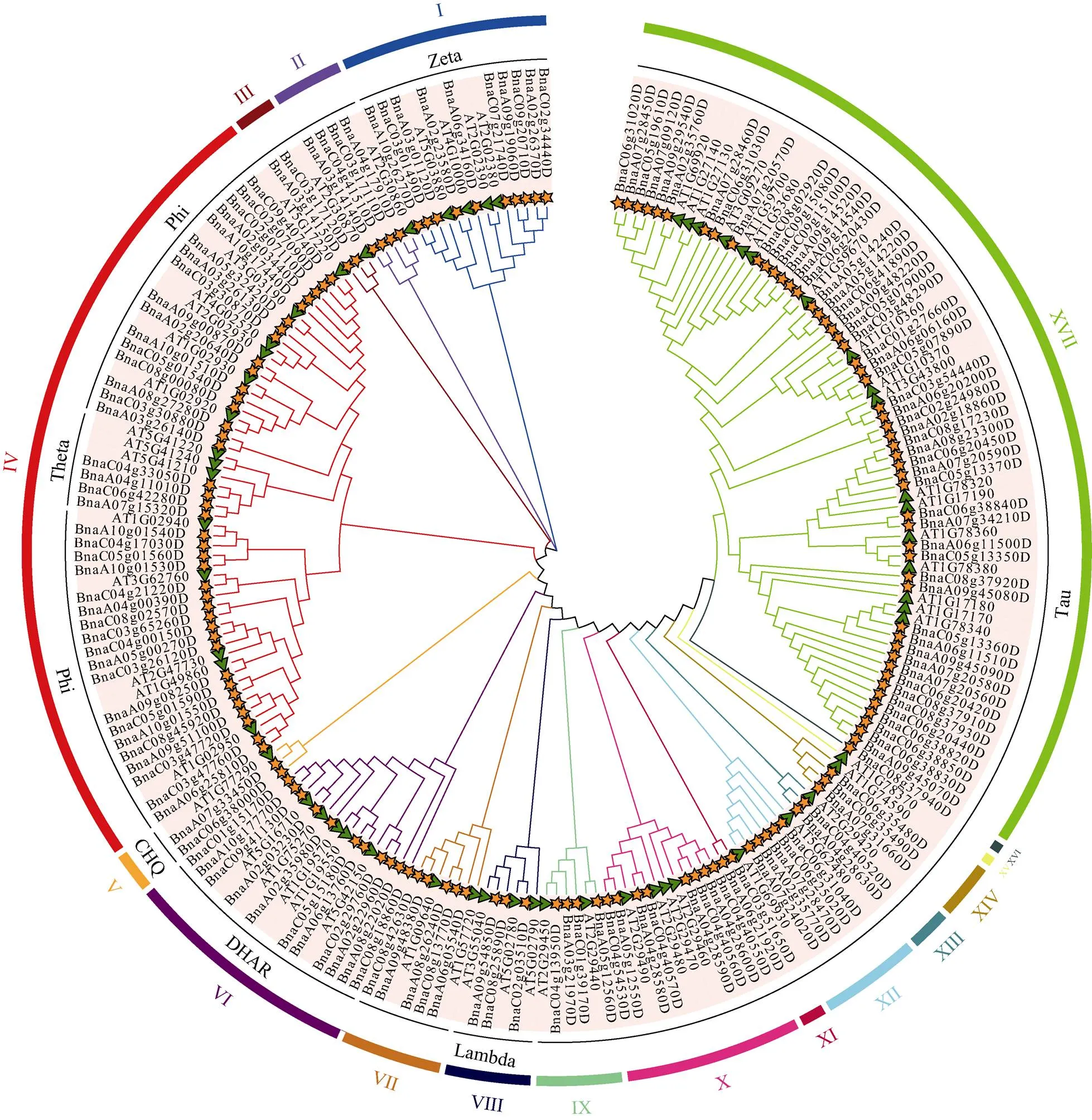
图6 甘蓝型油菜和拟南芥GST蛋白的系统发育树
亚家族I~XVII用不同的颜色标记。绿色三角形代表拟南芥, 黄色五角星代表甘蓝型油菜。
Subfamilies I–XVII are marked with different colors. Green triangles represent, and yellow stars represent.
2.6 BnGSTs基因结构、Motif组成与共线性分析
对甘蓝型油菜BnGSTs家族成员的基因结构分析表明, 153个的基因结构主要分为2类, 一类是不含UTR, 另一类是具有UTR的序列结构。外显子数目在1~33个之间, 第VII亚族的2个成员和只有1个外显子, 而外显子数目为33个, 各亚家族之间的外显子数目相似。利用MEME软件分析154个BnGSTs家族成员保守的蛋白质基序, 预测出10个保守的蛋白质基序(Motif), 每个Motif包含21~50个氨基酸。其中亚家族I~VII主要包含Motif 1、Motif 3、Motif 5、Motif 6、Motif 7、Motif 8和Motif 10, 亚家族VIII~XII主要包含Motif 2、Motif 4和Motif 9, 亚家族XII有3个成员没有匹配到Motif (图7)。利用MCScanX分析甘蓝型油菜基因组内共线性结果显示, BnGSTs基因家族中有99对基因存在共线性关系, 染色体A06和C07之间的共线性基因对最多, 为14对, 其余染色体之间的共线性基因对为1~4对, C01染色体上没有的共线性基因。进一步分析发现BnGSTs基因家族中131个来自基因复制事件, 均为片段重复, BnGSTs基因家族中没有串联重复事件, 片段重复事件在基因的进化中起着重要作用(图8)。

图7 甘蓝型油菜BnGSTs的系统进化、Motif组成和基因结构分析
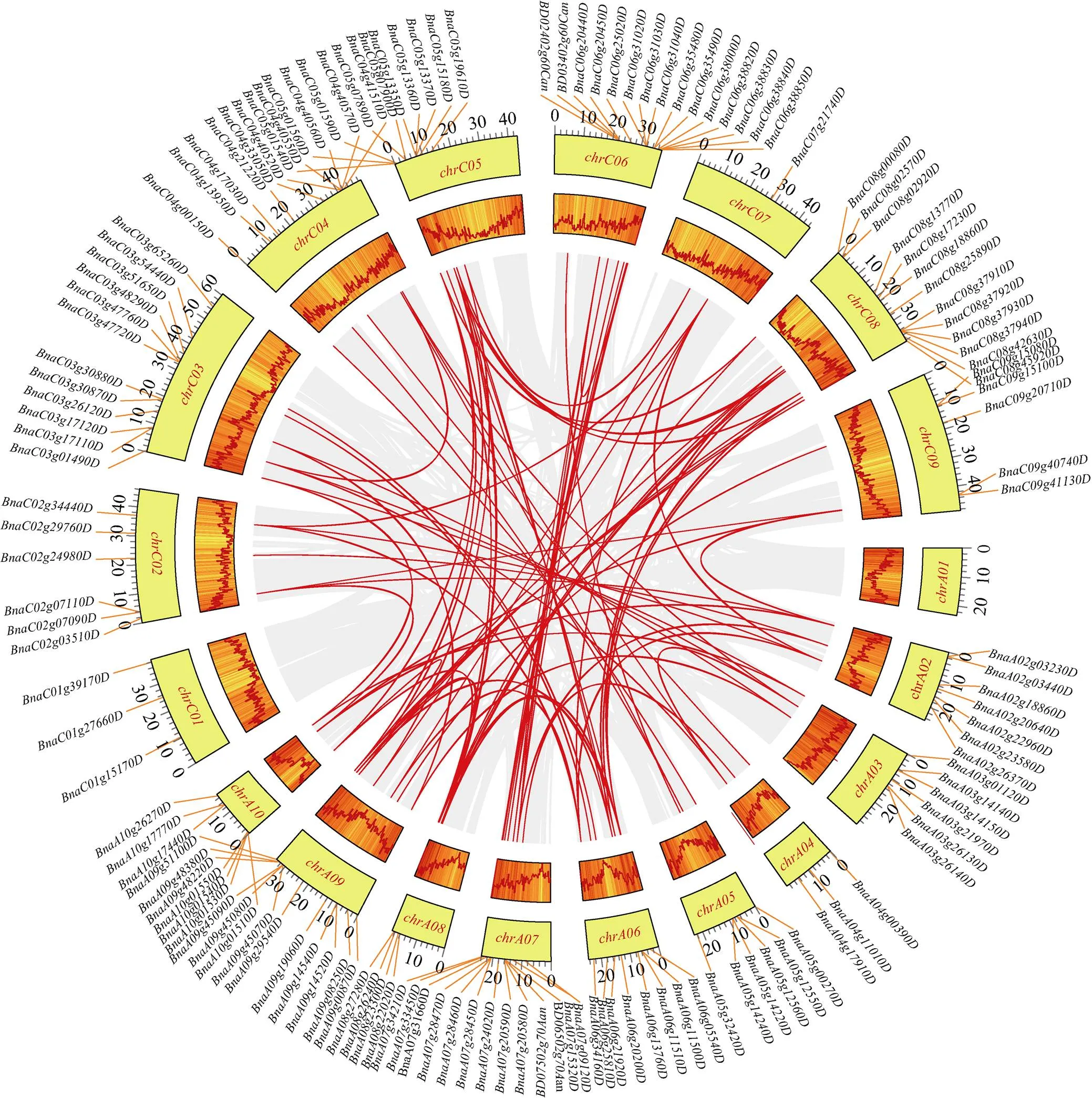
图8 甘蓝型油菜BnGSTs的共线性关系分析
2.7 BnGSTs基因家族在低温胁迫下的表达
利用转录组数据比较了冷(4℃)和冷冻(-2℃)胁迫下甘蓝型冬油菜BnGSTs家族成员的表达模式(图9)。在冷胁迫下, 强抗寒‘16VHNTS309’中有98个上调表达基因, 42个下调表达基因, 未表达基因13个, 显著上调表达基因50个, 显著下调表达基因13个(log2Fold Change≥1或≤1,-value≤0.05为显著); 在弱抗寒的‘天油2238’中有76个上调表达基因, 63个下调表达基因, 14个不表达基因, 显著上调表达基因41个, 显著下调表达基因27个。其中、、、和这个5个基因在‘16VHNTS309’中表达量达到7以上, 而在弱抗寒‘天油2238’中均显著下调表达,在‘16VHNTS309’的表达量为9.18, 而在天油2238中表达量为-9.53。和在强抗寒品种的表达量为12.46和12.44, 在弱抗寒品种中的表达量为11.79和3.01。另外在‘天油2238’中表达量为9.74, 是‘16VHNTS309’的7.79倍,在2个品种中均上调表达。在冷冻胁迫下上调表达、下调表达和未表达基因分别为44、50和59个。发现一些上调/下调表达基因(、、、、、、和)在1、3和24 h处理下的表达水平一致, 例如在3个时间段的表达量分别为4.02、4.22和4.14,的为–3.08、–3.22和–2.21。、和在1和3 h处理下显著上调表达, 而在24 h表达量下降,在3个时间段的表达量分别为4.42、5.69和1.21。各亚族中均有较高或较低表达的基因, 表明该家族基因在响应冷胁迫过程中的应答机制之间存在差异, 分工不同。这些特殊的差异表达基因为我们后续的研究提供了大量的候选基因资源。
3 讨论
蛋白质组学在植物响应低温胁迫研究中得到广泛应用, 已经在拟南芥、小麦、水稻等作物中鉴定出诸多与冷胁迫相关的蛋白质[23-25]。大多植物初次完全暴漏在非冰冻温度下, 即冷胁迫过程中, 会使自身抗寒性增加[26]。在‘16VHNTS309’中, 超氧化物歧化酶[Cu-Zn] (spot18)和谷胱甘肽S-转移酶DHAR1 (spot27)参与对超氧化物的反应途径, L-抗坏血酸过氧化物酶1 (spot15和25)参与L-抗坏血酸的代谢过程, Kunitz型丝氨酸蛋白酶抑制剂DrTI-like (spot 11、19、24和30)与对过氧化氢的反应有关。RNA结合蛋白CP29B (spot 2和9)参与冷适应, 烯醇酶2/转录激活剂(spot 6)参与对温度刺激的反应。研究发现溶氧增强(spot 8)和叶绿体核酮糖1,5-二磷酸羧化酶/加氧酶小亚基(spot 17、23和26)参与光合作用, 冷胁迫后这些蛋白表达增强, Rubisco通过光合作用碳还原周期来控制光通量, 以响应环境中的短期变化[27]。强抗寒甘蓝型冬油菜在冷胁迫下能正常进行光合作用, 可能与其苗期地上部生长旺盛有关[28]。冷胁迫下强抗寒甘蓝型冬油菜中上调表达或新出现的这些蛋白质, 反映出冷胁迫诱导了强抗寒甘蓝型冬油菜的抗氧化酶基因和冷诱导基因的表达, 并增加了抗氧化蛋白和酶的活性。
在生理层面上, 强抗寒甘蓝型冬油菜利用保护酶和调节物质来提高其抗寒能力[29]。植物受到冷胁迫后ROS大量积累并破坏细胞的结构, 在严重的情况下细胞过度失水, 导致叶片死亡。此时, 植物的抗氧化能力的强弱直接反映在其将产生的ROS恢复到正常水平的能力上[30-32]。本研究结果表明, 在冷胁迫下甘蓝型冬油菜的保护性酶、可溶性物质、Pro和APX均升高, 并且抗寒性强的品种生理水平均优于抗寒性弱的品种, 积累较少的MDA (图3-C)。谷胱甘肽是抗氧化系统的一个组成部分, 包括谷胱甘肽S-转移酶(GST)、谷胱甘肽过氧化物酶(GPX)和谷胱甘肽还原酶(GR), 保护植物免受诱导应激反应的某些外源性物质与活性氧(ROS)的伤害[12]。
本研究在甘蓝型油菜中鉴定到153个BnGSTs家族成员, 分为12个亚族, 在每个亚族中各成员都含有相似的基因结构和保守的Motif, 根据这些保守的Motif发现甘蓝型油菜BnGSTs家族成员大部分属于Phi和Tau类的GST, 并且研究证实这2类的基因在植物抵抗非生物胁迫过程中具有重要的功能[33]。不同的亚族之间的基因结构和Motif存在个别差异, 这些差异可能是造成基因家族功能分化的原因。基因家族和基因组进化机制的扩展主要取决于基因复制事件, 主要的复制模式是串联和片段复制[34]。本研究发现, BnGSTs基因家族中有131个属于基因复制事件, 片段复制是BnGSTs基因家族扩展过程中起主要推动作用[35]。并且片段复制事件对Tau分支的扩展贡献更大, 可能是由于其防御反应的作用, BnGSTs支系可以提高植物对各种环境胁迫的耐受性[36]。谷胱甘肽S-转移酶在植物生长发育和胁迫管理中具有至关重要的作用[37]。从哈密瓜中鉴定了39个具有典型结合位点的基因, 它们可分为7个亚家族。在低温胁迫下, 耐冷性品种具有较高的GST酶活性和28个胁迫相关基因的表达水平, 一些属于Tau、Phi和DHAR类别的在冷胁迫下发挥了重要作用[38]。Yang等[39]研究发现, 冷胁迫处理下核桃(L.)转基因系生理酶活性显著高于对照及RNAi沉默系, 表明冷胁迫下转核桃可通过提高基因的表达增加GST酶活性, 增强自身的耐冷性。
Solexa/Illumina RNA测序(RNA-seq)近年来被用来鉴定不同物种的耐寒基因[40]。植物对冷胁迫的反应的研究涉及许多植物物种的转录组分析, 包括拟南芥()、甘蓝型油菜(L.)、茶树()等其他诸多物种[41-44]。研究西葫芦果实对低温反应的RNA-Seq分析揭示了几种与低温耐受有关的分子机制, 并鉴定了可调节或促进低温耐受的候选基因, 包括转录因子MYB76、、DELLA蛋白、GAIP和AP2/ERF结构域的蛋白[45]。之前的研究通过转录组测序鉴定了甘蓝型冬油菜在低温胁迫下的132个基因, GO功能分析和启动子序列分析表明, 这些基因参与了诸多可能增强抗寒性的分子途径[41]。Basnet等[46]对2种不同形态的白菜型油菜种子进行基因共表达分析表明, 白菜型油菜种子的发育过程时间比其相关物种甘蓝型冬油菜要晚。本研究利用转录组数据分析了冷(4℃)和冷冻(–2℃)胁迫下甘蓝型冬油菜BnGSTs家族成员的表达模式。在冷胁迫下鉴定到、、、和在强抗寒甘蓝型冬油菜中显著上调表达, 在冷冻胁迫下鉴定到一些持续表达基因(、、、、、、和)和瞬时表达基因(、和), 这些基因在强抗寒甘蓝型冬油菜响应冷胁迫过程中发挥重要作用。另外, 部分BnGSTs在冷和冷冻胁迫下的表达趋势相反, 说明不同的基因响应不同的逆境胁迫, 而一些基因在2种胁迫下的表达趋势相似, 反应出基因功能的单一性和多效性[35,47]。表明BnGSTs在抵御低温胁迫方面扮演着重要的角色。
4 结论
‘16VHNTS309’基因序列与甘蓝型油菜序列相似性达99.22%, 共有5个碱基位点差异, 第127位氨基酸突变。从甘蓝型油菜中鉴定到153个BnGSTs家族基因, 大部分的具有Phi和Tau这2个类GST的功能, 冷胁迫下、、、和这5个在强抗寒甘蓝型冬油菜响应冷胁迫过程中具有重要作用。另外, 一些瞬时和持续表达基因在强抗寒甘蓝型冬油菜响应冷冻胁迫下发挥关键作用。这项研究结果从蛋白水平、生理变化和BnGSTs基因家族表达模式反映了强抗寒甘蓝型冬油菜响应冷胁迫的综合信息, 为解析强抗寒甘蓝型冬油菜抗寒分子机制提供了新的线索。
附图和附表 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical- zuowxb.aspx。
[1] 孙万仓, 刘海卿, 刘自刚, 武军艳, 李学才, 方彦, 曾秀存, 许耀照, 张亚宏, 董云. 北方寒旱区白菜型冬油菜安全越冬的临界指标分析. 作物学报, 2016, 42: 609–618.
Sun W C, Liu H Q, Liu Z G, Wu J Y, Li X C, Fang Y, Zeng X C, Xu Y Z, Zhang Y H, Dong Y. Critical index analysis of safe over-wintering rate of winter rapeseed () in cold and arid region in north China., 2016, 42: 609–618 (in Chinese with English abstract).
[2] 刘自刚, 孙万仓, 杨宁宁, 王月, 何丽, 赵彩霞, 史鹏飞, 杨刚,李学才, 武军艳, 方彦, 曾秀存. 冬前低温胁迫下白菜型冬油菜抗寒性的形态及生理特征. 中国农业科学, 2013, 46: 4679–4687.
Liu Z G, Sun W C, Yang N N, Wang Y, He L, Zhao C X, Shi P F, Yang G, Li X C, Wu J Y, Fang Y, Zeng X C. Morphology and physiological characteristics of cultivars with different levels of cold-resistance in winter rapeseed (L.) during cold acclimation., 2013, 46: 4679–4687 (in Chinese with English abstract).
[3] 蒲媛媛, 赵玉红, 武军艳, 刘丽君, 白静, 马骊, 牛早霞, 金姣姣, 方彦, 李学才, 孙万仓. 北方强冬性甘蓝型冬油菜品种(系)抗寒性评价. 中国农业科学, 2019, 52: 3291–3308.
Pu Y Y, Zhao Y H, Wu J Y, Liu L J, Bai J, Ma L, Niu Z X, Jin J J, Fang Y, Li X C, Sun W C. Comprehensive assessment on cold tolerance of the strong winterL. cultivated in northern China., 2019, 52: 3291–3308 (in Chinese with English abstract).
[4] 曹小东, 刘自刚, 米文博, 徐春梅, 邹娅, 徐明霞, 郑国强, 方新玲, 崔小茹, 董小云, 米超, 陈其鲜. 甘蓝型冬油菜北移种植的适应性分析. 中国农业科学, 2020, 53: 4164–4176.
Cao X D, Liu Z G, Mi W B, Xu C M, Zou Y, Xu M X, Zheng G Q, Fang X L, Cui X R, Dong X Y, Mi C, Chen Q X. Analysis on the adaptability of northward planting of., 2020, 53: 4164–4176 (in Chinese with English abstract).
[5] Li L J, Lu X C, Ma H Y, Lyu D G. Comparative proteomic analysis reveals the roots response to low root-zone temperature in., 2018, 131: 865–878.
[6] Xuan J P, Song Y F, Zhang H X, Liu J X, Guo Z R, Hua Y L. Comparative proteomic analysis of the stolon cold stress response between the C4 perennial grass speciesand., 2013, 8: e75705.
[7] Wang J C, Meng Y X, Li B C, Ma X L, Lai Y, Si E J, Yang K, Xu X L, Shang X W, Wang H J, Wang D. Physiological and proteomic analyses of salt stress response in the halophyte., 2015, 38: 655–669.
[8] 张雪, 陶磊, 乔晟, 杜秉昊, 郭长虹. 谷胱甘肽转移酶在植物抵抗非生物胁迫方面的角色. 中国生物工程杂志, 2017, 37(3): 92–98.
Zhang X, Tao L, Qiao S, Du B H, Guo C H. Roles of glutathione S-transferase in plant tolerance to abiotic., 2017, 37(3): 92–98 (in Chinese with English abstract).
[9] Lan T, Yang Z L, Yang X, Liu Y J, Wang X R, Zeng Q Y. Extensive functional diversification of the populus glutathione S-transferase supergene family., 2009, 21: 3749–3766.
[10] Anderson J V, Davis D G. Abiotic stress alters transcript profiles and activity of glutathione S-transferase, glutathione peroxidase, and glutathione reductase in., 2004, 120: 421–433.
[11] Dmitriev A A, Krasnov G S, Rozhmina T A, Kishlyan N V, Zyablitsin A V, Sadritdinova A F, Snezhkina A V, Fedorova M S, Yurkevich O Y, Muravenko O V, Bolsheva N L, Kudryavtseva A V, Melnikova N V. Glutathione S-transferases and UDP-glycosyltransferases are involved in response to aluminum stress in flax., 2016, 7: 1920.
[12] Yang G Y, Chen S W, Li D P, Gao X Q, Su L Y, Peng S B, Zhai M Z. Multiple transcriptional regulation of walnutgene in response to osmotic stress., 2019, 166: 748–761.
[13] Wang Z, Huang S Z, Jia C H, Liu J H, Zhang J B, Xu B Y, Jin Z Q. Molecular cloning and expression of five glutathione S-transferase (GST) genes from Banana (L. AAA group, cv.)., 2013, 32: 1373–1380.
[14] Jha B, Sharma A, Mishra A. Expression of(class glutathione S-transferase) gene isolated fromin tobacco for salt tolerance., 2011, 38: 4823–4832.
[15] 杨宁宁, 孙万仓, 刘自刚, 史鹏辉, 方彦, 武军艳, 曾秀存, 孔德晶, 鲁美宏, 王月. 北方冬油菜抗寒性的形态与生理机制. 中国农业科学, 2014, 47: 452–461.
Yang N N, Sun W C, Liu Z G, Shi P H, Fang Y, Wu J Y, Zeng X C, Kong D J, Lu M H, Wang Y. Morphological characters and physiological mechanisms of cold resistance of winter rapeseed in northern China., 2014, 47: 452–461 (in Chinese with English abstract).
[16] Sheffield J, Taylor N, Fauquet C, Chen S X. The cassava (Crantz) root proteome: Protein identification and differential expression., 2006, 6: 1588–1598.
[17] Mistry J, Finn R D, Eddy S R, Bateman A, Punta M. Challenges in homology search: HMMER3 and convergent evolution of coiled-coil regions., 2013, 41: e121.
[18] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33: 1870–1874.
[19] Bailey T L, Boden M, Buske F A, Frith M, Grant C E, Clementi L, Ren J Y, Li W W, Noble W S. MEME suite: Tools for motif discovery and searching., 2009, 37: W202–W208.
[20] Chen C J, Chen H, Zhang Y, Thomas H R, Frank M H, He Y H, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[21] Bustin S A, Benes V, Nolan T, Pfaffl M W. Quantitative real-time RT-PCR: a perspective., 2005, 34: 597–601.
[22] Pu Y Y, Liu L J, Wu J Y, Zhao Y H, Bai J, Ma L, Yue J L, Jin J J, Niu Z X, Fang Y, Sun W C. Transcriptome profile analysis of winter rapeseed (L.) in response to freezing stress, reveal potentially connected events to freezing stress., 2019, 20: 2771.
[23] Zhang L, Tian L H, Zhao J F, Song Y, Zhang C J, Guo Y. Identification of an apoplastic protein involved in the initial phase of salt stress response in rice root by two-dimensional electrophoresis., 2009, 149: 916–928.
[24] Wang X Q, Liu Y L, Yang P F. Proteomic studies of the abiotic stresses response in model moss-., 2012, 3: 258.
[25] He C Y, Gao G R, Zhang J G. Proteome profiling reveals insights into cold-tolerant growth in sea buckthorn., 2016, 14: 14.
[26] Kong F J, Oyanagi A, Komatsu S. Cell wall proteome of wheat roots under flooding stress using gel-based and LC MS/MS-based proteomics approaches., 2010, 1804: 124–136.
[27] Tian X, Liu Y, Huang Z G, Duan H P, Tong J H, He X L, Gu W H, Ma H, Xiao L T. Comparative proteomic analysis of seedling leaves of cold-tolerant and -sensitive spring soybean cultivars., 2015, 42: 581–601.
[28] Liu Z G, Sun W C, Zhao Y N, Li X C, Fang Y, Wu J Y, Zeng X C, Yang N N, Wang Y, He L. Effects of low nocturnal temperature on photosynthetic characteristics and chloroplast ultrastructure of winter rapeseed., 2016, 63: 451–460.
[29] Jędrzejuk A, Rabiza-świder J, Skutnik E, Łukaszewska A. Growing conditions and preservatives affect longevity, soluble protein, H2O2and MDA contents, activity of antioxidant enzymes and DNA degradation in cut lilacs., 2018, 228: 122–131.
[30] Apel K, Hirt H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction., 2004, 55: 373–399.
[31] Cao Y Y, Yang M T, Li X, Zhou Z Q, Wang X J, Bai J G. Exogenous sucrose increases chilling tolerance in cucumber seedlings by modulating antioxidant enzyme activity and regulating proline and soluble sugar contents., 2014, 179: 67–77.
[32] Yan S P, Zhang Q Y, Tang Z C, Su W A, Sun W N. Comparative proteomic analysis provides new insights into chilling stress responses in rice., 2006, 5: 484–496.
[33] Sappl P G, Oñate-sánchez L, Singh K B, Millar H. Proteomic analysis of glutathione S-transferases ofreveals differential salicylic acid-induced expression of the plant- specific phi and tau classes., 2004, 54: 205–219.
[34] Vision T J, Brown D G, Tanksley S D. The origins of genomic duplications in., 2000, 290: 2114–2117.
[35] Wei L J, Zhu Y, Liu R Y, Zhang A X, Zhu M C, Xu W, Lin A, Lu K, Li J N. Genome wide identification and comparative analysis of glutathione transferases (GST) family genes in., 2019, 9: 9196.
[36] He G, Guan C N, Chen Q X, Gou X J, Liu W, Zeng Q Y, Lan T. Genome-wide analysis of the glutathione S-transferase gene family in: identification, expression, and biochemical functions., 2016, 7: 1325.
[37] Islam S, Sajib S D, Jui Z S, Arabia S, Islam T, Ghosh A. Genome-wide identification of glutathione S-transferase gene family in pepper, its classification, and expression profiling under different anatomical and environmental conditions., 2019, 9: 9101.
[38] Song W, Zhou F K, Shan C H, Zhang Q, Ning M, Liu X M, Zhao X X, Cai W C, Yang X Q, Hao G F, Tang F X. Identification of glutathione S-transferase genes in hami melon (var.) and their expression analysis under cold stress., 2021, 12: 672017.
[39] Yang G Y, Xu Z G, Peng S B, Sun Y D, Jia C X, Zhai M Z. In planta characterization of a tau class glutathione S-transferase gene from() involved in chilling tolerance., 2016, 35: 681–692.
[40] Pagter M, Alpers J, Erban A, Kopka J, Zuther E, Hincha D K. Rapid transcriptional and metabolic regulation of the deacclimation process in cold acclimated., 2017, 18: 731.
[41] Du C F, Hu K N, Xian S S, Liu C Q, Fan J C, Tu J X, Fu T D. Dynamic transcriptome analysis reveals AP2/ERF transcription factors responsible for cold stress in rapeseed (L.)., 2016, 291: 1053–1067.
[42] Jeon J, Kim J. Cold stress signaling networks in., 2013, 56: 69–76.
[43] Li N N, Yue C, Cao H L, Qian W J, Hao X Y, Wang Y C, Wang L, Ding C Q, Wang X C, Yang Y J. Transcriptome sequencing dissection of the mechanisms underlying differential cold sensitivity in young and mature leaves of the tea plant ()., 2018, 224–225: 144–155.
[44] Zhu H S, Yu X J, Xu T, Wang T L, Du L X, Ren G H, Dong K H. Transcriptome profiling of cold acclimation in bermudagrass ()., 2015, 194: 230–236.
[45] Carvajal F, Rosales R, Palma F, Manzano S, Cañizares J, Jamilena M, Garridp D. Transcriptomic changes infruit after cold storage: Differential response between two cultivars contrasting in chilling sensitivity., 2018, 19: 125.
[46] Basnet R K, Moreno P N, Lin K, Bucher J, Visser R G F, Maliepaard C, Bonnema G. Genome-wide analysis of coordinated transcript abundance during seed development in differentmorphotypes., 2013, 14: 840.
[47] Kayum M A, Nath U K, Park J I, Biswas M K, Choi E K, Song J Y, Kim H T, Nou I S. Genome-wide identification, characterization, and expression profiling of glutathione S-transferase (GST) family in pumpkin reveals likely role in cold-stress tolerance., 2018, 9: 84.
Protein and physiological differences under cold stress, and identification and analysis ofinL.
MA Li1, BAI Jing2, ZHAO Yu-Hong3, SUN Bo-Lin3, HOU Xian-Fei4, FANG Yan1, WANG Wang-Tian1, PU Yuan-Yuan1, LIU Li-Jun1, XU Jia1, TAO Xiao-Lei1, SUN Wan-Cang1,*, and WU Jun-Yan1,*
1State Key Laboratory of Aridland Crop Science / Gansu Research Center of Rapeseed Engineering and Technology / College of Agronomy, Gansu Agricultural University, Lanzhou 730070, Gansu, China;2Zhangye Academy of Agricultural Sciences, Zhangye 734000, Gansu, China;3Gansu Yasheng Agricultural Research Institute Co. Ltd, Lanzhou 730030, Gansu, China;4Institute of Economic Crops, Xinjiang Academy of Agricultural Sciences, Urumqi 830091, Xinjiang, China
Glutathione S-transferases (GST) are involved in regulating many aspects of plant growth, development, and response to adversity stress. In this study, the differentially expressed proteins of ‘16VHNTS309’ under cold stress were analyzed using bidirectional electrophoresis and mass spectrometry, and proteins involved in the cold stress response, such as BE, APX, SOD, and GST, were identified based on GO and KEGG analyses. The key protein GST was identified using qRT-PCR and physiological indicators in response to cold stress, and thegene of ‘16VHNTS309’ was cloned using homologous cloning. This gene CDS length was 642 bp, and encoded 213 amino acids, which was an unstable protein and belonged to the GST_N_3 glutathione S-transferase family. The sequence similarity with‘ZS11’ was 99.22%, and a comparison of the amino acid sequences of both ‘ZS11’ and ‘Vision’ revealed a mutation from leucine (L) to proline (P) at position 127. The gene family analysis showed that 153 BnGSTs members were identified inand were classified into seven main types according to their functions: Zeta, Phi, Theta, CHQ, DHAR, Lambda, and Tau. Most BnGSTs belonged to Phi and Tau types. Phylogeny divided BnGSTs into 12 subfamilies, subfamilies I and VIII contained more members. BnGSTs were unevenly distributed on 18 chromosomes, and the number of BNGSTs genes on C06 chromosome was the largest, it contained 10 conserved protein motifs. There were 99 pairs of genes in the BnGSTs gene family that were colinearly related, 131 genes from gene duplication events, and segmental duplication events played an important role in the evolution of BnGSTs genes. Significantly higher expression of,,,, andin strong cold-resistant varieties was 7–12 times higher than that in weak cold-resistant varieties under cold stress. And the strong cold-resistant varieties had higher physiological enzyme activities. In addition, a number of key candidate genes were identified for transient and sustained expression under freezing stress. This result lays the foundation for further studies on the molecular regulation of BnGSTs genes for cold resistance in strong cold resistant for.
winterL.; cold stress; protein mass spectrometry; GST gene family; expression pattern
10.3724/SP.J.1006.2023.14207
本研究由甘肃省青年科技计划项目(21JR7RA835), 省部共建干旱生境作物学国家重点实验室(甘肃农业大学)开放基金课题(GSCS-2020-06)和财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-12)资助。
This study was supported by the Gansu Provincial Youth Science & Technology Department (21JR7RA835), the Research Program Sponsored by State Key Laboratory of Aridland Crop Science (Gansu Agricultural University) (GSCS-2020-06), and the China Agriculture Research System of MOF and MARA (CARS-12).
通信作者(Corresponding authors):武军艳, E-mail: wujuny@gsau.edu.cn; 孙万仓, E-mail: 18293121851@163.com
E-mail: mal@gsau.edu.cn
2021-11-04;
2022-05-05;
2022-05-19.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220518.1945.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
