肿瘤坏死因子-α基因-环境交互作用与不明原因复发性流产易感性的关系
2023-01-10李静裴建赢刘倩王文第金玉霞毛宝宏
李静 裴建赢 刘倩 王文第 金玉霞 毛宝宏
1甘肃省妇幼保健院(兰州 730050);2兰州大学第二医院(兰州 730020)
复发性自然流产(recurrent spontaneous abortion,RSA)病因复杂,发生率约占1%~5%[1],严重影响着我国育龄期女性的生殖健康[2-3]。其中仍有约50%,其发病机制至今尚未完全明确,称作原因不明复发性流产(unexpected recurrent spontaneous abortion,URSA)。肿瘤坏死因子-α(tumor necrosis factor-α,TNF-α)主要由单核巨噬细胞分泌的一类促炎因子,它与URSA的相关性是目前研究的热点且研究结果存在分歧[4]。对于基因-环境之间的潜在关系,广义多因子降维法(generalized multifactor dimensionality reduction,GMDR)[5-6]被越来越多地用来发现复杂疾病潜在的生物学途径。目前有关基因-基因交互作用与URSA相关性的研究较少。因此,本研究拟基于GMDR模型探讨 TNF-α 基因-308G/A(rs1800629)和-238G/A(rs361625)两个单核苷酸多态性位点与URSA人群危险性的关系,为兰州地区人群URSA的病因的寻找及后期制定干预措施提供科学依据。
1 资料与方法
1.1 一般资料 本研究收集2019年1月至2020年8月在我院门诊就诊的URSA患者150例为病例组,年龄19~41岁;纳入标准:(1)流产次数≥ 2次;(2)甲状腺激素、胰岛素、性激素等内分泌检查正常;(3)无高血压、糖尿病、风湿免疫性疾病、家族遗传病史;(4)夫妇双方无近亲结婚,染色体均未见异常;(5)免疫学相关抗体筛查(抗心磷脂抗体、抗核抗体、抗甲状腺球蛋白抗体、抗β2-糖蛋白Ⅰ抗体)正常。对照组为无自然流产史、早产史和妊娠高血压病史并且至少成功分娩1次一名健康足月儿,年龄20~40岁,两组比较年龄差异无统计学意义(P>0.05)。本研究已经获得本院伦理委员会批准[编号:[2018]院伦审研第(3号)],研究对象均签订知情同意书。
1.2 研究方法
1.2.1 一般情况调查、标本采集及基因组DNA提取 对纳入人群统一行面对面调查问卷,获取一般人口学特征、生育史、环境暴露史及相关生活行为特征。采集外周静脉血5 mL,提取基因组DNA,并检测提取DNA浓度和纯度。
1.2.2 TNF基因SNP测定 深圳华大有限公司设计本次实验PCR扩增引物(正向:5'-CTTTCTGAAGCCCCTCCCAGTT-3',22 bp;反向:5'-GTTGGGGACACACAAGCATCAA-3',22 bp)及多重单碱基延伸反应延伸引物(正向:5'-TGAAGACCCCCCTCGGAATC-3',20 bp;反向:5'-TTTTTTGGAGGCTGAACCCCGTCC-3',24 bp),并采用 SNaPshot技术进行基因分型实验。多重PCR反应体系(20 μL)包含去离子水 5 μL,2x GC-I buffer(Takara.)10 μL,3.0 mmol/L Mg2+0.4 μL,0.3 mmol/L dNTP 2.4 μL,1 U HotStarTaq polymerase(Qiagen Inc。)0.2 μL,样本DNA1 μL和多重PCR引物1 μL。PCR循环程序:95℃ 2 min;94℃ 20 s、65℃和-0.5℃/cycle 40 s、72℃1.5 min,共11个循环;94℃ 20 s、59℃ 30 s、72℃ 1.5 min,24个循环;72℃ 2 min、4℃保存。PCR产物用Qiagen公司的HotStarTaq进行多重PCR获得,然后用ABI公司的SNaPshot Multiplex kit进行延伸反应。延伸反应后纯化,经ABI3730XL测序检测,收集的原始数据用GeneMapper4.1(AppliedBiosystems,USA)来分析并输出结果。
1.2.3 统计学方法 通过SAS 9.4(SAS Institute Inc.,Cary,NC,USA)软件进行分析。正态分布计量资料用均数±标准差表示,计数资料比较用χ2检验。双侧检验水准α=0.05。采用二分类logistic回归分析法分析TNF基因型频率及其在显性模型和隐性模型下与uRSA的关联性。应用SNPStats在线平台(https://www.snpstats.net/start.htm.)进行连锁不平衡分析和单体型及Hardy-Weinberg遗传平衡检验。基因型与环境危险因素交互作用应用GMDR进行模型构建和分析。
2 结果
2.1 基本情况及Hardy-Weinberg遗传平衡检测
病例组学历水平低于对照组,且是从事体力活动者易发生uRSA,轻中重度趋势相同(P<0.05),两组在年龄、民族、家庭收入、居住环境、体质指数、吸烟、饮酒喝茶、环境危险因素暴露分布差异均无统计学意义(P>0.05)。见表1。TNF基因rs1800629、rs361525位点在病例组和对照组均符合Hardy-Weinberg遗传平衡定律(均P>0.05)。

表1 研究对象一般特征分布Tab.1 Distribution of basic characteristics of the study population 例(%)
2.2 TNF基因基因型和等位基因分布比较及遗传模式分析 二分类logistic分析结果显示,校正年龄、教育、居住地、职业、工作属性、吸烟、接触高危因素、运动等协变量后,在共显性、显性和隐性遗传模式以及等位基因模式下,TNF基因rs1800629、rs361525位点均未发现与URSA发病风险增加有关,差异无统计学意义(P>0.05),见表2。
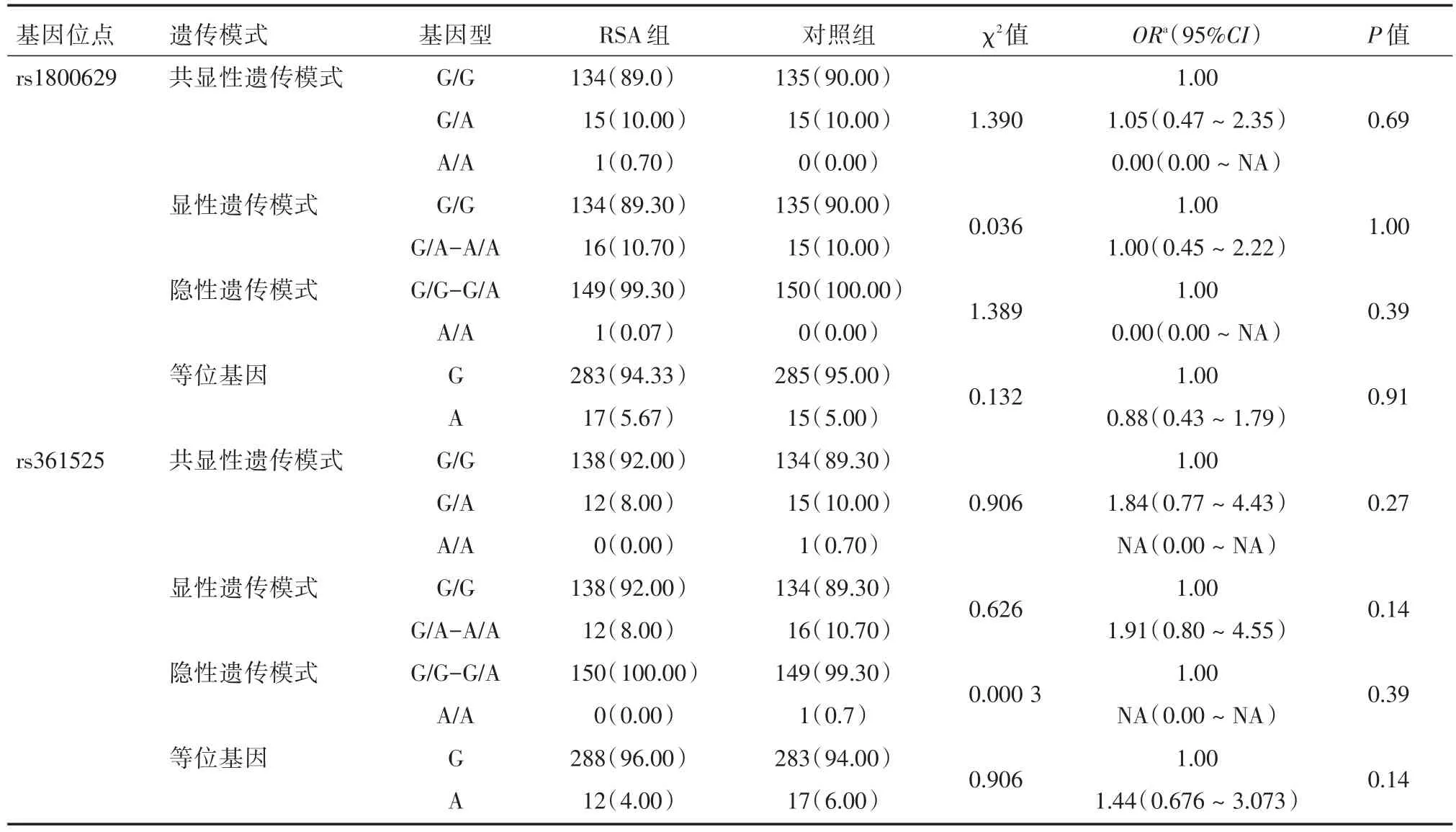
表2 两组多态性位点基因型和等位基因分布比较及遗传模式分析Tab.2 The comparison of SNPs's genotype,allele frequency and genetic model analysis in two groups例(%)
2.3 按流产次数进行分层分析 按流产次数进行分层分析,结果显示在所有遗传模型中,rs1800629位点不同流产次数下均未发现与URSA发病风险增加有关,差异无统计学意义(P>0.05)。在显性遗传模式中,rs361525位点G/A-A/A基因型携带者显著增加2次RSA的发生风险(ORa=2.526,95%CI:1.03~6.19);且在等位基因模式下,A等位基因携带者显著增加2次RSA的发生风险(ORa=2.402,95%CI:1.041~ 5.545),但≥ 3次RSA患者未见类似结果(表3)。
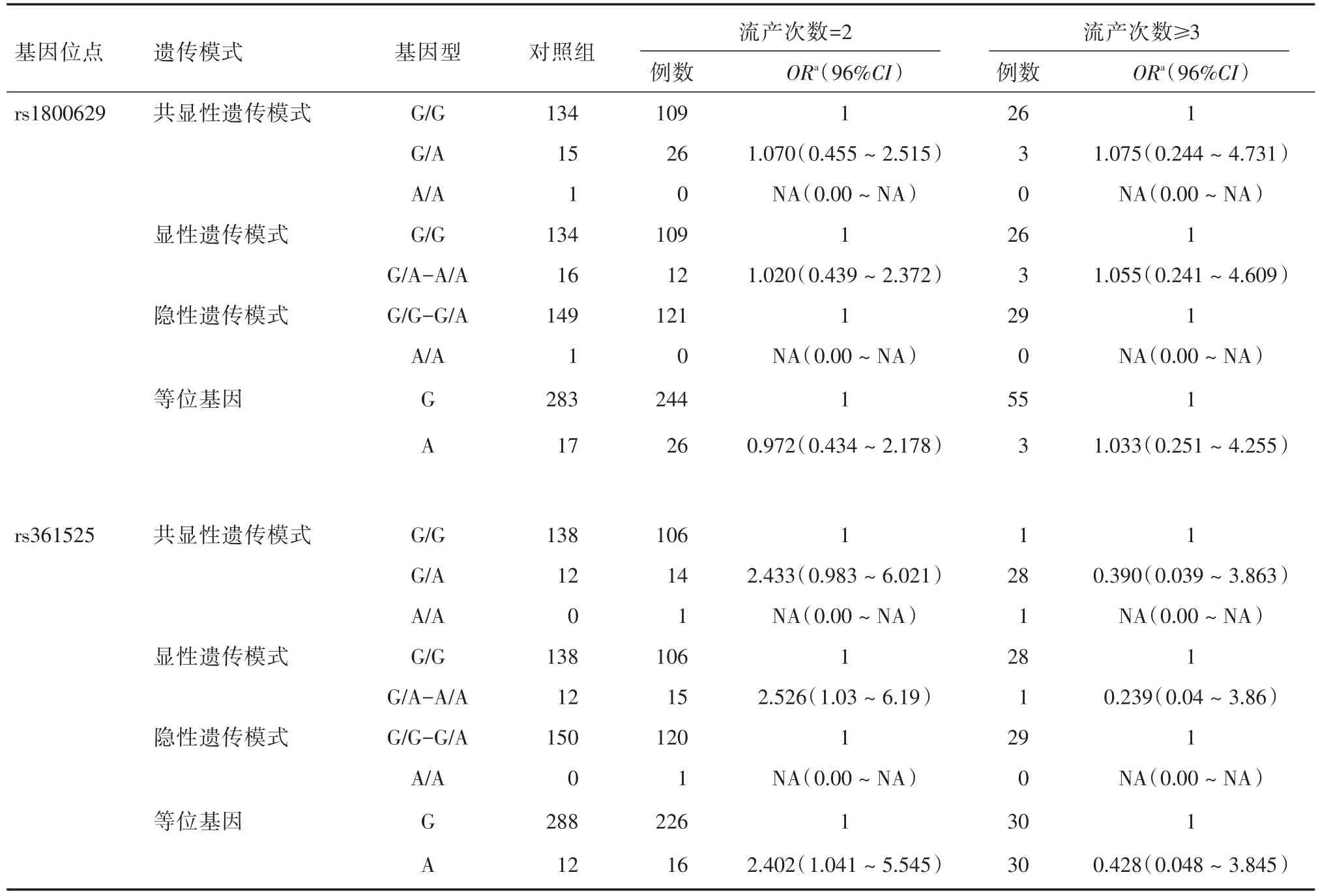
表3 按流产次数分层分析TNF基因SNPs与URSA易感性的关系Tab.3 Stratified analysis genetic model and URSA according the number of abortions in TNF locus
2.4 连锁不平衡检验和单体型分析 TNF基因rs1800629、rs361525两位点存在完全连锁不平衡(D'=0.979 8,r2=-0.052 4,P=0.199 2),排除分布频率<0.01的单体型,表4结果显示TNF基因两个位点形成3种单体型(AG、GA、GG),校正年龄等协变量因素后,各单体型在两组间分布差异无统计学意义(P <0.05,表4)。

表4 TNF基因位点单倍体型分布Tab.4 Haplotype distribution of TNF gene loci 例(%)
2.5 基因-环境交互作用分析 初筛出4个环境因素,教育水平、职业、工作属性、运动与否,最终只有工作属性这个环境因素能够建立较好的模型。从二阶交互最优模型是rs1805096与工作属性的交互作用,交叉一致性为10/10,测试集精准度为0.620 1,模型差异有统计学意义(P=0.004 7)。校正协变量因素后,在训练集中三因素交互显示三阶模型中rs1800629、rs361525及工作属性模型(训练集平衡精度=0.623 4,P=0.003 6),差异均有统计学意义,且十重交叉验证的一致率(比值)为10∶10,明显优于其他模型(表5)。
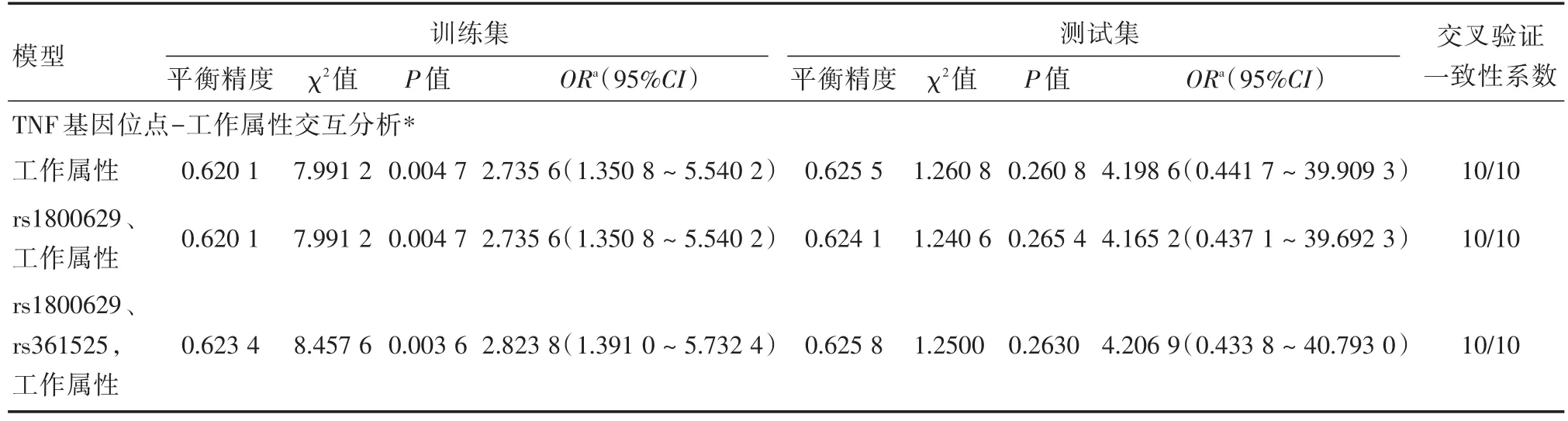
表5 TNF基因位点与环境交互作用GMDR分析结果Tab.5 GMDR analysis result of interaction between TNF gene loci and environment
3 讨论
TNF-α基因主要位于染色体6p21.3,该区域为高度多态的主要组织相容性复合体的Ⅲ类所在区域[7-8],其突变可能导致 TNF-α水平的变化,特别是在启动子区域[9-10],而且它的升高会诱导自然杀伤细胞的激活以及随后对胎盘生长的损害[11-12]。许多研究分析TNF-α多态性和RPL之间的关联。然而,结果仍然存在争议。
rs1800629位点是被研究的最多和最具有争议的一个位点。有些研究[13,15]发现该位点与RSA无关联,而有的研究[15-18]表明它与RSA正相关。此外,有研究[19-20]根据纳入人群的地理位置进行分层发现亚洲人群在该位点与RSA的关联性较欧洲人群更强。总之,引起这种结果争议可能原因是:(1)人种、地域等造成的遗传异质性;(2)选择标准差异:有的纳入的是流产3次以上人群,有的纳入2次以上人群;(3)排除标准差异:有的研究并未排除患者凝血、遗传、内分泌等较为明确的病因。为了系统的分析URSA的病因,本研究在排除了可能的造成RSA的原因后,同时校正年龄等协变量,发现rs1800629位点在共显性、显性和隐性遗传模式以及等位基因模式下其分布频率在两组间差异无统计学意义,与其他研究一致[13,15]。
对于rs36152位点,ZHAO等[21]系统评价研究表明,纯合子模型(AAvs.GG,OR=1.533,95%CI:1.022~2.301)和隐性模型(AAvs.GG+AG,OR=1.571,95%CI:1.050 ~ 2.350)与URSA风险具有相关性。但杂合子模型与URSA发病风险无关。而有的研究[13,23]未观察到此位点与RSA相关。在亚洲人群中与URSA相关的主要模型是(AGvs.GG;OR=7.155,95%CI:2.342 ~ 21.853)[22]。但是本研究结果显示,按流产次数进行分层分析,结果显示在共显性遗传模型、显性模型及隐性模型中,此位点A等位基因携带者显著增加发生2次流产的风险(OR=2.402,95%CI:1.041~5.545)。本研究结果和MA等[24]结果不一致。
URSA是多基因复杂性疾病,单体型分析比单个SNP分析更具有优势。STAVROS等[23]研究表明TNF-α-376G/-308A/-238G单倍型与低TNF-α水平和流产相关。但本研究单体型分析表明,TNF-α基因这两个位点形成3种单体型(AG、GA、GG),校正年龄等协变量因素后,各单体型在两组间分布差异无统计学意义。既往有研究[23]发现TNF-α基因无单体型与URSA的危险因素相关,与本报道类似。
GMDR是一种新颖而强大的检测和建模上位性的统计工具,它与logit模型相比,成比例的优势模型可以提高这些结果的统计能力,从而降低假阳性率的发生[26]。目前尚未见将GMDR方法应用于RSA的研究。本研究受样本量限制,将TNF-α基因的两个基因位点分别与环境因素(教育、居住地、职业性质、吸烟、接触高危因素、运动)引入模型,校正年龄等协变量,分析基因-环境交互作用,结果显示一阶交互最优模型是rs1800629与工作属性的交互作用,交叉一致性为10/10;二阶模型rs1800629、rs361525与工作属性模型存在交互作用。
综上所述,本研究发现兰州地区URSA人群TNF-α基因rs361525位点单核苷酸多态性与URSA(流产次数=2次)易感性相关,且研究对象的基因-环境交互作用中,模型(rs1800629、rs361525,工作属性)为最优模型,rs1800629、rs361525和工作属性之间的交互作用可能与URSA的发生有关联。基因与环境因素是URSA发病的重要因素,虽然既往研究表明了TNF基因与URSA密切相关,但该基因rs1800629、rs361525与URSA发生的关系,特别在兰州地区人群中并未明确。本研究存在一定的局限性,仅探讨了兰州地区人群,且样本量有限,但本研究说明URSA是由环境和遗传因素共同影响的复杂性疾病,在今后的研究中应进一步结合环境因素-内表型-基因进行交互分析,这将有助于从更广泛的角度去阐明URSA的病理机制。
