孤独症谱系障碍儿童肠道菌群多样性研究及功能预测分析
2022-12-08刘智程吴德屈爱娜王璐璐
刘智程 吴德 屈爱娜 王璐璐
(安徽医科大学第一附属医院小儿神经康复中心,安徽 合肥 230022)
孤独症谱系障碍(autism spectrum disorder,ASD)是一种以社交和沟通障碍、重复刻板行为及狭窄兴趣为核心症状的神经发育障碍性疾病。近年来,全球范围内ASD患病率快速上升。据统计,2018年美国8岁儿童中每44人就有1人患ASD[1]。2020年,我国6~12岁儿童ASD患病率为7‰[2]。目前,典型ASD尚无有效的治疗方法,给患儿家庭和社会带来了巨大负担。
ASD的病因尚不清楚,临床实践观察到近一半ASD儿童共患便秘、腹泻等胃肠道问题,因此,有研究者认为微生物-肠-脑轴(microbiota-gutbrain axis,MGBA)紊乱可能与ASD发病相关。已有为数不多的研究发现,ASD儿童肠道菌群的多样性或结构与正常发育(typically developing,TD)儿童不同,但具体研究结果并不统一。Lou等[3]及Kang等[4]研究均表明ASD儿童肠道菌群的多样性降低;Finegold等[5]研究则显示ASD儿童肠道菌群的多样性升高;此外,也有研究报道ASD儿童肠道菌群的α多样性无明显变化[6]。这些研究均基于操作分类单元(operational taxonomic units,OTUs)聚类的方法进行物种注释,其物种注释和多样性分析的准确性均低于基于扩增子序列变体(amplicon sequence variants,ASVs)的分析方法。本研究采用基于ASVs进行物种注释的16S rDNA高通量测序技术,前瞻性地分析ASD儿童肠道菌群的分布及多样性特征,并预测分析2组菌群代谢功能的差异,为进一步探索儿童ASD与肠道菌群的相关性,以及ASD的微生态疗法提供科学理论依据。
1 资料与方法
1.1 研究对象
选取安徽医科大学第一附属医院小儿神经康复中心门诊确诊为ASD的30例儿童纳入ASD组,同时招募20例TD儿童作为对照(TD组)。ASD组需符合美国精神障碍诊断与统计手册第5版中ASD的 诊 断 标 准[7],且 儿 童 孤 独 症 评 定 量 表(Childhood Autism Rating Scale,CARS)评 分≥30分。此外,2组需符合以下共同的纳排标准。纳入标准:(1)年龄≤12岁;(2)监护人同意此研究并签署知情同意书。排除标准:(1)早产及过期产儿童;(2)有神经系统或遗传代谢性疾病史;(3)有消化道畸形或消化道手术史;(4)合并胃肠道急慢性感染;(5)有粪便采样前1个月内抗生素、微生态制剂、质子泵抑制剂或激素类药物使用史。本研究经安徽医科大学第一附属医院伦理委员会批准(PJ2021-14-10)。
1.2 基本信息
受试者主要监护人填写问卷,包括受试者年龄、性别、出生方式和喂养方式、居住环境及婴儿期抗生素使用史等基本信息。
1.3 样本采集与保存
收集2组儿童上午10时之前的新鲜粪便2~3 g于无菌采样管中,立即低温转运至-80℃冰箱保存。
1.4 DNA提取、文库制备及测序
采用QIAamp Fast DNA Stool Mini Kit(QIAGEN)提取样本DNA,采用NanoDrop ND-1000(Thermo Fisher Scientific,Waltham,MA,USA)分光光度计测定DNA数量和质量,琼脂糖凝胶电泳检测DNA完整性。利用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')PCR扩 增16S rDNA V4区。用AMPure XP Beads(Beckman Coulter,Indianapolis,IN)试剂盒纯化PCR产物,使用Qubit(Thermofisher,Qubit 4.0)进行文库定量。采用Illumina NovaSeq6000平台进行双端测序。使用Vsearch 2.4.4组装拼接双端序列,质控并过滤低质量数据、接头序列或PCR错误等,再经过Vsearch 2.15.0去重、降噪、去嵌合体等步骤得到ASVs,样本中含量低于总序列0.001%的ASVs将被去除。通过Qiime2基于Silva138数据库对ASVs的代表序列进行物种注释。
1.5 生物信息学分析
主要使用Qiime2和R包3.2.0对测序数据进行分析。使用Qiime2计算α多样性指数(Chao1、Shannon和Simpson)评估物种的丰富度和均匀度。绘制稀释性曲线评价测序深度是否合理。采用Anosim分析检验组间差异是否显著大于组内,从而判断分组是否有意义。采用基于非加权UniFrac距离的主坐标分析(principal component analysis,PCoA)评估组间物种组成差异。采用Wilcoxon秩和检验分析选取2组样本在各分类水平上的差异物种。采用线性判别影响大小(linear discriminant analysis effect size,LEfSe)分析方法筛选组间生物标志物(即差异显著物种)。构建随机森林模型对样本进行分类预测,采用受试者工作特征(receiver operating characteristic,ROC)曲线验证模型的可靠性。采用Bugbase软件预测菌群表型特征。基于Picrust2 2.3.0-b软件预测菌群代谢功能。
1.6 统计学分析
使用SPSS 24.0软件进行统计学分析。非正态分布的计量资料以中位数(四分位数间距)[M(P25,P75)]表示,组间比较采用Wilcoxon秩和检验;计数资料采用例数和百分率(%)表示,组间比较使用卡方检验。P<0.05为差异有统计学意义。
2 结果
2.1 2组儿童基本信息比较
ASD组和TD组儿童入组年龄、性别、出生方式、喂养方式、婴儿期抗生素使用史、乡村居住史及宠物喂养史方面比较差异均无统计学意义(P>0.05),见表1。
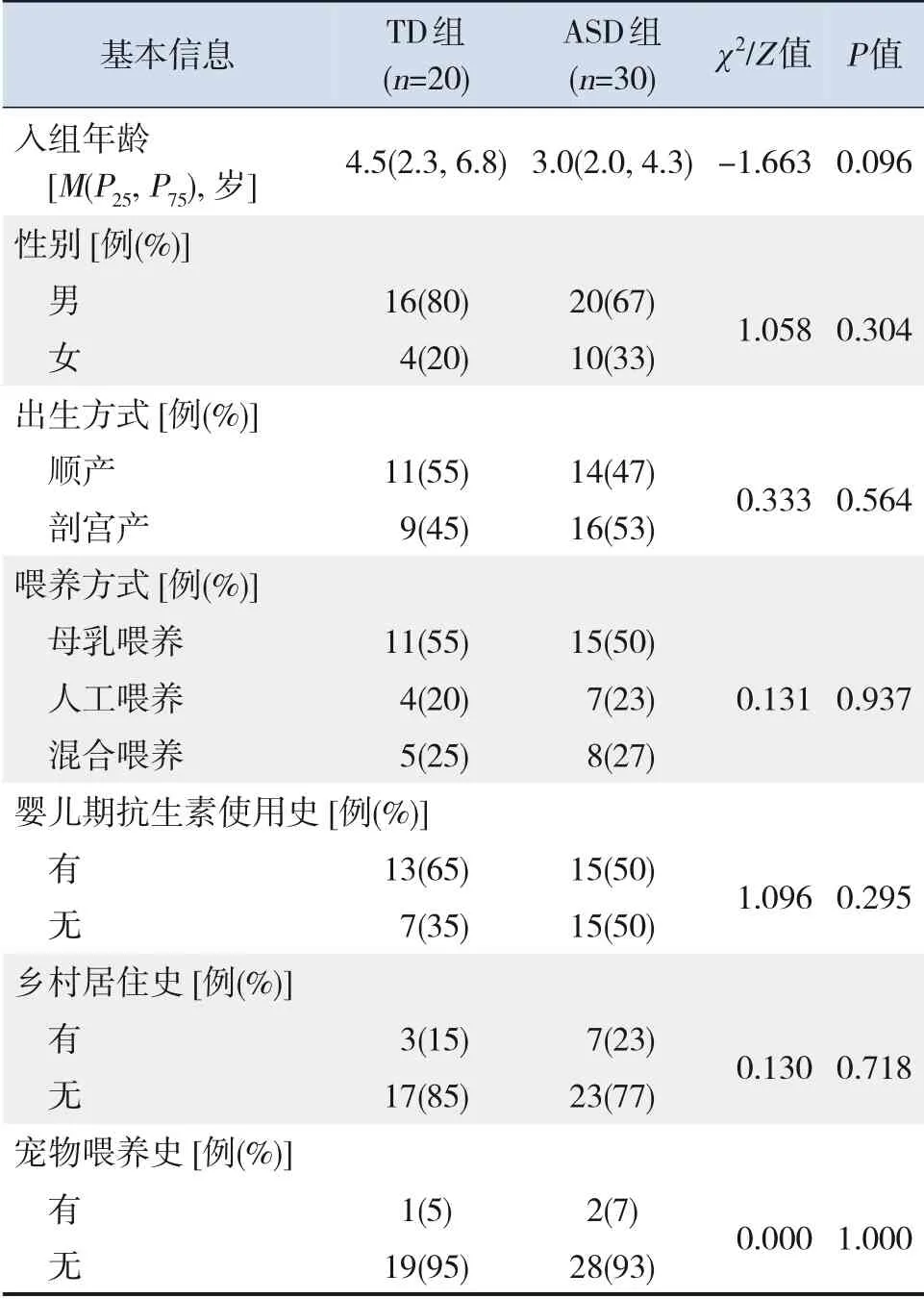
表1 2组儿童基本信息比较
2.2 测序数据统计
测序读长范围245~292 bp。50例样本测序得到总有效序列数为6 428 113。测得样本序列数最小为107 491,均数为128 562。稀释性曲线显示测序深度合理,见图1。得到ASVs总数为1 354,其中146个为ASD组特有,40个为TD组特有。

图1 ASVs稀释性曲线 曲线趋于平缓,提示测序深度已基本覆盖样本中所有物种。
2.3 物种多样性分析
α多样性指数中Chao1指数反映物种丰富度,Shannon和Simpson指数均反映物种的丰富度和均匀 度。ASD组 和TD组 样 本Chao1、Shannon及Simpson指数比较差异均无统计学意义(P>0.05),见表2。

表2 2组儿童肠道菌群α多样性分析[M(P25,P75)]
β多样性分析利用不同样本的丰度变化或进化关系来计算样本间距离,从而反映样本或组间微生物组成的相似性。Anosim分析可判断分组是否有意义,R>0说明组间差异大于组内,P值说明了差异的显著性。PCoA是一种非约束性数据降维分析方法,可用来研究样本群落组成的相似性。基于非加权UniFrac距离的Anosim及PCoA分析显示,2组儿童肠道菌群的分布不同,见图2。

图2 非加权UniFrac距离Anosim分析(A)和PCoA组间差异分析(B) A:Between代表组间差异,ASD、TD分别表示各组内差异;图中各箱线图的凹槽互不重叠,说明各组中位数差异显著。B:每个点代表一个样本,点间距反映样本相似性;图中ASD组和TD组散点部分分离,提示2组间存在一定差异。
2.4 物种分布及差异分析
ASVs经物种注释共得到8门、12纲、26目、44科、107属。ASD组和TD组样本中相对丰度位列前四的菌门相同,依次为厚壁菌门、拟杆菌门、放线菌门和变形菌门,这4种菌门的总丰度在各组中占比均超过99%,其中厚壁菌门和拟杆菌门总丰度在各组中占比均超过89%。2组样本中相对丰度位列前三的菌属均为拟杆菌属、粪杆菌属和双歧杆菌属。在门和纲水平,2组样本的菌群分布差异无统计学意义(P>0.05)。在属水平,ASD组中有6个菌属的相对丰度大于TD组,分别为巨单胞菌属(P<0.001)、巴恩斯氏菌属(P=0.004)、小杆菌属(P=0.006)、巨球菌属(P=0.020)、瘤胃球菌属扭链群(P=0.028)及梭杆菌属(P=0.040)。2组样本属水平主要菌群构成见图3。

图3 2组样本属水平物种相对丰度分布图 右侧图例列出了属水平相对丰度位列前十的物种名称,其余物种归为“其他”。
2.5 LEfSe筛选组间生物标志物
应用LEfSe分析方法选取各组优势物种,即组间生物标志物。结果显示,ASD组的优势物种为巨单胞菌属、小杆菌属、巨球菌属、消化链球菌科、巴恩斯氏菌属、瘤胃球菌属扭链群及链型杆菌属等;TD组优势物种为毛螺菌科、消化球菌科、Saccharimonadaceae、TM7x、阿里松氏菌属、帕拉普氏菌属、乳杆菌目_g_P5D1_392及Saccharimonadales等。见图4。

图4 LEfSe分析筛选各组生物标志物 柱状图的长度代表差异显著物种的影响大小(LDA score),其绝对值大于预设值(默认为2)表示该物种为差异有统计学意义的标志物。
2.6 随机森林组间预测
构建随机森林模型对ASD组和TD组样本进行鉴别,结果显示以巨单胞菌属、罕见小球菌属及小杆菌属等属水平细菌为特征可以较准确地区分2组样本(错误率为30.83%),其中巨单胞菌属对分类预测的效果影响最大,见图5。绘制ROC曲线评估随机森林分类模型的优劣,曲线下面积(area under curve,AUC)为0.82,说明该模型分组预测具有一定的准确性,见图6。

图5 随机森林物种重要性点图 横坐标(平均准确度下降)代表重要性水平,纵坐标为模型中对分类效果起主要作用的菌属,按作用从大到小排列。
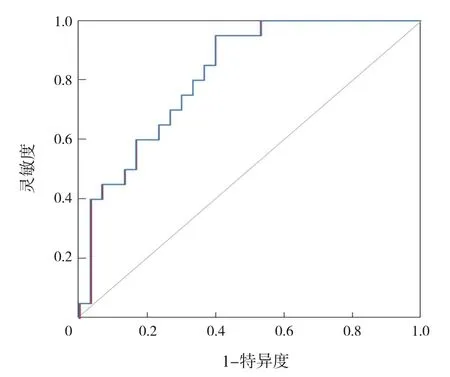
图6 评估随机森林分类模型的ROC曲线 AUC越接近于1,分类结果越准确;AUC在0.7~0.9时,分类结果有一定准确性。纵坐标(灵敏度)越高代表分类的准确率越高,横坐标(1-特异度)越低代表误判率越低,曲线越靠近左上角,说明模型的分类效果越好。
2.7 菌群表型特征预测
ASD组和TD组儿童肠道菌群的表型,如革兰阳性、革兰阴性、生物膜形成、致病性、移动元件含量、氧需求及氧化胁迫耐受等差异均无统计学意义(P>0.05),见表3。

表3 2组儿童肠道菌群表型特征预测分析[M(P25,P75)]
2.8 菌群功能预测分析
ASD组菌群的细胞内吞、鞘糖脂生物合成-乳糖和新乳糖系列代谢途径的丰度较TD组高(P值分别为0.031、0.018);而ASD组非同源末端连接代谢途径的丰度较TD组低(P=0.009)。
为了进一步发掘样本中与肠-脑轴模块相关的代谢信息,将测序数据基于KEGG数据库进行基因注释,并将注释信息转化为肠道代谢模块和肠-脑轴模块的分析结果。肠道代谢模块分析显示,ASD组菌群的谷氨酸降解Ⅰ(MF0030)、色氨酸降解(MF0025)、异化性硝酸盐还原(MF0101)、丙酮酸脱氢酶复合体(MF0072)、甘油降解Ⅱ(MF0061)及丁酸盐生成Ⅱ(MF0089)等代谢功能的丰度较TD组低,差异有统计学意义(P值分别为0.002、0.008、0.029、0.036、0.042、0.046)。肠-脑轴模块分析显示,ASD组菌群的谷氨酸降解Ⅰ(MGB050)、色氨酸降解(MGB049)、多巴胺降解(MGB023)及丁酸盐生成Ⅱ(MGB053)等代谢功能的丰度较TD组低(P值分别为0.002、0.008、0.022、0.046),γ-氨 基 丁 酸 降 解(MGB019)功能的丰度较TD组高(P=0.006)。见图7~8。
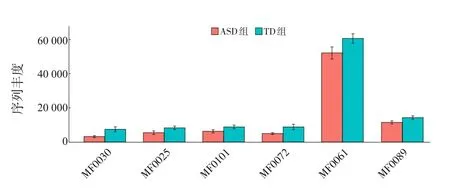
图7 肠道代谢模块菌群功能预测 MF0030:谷氨酸降解Ⅰ;MF0025:色氨酸降解;MF0101:异化性硝酸盐还原;MF0072:丙酮酸脱氢酶复合体;MF0061:甘油降解Ⅱ;MF0089:丁酸盐生成Ⅱ。ASD组和TD组各指标的比较均P<0.05。

图8 肠-脑轴模块菌群功能预测 MGB050:谷氨酸降解Ⅰ;MGB019:γ-氨基丁酸降解;MGB049:色氨酸降解;MGB023:多巴胺降解;MGB053:丁酸盐生成Ⅱ。ASD组和TD组各指标的比较均P<0.05。
3 讨论
肠道菌群是人体肠道微生态系统的主要成分,数量约为人体细胞总数的10倍,被称为人体的“第二基因组”[8]。肠道菌群参与人体营养物质代谢、维生素合成、肠道黏膜生物屏障形成、免疫系统成熟及免疫应答调节等多种生理活动,进而影响人体多系统功能[9]。研究表明,肠道菌群可能对脑功能有重要影响,并且与ASD密切相关[10]。
多数相关研究表明,ASD儿童肠道菌群的α多样性较健康儿童低[4]。而本研究则显示,2组样本的α多样性指数Chao1、Shannon及Simpson比较差异均无统计学意义,可能与样本数量少有关。本研究发现2组样本菌群结构存在差异,属水平共发现6个差异显著物种,分别为巨单胞菌属、巴恩斯氏菌属、小杆菌属、巨球菌属、瘤胃球菌属扭链群及梭杆菌属。
关于ASD儿童肠道巨单胞菌属含量的研究报道并不一致,部分研究结果相反[11]。Zou等[12]发表ASD儿童肠道巨单胞菌属的含量增多,与本研究相同。巨单胞菌属在肠道可以将葡萄糖发酵成乙酸和丙酸,用于合成脂质和胆固醇,也是宿主的能量来源之一[13]。此外,该菌属在肥胖者肠道中含量增多[14]。赵锐豪等[15]发现巴恩斯氏菌属是ASD儿童肠道的优势菌群之一,与本研究结果相似。另一项研究表明,相较于TD儿童,合并便秘的ASD儿童肠道巴恩斯氏菌属的丰度升高[16]。也有研究显示,ASD儿童肠道巴恩斯氏菌属的平均丰度降低[17]。巴恩斯氏菌属可竞争性抑制人体肠道致病菌,并与免疫调节及炎症性疾病相关[18-20]。但该菌属与ASD的具体相关性仍不清楚,需进一步探索。研究显示ASD患者肠道中小杆菌属的含量降低[11]。而本研究结果却与之相反,这可能与采用的统计方法不同或本研究样本量小有关。研究表明,对食物过敏的儿童肠道小杆菌属的含量降低[21]。因此,研究ASD与小杆菌属之间关系时尚需考虑过敏因素。研究显示ASD儿童肠道巨球菌属的含量增多[11],与本研究结果相同。而一项病例对照研究则显示,ASD男孩肠道中巨球菌属的丰度降低[22]。ASD儿童肠道巨球菌属的丰度是否受性别影响需进一步验证。巨球菌属可利用乳酸产生丁酸等短链脂肪酸[23]。此外,肥胖者肠道中该菌属的含量增多[24]。然而,巨球菌属是否参与ASD的病理过程仍未可知。本研究中,与TD儿童相比,ASD儿童肠道瘤胃球菌属扭链群的相对丰度升高,这与Ahmed等[25]研究结果相似。一项前瞻性研究显示,早期儿童肠道扭链瘤胃球菌的相对丰度与ASD儿童社交反应量表-2得分呈负相关[26]。可见,扭链瘤胃球菌与ASD具有相关性。本研究显示,ASD儿童肠道梭杆菌属的相对丰度升高。有研究表明,相较于TD儿童,有便秘症状的ASD儿童肠道梭杆菌属含量增多[15];母体免疫激活子代大鼠的肠道梭杆菌属丰度升高,并与重复行为和焦虑样症状相关[27]。由此可见,ASD可能与肠道梭杆菌属之间存在相关性。
尽管多数相关研究均认为ASD与肠道菌群密切相关,但进一步探索二者之间因果关系的研究却很少。相关研究多倾向于认为肠道菌群异常可能参与了ASD的病理过程,虽然具体机制尚不明确,但目前较统一的认识是基于MGBA理论。这一理论认为,微生物及其代谢产物可通过神经内分泌、神经免疫及神经通路等途径影响脑功能[28]。结合本研究,我们不仅发现ASD儿童肠道菌群结构上的异常,而且通过功能预测分析还发现,ASD儿童肠道菌群的代谢功能与TD儿童相比存在显著差异,如丁酸盐生成,色氨酸、谷氨酸及γ-氨基丁酸等活性物质的降解功能等,而这些物质均对脑功能有重要影响。因此,肠道菌群异常可能通过MGBA介导神经炎症、免疫异常及神经递质紊乱等病理过程,进而参与ASD的发生与发展。
本研究采用了高通量16S rDNA测序技术,创新之处在于使用了去噪方法获得ASVs,基于ASVs的物种注释相较于以往的OTUs聚类更加准确,且与其他研究的ASVs数据结果进行比较时,更具可比性和一致性。本研究也存在不足之处:首先,样本量相对较小,针对相关因素进行多组分析较困难;其次,未对血液及粪便代谢组学进行检测分析,以验证基于扩增子测序对肠道菌群代谢功能预测的结果。期望以后的研究采用大样本、考虑多因素并使用代谢组学等更完善的研究方法更加深入地探究肠道菌群与ASD的关系。
综上所述,ASD儿童和TD儿童肠道菌群的构成存在显著差异,临床实践中通过调节肠道菌群结构以改善微生态可能有助于ASD儿童获得更好的预后;预测2组儿童肠道菌群的代谢功能存在显著不同,其中肠道菌群的丁酸盐生成、谷氨酸及γ-氨基丁酸降解等代谢功能可能是未来相关研究需关注的重点。
利益冲突声明:所有作者均声明不存在利益冲突。
