影像组学及分析工具浅谈
2022-12-02王丽嘉
刘 鹏,王丽嘉,马 超
人工智能(artificial intelligence,AI)和医学成像技术发展迅速,使得医学图像可转换为适合深度挖掘的高维数据[1],影像组学(radiomics)应运而生,成为AI 领域的重要组成部分[2]。作为医工交叉的产物,影像组学有着广阔的临床应用前景,笔者就影像组学概念及相关分析工具等展开综述。
1 影像组学概念
影像组学属于计算机辅助诊断 (computer-aided diagnosis,CAD)的范畴。2012年由荷兰学者Lambin P等[3]提出,当时主要使用计算机体层摄影术(computed tomography,CT)数据。近几年影像组学的概念不断被完善,数据从CT 扩展到磁共振成像(magnetic resonance imaging,MRI)、超声(ultrasound,US)等[4],即高通量地从影像图像中提取并分析大量的影像学定量特征。影像组学从医学图像中提取特征,通过统计学方法建立用于疾病诊断、预后预测、疗效评价的模型,以指导治疗方式的选择、实现个性化和精准化治疗[5,6]。近年来,影像组学已用于多种疾病研究,如胶质瘤[7]、肺癌[8]、乳腺癌[9]、肝肿瘤[10]、胰腺肿瘤[11]、前列腺癌[12]等。
2 影像组学分析流程
影像组学分析步骤包括获取影像数据、感兴趣区(region of interest,ROI)分割、特征提取、特征选择、模型建立与结果分析。
2.1 获取影像数据
影像组学可用于CT、MRI、正电子发射计算机体层摄影术(positron emission tomography,PET)、X 射线和US 等多种模态影像数据[4],研究者要根据临床需求选择合适的成像方式。临床使用的影像设备种类繁多,图像采集和重建缺乏跨中心的标准协议,图像的信噪比、对比度和空间分辨率等会影响形状、直方图、纹理和高阶特征[13]。基于CT 研究发现体素大小对图像特征的可重复性影响显著,尤其是层厚,而管电压、管电流等对图像特征的可重复性影响较小,推荐使用1.0~2.5 mm 层厚的CT 图像进行影像组学分析。Dercle L 等[14]统计了161 项影像组学研究,发现多数PET-CT 研究使用18F-氟代脱氧葡萄糖 (18F-fluorodeoxyglucose,18F-FDG)作为示踪剂;40%MRI 研究中除了T1和T2序列外,还使用扩散加权成像(diffusion weighted imaging,DWI),患者数量的中位数为64,仅少数研究(9%)是多中心的。研究者可采用大队列数据进行影像组学研究,以降低成像参数引起的误差,也可使用相同参数影像数据的小队列开展相关研究[15]。
2.2 感兴趣区分割
ROI 分割是影像组学的关键步骤之一,需要较高的可重复率和准确度,因为影像组学特征主要是从ROI 中提取的。有手动分割、半自动分割和全自动分割3 种方式,手动分割精度高但耗时;半自动分割方法通过人机交互提高了分割速度,对边界模糊的区域可以通过人工二次矫正以提高分割的精度;全自动分割由计算机独立完成,重复性好、耗时短且工作量小,但对于边界模糊的肿瘤或病变的高精确度分割较为困难[16],目前尚无通用的肿瘤或病变的自动分割工具。传统自动分割方法有基于弱先验知识的阈值、区域生长和边缘检测算法等,基于强先验知识的可变形模型、多图谱等[17]。深度学习在自动分割中展示出优于传统方法的性能,常用的图像分割网络框架包括全卷积网络 (fully convolutional networks,FCN)、U 型网络(U-net)等,已应用于脑[18]、肺[19]、胰腺[20]、前列腺[21]和多器官[22]图像的分割。
2.3 特征提取
常用影像组学特征有4 类:形态特征,一阶统计特征,纹理特征,基于滤波或变换的特征。形态特征用于描述肿瘤的几何形态,包括形状和大小等;一阶统计特征用来分析ROI 内的体素强度分布;纹理特征用来描绘图像中灰度值的空间分布关系,量化肿瘤异质性,需要先提取图像纹理特性,保存到变换矩阵(例如灰度共生矩阵)后再计算相应特征;基于滤波或变换的特征是通过对原图进行变换后再提取变换图像的灰度或纹理特征,常用变换方法有小波变换、高斯滤波等。见表1。
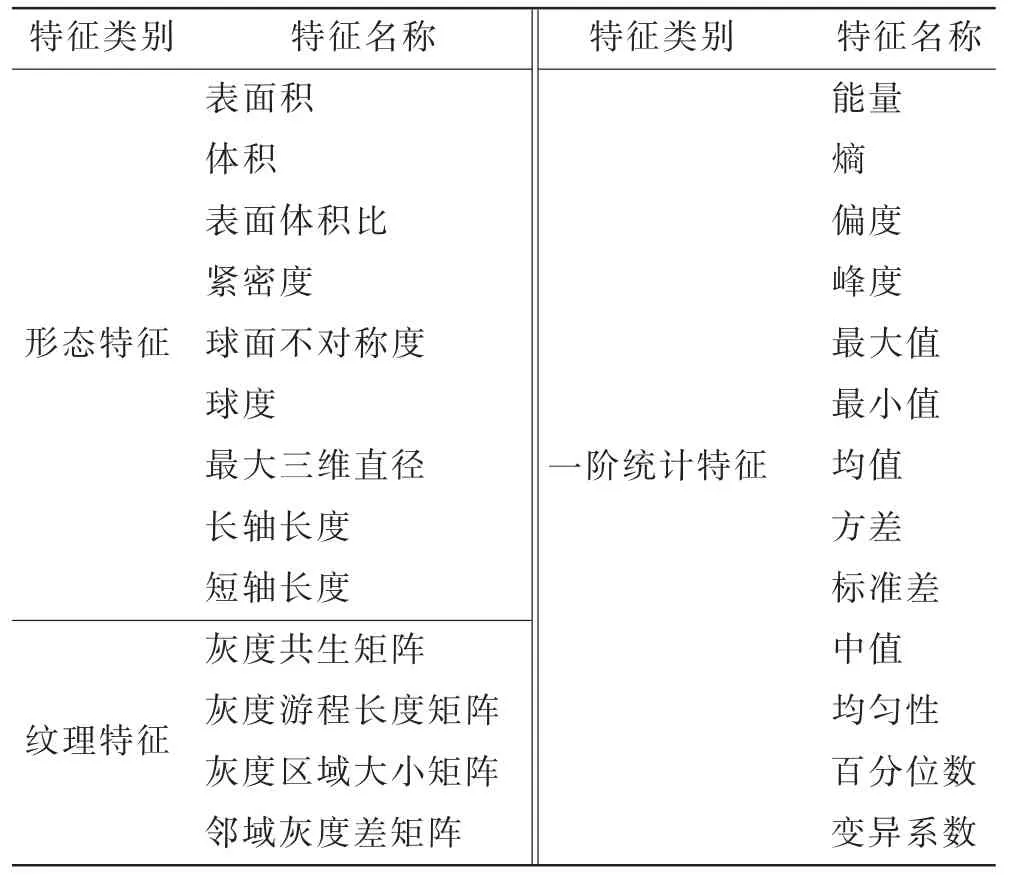
表1 常用影像组学特征Tab.1 Features of radiomics
国际成像生物标记物标准化倡议小组(Image Biomarker Standardization Initiative,IBSI) 提出了11种特征174 个参数作为影像组学特征提取的参考[23](表2)。纹理特征没有明确标准,不同的研究内容所需的图像处理方式多样,研究人员可以根据研究需要进行图像变换,探究不同变换下特征对研究目标的影响。
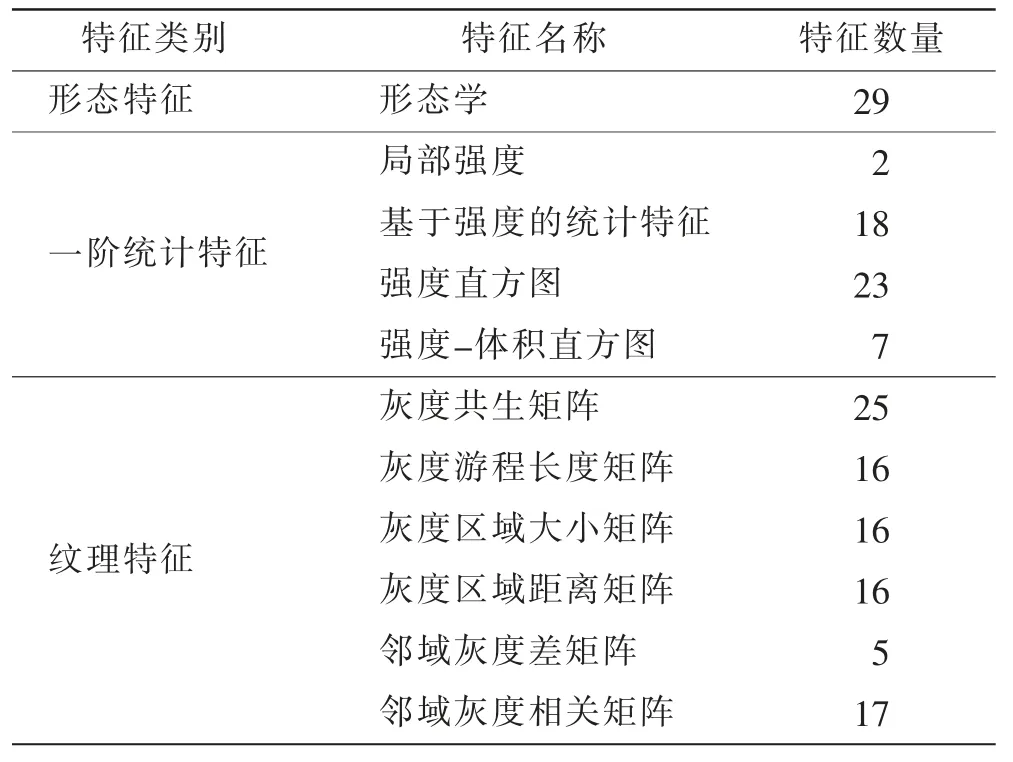
表2 IBSI 标准化特征Tab.2 Standardization features of IBSI
2.4 特征选择
影像组学提取的特征数量较多,样本量有限时会导致预测模型出现过拟合并增加特征分析和模型训练的时间,因此需要对特征进行降维以获取与结果相关性最大、彼此相关性最小的最优特征子集。根据数据具有的标签(ground truth),特征选择可以分为有监督(有标签)、无监督(无标签)和半监督(部分标签)3种方式[24](表3)。Parmar C 等[25]评估了14 种基于滤波的特征选择方法对两组肺癌患者的预测能力,其中Wilcoxon 检验法预测结果最好。在监督法中,嵌入式可以同时实现特征选择和模型构建,常用的如最小绝对收敛和选择算子(least absolute shrinkage and selection operator,LASSO)[26],封装式根据不同特征子集得到的模型性能选择特征;特征选择后需要重新构建模型,常用的有回归特征消除(recursive feature elimination,RFE)等。在实际研究中,获得完整的标签数据较为困难,此时可以同时使用有标签和无标签的数据进行特征选择,即半监督方法[27]。无监督方法常用的有主成分分析 (principal components analysis,PCA)、聚类等;在实际应用中,研究人员可以通过试验不同方法以获得最佳结果。

表3 常用的特征选择方法Tab.3 Methods of features selection
2.5 模型建立与结果分析
影像组学通常使用监督学习算法来构建模型,即通过分析特征分布,建立从特征到目标问题的映射模型,以达到研究目的[28]。模型构建方法的选择通常是根据研究任务及研究人员的经验,在模型的预测能力和解释能力两个方面进行权衡,最终选择适合目标问题的方法进行建模。Parmar C 等[25]评估了12 种分类方法对两组肺癌患者的预测能力,其中随机森林分类器结合大多数特征选择技术的表现最好,贝叶斯模型最稳定,而在临床研究中,通常对建立的模型有性能好、稳定、可复现性高的要求。
机器学习模型通常可以分为分类模型和回归模型。在分类任务中,可选择逻辑回归进行疾病的二分类;支持向量机求解划分数据集;随机森林分类器利用多个决策树进行分类,通过统计每个决策树的结果,选择投票数最多的结果作为其最终结果。在生存分析任务中,可选择比例风险回归模型(cox proportional hazards model,Cox),该模型能同时分析众多因素对生存期的影响。
除了常用于分类回归的模型外,深度学习也是重要方法,其使用多次线性加权和非线性激活函数的组合来对特征进行映射,得到目标问题。卷积神经网络(convolutional neural network,CNN)可从图像中学习特征并进行预测,不需要特征选择。深度学习也分为监督、半监督和无监督3 种方式,研究人员可自行设计CNN,也可以使用现有的网络,如视觉几何群网络 (visual geometry group network,VGG)[29]、残差网络(residual network,ResNet)[30]等,根据数据大小,可以选择固定参数或使用迁移学习对网络参数微调。
影像组学的目标是得到一个高精度和高效率的预测模型,模型计算结果和泛化性等需要通过性能指标来衡量,因此需要数据来训练和验证模型(内部验证),通常使用交叉验证的方法优化模型,也可提供其他来源数据评估模型的准确度(外部验证)。在分类任务中,性能指标包括受试者工作特性(receiver operating characteristic,ROC)曲线和曲线下面积(area under curve,AUC)等,其中AUC 越接近1 说明结果越好。也可使用临床相关指标,如准确度、灵敏度、特异度等。对于预测风险比和生存曲线等任务,主要衡量一致性指数,即比较观察结果与预测结果之间的一致性,越接近1 说明结果越好。生存分析还可绘制Kaplan-Meier(KM)曲线,通过对数秩检测(Log-rank test)等方法进行评估[31]。
3 影像组学分析工具
多个研究小组开发了软件/工具包用于影像组学研究(表4)。ITK-SNAP 是一款医学图像处理软件,能对数据可视化和分割,支持多种格式的数据,如医学数字成像与通信 (digital imaging and communications in medicine,DICOM)、神经成像信息技术倡议(neuroimaging informatics technology initiative,NiFTI)、近原始光栅数据(nearly raw raster data,NRRD)、可视化工具箱(visualization Tool kit,VTK)等,对计算机配置要求较低,软件提供了手动和自动分割方式。自动化方法使用的是可调参数的活动轮廓方法,在冠状面、矢状面及横断面三个方向上进行分割并可以三维显示。三维slicer 可在Linux、MacOSX 和Windows 等多种操作系统上使用,支持DICOM、NiFTI、NRRD、VTK、位图(Bitmap,BMP)等格式;同样支持三个方向的分割,可实现配准及二维、三维和四维数据集的可视化功能,其半自动分割方法有追踪相同强度值、区域生长、选定阈值等;其扩展的PyRadiomics 包可用于图像特征的提取。影像标记物开发软件(imaging biomarker explorer,IBEX) 使用MATLAB 和C/C++编程语言开发,可以在Windows 系统和1 GB 内存的计算机上运行,支持DICOM 和Pinnacle 数据,能构建特征集,方便用户进行特征提取,支持在三个方向上的分割,但不能显示三维结果。LIFEx 可以从PET、单光子发射计算机体层摄影术 (single-photon emission computed tomography,SPECT)、MRI、CT 和US 图像或任何成像模式的组合中计算纹理和形状特征,使用该软件不需要任何编程技能,适用于Windows、Linux 和Mac 操作系统,支持DICOM、NiFTI、联合图像专家组(joint photographic experts group,JPEG)、标签图像文件格式(tag image file format,TIFF)、可移植网络图形格式(portable network graphic format,PNG)和BMP 等格式的数据。MaZda 是一个广泛用于图像分析任务的二维和三维图像纹理分析软件,包括简单的轮廓工具和一些用于特征降维及分析的工具。长庚图像纹理分析工具(Chang Gung image texture analysis,CGITA)是长庚纪念医院研究人员基于MATLAB 开发的纹理分析软件,可用于分子影像、CT、MRI 数据的分析。PyRadiomics 是一个开源python 包,可扩展到其他软件,旨在为影像组学分析建立一个参考标准,并提供一个经过测试和维护的易于重复的特征提取平台。特征探索者(FeAture Explorer,FAE)由华东师范大学上海市磁共振重点实验室开发,可用于影像组学特征提取及模型参数的建立,支持NiFTI 格式文件,其提供了一个超参数探索平台,包括多种数据处理方法及模型建立方法,方便研究人员横向比较不同方法在处理特定数据时的效果。

表4 影像组学分析开源软件/工具包Tab.4 Open source softwares/toolkits of radiomics analysis
3.1 分割工具
选择分割工具首先要确定软件支持的数据格式和ROI 保存格式,确保数据的成功导入和保存;其次,软件界面应简洁友好,以便用户能快速正确地进行相应操作;另外,软件对计算机的硬件配置需求也在考虑范围内[40]。分割工具使用流程如图1所示。
ITK-SNAP 是应用广泛的ROI 分割软件,对计算机要求较低,操作简便,且支持保存的格式较多,图2展示了ROI 分割具体操作流程。
3.2 特征提取工具
影像组学特征提取时首先要确定特征数量及类别,选择特征提取工具时应考虑是否能导入相应的图像和ROI 格式,还要注意结果的保存格式和对计算机的配置要求等。不同软件之间特征的命名及计算方式不尽相同,即便在使用同一软件进行特征提取时,研究人员也应确认其使用的参数是否相同。
FAE 兼具特征提取及建模工具,包括特征提取、数据清洗、模型建立和结果描述4 个模块。图3展示了其特征提取功能,在导入数据前,每例数据需要把原始图像和ROI 图像放在一个单独文件夹中。FAE 可选择图像的变换及特征类型,结果保存为数据表格形式,进一步分析前需手动添加标签信息来标记临床结论或分类。
3.3 建模及数据分析
影像组学提取的特征较多,但并不是所有提取的特征都对研究任务有意义,因此数据分析和任务模型建立是重要的步骤。FAE 软件有多种数据处理和建模方法可供选择,图4为模型建立模块,FAE 对不同的建模方法进行组合以横向比较各方法的效果,建模结果保存为字符分隔值 (comma-separated values,csv)格式文件。
3.4 结果展示和分析
模型结果的好坏需要通过性能指标来衡量。图5展示了FAE 对分类模型结果的描述,以表格方式显示了不同模型在训练集、验证集和独立测试集上的临床指标(如AUC、灵敏度、特异度等),并展示了ROC曲线及不同模型的AUC 比较,以及特定模型的特征贡献程度。同时FAE 以csv 格式保存了模型建立过程中的所有中间结果,从而允许研究者进行后续的分析。
4 影像组学质量评估及挑战
统一的评价标准和报告准则可以有效地提高影像组学模型的质量和准确度。Lambin P 等[6]提出了影像组学质量评分(radiomics quality score,RQS)来评估研究结果,其包含16 个评分准则,满分36 分。目前有多个组织正努力构建影像组学研究的标准化,如前文所述的IBSI 为影像组学特征提供标准定义、命名和计算方法、报告指南等。另外有定量成像网络[41]、北美放射学会、定量成像生物标志物联盟[42]、中国医学影像AI 产学研用创新联盟等都致力于建立标准化成像协议。
影像组学研究增长显著,大多数影像组学的研究结果并没有得到充分验证,患者人群的普适性不高[43],其数据来源多为单一机构且样本数量较少,缺乏有效的外部验证。量大高质的数据集和共享对提高研究的有效性有巨大帮助,也是影像组学研究需要努力的方向[44]。
对于回顾性影像组学研究,需要进行独立的外部验证,以保证结果的准确度;在前瞻性影像组学研究中,研究者在试验设计时需制定完善和统一的影像采集标准和临床数据搜集方案,以获得高质量的图像和临床信息,避免因设计缺陷或人工失误带来的数据损失,提高处理效率,推荐使用标准化的电子病历系统和结构化报告等,以建立数据间的有效链接和数据的深度挖掘。应加强学术交流,建立多中心间的合作,推动标准化成像协议发展,促进数据共享、研究过程及结果共享,以便于影像组学模型的对比和改进。
5 总结与展望
笔者介绍了影像组学的分析流程和相关软件,旨在使研究人员能便捷地开展影像组学研究,还有许多影像组学分析软件没有涉及,且每个软件都有各自的优缺点,研究人员应根据实际需求进行权衡和选择。影像组学具有广阔的临床应用前景,临床医生和影像学家应共同合作,快速掌握分析方法。当然,有条件的小组或实验室也可以招募一些计算机领域或生物医学工程背景的专业人员进行相关软件的开发等,以提高模型的鲁棒性,不断为实现精准医疗和临床实践而努力。
