植物免疫激酶AtCERK1的重组表达与结晶研究
2022-11-16牛成群杨芷茜明振华
牛成群,杨芷茜,林 洁,明振华
(广西大学生命科学与技术学院亚热带农业生物资源保护与利用国家重点实验室广西甘蔗生物学重点实验室,中国广西 南宁 530004)
为抵御自然界病原微生物的侵染,植物在漫长进化过程中,形成了特有的两道免疫防线[1]。第一道免疫防线是病原体相关分子模式(pathogen associated molecular pattern,PAMP)触发的免疫反应(PAMP-triggered immunity,PTI)[2],该免疫反应一般是通过位于植物细胞表面的模式识别受体(pattern recognition receptor,PRR)识别PAMPs,并将免疫信号传递至细胞内[3~5],最终引起活性氧(reactive oxygen species,ROS)含量上升、丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)的磷酸化、内质网膜上离子通道的激活、胼胝质的沉积、生长抑制和防御相关基因的表达等免疫相关反应[6];第二道免疫防线是病原微生物逃逸PTI过程中产生的效应因子所触发的免疫反应(effectortriggered immunity,ETI)。触发该免疫反应的是病原微生物向宿主植物细胞注入的毒力因子(virulence factors),也被称为效应因子(effector molecules),这类毒力因子通过对PTI途径的关键信号组分进行修饰、降解、结合或者活性调控,抑制植物的PTI[7]。毒力因子对宿主细胞的破坏作用如果被定位在植物细胞内的抗性蛋白(R蛋白)所感知,一般会引起剧烈的免疫防御反应(通常伴随超敏反应)[8~9]。
CERK1(chitin elicitor receptor kinase 1)是植物细胞膜上存在的一种类受体激酶(receptor-like kinase,RLK),因其可以感知真菌细胞壁含有的几丁质并磷酸化下游蛋白质引发免疫反应而被发现并命名,在植物感知并抵御真菌侵染方面有重要作用[10~13]。同时,CERK1在介导植物与共生真菌互作过程中发挥关键作用[14]。CERK1在拟南芥、水稻、人、小鼠等物种内广泛存在,并具有较高的同源性[15]。CERK1识别的PAMPs或共生因子包括真菌几丁质、细菌肽聚糖、真菌线性β-1,3-葡聚糖和菌根真菌的共生因子等微生物来源的N-乙酰葡萄糖胺多聚物[16~17]。根据蛋白质与细胞膜的相对位置,CERK1可分为3个组成部分:细胞外负责感知PAMPs的LysM结构域[18]、跨膜疏水性结构域、细胞内负责磷酸化下游蛋白质的丝氨酸/苏氨酸激酶区(kinase domin,KD)结构域[19]。在拟南芥中,AtCERK1胞外结构域一般通过组成同源二聚体[20],或和LYK5胞外区组成异源二聚体来感知PAMPs[21]。AtCERK1胞外LysM结构域感知真菌几丁质这一PAMP后[22],引发胞内KD区的自磷酸化及其对PBL27蛋白的转磷酸化,随后PBL27磷酸化MAPKKK5(MAPK kinase kinase 5),引起级联免疫反应[23]。鉴于AtCERK1在识别和感知真菌几丁质、将免疫信号传递至胞内并触发PTI免疫反应中的重要性,一些病原菌通过分泌毒力因子攻击AtCERK1途径以实现免疫逃逸。例如,细菌Ⅲ型效应蛋白AvrPtoB可以特异识别并泛素化AtCERK1的KD区,进而靶向降解AtCERK1,使植物丧失由AtCERK1诱导引起PTI的能力[24]。目前,CERK1胞外感知几丁质的机制已经比较清楚,但其如何发挥下游信号传递的功能及如何被AvrP-toB特异性修饰有待进一步探索,故CERK1胞内KD区的结构解析显得尤为关键。
本课题旨在利用晶体学手段对AtCERK1胞内KD区进行晶体筛选以获得高分辨率的蛋白质结构。实验成功构建了AtCERK1的体外原核表达系统,并经纯化获得纯度为95%以上的野生型及突变型蛋白质样品,成功筛选到可以结晶的条件,经X射线检测收集,获得分辨率为3.2 Å的AtCERK1和ADP复合物的衍射数据,为该蛋白质进一步的结构与功能研究进行了实验探索。
1 材料与方法
1.1 材料
AtCERK1胞内区原核表达载体pRSFDuet1-CERK1为实验室构建;大肠杆菌BL21感受态购于北京全式金生物技术股份有限公司;LB培养基、卡那霉素(kanamycin,Kana)、诱导剂异丙基硫代-β-D-半乳糖苷(isopropylthio-β-D-galactoside,IPTG)购于北京索莱宝科技有限公司;质粒小提试剂盒、胶回收试剂盒购于上海碧云天生物技术有限公司;蛋白质纯化相关试剂:镍柱(Ni NTA beads)、阴离子交换柱(Capto Q)和Superdex75分子筛,购于GE Healthcare公司(美国);蛋白质结晶相关试剂:结晶试剂盒(包括 Index、Crystal screen1&2、Sa-ltRx1&2、Natrix1&2、PEGRx1&2 和 PEGion1&2)购于Hampton Research公司(美国),防冻液相关试剂乙二醇(ethylene glycol,EG)、聚乙二醇(polyethylene glycol,PEG)和NaCOOH购于Sigma公司(美国)。
1.2 溶液的配制
AtCERK1纯化缓冲液:称取2.423 g三羟甲基氨基甲烷(trihydroxymethyl aminomethane,Tris)、0.681 g咪唑和8.766 g NaCl于Ⅰ级水中,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L,过滤除菌后4℃储存备用(Buffer 1);称取8.000 g NaCl、0.200 g KCl、1.440 g Na2HPO4、0.240 g KH2PO4和0.681 g咪唑,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L(Buffer 2,即PBS缓冲液);称取2.423 g Tris、20.424 g咪唑和8.766 g NaCl于Ⅰ级水中,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L,过滤除菌后4℃储存备用(Buffer 3,即洗脱缓冲液)。
AtCERK1离子交换缓冲液:称取2.423 g Tris于Ⅰ级水中,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L,过滤除菌后4℃储存备用(Buffer A);称取2.423 g Tris、58.440 g NaCl于Ⅰ级水中,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L,过滤除菌后4℃储存备用(Buffer B)。
AtCERK1凝胶过滤缓冲液:称取2.423 g Tris、0.406 g MgSO4·12H2O 和 8.766 g NaCl于Ⅰ级水中,用HCl调节pH到8.0,随后用Ⅰ级水定容至1 L,过滤除菌后4℃储存备用(Buffer C)。
1.3 生物信息学分析
拟南芥CERK1(AtCERK1)蛋白的CDS序列(NP_566689.2)从NCBI上查找并下载得到。使用TMHMM Server v2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0;2020-09-11)在线网站对蛋白质的跨膜结构域进行预测以确定蛋白质边界。使用Phyre 2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index;2020-09-11)在线网站对AtCERK1的二级结构进行预测。利用ExPASy网站的在线工具ProtParam(https://web.expasy.org/protparam/;2020-09-18)预测AtCERK1的等电点(isoelectric point,PI)、相对分子质量等理化性质。利用HHpred(https://toolkit.tuebingen.mpg.de/tools/hhpred;2020-11-20)在线网站搜索PDB数据库里已经解析出的同源性高的蛋白质结构用于分子置换结构解析。
1.4 AtCERK1的野生型及突变型构建
经由PCR获得AtCERK1(301~599)的目的基因(引物序列见表1),PCR产物采用1%的琼脂糖凝胶进行电泳,随后经胶回收分离纯化出所需目的基因片段。用EcoRⅠ和XhoⅠ分别对目的基因和pRSFDuet1载体进行双酶切,酶切产物采用1%的琼脂糖凝胶进行电泳,分别分离纯化出酶切后具有相同黏性末端的AtCERK1(301~599)和pRSFDuet1片段。用T4 DNA连接酶于16℃进行连接,2 h后连接产物转入DH5α感受态细胞中,冰上静置30 min,经42℃热激90 s后再次静置于冰上5 min,37℃、210 r/min培养1 h后涂于含Kana抗性的LB固体平板上,倒置放入37℃恒温培养箱,等待14~16 h。挑取单克隆于含Kana的LB液体培养基中,37℃培养6~8 h,用质粒小提试剂盒提取质粒,用EcoRⅠ和XhoⅠ快切酶进行双酶切验证,取阳性样本送到广州睿博生物科技有限公司进行测序。

表1 质粒构建所需的引物Table 1 Primers used for construction of plasmids
基于重叠延伸PCR技术进行点突变扩增,获得未甲基化的突变重组质粒AtCERK1F460V和At-CERK1K349R。用DpnⅠ酶(北京全式金生物技术股份有限公司)消化后,转入DMT感受态(北京全式金生物技术股份有限公司)中,14 h后在LB固体培养板上挑取单克隆,接入LB培养基中培养,提取质粒后送往广州睿博生物科技有限公司进行测序。
1.5 SDS-PAGE检测
配置SDS-PAGE胶,将所制备样品加入上样孔里,120 V恒压电泳1 h,考马斯亮蓝染色15 min,脱色过夜后进行拍照分析。
1.6 野生型AtCERK1蛋白的表达及亲和层析纯化
pRSFDuet1-CERK1质粒转化至感受态细胞E.coli BL21(DE3)后,挑取菌落接种至含Kana(50 μg/mL)的10 mL LB培养基中培养6~8 h(37 ℃、210 r/min),接入1 L含有Kana的LB培养基中,振荡培养至OD600到0.8(37℃、210 r/min)。加入诱导剂IPTG(终浓度0.5 mmol/L),16℃诱导20 h后,蛋白质表达完成。4 000 r/min离心15 min收集菌体,用 Buffer 1(20 mmol/L Tris、150 mmol/L NaCl、10 mmol/L咪唑,pH 8.0)重悬,高压破碎后离心1 h(4℃、12 000 r/min);取上清与镍柱结合进行亲和层析,Buffer 1冲洗30 mL后,取柱上样品R,采用咪唑对纯化的蛋白质进行梯度洗脱(10~70 mmol/L咪唑),并收集梯度洗脱样品E10~E70,再由含300 mmol/L咪唑的洗脱缓冲液(Buffer 3)进行洗脱,同时收集洗脱样品E300。所有样品采用SDSPAGE进行分析。
缓冲液纯化效果比较:蛋白质表达完成后,用Buffer 1和Buffer 2(PBS缓冲液,10 mmol/L咪唑,pH 8.0)分别重悬菌体,高压破碎后离心1 h(4℃、12 000 r/min)。取上清与镍柱结合进行亲和层析,收集柱上样品R,用提高了咪唑浓度(20 mmol/L)的Buffer 1及Buffer 2对镍柱进行洗杂,收集洗下的样品E20。采用SDS-PAGE对收集的样品进行分析。
1.7 AtCERK1的深度纯化
离子交换层析:将用20 mmol/L咪唑洗杂、300 mmol/L咪唑洗脱的初步纯化的AtCERK1蛋白浓缩到2 mL,置于冰上备用。阴离子交换柱(Capto Q)用 Buffer A(20 mmol/L Tris,pH 8.0)、Buffer B(20 mmol/L Tris、1 mol/L NaCl,pH 8.0)平衡,平衡程序为7.5%Buffer B,即用75 mmol/L NaCl平衡。将2 mL浓缩蛋白质和2 mL Buffer A混合进行降盐处理,4℃、12 000 r/min离心10 min,去沉淀后通过5 mL Loop环上样,同时收集样品,等UV(ultraviolet)峰走平后,逐渐升高盐浓度(20 min内从75 mmol/L升高到600 mmol/L),停止收集。取收集的样品进行SDS-PAGE分析。
凝胶过滤层析:将离子交换层析纯化到的蛋白质样品浓缩到1 mL以下,置于冰上备用。分子筛柱(Superdex75)用 Buffer C(20 mmol/L Tris、150 mmol/L NaCl,pH 8.0)平衡。将1 mL浓缩蛋白质样品在4℃、12 000 r/min条件下离心10 min,去沉淀后通过1 mL Loop环上样,并收集样品,待UV峰平稳后,停止收集。取收集的样品进行SDSPAGE分析。将分离纯化的蛋白质进行浓缩,利用Nanodrop One紫外分光光度计进行蛋白质浓度测定。
1.8 突变型AtCERK1蛋白的纯化
AtCERK1突变型的纯化方案参考野生型,用20 mmol/L咪唑竞争性洗杂纯化后,采用含300 mmol/L咪唑的Tris-NaCl缓冲液进行充分洗脱,随后将洗脱蛋白质用Superdex75分子筛纯化柱进行凝胶过滤分选。SDS-PAGE分析后进行蛋白质浓缩。
1.9 AtCERK1野生型及突变型的结晶条件筛选
经纯化得到的野生型及突变型蛋白质均浓缩到10 mg/mL,分别与终浓度为5 mmol/L的ADP或ATP冰上孵育1 h。对每种蛋白质的3种不同状态:单独蛋白质、与ADP的复合物、与ATP的复合物,进行蛋白质晶体初步筛选。晶体初筛采用气象悬滴扩散法进行,采用Hampton Research公司的6×96种晶体学条件:Crystal screen1&2、Index、PEGRx1&2、PEGion1&2、SaltRx1&2、Natrix-1&2,在硅化玻片上加入1 μL蛋白质溶液和1 μL池液(即试剂盒溶液),并将其混合,琼脂密封好后将晶体板放置于16℃,每隔24 h在光学显微镜下观察晶体生长情况。
1.10 晶体优化
有晶体初步析出后,对析出晶体的条件进一步优化,优化方案包括:1)4∶1优化。即把初筛条件中的池液改为4/5的量,加入1/5份其他条件的池液,以研究不同溶质组分对晶体生长的影响;2)梯度对拉优化。即把初筛得到的池液条件中所含的某一溶质成分按照线性梯度进行溶液的配置;3)添加剂及去垢剂优化。将1 μL蛋白质溶液加入1 μL池液,再加入0.2 μL添加剂或去垢剂。优化后每隔24 h观察晶体。
1.11 晶体衍射分析
将晶体在显微镜下用合适直径的带底座晶体环捞取后,放置于提前准备好的防冻液(由原池液基础上添加20%的甘油配置)中,脱水后迅速放入液氮中冻存。在上海同步辐射光源中心(Shanghai Synchrotron Radiation Facility,SSRF)大分子线站BL17U1进行晶体衍射。收集数据后用XDS处理并用软件CCP4以及Phenix进行分子置换以及结构解析。
2 结果
2.1 AtCERK1的生物信息学分析
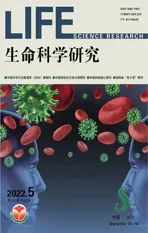
在NCBI上查找到AtCERK1的CDS序列,在TMHMM Server v2.0在线网站上对序列进行跨膜区域分析,从图1的分析结果可知胞内区边界为255~617。结合Phyre2的二级结构预测结果(图2),删除一些可能影响蛋白质结晶的柔性结构域,确定克隆边界为301~599。在ExPASy上预测蛋白质的基本理化性质,得到AtCERK1的基本信息:AtCERK1由299个氨基酸组成,预测相对分子质量为33 kD,pI为6.22,消光系数Abs(0.1%=1 g/L)为 0.802。

图1 AtCERK1全长的跨膜区域预测结果Fig.1 Transmembrane domain prediction of AtCERK1
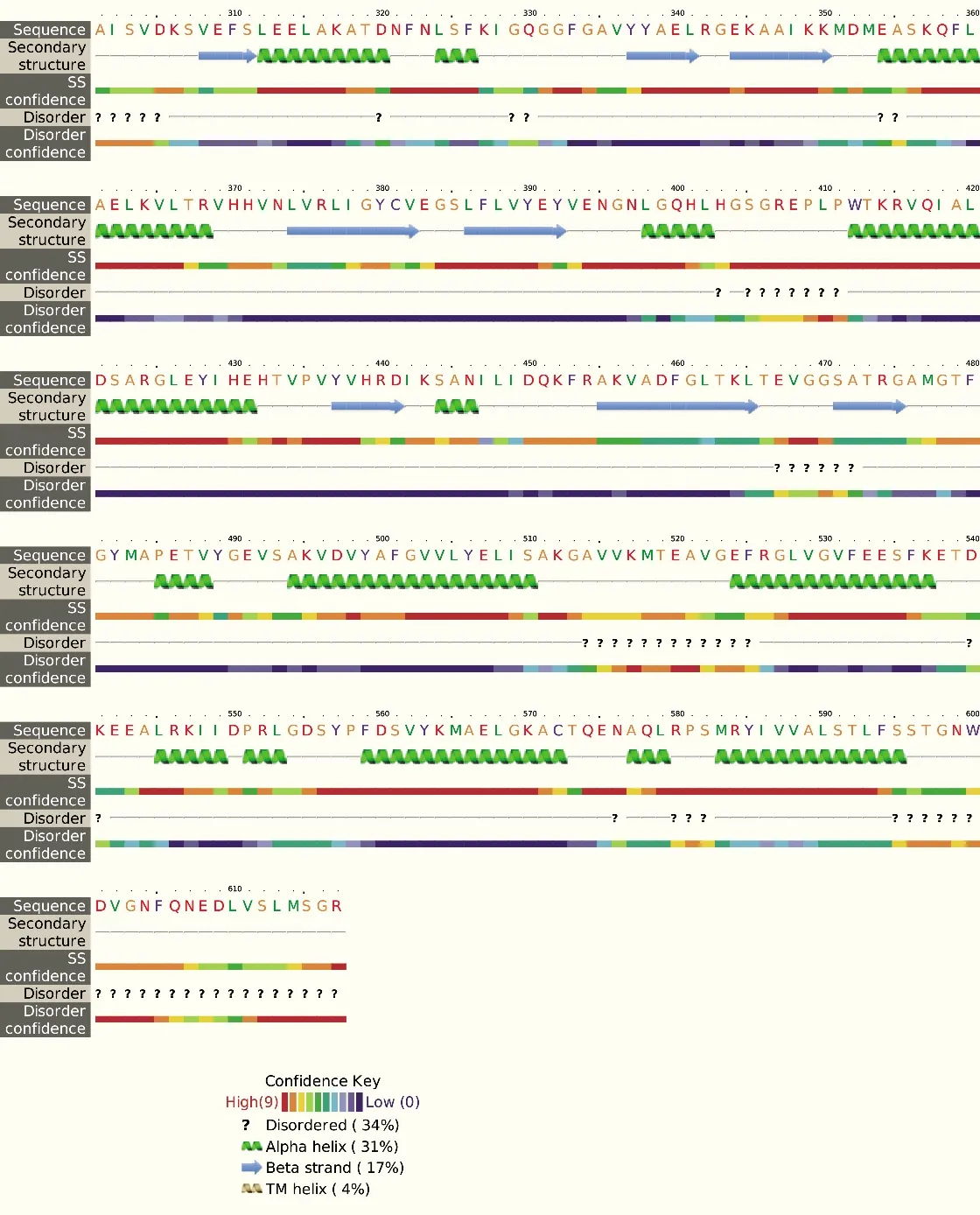
图2 AtCERK1胞内区的二级结构预测结果Fig.2 Secondary structure prediction of AtCERK1 intracellular region
2.2 野生型AtCERK1蛋白的表达纯化
pRSFDuet1载体的融合表达蛋白在其N端带有6个His标签,故使用镍柱亲和层析进行第1轮蛋白质纯化。分别选用Tris-NaCl缓冲液(Buffer 1)以及PBS缓冲液(Buffer 2)对诱导表达的菌体进行重悬,随后进行高压破碎、镍柱亲和层析纯化。取匀浆液中的上清样品(S)、沉淀样品(P),以及用含20 mmol/L咪唑的不同缓冲液洗下的样品(E20)和洗杂后与镍介质结合的样品(R),进行SDSPAGE分析。从图3的电泳结果可知:在PBS缓冲体系(Buffer 2)下纯化出的AtCERK1与镍柱结合得更牢,不会轻易被低浓度咪唑洗脱,但最后留在镍柱上的杂蛋白很多;而Tris-NaCl缓冲体系(Buffer 1)下纯化出的AtCERK1蛋白只有两条主要的杂蛋白需要去除,且与PBS缓冲体系(Buffer 2)相比,最后留在镍柱的AtCERK1蛋白含量相差不大。综合分析后选用Tris-NaCl缓冲液(Buffer 1)进行后续的纯化实验。
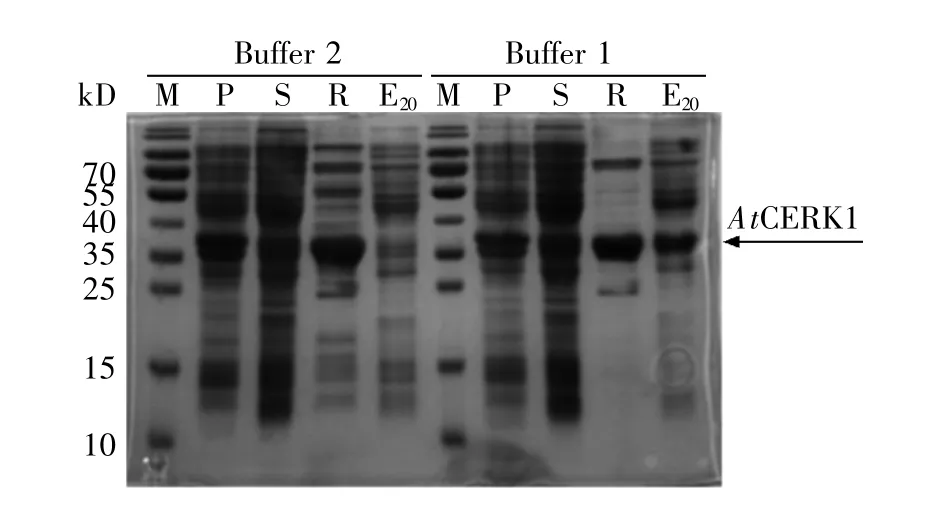
图3 不同缓冲体系对AtCERK1初步纯化的影响M:蛋白质分子量标准;P:沉淀样品;S:上清样品;R:介质样品;Ex:咪唑洗脱样品(x为咪唑浓度)。Fig.3 Effects of different buffers on purification of AtCERK1M:Protein marker;P:Precipitation sample;S:Supernatant sample;R:Medium sample;Ex:Imidazole elution sample(x represents the concentration of imidazole).
确定蛋白质纯化的缓冲体系之后,为进一步探索洗脱蛋白质的咪唑浓度,用不含咪唑的Tris-NaCl缓冲液对诱导表达的菌体进行重悬,并进行高压破碎和离心,上清与镍介质结合后取样品(R),同时,依次用含10~70 mmol/L咪唑的Tris-NaCl溶液洗涤镍柱并收集洗脱样品(E10~E70)。对上述样品进行SDS-PAGE分析,从图4的电泳结果可知:用20 mmol/L咪唑可竞争性洗脱大量杂蛋白。故后期的初步纯化方案为含20 mmol/L咪唑的Tris-NaCl缓冲液洗杂,含300 mmol/L咪唑的Tris-NaCl缓冲液洗脱,这样即得到初步纯化的AtCERK1。

图4 不同咪唑浓度对AtCERK1的纯化效果M:蛋白质分子量标准;R:介质样品;Ex:咪唑洗脱样品(x为咪唑浓度)。Fig.4 Effects of different imidazole concentrations on purification of AtCERK1M:Protein marker;R:Medium sample;Ex:Imidazole elution sample(x represents the concentration of imidazole).
使用离子交换和凝胶过滤层析方法对初步纯化的蛋白质进行进一步纯化。利用阴离子交换柱(Capto Q)进行离子交换层析,纯化结果如图5所示:随着盐浓度的升高(红色曲线为电导值,可表示盐浓度),AtCERK1蛋白先被洗脱下来,随后25 kD左右的一条杂蛋白才被洗脱下来,故离子交换层析处理可去除25 kD的一条主要的杂蛋白。取离子交换层析洗脱的目的蛋白液,浓缩后用Superdex75分子筛进行凝胶过滤层析,分离纯化结果如图6所示:绝大多数的杂蛋白会先从分子筛上洗脱下来,而目的蛋白AtCERK1主带在11.5 mL处才开始被洗脱下来,对称的峰形说明蛋白质比较均一。收集并浓缩AtCERK1的蛋白质溶液,得到高浓度、高纯度的目的蛋白,以用于后续结晶实验。
图5 AtCERK1的离子交换层析(A)离子交换色谱图;(B)对应的蛋白质凝胶电泳图。Fig.5 Ion exchange chromatography of AtCERK1(A)The ion exchange chromatogram;(B)The protein gel electrophoresis diagram.

图6 AtCERK1的凝胶过滤层析(A)凝胶过滤色谱图;(B)对应的蛋白质凝胶电泳图。Fig.6 Size exclusion chromatography of AtCERK1(A)The size exclusion chromatogram;(B)The protein gel electrophoresis diagram.
2.3 AtCERK1突变型的表达纯化
用亲和层析、凝胶过滤层析手段纯化出针对植物免疫激酶的ATP结合位点设计的两个突变型AtCERK1K349R和AtCERK1F460V。纯化结果如图7所示,突变型AtCERK1K349R和AtCERK1F460V的纯度已达到结晶要求,故将其浓缩到10 mg/mL待用。
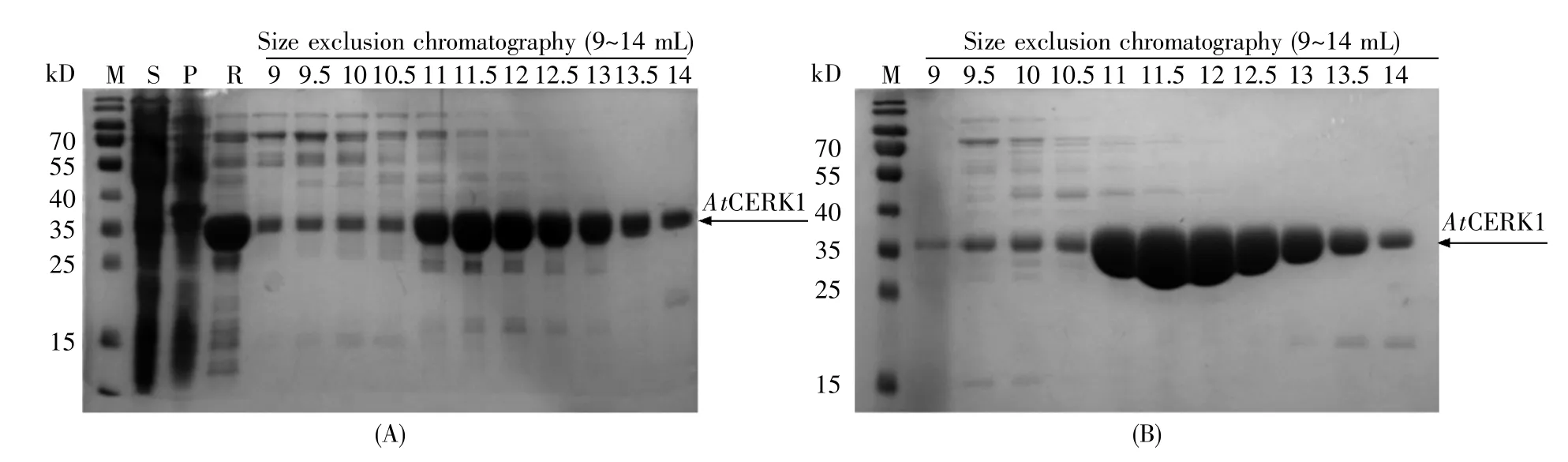
图7 AtCERK1K349R(A)和AtCERK1F460V(B)的纯化结果Fig.7 Purification of AtCERK1K349R(A)and AtCERK1F460V(B)
2.4 AtCERK1野生型和突变型的晶体筛选
除了单独的野生型或突变型蛋白质,At-CERK1、AtCERK1F460V和 AtCERK1K349R还分别与终浓度为5 mmol/L的ATP或ADP冰上孵育1 h,以制备复合体样品。将一共9种样品进行蛋白质晶体初步筛选,在16℃恒温培养3 d后,于显微镜下观察到突变型AtCERK1F460V与ADP的复合物在PEGion1试剂盒的3号条件(0.2 mol/L氟化铵、20%PEG3350)出现蛋白质结晶,晶体呈金刚石状(图 8)。

图8 AtCERK1F460V与ADP复合物的晶体Fig.8 Crystals of the AtCERK1F460V-ADP complex
2.5 AtCERK1F460V复合物晶体的衍射分析
晶体X射线衍射获得的最高衍射分辨率为3.2 Å(图9),具体数据资料见表2。利用HHpred搜索PDB数据库里同源性最高的结构模型作为分子置换模版,可以得到正确的解。但由于晶体衍射分辨率不高,电子密度不理想,未来还需要对晶体筛选条件进行进一步优化。
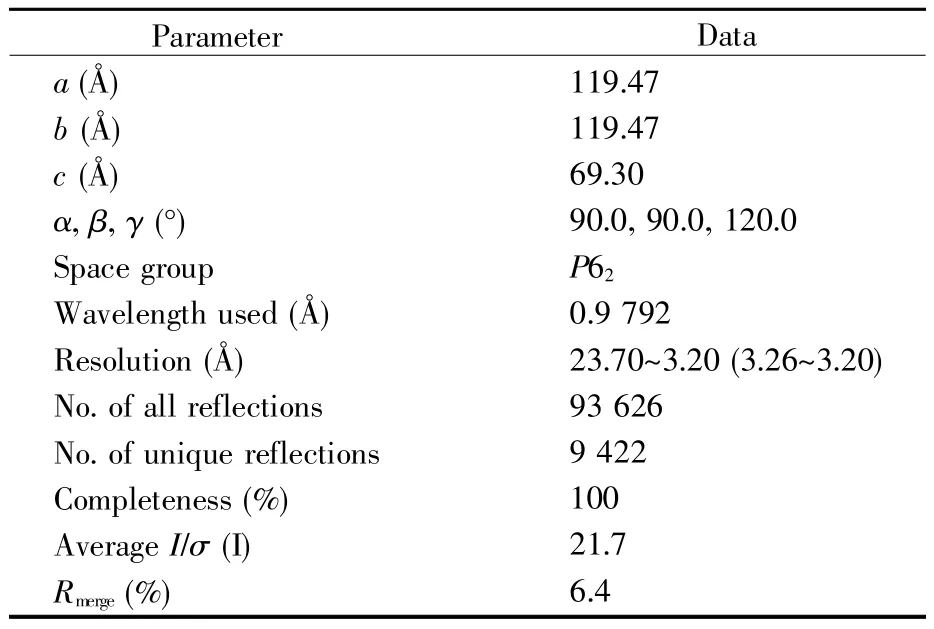
表2 AtCERK1F460V与ADP复合物的X射线衍射数据Table 2 X-ray diffraction data of AtCERK1F460V-ADP complex

图9 AtCERK1F460V与ADP复合物的X射线衍射图谱Fig.9 X-ray diffraction pattern of the AtCERK1F460VADP complex
3 讨论
植物的固有免疫分为PTI和ETI两种类型,对植物生长发育和抗性至关重要。CERK1作为能够特异性识别几丁质类多糖分子并向细胞内传递信号触发PTI免疫反应的模式识别受体,对植物抵御致病真菌和细菌感染至关重要。在拟南芥中,AtCERK1通过胞外LysM区识别几丁质来感知真菌的侵染,然后由胞内激酶区通过磷酸化下游蛋白质PBL27传递信号,再由PBL27磷酸化MAPKKK5引起级联免疫反应[25]。区段交换与嵌合体设计实验表明,胞外区感知信号和胞内区传递信号是两个相对独立的过程[26],但目前AtCERK1胞内信号传递结构域的作用机理尚不清楚,因此,解析AtCERK的胞内激酶区结构,深入分析其信号激活与信号传递的分子机理,十分必要。
为了解析AtCERK1胞内激酶区的三维结构,首先需要对该蛋白质片段进行晶体学研究。本实验针对AtCERK1蛋白的KD区基因片段序列,构建了E.coli-pRSF表达系统和两种关键突变型,最终获得3种纯度在95%以上的蛋白质样品,并通过与ATP底物或ADP产物组装复合体,最终使用气象悬滴扩散法成功获得可以产生X射线衍射的蛋白质晶体。在本研究中,野生型AtCERK1析出的晶体经X射线衍射分辨率很低,在8~10 Å之间。我们猜想可能是受野生型AtCERK1酶活力的影响,其每个分子空间结构间不能很有规律的排布。之后的实验中我们根据激酶经典的ATP结合位点(K)及关键催化位点(F)[27]进行突变,最终成功提高了蛋白质晶体的分辨率。蛋白质晶体学研究的难点是获得高质量蛋白质晶体。激酶发挥功能的过程中酶先与ATP结合,将ATP中γ位的磷酸基团转移到底物的羟基,最后释放ADP。反应涉及酶与ATP结合以及酶与ADP结合的两种过渡态,不同状态下样品的结晶能力有所差别[28]。为了提高AtCERK1胞内激酶区结晶成功率,我们将野生型和突变型蛋白质与ATP或ADP组装复合体,最终成功实现了复合体结晶。复合体结晶比单独蛋白质的结晶更有意义,解析蛋白质与小分子相互作用的细节有助于之后的研究深入理解其工作机理[29]。
在0.2 mol/L氟化铵、20%PEG3350条件下,突变型AtCERK1F460V和ADP的复合物成功析出晶体,经X射线衍射得到的最高分辨率为3.2 Å。衍射图像分析显示,晶体属于P62空间群;晶胞参数是:a=119.47 Å,b=119.47 Å,c=69.30 Å,α=90.0°,β=90.0°,γ=120.0°。根据蛋白质的氨基酸数目,计算得到该晶体最有可能的马修斯系数为2.12,暗示一个不对称单位中可能有两个复合体分子。初步的结构解析显示,蛋白质电子密度不太理想,对比以往的研究分析[30],原因可能有:1)晶体是由大量微观物质单元按一定规律有序排列的结构,衍射时,X衍射也是通过晶体内部原子有规律的排列才出现衍射点,而本研究中所获得的蛋白质晶体析出的速度比较快,晶体内部原子堆积排列可能不规律;2)晶体结晶的条件仍需要进一步优化,例如:改变盐离子、缓冲液成分、沉淀剂以及pH值,或者添加一些其他可能促进晶体质量的元素,或者用添加剂、去垢剂试剂盒进行优化等;3)防冻液没有配置适宜,晶体质量在液氮环境中发生了退化。虽然刘婷婷等[31]解析出了CERK1胞外区的三维结构,但CERK1胞内结构一直没有得到解析研究。因此,实验最终成功拿到中等衍射分辨率的蛋白质晶体,是向解析CERK1激酶区的空间结构迈出的关键一步,为后续AtCERK1结构解析提供了重要信息和宝贵经验。在后续的实验中,课题组将继续优化AtCERK1激酶区的蛋白质晶体,并在解析出结构后对其关键氨基酸残基定向改造,以获得酶活力更高、热稳定性更强的重组酶分子,从而强化植物免疫,提高植物存活率及生长质量。