基于DNA条形码和SRAP的太子参种质遗传多样性分析
2022-11-11李萍萍王泽榕莫晶晶陈美霞叶祖云
李萍萍,王泽榕,王 丁,莫晶晶,陈美霞,3,叶祖云,3*
基于DNA条形码和SRAP的太子参种质遗传多样性分析
李萍萍1,2,王泽榕1,2,王 丁1,2,莫晶晶1,陈美霞1,3,叶祖云1,3*
1. 宁德师范学院生命科学学院,福建宁德 352100;2. 福建农林大学农学院,福建福州 350000;3. 福建省特色药用植物工程技术研究中心,福建宁德 352100
应用DNA条形码和SRAP分子标记技术,开展15份太子参种质资源遗传差异分析。太子参DNA条形码显示:序列的突变位点占比最多为2.16%,序列的突变位点占比最少为1.23%;序列缺失位点占比最多为4.88%;序列的GC含量最高,达55.52%;序列的GC含量最低,仅为24.71%。太子参SRAP标记筛选出9对引物,扩增出215个位点,其中42个为多态性位点,多态性位点百分率(PPL)为19.53%;从扩增位点总数看,多态性条带丰富、清晰,既有共同位点又有特异性位点;7个居群Nei’s遗传多样性指数()范围为0.3466~0.4985,Shannon信息指数()范围为0.5301~0.6917,显示出较高的遗传多样性水平,太子参种群基因多样性(t)为0.4004,其中种群内基因多样性(s)和遗传分化系数(st)分别为0.0901、0.3103,分别占t的77.50%、22.50%。种源间st为0.7506,即有75.06%的遗传变异存在于种源间,24.94%的遗传变异存在于种源内,表明太子参种源间遗传变异大于种源内遗传变异,存在较低程度的遗传分化。太子参种源的平均基因流(m)为0.1929,表明太子参各种源之间的基因交流顺利。15份太子参种质间的K2P遗传距离范围为0.0005~0.0056,而遗传相似系数在0.3913~0.8695之间,遗传相似系数在0.5900时,可将15份种质分为3个类群,其中杂交种质为独立类型。15份不同种质太子参DNA条形码和SRAP分子标记的遗传距离、遗传相似系数及聚类分析结果相近,以这2种方法相结合,能更准确有效鉴定太子参种质,这对太子参种质资源的利用及杂交新品种的选育具有重要意义。
太子参;DNA条形码技术;SRAP分子标记技术;遗传多样性
太子参[(Miq.) Pax ex Pax et Hoffm]为石竹科草本药用植物,以块根入药,具益气健脾、生津润肺之功效,是我国常用道地中药材[1]。太子参在我国从华东到西北,从华中到东北的山林里均有生长,随着野生太子参资源日渐减少,需求量不断增大,在全国多地均有种植,主产于福建柘荣、贵州施秉、安徽宣城等地,入选福建省“福九味”闽产中药材品种名单。太子参人工栽培发展至今,品种较为单一、种质退化、病害严重等成为制约太子参种植发展的重要因素[2]。目前,太子参栽培品种较少,已通过省级认定的栽培品种只有6个,其中5个是系统选育,1个是诱变选育,未见太子参杂交品种。杂交育种通过杂交、选择和鉴定,不仅能够获得结合亲本优良性状于一体的新类型,而且由于杂种基因的超亲分离,在杂种后代群体中还可能出现性状超越任一亲本,或通过基因互作产生亲本所不具备的新性状的类型,该技术在我国育种工作中占据很重要的地位。国内杂交育种技术在主栽作物如水稻、小麦、甘薯等的新品种选育工作中应用非常广泛[3-4],但在我国药用植物良种选育方面的研究现状不容乐观[5-6],关于太子参杂交育种的报道也仅见本实验室[7-8]。
近些年,相继出现了RAPD[9]、ISSR[10-11]、DNA条形码[12-17]等标记技术在太子参遗传多样性分析、种质资源鉴定等方面的研究,加快了太子参遗传育种的步伐。DNA条形码技术是以染色体组为基础,用一段标准DNA序列作为标记,实现快速、准确和自动化的物种鉴定技术[18],2003年由HEBERT等[19]率先提出;2009年,国际条形码协会植物工作组提出叶绿体基因作为植物核心DNA条形码,随后又以rDNA、作为补充[20-21]。余永邦等[12]利用引物对14个产区的太子参进行测序,其中大部分种质的序列存在变异。赵海等[13]以和序列多片段组合方式可准确鉴定太子参品种‘黔太子参1号’和‘太子参丰抗1号’。朱艳等[14]通过对太子参种质及其混伪品rDNA区进行序列测定,可准确鉴定出太子参种质。
相关序列扩增多态性(SRAP)分子标记方法是2001年在芸薹属植物中开发出来的一种随机引物标记系统[22-24]。近年来,SRAP标记在植物遗传多样性分析、种质资源鉴定、遗传连锁图的构建与基因定位中得到了广泛应用,与RAPD、ISSR、SSR等标记相比,SRAP具有更丰富的多态性、高共显性、操作简便和费用低等优点,并且省去了引物开发的过程[25-29]。本研究采用DNA条形码和SRAP分子标记技术相结合的方法,对来源于福建、江苏、贵州、山东、安徽及湖南等主要产地的太子参种质进行遗传差异分析,研究各产地不同种质间的遗传关系,鉴定太子参杂交种质,推进杂交新品种的选育,并为建立太子参基因资源库、品种鉴定、种质资源保护和创新利用奠定理论基础。
1 材料与方法
1.1 材料
供试材料共15份,包括从全国收集的8份太子参种质及本实验室选育的7份杂交种质,均栽培保存于福建省宁德市柘荣县凤洋村的太子参良种繁育基地(27°15¢30²N,119°50¢15²E)(表1)。

表1 实验材料及来源
1.2 方法
1.2.1 提取DNA 剪取新鲜幼嫩太子参叶片0.1 g,采用植物全基因组DNA提取试剂盒(北京康为)提取叶片DNA,–20℃保存备用。
1.2.2 PCR扩增 以15份太子参的DNA作为模板,应用ITS1、ITS5、psbA-trnH、rbcL、rbcL2、rbcL6共6对DNA条形码通用引物(表2)进行PCR扩增[30-33]。PCR反应体系:DNA模板2 μL,10×ex缓冲液2 μL,dNTPMix 2 μL,上下游引物各1 μL,ex酶0.2 μL,ddH2O补足至20 μL。(1)以ITS为引物的PCR反应程序:94℃预变性5 min;94℃变性30 s,53℃退火1 min,72℃延伸1.5 min,35个循环;72℃延伸10 min。(2)以psbA-trnH为引物的PCR反应程序:94℃预变性5 min;94℃变性30 s,58℃退火1 min,72℃延伸1.5 min,30个循环;72℃延伸10 min。(3)以rbcL为引物的PCR反应程序:95℃预变性5 min;94℃变性1 min,57℃退火1 min,72℃延伸1.5 min,33个循环;72℃延伸10 min。
1.2.3 克隆子验证 凝胶回收操作按照微柱浓度DNA凝胶试剂盒说明书进行(庄盟生物);连接和转化按照M5 HiPer pTOPO-TA Cloning Kit操作(北京聚合美),克隆子验证后送样测序(铂尚生物)。菌落PCR反应体系:模板 1 μL,10×PCR buffer 2.5 μL,dNTPMix 1 μL,上下游引物各0.5 μL,r酶0.15 μL,ddH2O补足至25 μL。菌落PCR反应程序:95℃预变性5 min;94℃变性30 s,54℃退火30 s,72℃延伸100 s,35个循环;72℃延伸7 min,4℃保存。
1.2.4 序列分析、遗传距离分析及构建进化树 利用Sequencher 5.4.5和DNAMAN对已完成测序的序列进行拼接、比对和校正[12],统计序列长度、变异位点和GC含量等数据,利用Mega 7.0软件计算太子参不同种质资源之间的K2P遗传距离,并构建NJ系统进化树。
1.2.5 SRAP-PCR扩增 太子参基因组DNA的提取同1.2.1,SRAP引物序列参照BUDAK等[24]的方法,筛选出9对多态性引物(M1E3、M1E8、M1E18、M1E19、M2E13、M2E14、M2E16、M3E3、M4E16),引物由公司合成。SRAP-PCR体系:DNA模板1 μL,10×exbuffer 2 μL,dNTPMix 2 μL,上下游引物各1 μL,ex酶 0.3 μL,ddH2O补足至20 μL。SRAP-PCR程序:94℃预变性5 min;90℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,51℃退火1 min,72℃延伸1 min,30个循环;72℃延伸10 min,12℃保存。
1.2.6 聚丙烯酰胺凝胶电泳及数据分析 配制6%聚丙烯酰胺凝胶电泳检测,经固定、洗涤、银染、显影和拍照。对电泳图谱每个样品的扩增条带进行记录,相同迁移率位置上,用1来表示有条带,0表示无条带。采用POPGENE 32软件[34]计算材料间的Nei’s遗传多样性指数(),Shannon’s多态性指数(),种群的基因多样性(t),种群内基因多样性(s),遗传分化系数(st),种源间基因流(m),并利用NTSYS-PC 2.1软件[35]采用UPGMA方法构建聚类分析图。
2 结果与分析
2.1 DNA提取结果
太子参测试样品DNA主条带清晰整齐、无杂带和拖带情况,浓度均超过40 ng/μL,260/280在1.80左右,是理想的PCR扩增模板(图1A)。
2.2 DNA条形码PCR扩增结果
应用引物ITS1、ITS5、psbA-trnH、rbcL、rbcL2、rbcL6对太子参测试样品进行PCR扩增,扩增片段长度分别为648、685、389、703、327、815 bp,条带清晰明亮。PCR扩增结果以引物ITS1和rbcL2为例(图1B、图1C)。

1~15:与表1中的材料对应。A:全DNA电泳图谱,M1:15 000 bp DNA marker;B:ITS1引物PCR扩增电泳图谱-DNA条形码,M2:DL2000 DNA marker;C:rbcL2引物PCR扩增电泳图谱-DNA条形码,M3:DL2000 DNA marker;D:M1E18引物PCR扩增部分聚丙烯酰胺凝胶电泳图谱-SRAP,M4:100 bp DNA marker。
2.3 DNA条形码片段序列信息
太子参DNA条形码长度、变异位点和GC含量等序列信息见表2。各候选条形码序列的测序成功率均为100%,序列质量均较高,6对候选条形码片段的序列长度从327~815 bp不等;以rbcL6作为引物的样本序列最长,为815 bp;以rbcL2作为引物的样本序列最短,为327bp;rbcL6序列的保守位点占比最多为98.65%;ITS1序列的突变位点占比最多为2.16%,rbcL6序列的突变位点占比最少为1.23%;psbA-trnH序列缺失位点占比最多为4.88%,ITS1、ITS5和rbcL2序列没有缺失位点。各候选条形码片段序列的GC含量均不相同,ITS1序列的GC含量达55.52%;psbA-trnH序列的GC含量仅为24.71%。
2.4 SRAP引物筛选及PCR扩增结果
太子参SRAP引物筛选结果见图1D和表3,共有9对引物可扩增出多态性丰富、重复性高、稳定性好的条带。15个样本共扩增出215个位点,其中42个多态性位点,平均每个引物扩增24个位点,多态性位点百分率(PPL)为19.53%,扩增片段在80~650 bp之间。引物M4E16扩增出的条带最多,有29条;引物M3E3扩增出的多态性条带较多,有7条;引物M2E13扩增出的多态性条带最少,只有2条;从扩增位点总数看,多态性条带丰富、清晰,既有共同位点又有特异性位点。
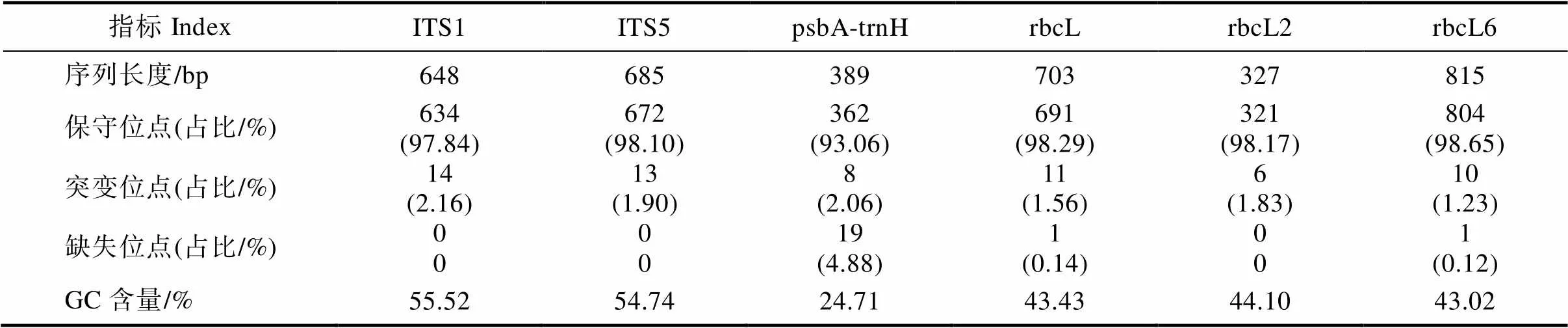
表2 太子参DNA条形码片段序列信息
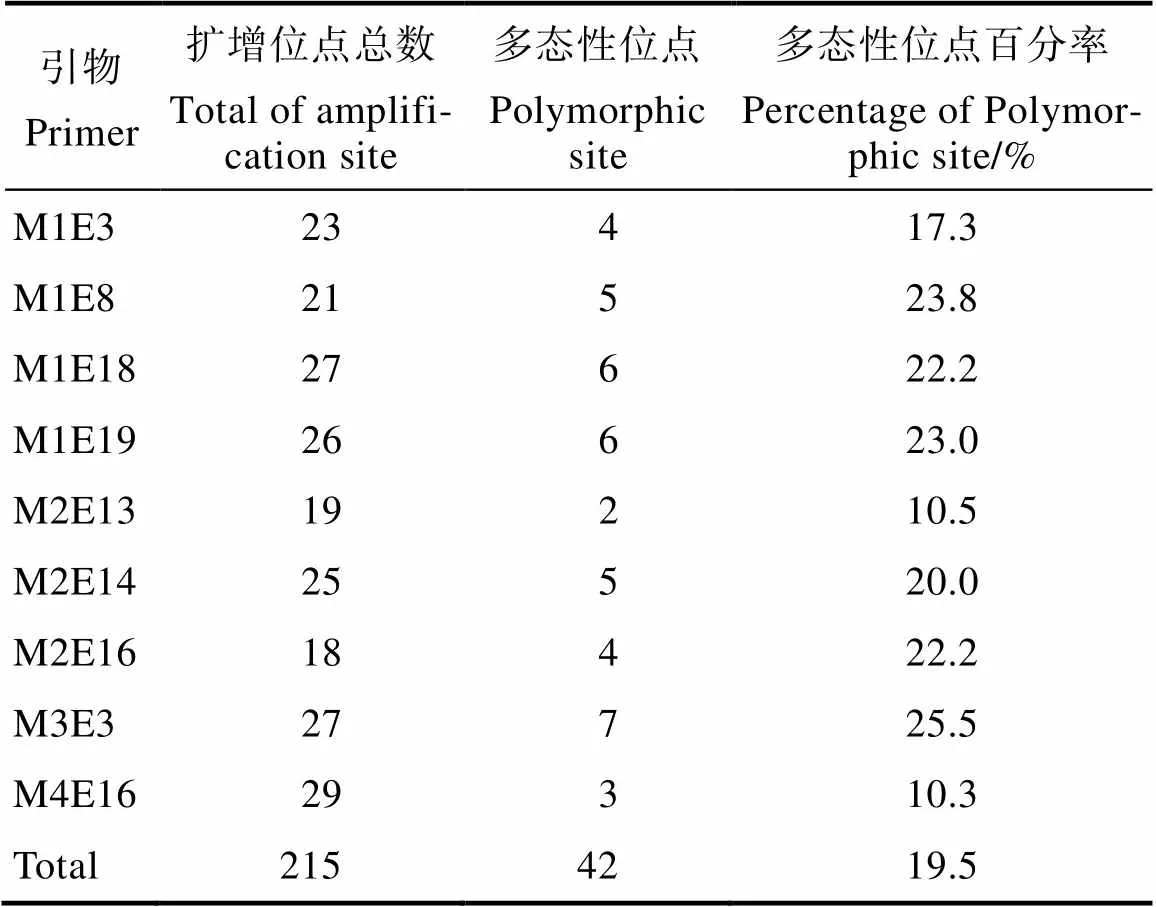
表3 以SRAP引物对太子参种质PCR扩增的DNA片段和序列多样性
2.5 不同种质太子参的遗传多样性和遗传分化分析
15份太子参种质可分为福建种、江苏种、贵州种、湖南种、山东种、安徽种、杂交种质7个居群,Nei’s遗传多样性指数()范围在0.3466~0.4985之间,Shannon信息指数()范围在0.5301~0.6917之间,平均值为0.4173,平均值为0.6052,显示出较高的遗传多样性水平。由表4可知,太子参种群基因多样性(t)为0.4004,其中种群内基因多样性(s)和遗传分化系数(st)分别为0.0901、0.3103,分别占t的77.50%、22.50%。种源间st为0.7506,即有75.06%的遗传变异存在于种源间,24.94%的遗传变异存在于种源内,表明太子参种源间遗传变异大于种源内遗传变异,存在较低程度的遗传分化。太子参种源的平均基因流(m)为0.1929,表明太子参各种源之间的基因交流顺利[36]。

表4 不同种质太子参遗传多样性
2.6 太子参测试样品遗传距离和遗传相似系数分析
太子参测试样品复合片段序列的K2P遗传距离范围在0.0005~0.0056之间,平均值是0.0028,遗传相似系数是在0.3913~0.8695,表明样品间遗传差异较小,亲缘关系较近,具有较高的遗传多样性,但各样品之间仍存在一定的遗传距离,DNA条形码和SRAP分子标记可对样品进行有效区分,能清晰划分太子参种质之间的亲缘关系,适用于太子参遗传多样性的分析和不同种质的鉴别(表5,表6)。
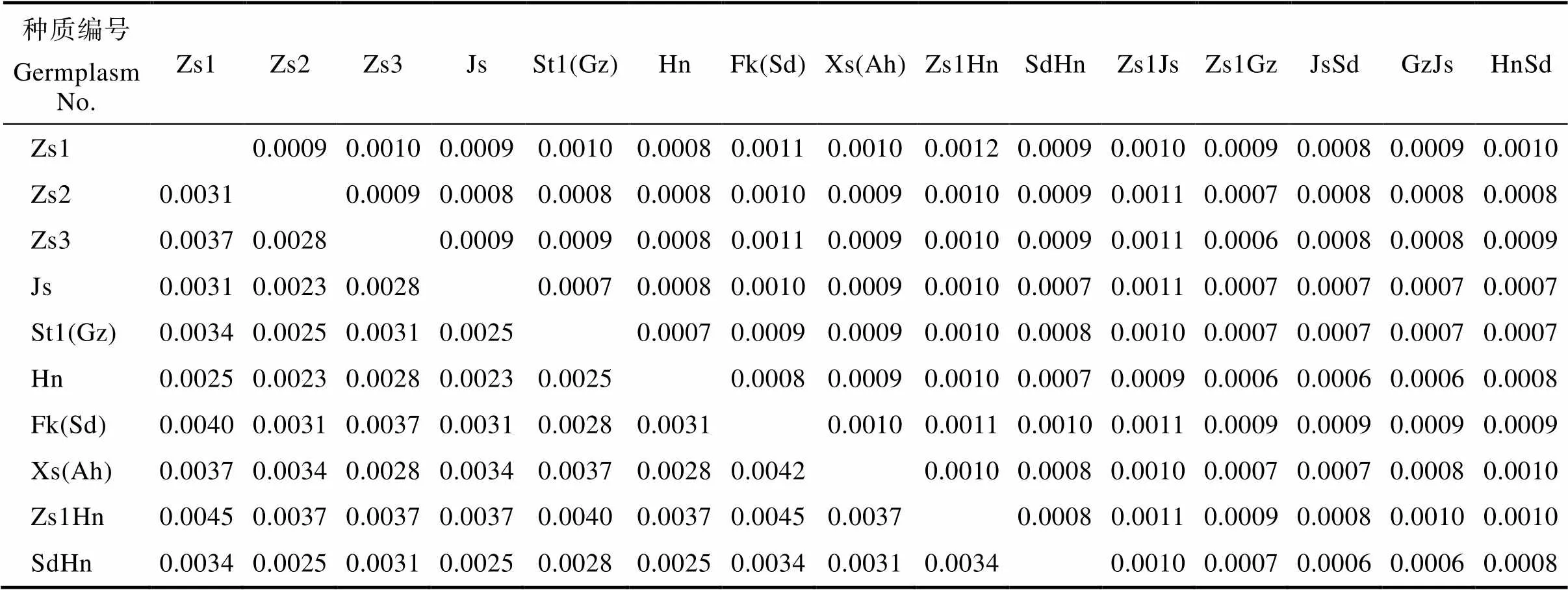
表5 15份太子参种质K2P遗传距离分析-DNA条形码
续表5 15份太子参种质K2P遗传距离分析-DNA条形码
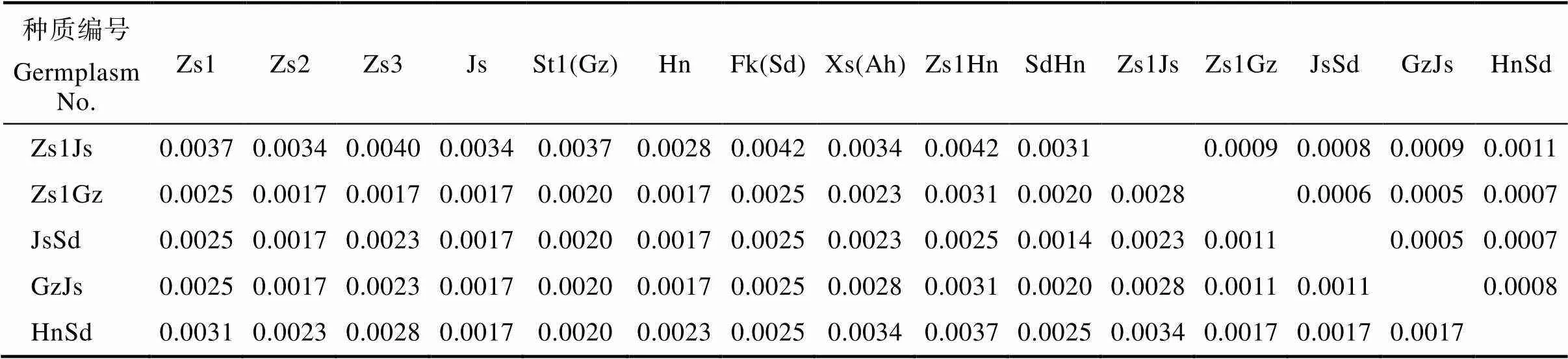
Tab. 5 Genetic distance analysis of 15 P.heterophylla germplasms with K2P-DNA barcodes (continued)
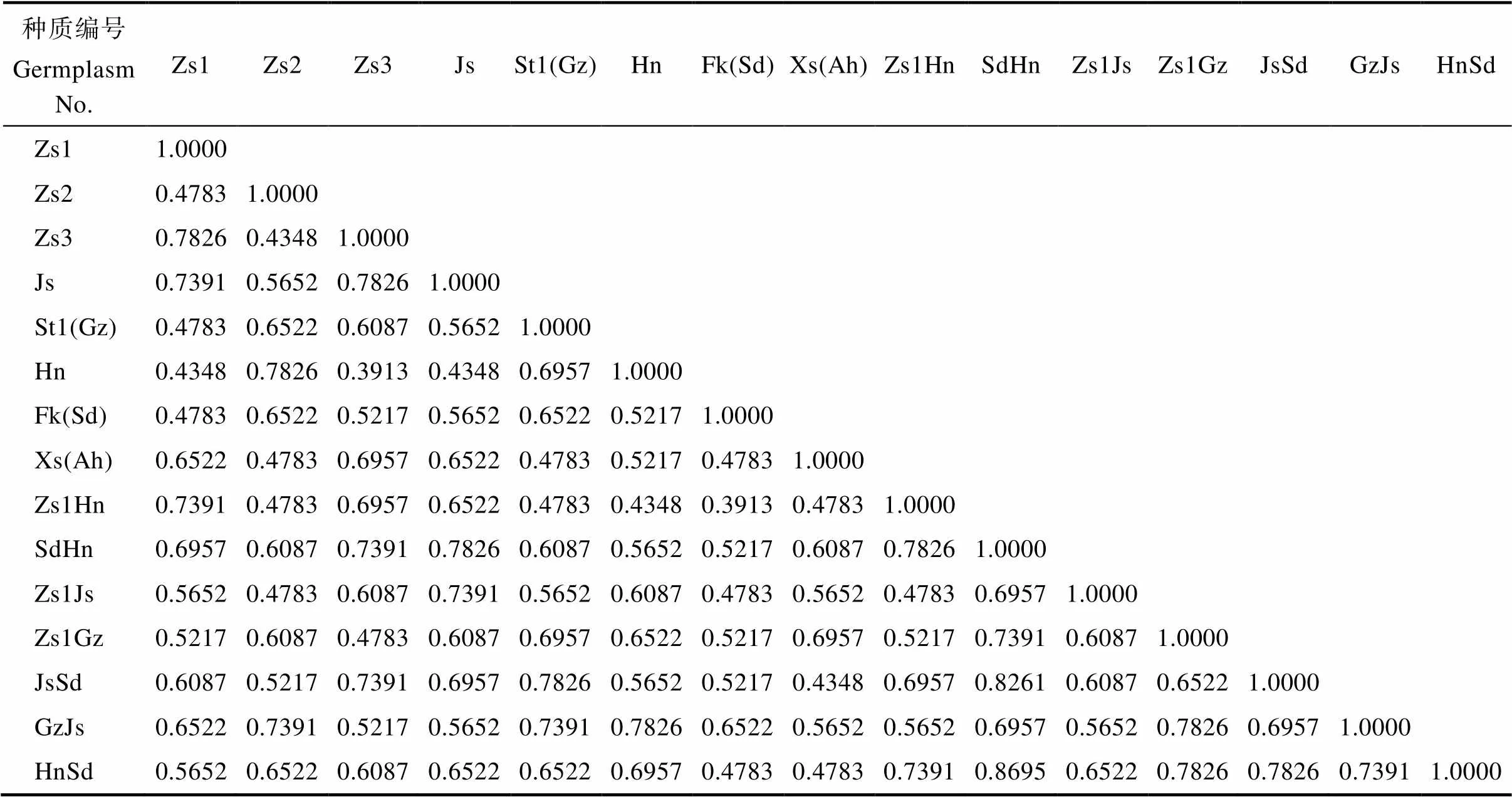
表6 15份太子参种质遗传相似系数分析-SRAP分子标记
2.7 构建NJ系统进化树和聚类分析
在太子参DNA条形码中,可将各省的种质分为3个类群,其中江苏种、山东种、贵州种和福建种的‘柘参2号’聚为一类,福建种的‘柘参3号’和安徽种聚为一类,湖南种和福建种的‘柘参1号’聚为一类;太子参SRAP标记中遗传相似系数在0.590时,可将各省的种质分为3个类群,其中江苏种和福建种的‘柘参1号’‘柘参3号’聚为一类,安徽种为一类,福建种的‘柘参2号’、湖南种、贵州种和山东种聚为一类。2种分子标记方法显示山东种和贵州种均聚为一类,亲缘关系较近,可能是由于相互引种,福建种的3个品种均未聚为一类,说明太子参种质资源间遗传多样性丰富,但并不完全按照地理分布聚类,各地太子参材料存在一定程度的基因交流。
在太子参杂交种质中,2种标记方法结果基本一致:F1(湖南种♀×山东种♂)与父本山东种聚为一类;F1(贵州种♀×江苏种♂)与母本贵州种聚为一类;F1(江苏种♀×山东种♂)与母本江苏种聚为一类;F1(山东种♀×湖南种♂)与母本山东种聚为一类;F1(柘参1号♀×贵州种♂)、F1(柘参1号♀×湖南种♂)和F1(柘参1号♀×江苏种♂)均与母本‘柘参1号’聚为一类(图2)。
3 讨论
3.1 DNA条形码和SRAP相结合可较准确鉴定太子参的亲缘关系
本研究采用DNA条形码和SRAP分子标记相结合的方法,开展数量较多的太子参种质遗传差异分析。15份太子参种质DNA条形码和SRAP分子标记的聚类分析结果基本一致,均能快速准确鉴定出太子参杂交新种质。DNA条形码显示,采用太子参叶绿体和核糖体的相对保守的多对引物,转化子测序成功率可达100%,质量较高,太子参杂交种质复合序列的差异位点较多,存在一定的遗传距离,对种质资源鉴别能力较好;SRAP分子标记显示太子参扩增条带丰富,差异性条带明显、清晰、重复性好,呈现较高的遗传多样性水平,种源间遗传变异大于种源内遗传变异,存在较低程度的遗传分化,太子参各种源之间的基因交流顺利。采用SRAP分子标记开展太子参各种质遗传多样性研究,太子参SRAP分子标记引物扩增的多态性条带,最多的是7条,最少的仅2条,多态性条带平均占比也较低,还需要进一步从SRAP标记引物中筛选出核心标记,以便更准确可靠分析太子参种质资源的遗传差异。2种方法结合,更好地显示太子参种质之间丰富的遗传差异信息,这对加快太子参新种质的选育鉴定、种质资源遗传多样性研究和开发利用是非常重要的。
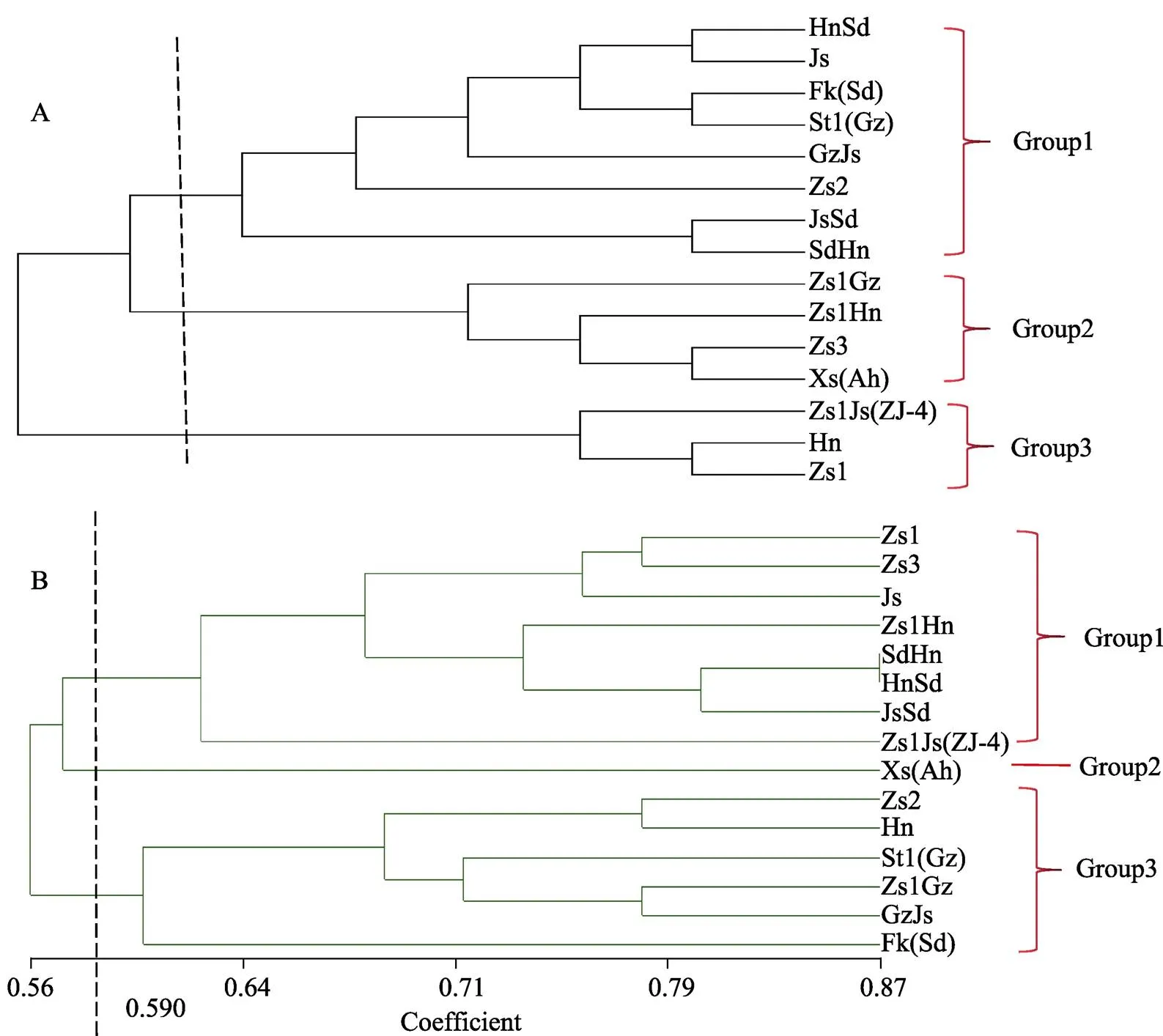
A:DNA条形码;B:SRAP分子标记。
3.2 DNA条形码和SRAP相结合可为杂交后代的鉴定提供依据
在太子参种质选育过程中,由于种种原因,可能存在不确定其亲本组合的情况,通过这2种方法进行聚类分析,可推测其遗传背景。如F1(湖南种♀×山东种♂)与父本山东种聚为一类;F1(贵州种♀×江苏种♂)与母本贵州种聚为一类;F1(江苏种♀×山东种♂)与母本江苏种聚为一类;F1(山东种♀×湖南种♂)与母本山东种聚为一类。
3.3 DNA条形码和SRAP使用多对引物相结合鉴定太子参种质
根据聚类结果,筛选出6对DNA条形码和9对SRAP多态性标记,虽然这些引物单独使用时无法准确鉴定所有太子参种质,但共同使用时可增加鉴定太子参种质的准确性。相比通过单一引物来鉴定太子参种质的方法,多对引物鉴定具有一定的创新性,这也为鉴定其他太子参种质提供新的思路。鉴于本研究仅分析了15份太子参种质,增加样本量并结合其形态特性,采用多对引物鉴定太子参种质,有望提高种质鉴定的准确度。本课题已开展了太子参杂交授粉及杂交种子种胚离体诱导培养[7-8],下一步将加大样品数量,对太子参杂交F1代遗传信息进行更加深入的研究。
4 结论
本研究利用DNA条形码和SRAP标记技术相结合的方法,对15份太子参材料进行亲缘关系分析,从聚类结果来看,2种分子标记方法显示出太子参种质资源间遗传多样性丰富,但并不完全按照地理分布聚类,遗传背景比较复杂,各地太子参材料存在一定程度的基因交流。2种标记方法在这些材料的亲缘关系分析中表现的可信度较高,且可根据聚类结果推测种质的来源。此外,本研究发现结合6对DNA条形码和9对SRAP多态性标记能更准确有效地鉴定太子参种质,这为太子参种质资源的利用及杂交新品种的选育等提供依据。
[1] 中华人民共和国卫生部药典委员会. 中华人民共和国药典(一部)[M]. 北京: 北京化学工业出版社, 2020: 69.
Pharmacopoeia Commission, Ministry of Health, People’s Republic of China. Pharmacopoeia of the PRC (Part 1)[M]. Beijing: Beijing Chemical Industry Press, 2020: 69. (in Chinese)
[2] 温学森, 霍德兰, 赵华英. 太子参常见病害及其防治[J]. 中药材, 2003, 26(4): 243-245.
WEN X S, HUO D L, ZHAO H Y. Common diseases ofand their control[J]. Journal of Chinese Medicinal Materials, 2003, 26(4): 243-245. (in Chinese)
[3] 程式华. 中国水稻育种百年发展与展望[J]. 中国稻米, 2021, 27(4): 1-6.
CHENG S H.One-hundred years’ development and prospect of rice breeding in China[J]. China Rice, 2021, 27(4): 1-6. (in Chinese)
[4] 谢一芝, 贾赵东, 边小峰, 马佩勇, 禹 阳, 郭小丁. 食用甘薯品种选育及系谱分析[J]. 中国农学通报, 2021, 37(30): 32-38.
XIE Y Z, JIA Z D, BIAN X F, MA P Y, YU Y, GUO X D. Breeding and pedigree of edible sweet potato varieties[J]. Chinese Agricultural Science Bulletin, 2021, 37(30): 32-38. (in Chinese)
[5] 孙莹莹, 罗 睿, 杜禹珊, 朱燕燕. 药用植物杂交育种技术研究进展[J]. 中药材, 2016, 39(2): 442-446.
SUN Y Y, LUO R, DU Y S, ZHU Y Y. Research progress on cross breeding technology of medicinal plants[J]. Journal of Chinese Medicinal Materials, 2016, 39(2): 442-446. (in Chinese)
[6] 董 玲, 李卫文, 宁志怨, 廖华俊, 江 芹, 姚其盛. 我国药用植物育种工作的现状与展望[J]. 现代中药研究与实践, 2014, 28(6): 3-6.
DONG L, LI W W, NING Z Y, LIAO H J, JIANG Q, YAO Q S. Status and prospect of medicinal plant breeding in China[J]. Research and Practice of Modern Chinese Medicine, 2014, 28(6): 3-6. (in Chinese)
[7] 叶祖云, 吴文标, 缪霖辉, 苏志杭, 鞠建文. 一种太子参人工杂交授粉方法: 201710055724. X[P]. 2018-11-13.
YE Z Y, WU W B, MIU L H, SU Z H, JU J W.An artificial cross pollination method of: 201710055724. X[P]. 2018-11-13. (in Chinese)
[8] 叶祖云, 何绿洪, 董晓菲, 刘 群. 一种太子参杂交种子人工培养选育方法: 201610972933. 6[P]. 2016-11-07.
YE Z Y, HE L H, DONG X F, LIU Q.A method for artificial culture and breeding of hybrid seeds of: 201610972933. 6[P]. 2016-11-07. (in Chinese)
[9] 朱 艳, 秦民坚, 杭悦宇, 郑晓清. 不同种源太子参的RAPD分析[J]. 植物资源与环境学报, 2007, 16(3): 19-22.
ZHU Y, QIN M J, HANG Y Y, ZHENG X Q. RAPD analysis onfrom different provenances[J]. Journal of Plant Resources and Environment, 2007, 16(3): 19-22. (in Chinese)
[10] 侯 娅, 马 阳, 邹立思, 谷 巍, 刘训红, 兰才武, 袁济端. 不同种质太子参无机元素测定及其遗传多样性分析[J]. 时珍国医国药, 2015, 26(5): 1236-1239.
HOU Y, MA Y, ZOU L S, GU W, LIU X H, LAN C W, YUAN J D. Determination of inorganic elements and analysis of genetic diversity indifferent idio-plasm of[J]. Lishizhen Medicine and Materia Medica Research, 2015, 26(5): 1236-1239. (in Chinese)
[11] 肖承鸿, 周 涛, 江维克, 艾 强, 杨昌贵, 熊厚溪, 廖明武. 栽培太子参的遗传多样性与质量分析[J]. 中草药, 2014, 45(9): 1319-1325.
XIAO C H, ZHOU T, JIANG W K, AI Q, YANG C G, XIONG H X, LIAO M W. Genetic diversity and quality analysis of cultivated[J]. Chinese Traditional and Herbal Drugs, 2014, 45(9): 1319-1325. (in Chinese)
[12] 余永邦, 秦民坚, 梁之桃, 余国奠, 谭宁华. 不同产区太子参的rDNA ITS区序列的比较[J]. 植物资源与环境学报, 2003, 12(4): 1-5.
YU Y B, QIN M J, LIANG Z T, YU G D, TAN N H. Ribosomal DNA ITS sequence comparisons offrom different geographical[J]. Journal of Plant Resources and Environment, 2003, 12(4): 1-5. (in Chinese)
[13] 赵 海, 李园园, 刘红昌, 王华磊, 侯 峰, 兰才武. 基于ITS2和psbA-trnH序列的黔太子参品种DNA条形码鉴定研究[J]. 中药材, 2018, 41(4): 855-858.
ZHAO H, LI Y Y, LIU H C, WANG H L, HOU F, LAN C W. DNA barcoding identification ofvarieties based on ITS2 and psbA-trnH sequences[J]. Journal of Chinese Medicinal Materials, 2018, 41(4): 855-858. (in Chinese)
[14] 朱 艳, 秦民坚, 杭悦宇, 王丽琴. 太子参及其混伪品的rDNA ITS区序列分析及鉴别[J]. 中国天然药物, 2007, 5(3): 211-215.
ZHU Y, QIN M J, HANG Y Y, WANG L Q. Sequence analysis and identification of rDNA ITS region ofand its adulterants[J]. Chinese Journal of Natural Medicines, 2007, 5(3): 211-215. (in Chinese)
[15] 赵 丹, 周 涛, 江维克, 袁 媛, 肖承鸿, 郑 伟. 太子参药材的快速分子鉴定[J]. 中国国药杂志, 2014, 39(19): 3689-3693.
ZHAO D, ZHOU T, JIANG W K, YUAN Y, XIAO C H, ZHENG W. Rapid molecular identification ofRadix[J]. China Journal of Chinese Materia Medica, 2014, 39(19): 3689-3693. (in Chinese)
[16] 刘宇婧, 刘 越, 黄耀江, 龙春林. 植物DNA条形码技术的发展及应用[J]. 植物资源与环境学报, 2011, 20(1): 74-82.
LIU Y J, LIU Y, HUANG Y J, LONG C L. Progress and application of DNA barcoding technique in plants[J]. Journal of Plant Resources and Environment, 2011, 20(1): 74-82. (in Chinese)
[17] 方强强, 王 燕, 彭 春, 唐 玲. 中药DNA条形码分子鉴定技术的应用与展望[J].中国实验方剂学杂志, 2018, 24(22): 197-205.
FANG Q Q, WANG Y, PENG C, TANG L. Application and prospect of DNA barcode molecular identification technology in traditional Chinese medicine[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2018, 24(22): 197-205. (in Chinese)
[18] 贺水莲, 吴红芝, 孟 静, 杜 娟, 杨 扬. 应用DNA条形码鉴定滇西北4种传统藏药绿绒蒿[J]. 西部林业科学, 2017, 46(5): 1-5.
HE S L, WU H Z, MENG J, DU J, YANG Y. Identification of four species of Tibetan medicinefrom northwest Yunnan by DNA barcoding[J]. Journal of West China Forestry Science, 2017, 46(5): 1-5. (in Chinese)
[19] HEBERT P D N, CYWINSKA A, BALL S L, DEWAARD J R. Biological identifications through DNA barcodes[J]. The Royal Society, 2003, 270: 313-321.
[20] LI D Z, LIU J Q, CHEN Z D. Plant DNA barcoding in China[J]. Journal of Systematics and Evolution, 2011, 49(3): 165-168.
[21] CBOL Plant Working Group. A DNA barcode for land plants[J].Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(31): 12794-12797.
[22] BECKMANN J S, SOLLER M. Restriction fragment length polymorphism in genetic improvement: methodologies, mapping and costs[J]. Theoretical and Applied Genetics, 1983, 67(1): 35-43.
[23] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in[J]. Theoretical and Applied Genetics, 2001, 103(2/3): 455-461.
[24] BUDAK H, SHEARMAN R C, PARMAKSIZ I, DWEIKAT I. Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationships using ISSRs, SSRs, RAPDs, and SRAPs[J]. Theoretical and Applied Genetics, 2004, 109(2): 280-288.
[25] JING Z B, CHENF J M, GUO C H, WANG X P. Seed traits, nutrient elements and assessment of genetic diversity for almond (spp.) endangered to China as revealed using SRAP markers[J]. Biochemical Systematics and Ecology, 2013, 49: 51-57.
[26] 林宗铿, 张天翔, 杨俊杰. 基于SRAP的辣木种质资源遗传多样性和亲缘关系分析[J]. 热带作物学报, 2021, 42(4): 945-950.
LIN Z K, ZHANG T X, YANG J J. Assessment of genetic diversity and relationship among 18species based on SRAP marker[J]. Chinese Journal of Tropical Crops, 2021, 42(4): 945-950. (in Chinese)
[27] 刘丽娟, 刘灶长, 陈海荣, 罗利军. SRAP标记技术及其在蔬菜作物遗传多样性分析中的应用[J]. 中国农学通报, 2009, 25(21): 43-54.
LIU L J, LIU Z Z, CHEN H R, LUO L J. SRAP marker technique and its application in genetic diversity analyses of vegetable crops[J]. Chinese Agricultural Science Bulletin, 2009, 25(21): 43-54. (in Chinese)
[28] 胡福初, 吴小波, 陈 哲, 吴凤芝, 周文静, 冯学杰, 范鸿雁, 周瑞云, 王祥和. 基于SRAP分子标记的特早熟荔枝种质资源遗传多样性分析[J]. 热带作物学报, 2021, 42(4): 920-926.
HU F C, WU X B, CHEN Z, WU F Z, ZHOU W J, FENG X J, FAN H Y, ZHOU R Y, WANG X H. Genetic diversity analysis of very early maturing Litchi germplasm resources based on SRAP molecular markers[J]. Chinese Journal of Tropical Crops, 2021, 42(4): 920-926. (in Chinese)
[29] 袁 媛, 曹 彬, 张咏琪, 陈清西, 陈南川. 基于SRAP标记的国兰种质资源遗传多样性分析[J]. 热带作物学报, 2020, 41(5): 929-938.
YUAN Y, CAO B, ZHANG Y Q, CHEN Q X, CHEN N C. Genetic diversity analysis ofgermplasms based on SRAP markers[J]. Chinese Journal of Tropical Crops, 2020, 41(5): 929-938. (in Chinese)
[30] KRESS W J, ERICKSON D L, SHIN S H. A two-locus global DNA barcode for land plants: the codingL gene complements the non-coding trnH-psbA spacer region[J]. PLoS One, 2007, 2(6): e508.
[31] LAHAVE R, VAN DER Bank M, BOGARIN D, WARNER JPOPULIN F, GIGOT G, MAURIN O, DUTHOIT S, BARRACOLUGH T G, SAVOLAINEN V. DNA barcoding the floras of biodiversity hotspots[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(8): 2923-2928.
[32] SONG J Y, YAO H, LI Y, LI X W, LIN Y L, LIU C, HAN J P, XIE C X, CHEN S L. Authentication of the family Polygonaceae in Chinese pharmacopoeia by DNA barcoding technique[J]. Journal of Ethnopharmacology, 2009, 124(3): 434-439.
[33] YAO H, SONG J Y, MA X Y, LIU C, LI Y, XU H X, HAN J P, DUAN L S, CHEN S L. Identification ofspecies by a candidate DNA barcode sequence: the chloroplast psbA-trnH intergenic region[J]. Planta Medica, 2009, 75(6): 667-669.
[34] YEH F C, YANG R C, MAO J, YE Z, BOYLE T J. POPGENE, the microsoft windows-based user-friendly software for population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany, 1996, 129: 157.
[35] POWELL W, MORGANTE M, ANDRE C, HANAFEY M, VOGEL J, TINGEY S, RAFALSKI A. The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis[J]. Molecular Breeding, 1996, 2(3): 225-238.
[36] WRIGHT S. Evolution in Mendelian population[J]. Bulletin of Mathematical Biology, 1990, 52(1/2): 241-295.
Genetic Diversity Analysis ofGermplasms Based on DNA Barcoding and SRAP
LI Pingping1,2, WANG Zerong1,2, WANG Ding1,2, MO Jingjing1, CHEN Meixia1,3, YE Zuyun1,3*
1. College of Life Sciences, Ningde Normal University, Ningde, Fujian 352100, China; 2.College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350000, China; 3. Fujian Engineering Technology Research Center of Characteristic Medicinal Plants, Ningde, Fujian 352100, China
DNA barcoding and SRAP (sequence-related amplified polymorphism) molecular markers were used to analyze the genetic differences of 15 germplasms of.The mutation sites ofsequence accounted for 2.16% at most, and the mutation sites ofsequence accounted for 1.23% at least.sequence deletion sites accounted for 4.88% in the maximum. The highest GC content ofsequence was 55.52%, and the lowest GC content ofsequence was only 24.71%. Nine pairs of primers were screened out for the generation of SRAP markers by PCR amplification, and hereafter, 215 genomic DNA loci were amplified, including 42 polymorphic loci, and the percentage of polymorphic genomic DNA loci (PPL) was 19.53%,in terms of the total number of amplification sites, the polymorphic bands were rich and clear, with both common and specific sites. Nei’s genetic diversity index () ranged from 0.3466 to 0.4985 and Shannon information index () ranged from 0.5301 to 0.6917, showing a high level of genetic diversity. Population genetic diversity (t) ofwas 0.4004, in which the genetic diversity within populations (s) and genetic differentiation coefficient (st) was 0.0901 and 0.3103, accounting for 77.50% and 22.50% oft, respectively. Thestbetween provenances was 0.7506, meaning 75.06% of the genetic variation existed between provenances and 24.94% within provenances, indicating that the genetic variation among provenances was greater than that within provenances, and there was a low degree of genetic differentiation. The average gene flow (m) of the provenances ofwas 0.1929, and the result implied the gene exchange between provenances ofwas smooth. The genetic distance of K2P among 15 germplasms ofranged from 0.0005 to 0.0056, while the genetic similarity coefficient was between 0.3913 and 0.8695. When the genetic similarity coefficient was confined in superior 0.5900. 15 germplasms could be divided into three groups, among which the hybrid germplasm was an independent category. The results of genetic distance, genetic similarity coefficient and cluster analysis of 15 different germplasms ofwith DNA barcoding and SRAP molecular markers were similar. The combination of these two methods could identifygermplasms more accurately and effectively, which is of great significance for the utilization of germplasm resources and the breeding of new hybrid varieties of.
; DNA barcoding technology; SRAP molecular marker technology; genetic diversity
S567.21
A
10.3969/j.issn.1000-2561.2022.10.009
2022-01-20;
2022-03-11
国家重点研发计划中医药现代化研究专项(No. 2019YFC1710500);中央引导地方科技发展专项(No. 2021L3030);宁德师范学院科研项目(No. 2019FZ04)。
李萍萍(1996—),女,硕士研究生,研究方向:作物遗传育种。*通信作者(Corresponding author):叶祖云(YE Zuyun),E-mail:zyye0593@163.com。
