细菌3-酮脂酰ACP还原酶的研究现状与展望
2022-10-31王海洪
王海洪,胡 喆
(华南农业大学 生命科学学院/广东省农业生物蛋白质功能与调控重点实验室, 广东 广州 510642)
脂肪酸是细胞生物主要的组成成分之一,是细胞膜磷脂、三酰甘油酯(脂肪)和糖脂的重要组分[1]。按碳链是否有支链可分为直链脂肪酸和支链脂肪酸[2];按照主碳链的碳原子数目,可以分为偶数脂肪酸和奇数脂肪酸,其中细胞膜的主要成分多为偶数脂肪酸[3];按照含不饱和碳碳双键的数目,可以分为饱和脂肪酸、单不饱和脂肪酸和多不饱和脂肪酸,生物体中单不饱和脂肪酸居多[4],按照双键构型,不饱和脂肪酸又可分为顺式脂肪酸和反式脂肪酸,生物体的不饱和脂肪酸主要为顺式不饱和脂肪酸[5]。
细菌通过调节脂肪酸的种类和组成,调节细胞膜的流动性,适应不同的生长环境[6-7]。此外脂肪酸还可以通过β-氧化途径为细菌提供大量的能量,是一种重要的储能物质[8-9]。
细菌能够从头合成脂肪酸,除了为膜磷脂和糖脂的合成提供底物外,还可为硫辛酸、生物素、鼠李糖脂、聚羟基脂肪酸(PHA/PHB)、类脂A、N-脂酰高丝氨酸内酯类(AHLs)和可扩散信号分子(Diffusible signal factor, DSF)等的合成提供前体[3,10-14]。
目前对细菌脂肪酸合成系统已经进行了深入的研究,对一些关键酶的催化机理、生物学功能及其多样性也有众多的综合性论述,但鲜见3-酮脂酰ACP还原酶(3-oxoacyl-ACP-reductase,OAR)的系统性总结。本文对细菌3-酮脂酰ACP还原酶的研究进行系统性总结,以便为今后的科学研究提供参考。
1 脂肪酸合成系统
细胞生物有2种类型的脂肪酸合成系统。哺乳动物、真菌以及分枝杆菌采用I型脂肪酸合成系统(Fatty acid synthesis I, FAS I),特点为脂肪酸的合成由1个多结构域的复杂脂肪酸合酶催化,其中每个结构域催化1个特定的生化反应,而脂酰基团由酰基载体蛋白(Acyl carrier protein,ACP)结构域携带,在各结构域间传递脂酰基团[15-16]。FAS I的最终产物一般为棕榈酸。另外,该系统不合成不饱和脂肪酸[17]。
细菌和植物采用II型脂肪酸合成系统(Fatty acid synthesis II, FAS II)合成脂肪酸(图1A)[18]。FAS II的每一步反应均由独立的酶催化完成,脂酰基团由酰基载体蛋白ACP携带,可产生各种类型的脂酰ACP,为不同的最终产物提供前体[19-20]。同时,FAS II也可产生顺-3烯脂酰ACP,合成不饱和脂肪酸[5,21-22]。因此,虽然I型和II型脂肪酸合成系统的生化反应相似,但参与催化的酶在结构和组成上有很大差异,这使得细菌脂肪酸合成途径成为开发新型抗菌药物的热门靶点[23-24]。
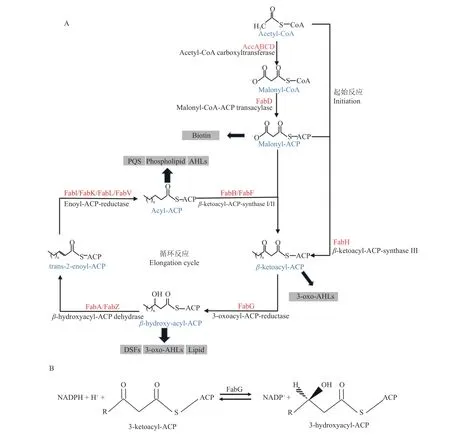
图1 细菌脂肪酸合成途径(A)与OAR催化反应机制(B)Fig. 1 The pathway of fatty acid biosynthesis in bacterial (A) and the reduction of 3-oxoacyl-ACP catalyzed by 3-oxoacyl-ACP reductase (B)
FAS II可分为起始反应和循环反应2部分(图1A)[18]。以大肠埃希菌Escherichia coli脂肪酸合成途径为例,起始反应的底物为乙酰-CoA,由乙酰辅酶A羧化酶(AccABCD)将其转化为丙二酸单酰-CoA,接着在丙二酸单酰辅酶A:ACP酰基转移酶(FabD)的催化下生产丙二酸单酰ACP,完成脂肪酸合成的起始反应[25]。循环反应的第1次循环由3-酮脂酰ACP合成酶III(FabH)将丙二酸单酰ACP和乙酰-CoA缩合,生成3-酮基丁酰ACP开始[26];接下来由3-酮脂酰ACP还原酶(FabG)催化,将3-酮基丁酰ACP还原为3-羟基丁酰ACP;然后由3-羟脂酰ACP脱水酶(FabA或FabZ)催化3-羟基丁酰ACP生成反-2-丁烯酰ACP[27-28];最后由烯脂酰ACP还原酶(FabI)催化反-2-丁烯酰ACP,生成丁酰ACP,至此完成第1次循环[29-30]。接下来的循环反应,由3-酮脂酰ACP合成酶I或II(FabB或FabF)催化脂酰ACP与丙二酸单酰ACP缩合开始,之后经FabG还原,FabA或FabZ脱水和FabI再次还原完成[31-32]。每一轮循环增加2个碳原子,直到合成棕榈酰ACP(C16-ACP)或硬脂酰ACP(C18-ACP)[33]。在大肠埃希菌中不饱和脂肪酸也可由FAS II酶催化合成。当碳链延伸到反-2-癸烯酰ACP时,双功能酶FabA行使其癸烯酰ACP异构酶的功能将反-2-癸烯酰ACP异构为顺-3-癸烯酰ACP,顺3-癸烯酰ACP不能被FabI催化,而是被FabB缩合进入下个循环反应,最终保留双键形成棕榈油酰ACP(C16:1)或异油酰ACP(C18:1)[34-35]。
2 细菌脂肪酸合成中的3-酮脂酰ACP还原酶
细菌脂肪酸合成反应中,3-酮脂酰ACP还原酶(OAR)催化3-酮基脂酰ACP还原为3-羟基脂酰ACP。OAR是氧化还原酶,以NADPH为辅因子,极少数的OAR也可以利用NADH[23,36-37]。与参与细菌脂肪酸合成的其他酶不同,细菌OAR较为保守,目前已经报道的参与细菌脂肪酸合成的OAR均为大肠埃希菌FabG的同源蛋白,且编码基因均处在脂肪酸合成簇中。
大肠埃希菌FabG是首个报道的OAR,也是研究最为透彻的OAR。fabG基因位于脂肪酸合成基因簇plsX-fabH-fabD-fabG-acpP-fabF中。Zhang等[38]证明,fabG与上游的fabD基因共转录,在fabD与fabG的间隔序列中插入转录终止元件可使fabG无法转录。此外,转录终止元件的插入对大肠埃希菌是致死的,也证明了fabG是必需基因[39]。Lai等[40]构建了大肠埃希菌fabG的温度敏感突变菌株CL104。CL104在30 ℃时能够正常生长,在42 ℃时,由于FabG蛋白稳定性降低,菌体不能生长,这再次证明FabG是大肠埃希菌唯一参与脂肪酸合成的OAR。
类似的脂肪酸合成基因簇在细菌中广泛存在。王海洪课题组先后对乳酸乳球菌Lactococcus lactis、茄科雷尔氏菌Ralstonia solanacearum、苜蓿中华根瘤菌Sinorhizobium meliloti、野油菜黄单胞菌Xanthomonas campestrispv.campestris和铜绿假单胞菌Pseudomonas aeruginosa脂肪酸合成基因簇中的fabG基因开展研究,结果证明这些fabG均为必需基因,且参与脂肪酸合成(图2)[41-45]。

图2 不同细菌中的脂肪酸合成基因簇与OAR编码基因Fig. 2 The fatty acid biosynthesis gene cluster and gene enconding 3-oxoacyl-ACP reductase in different bacteria
除脂肪酸合成基因簇中的fabG被注释为OAR编码基因外,细菌基因组还有多个基因被注释为OAR编码基因。例如乳酸乳球菌基因组中有2个OAR编码基因:fabG1位于脂肪酸合成基因簇,fabG2所处的基因簇也与脂肪酸合成相关。但是研究表明只有fabG1编码的蛋白参与脂肪酸合成。茄科雷尔氏菌基因组包含1条染色体和1个巨质粒,生物信息学分析至少有4个基因被注释为OAR编码基因。fabG1(RSc1052)、fabG3(RSc0435)、fabG4(RSc05 36)位于染色体上,而fabG2(RSp0359)位于巨质粒上。Feng等[42]研究表明,fabG2虽然能被敲除,突变菌株的脂肪酸组成显著改变,3-羟基脂肪酸合成量减少,而不饱和脂肪酸合成量增加,但是发现只有fabG1是必需基因,不能被敲除。在野油菜黄单胞菌基因组有4个被标注为OAR家族的编码基因:fabG1(XCC1018)、fabG 2(XCC0416)、fabG3(XCC4003)和fabG4(XCC0384)。研究显示FabG1是一个典型的OAR,与大肠埃希菌FabG的序列一致性达到69.1%。FabG1在体内和体外均能够将3-酮脂酰ACP还原为3-羟脂酰ACP,fabG1是必需基因,只有在过表达其他OAR时才能将其敲除[44]。同样,铜绿假单胞菌中有12个基因被标注为OAR家族基因,然而只有位于脂肪酸合成基因簇的PA2967(fabG)参与脂肪酸合成[45];Cukier等[46]也通过构建条件突变菌株证明fabG是铜绿假单胞菌的必需基因。
2.1 细菌3-酮脂酰ACP还原酶结构特点
细菌脂肪酸合成中的OAR属于短链醇脱氢酶/还原酶(Short chain dehydrogenase/reductase,SDR)超家族[47]。SDR超家族是最大的蛋白质超家族之一,至少有160000的蛋白属于此家族[48]。根据催化底物和关键活性残基的不同,SDR超家族可分为7个大类,464个家族。SDR超家族蛋白一级结构的序列一致性仅为15%~30%,但在序列和结构上有一些共同的特征。SDR超家族蛋白具有典型的Rossmann折叠结构,中心为6~7个β折叠,两侧各有3~4个α螺旋[49];催化活性残基为Tyr、Lys、Ser和Asn;辅因子为NADH或NADPH,辅因子结合位点位于N端[50]。与中链醇脱氢酶/还原酶(Medium chain dehydrogenase/reductase, MDR)超家族蛋白催化需要金属离子作为辅因子不同,SDR超家族蛋白催化不需要金属离子[51]。
OAR单体含有1个典型的Rossmann折叠结构,核心为7~8个β链构成的扭曲、平行的β折叠,8个α螺旋依附于两侧。OAR的辅酶结合位点Gly-Xaa3-Gly-Xaa-Gly位于N端,中部含有Ser-Xaa12-Tyr-Xaa3-Lys催化基序。以上信息表明,OAR的序列和结构符合SDR超家族蛋白典型特征,属SDR家族中的129C亚家族[48]。
科学家们已经对大肠埃希菌、鼠伤寒沙门氏菌Salmonella entericaserovar Typhimurium、炭疽杆菌Bacillus anthracis、金黄色葡萄球菌Staphylococcus aureus、肺炎克雷伯菌Klebsiella pneumoniae、鲍曼不动杆菌Acinetobacter baumannii、结核分枝杆菌Mycobacterium tuberculosis等超过20个细菌的OAR进行了结构分析,所有的OAR都有着类似的三级结构[50,52-56]。OAR一般为D2对称性的四聚体,即2个单体首先形成二聚体,2个二聚体再形成对称的四聚体。OAR结合NADPH后,形成3-酮基脂酰ACP-OAR-NADPH的复合体,OAR的构象发生改变,以容纳底物和辅因子,并传递电子[57]。
大肠埃希菌FabG是研究最为透彻的OAR,OAR的众多关键活性位点均是在FabG中报道的。Price等[54]首先解析了大肠埃希菌FabG的蛋白结构,FabG是典型的四聚体结构,4个单体形成2个二聚体界面,进而形成四聚体。FabG单体含有1个典型的Rossmann折叠结构,核心为7个β链构成的扭曲、平行的β折叠,通过8个α螺旋依附于亚基的两侧。2个单体的α4,α5螺旋和α4′,α5′螺旋形成疏水口袋,容纳3-酮基脂酰ACP的脂酰链。2个单体的β7折叠和α6/α7螺旋形成亲水的口袋,在结合NADPH及后续的构象改变中起着非常重要的作用(图3)。

图3 大肠埃希菌FabG的单体(A)和四聚体(B)晶体结构Fig. 3 The crystal structures of Escherichia coli FabG monomer (A) and tetramer (B)
Price等[54]进一步解析了FabG的活性位点,Tyr229在空间上位于α6/α7螺旋相对的亲水口袋中,参与了氢键网络的形成,Arg129和Arg172在脂酰ACP和FabG的相互作用上起着重要作用。Price等[54]证明 Ser138、Tyr151和Lys155的突变体蛋白与NADPH的结合能力减弱、对乙酰乙酰辅酶A的催化活性急剧降低。保守基序Ser 138,Tyr151和Lys155是必需氨基酸,均集中在α5/β5催化口袋附近,在NADPH结合和电子传递中起着重要的作用。NADPH的结合会增强FabG对脂酰ACP的亲和力,降低最大结合浓度,引起蛋白构象的变化,导致Ser138、Tyr151和Lys155的重排。NADPH中核糖结构上的羟基、Ser-Tyr-Lys基序和四分子水形成质子传递通路。质子由NADPH提供,Tyr151羟基介导,最终完成3-酮脂酰ACP还原(图4A)[58]。
Ser138、Tyr151和Lys155突变体蛋白虽然对乙酰乙酰辅酶A的催化活性急剧降低,但FabG的天然底物是3-酮脂酰ACP而非脂酰辅酶A[59]。对茄科雷尔氏菌FabG1的研究表明突变Ser-Tyr-Lys任意残基并不会丧失OAR的功能,在42 ℃依然能够恢复大肠埃希菌CL104生长[42]。Hu等[60]对大肠埃希菌FabG催化活性位点展开研究,发现Ser138Ala、Tyr151Phe和Lys155Ala突变体蛋白在体外和体内都保有足够的OAR催化活性(图4B)。突变体蛋白不仅能互补大肠埃希菌CL104菌株,而且能互补鼠伤寒沙门氏菌温度敏感突变菌株CL65。在CL65中纯化表达突变体蛋白,突变体蛋白在体外能够在起始反应和延伸反应中完成脂肪酸合成。进一步以3-酮脂酰ACP为底物测定突变体蛋白活性,发现相对于野生型FabG,Ser138Ala、Tyr151Phe和Lys155Ala分别有82%、59%和50%的活力。这表明Ser138、Tyr151和Lys155并不是EcFabG的催化活性中心,更可能是作为维持催化中心结构的氨基酸存在。以上研究结果表明,OAR可能存在不同于典型SDR超家族蛋白的电子传递机制和保守催化活性中心。

图4 大肠埃希菌FabG假定催化机制(A)与突变体蛋白重建脂肪酸合成反应(B)Fig. 4 The postulated catalytic mechanism of Escherichia coli FabG (A) and reconstruction of fatty acid by mutant protein (B)
2.2 细菌3-酮脂酰ACP还原酶的抑制剂研究
相比于细菌脂肪酸合成系统的其他成员,OAR在不同细菌间较为保守,是非常优良的广谱抗生素开发靶位点[61]。有关筛选细菌OAR抑制剂的工作也取得积极进展[62-63]。
植物多酚类物质,尤其是黄酮类化合物对O A R具有抑制效果,如绿茶中的焙儿茶素(Epigallocatechin gallate,EGCG)及儿茶素是优良的抑菌剂。EGCG及儿茶素以FAS II系统作为抑制靶点,有效地抑制了脂肪酸延伸过程中FabG和FabI所催化的还原反应[64]。单宁酸及枫叶提取物革兰阳性菌的抗菌活性也来自对OAR的抑制[65]。有研究表明,类黄酮和大环内酰亚胺对FabG有抑制作用,且为非特异性抑制,所以不能应用于临床[66];鲎素能够抑制4种革兰阴性细菌的OAR,但存在细胞毒性和溶血作用[67]。反式肉桂酸及其衍生物也有很显著的OAR抑制能力,这些化合物不仅能阻遏NADPH结合,还能够与多个重要的氨基酸活性残基结合,阻止底物的靠近[68]。
Cukier等[46]筛选到16个铜绿假单胞菌FabG蛋白抑制物,其IC50仅为nmol级,是极具潜力的铜绿假单胞菌抗菌药物,对这些抑制物的结合位点进行分析,发现抑制剂均结合在FabG亚基相邻界面的变构位点;此外他们还发现抑制剂在FabG的结合主要依赖于疏水相互作用,但也有抑制剂依赖于特定的氢键与FabG结合。抑制剂一旦与FabG结合,蛋白-抑制剂复合物的构象发生改变,无法与NADPH结合[24]。然而这些化合物对其他OAR的抑制效果并不好。
Vella等[56]筛选出一系列IC50在μmol级的抑制物,并且证明了这些抑制物与铜绿假单胞菌筛选到的抑制物一样,结合在亚基界面的变构位点上,尤其是α4/α5螺旋;通过对变构位点的研究证明Asn86羰基、侧链基团Tyr151和Lys155参与结合NAPDH的氢键形成,而多肽链骨架上的Gly182和Ile184也可能参与;抑制剂的结合位点主要由疏水残基组成,通过一个额外的极性相互作用作用于亚基表面,抑制物主要阻遏NADPH和OAR的相互作用。
Varakala等[67]通过高通量从细菌中筛选出43个可以抑制多个FabG的合成物,其中14个为吲哚类,29个为酰胺类;对药物动力学的研究表明,其中7个可以在μmol级抑制多个FabG的活性;5-溴-2-(噻吩-2-甲酰胺)苯甲酸和6-溴-2-(二甲氨基)甲基)-5-羟基-1-苯基-1H-吲哚-3-甲酸乙酯能够抑制6个FabG,且IC50为7~70 μmol/L,2-(2,6-二氯苄基)硫代)吡唑[1,5-a][1,3,5]三嗪-4(3H)-酮能够抑制4个FabG。
3 其他类型的3-酮脂酰ACP还原酶
许多细菌基因组中,除了fab基因簇的fabG基因外,还有多个基因被标注为OAR编码基因。这些基因大体可分为2类:一类编码的蛋白在体外和体内均具有OAR活性,但它们不参与脂肪酸合成,而是专一地参与一些次生代谢物的合成,执行特殊的生物学功能;另一类编码的蛋白没有OAR活性,具体的生物学功能有待深入研究。
3.1 分枝杆菌的MabA参与分枝酸的合成
分枝酸是分枝杆菌属细菌细胞壁的特殊成分,是一类含60~90个碳原子的长链3-羟基脂肪酸。研究证明分枝杆菌的膜磷脂脂肪酸由I型脂肪酸合成系统(FAS I)生成,而分枝酸的合成则由FAS II催化。分枝杆菌首先通过FAS I以乙酰-CoA为底物合成16~26碳的脂肪酸链,接下来利用FAS II将碳链进一步延伸到48~60个碳,进而形成分枝酸。分枝杆菌的FAS II 相关基因包括fabD-acpMkasA-kasB-accA基因簇和mabA-inhA基因簇。
Banerjee等[69]克隆了结核分枝杆菌的mabA(Rv1483,MtFabG1),并纯化得到MabA蛋白,MabA蛋白能够消耗NADPH,并还原乙酰乙酰辅酶A。这表明MabA蛋白具有O A R活性。Marrakchi等[70]证实MabA参与结核分枝杆菌的分枝酸合成,对短链的3-酮脂酰脂肪酸的亲和力很低,而对长链(C8~C16)的3-酮脂酰脂肪酸的亲和力较高。Parish等[71]证实inhA与mabA共转录;且只有在外源携带有编码mabA-inhA基因簇的质粒存在时才能将染色体上的mabA敲除,这表明mabA是必需基因。此外Parish等[71]也发现大肠埃希菌fabG并不能替换结核分枝杆菌的mabA,而耻垢分枝杆菌Mycobacterium smegmatis的mabA可以替换结核分枝杆菌mabA,并且替换菌株的分枝酸和细胞通透性没有改变。这表明分枝杆菌FAS II系统有独特的OAR,参与分枝酸的合成。
3.2 苜蓿中华根瘤菌NodG参与结瘤因子合成
除染色体外,苜蓿中华根瘤菌还有1个巨质粒SymA,与共生结瘤相关的nod基因簇位于该巨质粒上。nod基因簇中的nodG被标注为OAR,其编码的蛋白与大肠埃希菌FabG的序列一致性达到53%。nodG不是苜蓿中华根瘤生长的必需基因,敲除后并不影响菌体脂肪酸生成。但是体外和体内试验显示NodG具有OAR活性(图5A)。同时证明,在过表达nodG的情况下可以敲除fabG,且突变菌株的脂肪酸组成与野生菌株一致。这表明nodG在苜蓿中华根瘤菌中行使与fabG相似的功能。然而遗传学的研究发现nodG缺失会导致苜蓿结瘤效率降低,并证明其专一性地参与结瘤因子的合成(图5B)[43]。

图5 苜蓿中华根瘤菌NodG具有OAR活性并参与苜蓿结瘤Fig. 5 The Sinorhizobium meliloti NodG maintains OAR activity and involves in alfalfa nodulation
3.3 野油菜黄单胞菌FabG2是一种新型OAR
可扩散信号分子DSF介导黄单胞菌的群体感应调控,目前在黄单胞菌中已经鉴定到4种DSF类信号,它们的化学本质是主碳链含12个碳原子的顺-2-不饱和烯酸[72-73]。Yu等[13]研究表明野油菜黄单胞菌的3-酮脂酰ACP合成酶III能以支链CoA为底物合成支链脂肪酸,同时决定了野油菜黄单胞菌产生多种类型的DSF类信号。
在野油菜黄单胞菌中,与fabG1不同,fabG2是1个独立基因,其编码的蛋白与大肠埃希菌FabG的序列一致性仅为32.4%。Hu等[44]的研究发现FabG2对短链3-酮脂酰ACP的催化活性低,单独fabG2不能恢复大肠埃希菌fabG温度敏感突变菌CL104的生长,也不参与野油菜黄单胞菌脂肪酸的从头合成。然而在与哈氏弧菌Vibrio harveyi脂酰ACP合成酶基因(aasS)共转录,并外源添加辛酸后,fabG2可以使CL104在非许可温度生长,表明FabG2对中长链的3-酮脂酰ACP具有较强的催化活性(图6A)。另外研究发现,在添加有辛酸时,过表达fabG2能够获得fabG1敲除突变菌株(图6B)。更为重要的是敲除XccfabG2导致野油菜黄单胞菌的DSF类信号合成量显著降低(图6C)。这表明FabG2是1个专一性参与DSF类信号合成调控的新型OAR。

图6 XccfabG2互补OAR突变菌株及参与Xcc DSF类信号分子合成Fig. 6 XccfabG2 complements OAR mutant strain and invovles in DSF signal synthesis in Xcc
3.4 野油菜黄单胞菌FabG3参与菌黄素合成
在野油菜黄单胞菌野油菜致病变种中,已经鉴定了3个OAR的生物学功能。FabG1参与脂肪酸合成,FabG2参与DSF类信号分子合成调控,而fabG3位于菌黄素合成基因簇中。Yu等[74]的研究结果表明,FabG3在体外和体内均能行使OAR的功能(图7A),但fabG3的缺失并不会导致Xcc的生长或脂肪酸组成发生改变;然而突变菌株菌黄素合成受阻,同时证明这一性状不能由其他OAR编码基因互补恢复,只能由fabG3互补恢复(图7B)。这些研究结果表明,FabG3是一种专一性参与菌黄素合成的OAR。

图7 XccfabG3互补CL104并参与菌黄素合成Fig. 7 XccfabG3 complements CL104 and invovles in xanthomonadin synthesis in Xcc
3.5 其他OAR编码基因功能
铜绿假单胞菌是一种重要的条件致病菌,脂肪酸合成系统在铜绿假单胞菌的多种致病过程中起重要作用。在PAO1的基因组中,有42个SDR超家族基因,12个标注为OAR家族基因。Guo等[45]对铜绿假单胞菌基因组中的候选基因进行了系统研究,结果发现除fabG外,只有PA4389和PA4786具有OAR的活性;PA4389和PA4786在体内和体外均能行使OAR功能。然而,PA4389和PA4786并不参与铜绿假单胞菌的脂肪酸合成,这2个基因突变后,铜绿假单胞菌的脂肪酸组成并没有显著改变,但信号分子2-庚基-3-羟基-4-喹诺酮类(PQS)合成量减少50%以上。这表明PA4389和PA4786虽然不直接参与铜绿假单胞菌的脂肪酸合成,却影响其信号分子的合成。
Guo等[45]研究表明其他的OAR家族候选基因虽然不具备OAR活性,但在其他代谢途径中发挥作用。PA3128与苜蓿中华根瘤菌参与TCA循环的关键基因SMc02486的氨基酸序列有60%的一致性。PA3128被敲除后,突变菌株的PQS合成量提高了20%,弹性蛋白酶水解酶LasB的活性降低20%。在基础培养基上,以L-丙氨酸或D-海藻糖为碳源时,PA3128的突变菌株生长缓慢,最终得到的生物量仅为野生型的20%~40%,而以D-天冬氨酸为唯一碳源时,PA3128的突变菌株完全不能生长。以上结果表明PA3128可能也参与铜绿假单胞菌的TCA循环。而PA0182和PA1470在铜绿假单胞菌利用L-丝氨酸、L-天冬氨酸、L-谷氨酸和L-组氨酸时发挥重要作用,任何单突变菌株均不能利用以上氨基酸。PA3387(RhlG)曾认为参与铜绿假单胞菌重要致病因子鼠李糖脂的合成,然而Zhu等[33]的研究表明无论在体外还是体内RhlG都不能催化3-酮基癸酰ACP还原为3-羟基癸酰ACP,并不参与鼠李糖脂的合成。
在天蓝色链霉菌Streptomyces coelicolor中,脂肪酸可以用于次级代谢产物十二烷基二苷酸的生物合成。天蓝色链霉菌基因组的3个OAR候选基因编码的蛋白均能够催化β-酮脂酰-N-乙酰半胱氨酸。其中SOC1815是脂肪酸合成的关键酶;而SOC1345能够区分β-酮脂酰-N-乙酰半胱氨酸-ACP和β-酮脂酰-ACP;SOC1846的生物学功能尚不清楚[75]。
4 总结与展望
细菌脂肪酸合成机制高度保守,这不仅表现在不同细菌脂肪酸合成相关酶的蛋白序列高度相似,而且相关编码基因在染色体上的排列顺序也非常相似。但是研究发现不同细菌的脂肪酸合成机制存在差异,主要体现在:1)同种细菌中催化同一生化反应的酶有多种;2)不同细菌催化同一生化反应的酶的结构不同;3)不同细菌合成同样产物的方式和机制不同。
生物信息学分析显示细菌基因组中有许多注释为OAR的编码基因,这些基因编码的蛋白有些具有OAR活性,且能够遗传互补大肠埃希菌fabG温敏突变菌株,但是研究证明真正参与细菌脂肪酸合成的OAR由位于脂肪酸合成基因簇(fab)中的fabG编码。fabG是细菌的必需基因,突变该基因致死细菌。虽然有些OAR的编码基因,例如中华苜蓿根瘤菌nodG和野油菜黄单胞菌fabG2,在某些特殊条件下能够替代fabG,合成脂肪酸,维持菌体生长,但是NodG和FabG2不是FabG的同工酶,它们各自有自己的生物功能,催化不同物质的合成。因此,与参与细菌脂肪酸合成的其他酶不同,OAR只有FabG类同工酶,因此,FabG是一个理想的抗菌药物筛选靶标,可用于广谱抗菌药物的筛选。
除了参与细菌脂肪酸合成的FabG外,细菌中还有一些具有OAR活性的蛋白,目前研究证明这些OAR往往专一地参与脂肪酸相关物质的合成,如分枝酸、结瘤因子、菌黄素等。这表明这些酶对3-酮基脂酰基团具有还原能力。因此,对这些OAR功能的鉴定将有助于研究一些与脂肪酸相关物质合成,至少可以推测这些物质合成中可能有3-酮基脂酰基团的还原反应。
FabG作为一个潜在的优良广谱抗菌药物的开发靶点,虽然引起了科学家们的高度关注,并据此筛选或设计了一些药物,但目前鲜见临床应用或工业化生产的报道。Ser-Tyr-Lys基序是SDR超家族蛋白的结构特征之一,也是该类蛋白的催化活性中心,然而大肠埃希菌FabG催化机制研究证明,Ser-Tyr-Lys基序在FabG催化3-酮基脂酰ACP还原中并不起关键作用,这表明FabG可能采用有别于一般S D R家族蛋白的催化机制。这也要求对FabG更深入地研究,进一步解析FabG的结构特点和催化特性,为以FabG为靶点开展药物设计奠定基础。
