结合GWAS先验标记信息的肉鸡RFI性状全基因组选择研究
2022-10-29杜永旺王一东陈智武赵桂苹郑麦青
杜永旺,黄 超,王一东,李 森,文 杰,陈智武,赵桂苹,郑麦青*
(1.中国农业科学院北京畜牧兽医研究所,北京 100193; 2.广西金陵农牧集团有限公司,南宁 530049)
饲料成本占据肉鸡养殖成本的70%左右,而遗传选育是改进饲料利用效率的主要途径。家禽育种由最初的表型选择、家系选择正逐渐过渡到分子育种。随着单核苷酸多态性(single nucleotide polymorphism, SNP)等分子标记的开发与应用,将部分功能验证的候选标记联合BLUP计算育种值的分子标记辅助选择不仅可以提高选择准确性,还可以实现早期选择,缩短世代间隔。但是大多数畜禽的经济性状都是受到微效多基因控制的数量性状,获取具有大效应的变异位点较为困难,这也是分子标记辅助选择存在的局限性。而基因组选择(genomic selection, GS)覆盖了全基因组范围内的分子标记,能更好的解释遗传变异。2006年在奶牛上的研究发现了GS在育种工作中的巨大潜力。GS方法主要包括以GBLUP为代表的直接法和以贝叶斯类方法为代表的间接法。间接法虽具有计算准确性较高的优势,但是计算时间长,占用计算资源大的问题导致其在生产中的应用受限。而GBLUP凭借计算时间短,使用简单的优势广泛应用在猪、牛、羊、鸡等畜禽的实际选育当中。在GBLUP模型中,默认所有的SNPs位点对性状均有效应且效应相同,即未考虑先验信息。但是实际上不同的数量性状的遗传机制不同,复杂程度也不同,因此GBLUP方法存在一定的改进空间。全基因组关联分析作为挖掘畜禽经济性状候选位点的重要手段,是基因组水平上最常用的先验信息获取方式。对人类复杂性状、畜禽重要经济性状以及模拟数据的研究结果均表明,整合GWAS先验信息可以提高GS的准确性。
金陵花鸡属于快大型黄羽肉鸡,对生长速度和饲料消耗的选育要求较高,其出栏日龄为56日龄。出栏前两周为金陵花鸡的快速生长期,选育快速生长期的剩余采食量对生产具有重要的经济价值。本研究以广西金陵花鸡为试验素材,以42~56日龄的剩余采食量为目标性状,将试验群体随机分为两组,其中一组作为先验标记信息发现群体,用于GWAS分析并筛选出最显著的top5%、top10%、top15%和top20%的SNPs作为先验标记信息;另外一组分别结合不同合集的先验标记信息进行遗传参数估计并比较基因组育种值的预测准确性,使用重复10次的五倍交叉验证法获取准确性,随后两组群体再进行交叉验证。比较两种策略的预测准确性,为在金陵花鸡剩余采食量性状选育中实施基因组选择提供理论依据。
1 材料与方法
1.1 试验材料
本试验所使用的材料是由广西金陵农牧集团提供的快速型黄羽肉鸡金陵花鸡E系。该群体在0~42日龄采用群体饲养,在42~56日龄进行剩余采食量测定,测定期间使用独立的饮水和采食装置,所有鸡只自由采食,按照正常程序免疫和饲养管理。记录42~56日龄期间的采食量和体重增长量,并计算剩余采食量。剩余采食量的计算方法为:
=+++++
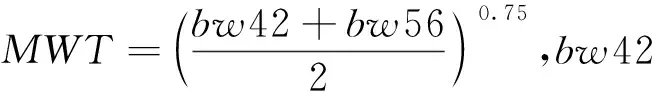
本研究使用3个世代共2 510个个体,其中公鸡1 648只,母鸡862只,均具有完整的系谱和表型记录。将群体随机分为两组,分组1和分组2各包含公鸡824个,母鸡431个,两组RFI的性状描述性统计见表1。
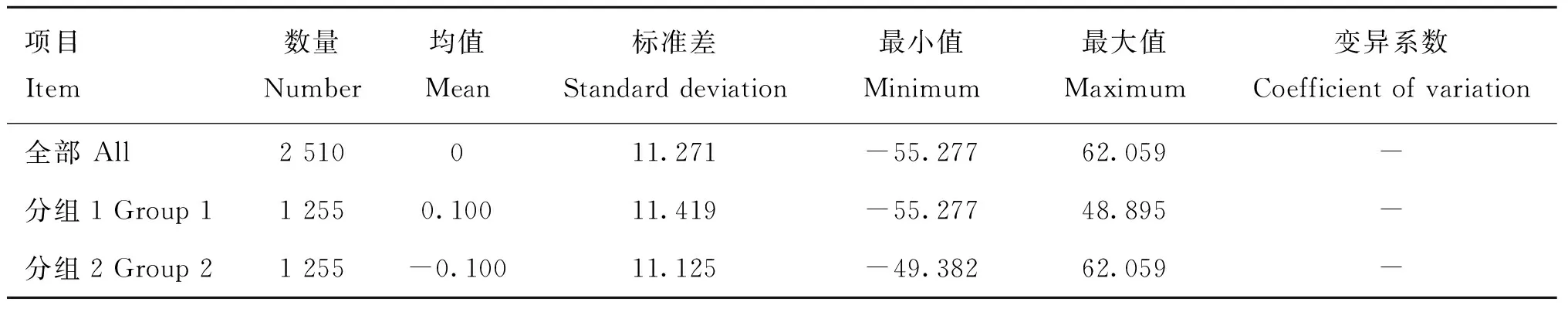
表1 剩余采食量的描述性统计Table 1 Descriptive statistics of RFI
1.2 基因分型与质控填充
试验群体在56日龄翅下静脉采血用于基因组DNA提取,并基于Illumina测序平台使用鸡55K SNP芯片对2 510个个体进行基因型检测,共得到44 561个SNPs,使用PLINK v1.9软件对数据进行质量控制处理,质控标准为:1)个体检出率大于0.95;2)最小等位基因频率(MAF)大于0.01;3)位点检出率大于0.95,缺失的SNPs使用Beagle v5.0软件进行填充,最终保留2 510个个体和40 492个SNPs用于后续研究。
1.3 全基因组关联分析
两组试验群体互相作为先验信息发现群体和先验信息验证群体。使用先验信息发现群体进行GWAS分析。所用模型为一般线性模型,具体为:
=++
其中,为所测定的性状,即RFI,为基因型数据,这里指每一个SNP,为SNP的系数,为世代、性别等因素,为性别的系数,为残差。将每个SNP作为固定因子进行回归分析,进行显著性检验,获取每一个SNP的值,并分别提取值较小的top5%、top10%、top15%、top20%的SNPs作为另外一组的先验标记信息。
1.4 基因组选择
获取的先验标记信息在先验信息验证群体上进行验证。GS使用的GBLUP模型为:
=b+μ+
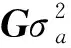
GBLUP使用的亲缘关系矩阵是由VanRaden提出的,计算公式为:

其中,为×标准化的基因型矩阵,为标记个数,为分型个体数,为第个位点的最小等位基因频率。
结合GWAS先验信息的GS所用的亲缘关系矩阵构建方式为:使用GWAS结果中的先验标记信息构建亲缘关系矩阵,剩余的位点构建亲缘关系矩阵,分别计算和对RFI解释的遗传方差,并以解释遗传方差的比例为权重拟合出亲缘关系矩阵:
=λ+(1-)

1.5 预测准确性评估标准
本研究使用重复10次的5倍交叉验证来评估基因组育种值的预测准确性,将GS分析群体随机分为5组,其中4组合并为参考群,剩下一组作为验证群,将得到的基因组育种值与表型值的皮尔逊相关系数除以遗传力开方作为预测准确性,最后将重复10次得到的均值作为评价预测准确性的标准。
2 结 果
2.1 GWAS结果
基于测定的RFI性状,将3个世代2 510个个体随机分为两组,分组1和分组2各包含824只公鸡和431只母鸡,分别对两个分组做GWAS分析,曼哈顿图见图1。获取最显著的top5%、top10%、top15%、top20%的SNPs作为另外一组的先验标记信息。结果中最显著的top5%有2 000个SNPs;top10%有4 000个SNPs;top15%有6 000个SNPs;top20%有8 000个SNPs,均散乱的分布在每条染色体上。
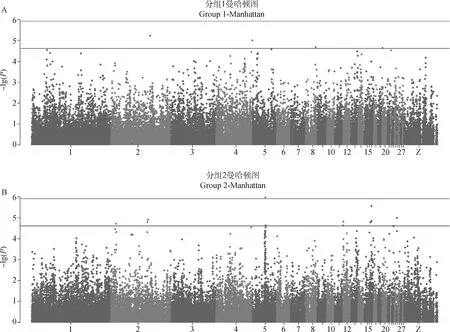
图1 曼哈顿图Fig.1 Manhattan plot
2.2 GS结果
分组1的遗传参数见表2,其中矩阵计算的遗传方差和环境方差分别为19.859和109.692,遗传力为0.153。五倍交叉验证的预测准确性结果为0.387。
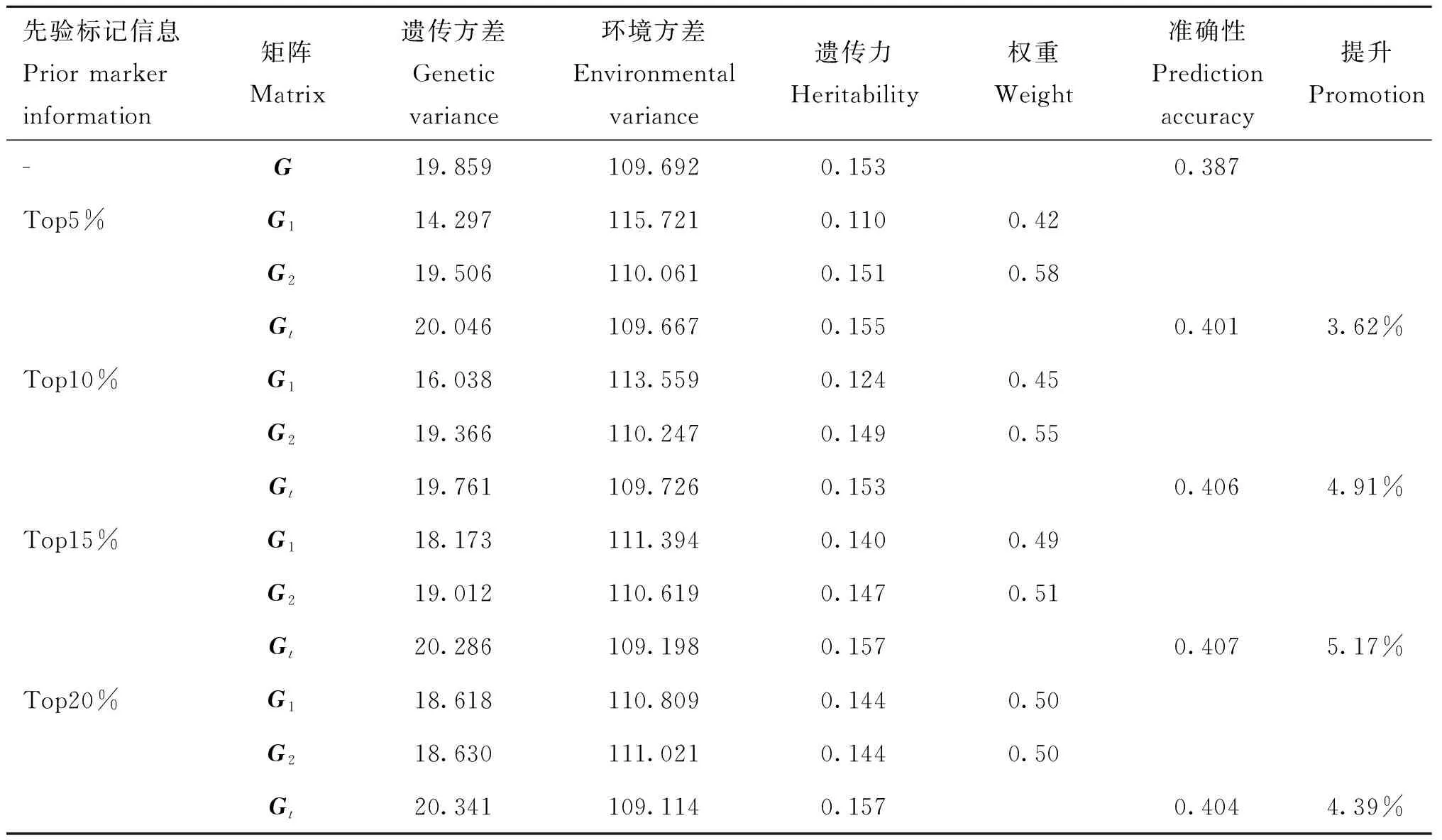
表2 分组1遗传参数Table 2 Genetic parameters of group 1
基于分组2的GWAS结果,筛选最显著的top5%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为14.297和115.721,遗传力为0.110;剩余95%的SNPs构建矩阵,解释的遗传方差和环境方差分别为19.506和110.061,遗传力为0.151。计算权重为0.42,权重为0.58。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为20.046和109.667,计算遗传力为0.155,预测准确性为0.401,准确性提升3.62%。
筛选最显著的top10%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为16.038和113.559,遗传力为0.124;剩余90%的SNPs构建矩阵,解释的遗传方差和环境方差分别为19.366和110.247,遗传力为0.149。计算权重为0.45,权重为0.55。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为19.761和109.726,计算遗传力为0.153,预测准确性为0.406,准确性提升4.91%。
筛选最显著的top15%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为18.173和111.394,遗传力为0.140;剩余85%的SNPs构建矩阵,解释的遗传方差和环境方差分别为19.012和110.619,遗传力为0.147。计算权重为0.49,权重为0.51。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为20.286和109.198,计算遗传力为0.157,预测准确性为0.407,准确性提升5.17%。
筛选最显著的top20%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为18.618和110.809,遗传力为0.144;剩余80%的SNPs构建矩阵,解释的遗传方差和环境方差分别为18.630和111.021,遗传力为0.144。计算权重为0.50,权重为0.50。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为20.341和109.114,计算遗传力为0.157,预测准确性为0.404,准确性提升4.39%。
分组2的遗传参数见表3,其中矩阵计算的遗传方差和环境方差分别为18.754和103.975,计算遗传力为0.153。五倍交叉验证的预测准确性结果为0.429。
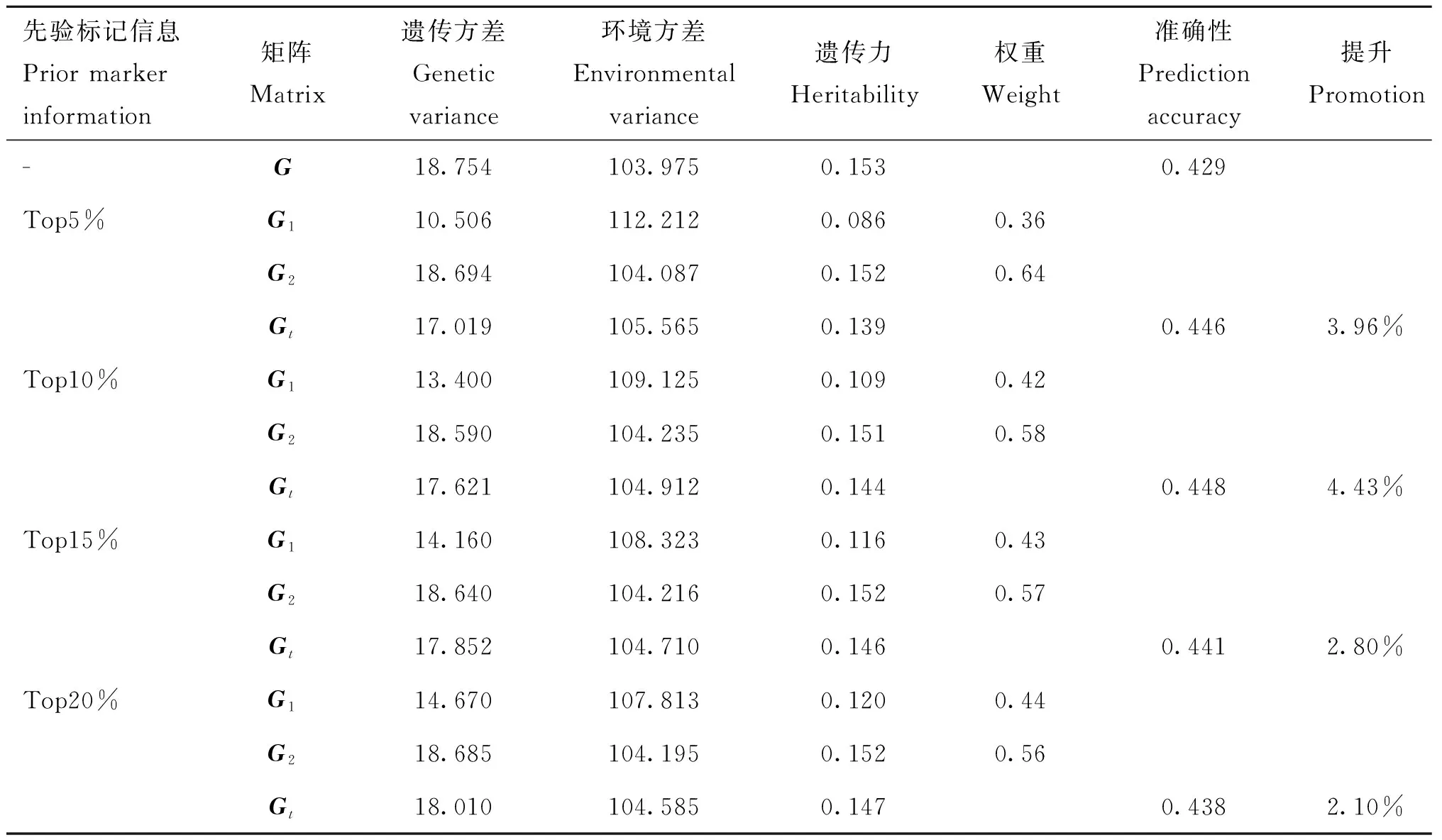
表3 分组2遗传参数Table 3 Genetic parameters of group 2
基于分组1的GWAS结果,筛选最显著的top5%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为10.506和112.212,遗传力为0.086;剩余95%的SNPs构建矩阵,解释的遗传方差和环境方差分别为18.694和104.087,遗传力为0.152。计算权重为0.36,权重为0.64。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为17.019和105.565,计算遗传力为0.139,预测准确性为0.446,准确性提升3.96%。
筛选最显著的top10%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为13.400和109.125,遗传力为0.109;剩余90%的SNPs构建矩阵,解释的遗传方差和环境方差分别为18.590和104.235,遗传力为0.151。计算权重为0.42,权重为0.58。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为17.621和104.912,计算遗传力为0.144,预测准确性为0.448,准确性提升4.43%。
筛选最显著的top15%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为14.160和108.323,遗传力为0.116;剩余85%的SNPs构建矩阵,解释的遗传方差和环境方差分别为18.640和104.216,遗传力为0.152。计算权重为0.43,权重为0.57。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为17.852和104.710,计算遗传力为0.146,预测准确性为0.441,准确性提升2.80%。
筛选最显著的top20%的SNPs构建亲缘关系矩阵,计算得到的遗传方差和环境方差分别为14.670和107.813,遗传力为0.120;剩余80%的SNPs构建矩阵,解释的遗传方差和环境方差分别为18.685和104.195,遗传力为0.152。计算权重为0.44,权重为0.56。使用结合GWAS先验标记信息的亲缘关系矩阵计算遗传方差和环境方差分别为18.010和104.585,计算遗传力为0.147,预测准确性为0.438,准确性提升2.10%。
3 讨 论
在本研究中,基于矩阵和矩阵计算的RFI遗传力范围处于0.139~0.157之间,为低遗传力性状,与李森等和Li等的研究结果相近,而在刘天飞等的研究结果中,RFI为中高遗传力,可能是由于本群体和其他研究群体的品种、饲养方式以及遗传背景不同导致。与传统的育种方法相比,基因组选择能够提高低遗传力性状的选育效果,在金陵花鸡RFI性状中应用基因组选择的选育效果较好。
在GBLUP模型中,默认所有的SNPs具有相同的效应且服从相同的分布,这显然是不合理的。有学者使用整合先验信息的GS模型进行了不同的改进,如基因组特征BLUP、BayesRC等。Zhang等提出对基因组关系矩阵进行优化,构建性状特异关系矩阵,即TABLUP,其基于间接法Bayes及岭回归最佳线性无偏预测方法给所有标记分配不同的权重,相较于GBLUP有显著的提升,但是BayesB计算时间较长、复杂程度高,预测准确性较高,基于BayesB提升GBLUP的准确性意义不大。2015年,Zhang等基于rrBLUP计算每个标记的效应值,并按照绝对值大小进行排序,分别构建两个不同的矩阵并相加,显著提升了预测准确性。同时,使用全基因组关联分析对标记分配权重也能提高预测准确性。而本研究基于GWAS分析,将SNP位点按照值大小进行排序,筛选对表型影响显著的SNPs,并将其作为先验信息整合至GBLUP模型中。
RFI作为受到微效多基因调控的低遗传力性状,在GWAS分析中没有定位到显著的SNPs。本研究从最显著的top5%的SNPs到top20%的SNPs中依次筛选并作为先验信息。研究结果中,随着top合集的扩大,矩阵解释的遗传方差逐渐扩大;在将top20%的SNPs作为先验信息的策略中,矩阵解释的遗传方差逐渐接近矩阵解释的遗传方差。这与Romé等认为20%的SNPs足以解释群体的连锁不平衡结果相近。
在本研究中,结合GWAS先验标记信息的GS相较于GBLUP提升了2.10%到5.17%。随着先验标记数量的增加,GWAS捕获到更多与RFI相关的SNPs位点,先验标记信息解释的遗传方差和遗传力也在不断增大,构建的矩阵权重也在逐渐增加。GS的预测准确性逐渐上升。当捕获到所有与RFI性状有关的位点时,再增加SNPs数量不会扩大遗传方差和遗传力,但是GS预测准确性会随着无关位点的干扰呈现下降趋势。整体来看,随着先验标记数量的增加,解释的遗传方差和遗传力逐渐增加,而预测准确性则呈现先上升再逐渐下降的趋势。在分组1中,先验SNP标记为top15%时取得最高预测准确性,提升5.17%;分组2中是在top10%取得最大提升,为4.43%。Gao等在黄羽肉鸡群体中,通过GWAS分析构建单倍型并整合基因注释信息到GS模型中,将RFI的预测准确性从0.464提升到了0.468,提升了0.86%。Zhang等利用GWAS检测到的QTL,构建性状特异性亲缘关系矩阵,即BLUP|GA,并在奶牛和水稻的真实数据中证明BLUP|GA提升了GS的预测准确性。袁泽湖在绵羊肉用性状中整合GWAS先验信息的GS准确性比基于芯片数据的GS准确性更高。Schrag等在玉米上的研究表明,结合基因组、转录组和代谢组等其他组学的先验信息也可以提高GS的预测准确性。目前,基因组选择已经广泛应用在实际生产中,对模型的改进也在不断进行,但是不同群体的选育目标和遗传背景均不相同。因此,在生产中应用基因组选择应该选择使用整合本群体生物学先验信息的GS模型并调整相关参数。
4 结 论
本研究以金陵花鸡为试验素材,基于GWAS分析结果中的值较小的top5%~20%的SNPs作为先验信息,并结合至GS模型中,对RFI进行了遗传参数估计和预测准确性分析。金陵花鸡RFI为低遗传力性状,将GWAS结果中最显著的top10%~top15%的SNPs作为先验信息,并结合至GS的策略可以将基因组育种值的预测准确性提高2.10%~5.17%,更适合本群体RFI性状的选育。
