一株来源于豆豉的地衣芽孢杆菌全基因组学及其风味物质形成分析
2022-10-17张盼文李浩徐钰红杨慧林王筱兰
张盼文,李浩,徐钰红,杨慧林,王筱兰
(江西师范大学 生命科学学院,江西 南昌,330022)
地衣芽孢杆菌(Bacilluslicheniformis)是一种革兰氏阳性兼性厌氧菌,广泛分布于自然界中,具有抗逆境能力强、抑制病原菌、产酶种类丰富等优良特性[1-2],非常适用于食品发酵工业生产。在传统食品曲霉型豆豉发酵过程中,地衣芽孢杆菌在大豆发酵过程中发挥着至关重要的作用,该菌具有良好的耐盐性能[3],发酵大豆的特征性芳香物质及关键的挥发性风味化合物和地衣芽孢杆菌息息相关[4]。这类菌可产生多种酶类分解生物大分子物质形成风味物质前体[5],从而促进了豆豉风味物质的产生。其分泌的糖苷酶[6]主要降解豆豉中的纤维素等糖类物质生成葡萄糖以及代谢途径中的其他小分子化合物,进而和氨基酸发生美拉德反应[7],赋予豆豉独特的香气。
杨林子等[8]基于GC-MS探究了郫县豆瓣酱中的地衣芽孢杆菌在化学体系中的酯合成途径和能力;SHAH等[9]对海洋地衣芽孢杆菌生产更高纤维素酶的发酵条件进行了优化;KURIBAYASHI等[10]对来自地衣芽孢杆菌的β-半乳糖苷酶在离子交换树脂中的固定化进行研究,以利于生产低乳糖或无乳糖乳制品。上述研究表明地衣芽孢杆菌有丰富的酶类资源和良好的发酵潜能,其代谢潜力有待进一步研究。目前已有一些学者利用基因工程和顶空固相微萃取-气相色谱-质谱法等技术探究及改善该菌的产酶及发酵性能[11],但是从全基因组角度上对于该物种在大豆发酵过程中风味物质产生机理的相关研究未见报道。
本课题组先前研究发现,地衣芽孢杆菌存在于传统豆豉发酵过程中的各个阶段,并且在发酵中后期占据优势地位[12]。豆豉发酵过程中,因为高温和拌盐的影响,葡萄球菌等大部分微生物含量都逐渐下降,而地衣芽孢杆菌的耐盐和抗菌特性及其分泌蛋白酶和糖苷酶的能力对豆豉后发酵阶段风味的产生仍有重要作用。因此,本研究利用全基因组测序技术获取地衣芽孢杆菌JXNUWL7完整基因组序列,分析预测该菌株可能的功能基因和风味物质代谢机理,同时通过GC-MS技术对其纯种发酵豆豉产生的挥发性物质进行测定分析,为地衣芽孢杆菌在豆制品发酵中的功能性作用及潜在应用价值方面的研究提供理论基础。
1 材料与方法
1.1 材料与试剂
JXNUWL7菌株,江西省南昌市稻香园调味食品有限公司生产的曲霉型豆豉中分离筛选获得;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;蛋白胨,北京奥博星生物技术有限责任公司;葡萄糖、NaOH、HCl、NaCl(分析纯),西陇科学股份有限公司;琼脂粉,北京索莱宝科技有限公司;酵母浸粉,安琪酵母股份有限公司。
1.2 培养基
脑心浸液肉汤BHI Broth,青岛高科技工业园海博生物技术有限公司;羧甲基纤维素钠(CMC-Na)培养基[13](g/L):CMC-Na 10,蛋白胨10,酵母浸粉5,NaCl 10,琼脂20,pH (7.2±0.2)。
1.3 仪器与设备
HICO21台式离心机,生工上海股份有限公司;PowerEase 90 W核酸凝胶电泳仪、HWS26电热恒温水浴锅,上海申安器械厂;QL-901旋涡振荡器,海门市其林贝尔仪器制造有限公司;T100型PCR仪,Bio-Rad公司;固相微萃取装置(包括手柄和DB/CAR/PDMS萃取头),Supelco公司;7890A/5975气相色谱-质谱联用仪,Agilent公司。
1.4 实验方法
1.4.1 全基因组测序样品制备
将实验室低温(-80 ℃)保藏的JXNUWL7菌株复苏后接种于100 mL BHI液体种子培养基中,35 ℃、170 r/min培养24 h后,8 000 r/min离心10 min,弃去上清液;参照细菌基因组DNA提取试剂盒说明书上的方法提取菌株的基因组。
1.4.2 全基因组测序和组装
地衣芽孢杆菌JXNUWL7的基因组DNA经提取后通过Illumina HiSeq×10测序平台完成全基因组测序,对符合要求的Clean data进行从头组装得到基因组序列,进行局部组装和优化后利用短序列组装软件SOAPdenoo2进行拼接。
1.4.3 菌种种属鉴定
仅通过16S rRNA基因序列分析的方法难以鉴别枯草芽孢杆菌菌群中的物种,编码DNA回旋酶B亚单位蛋白的gyrB基因[14]被选中作为一种替代的系统发育标记用于物种鉴定。根据菌株JXNUWL7全基因组注释结果,选取其中16S rRNA和gyrB基因序列提交至NCBI数据库进行BLAST比对分析,利用MEGA 7.0.26软件的邻位连接法构建系统发育树。
1.4.4 基因的预测与注释
利用tRNAscan-SE 2.0、Barrnap、Glimmer软件分别对该菌株基因组中包含的tRNA、rRNA和编码序列(coding sequence, CDS)进行预测;将该菌株全基因组序列与6大数据库(NR、Swiss-Prot、Pfam、EggNOG、GO和KEGG)比对进行功能注释,使用Diamond软件比对eggNOG数据库和KEGG数据库对菌株进行COG和KEGG功能注释,使用BLAST2 go软件比对GO数据库对菌株进行GO功能注释。
1.4.5 CAZymes酶类注释及验证
利用Diamond、hmmscan软件对菌株JXNUWL7的碳水化合物活性酶(carbohydrate-actie enzymes, CAZymes)进行预测。将活化的JXNUWL7菌株分别接入CMC-Na培养基中,在35 ℃恒温培养箱中培养3 d。用0.2%刚果红染色15 min,然后用1 mol/L NaCl溶液清洗2次后观察是否产生透明圈。
1.4.6 次级代谢产物合成基因簇分析
通过antiSMASH软件(http://antismash.secondarymetabolites.org)对样本的次级代谢产物合成基因簇进行预测。
1.4.7 菌株培养及单菌发酵豆豉
将实验室保藏的菌株JXNUWL7复苏后转接到100 mL液体种子培养基中,于37 ℃,170 r/min培养24 h,振荡培养到菌悬液的OD600为0.8时停止培养。将培养好的菌悬液和灭菌(121 ℃,20 min)处理的黑豆按1∶10(mL∶g)在超净工作台内进行接种,均匀混合后覆上灭菌过的保鲜膜,在42 ℃发酵25 d。培养结束后,用GC-MS分别测定3组平行样品挥发性风味物质。
1.4.8 风味化合物萃取
分别取发酵25 d的3组平行样品进行研磨,过40目筛后称取3 g样品加到15 mL顶空进样瓶中,60 ℃恒温水浴平衡20 min,将SPME装置置于进样瓶上方,60 ℃萃取30 min,通过GC-MS进行分析。
1.4.9 GC-MS分析条件
色谱条件:Agilent 19091S-433毛细管柱(30 m×250 μm×0.25 μm);进样口温度250 ℃,载气He,流速1 mL/min,分流进样;升温程序:起始40 ℃,保持5 min,5 ℃/min升至60 ℃,10 ℃/min升至250 ℃,保持5 min。
质谱条件:离子源EI,电离电压70 e;离子源温度230 ℃;四极杆温度150 ℃;MS1全扫描模式。
1.4.10 风味化合物的鉴定和含量计算
将各组分质谱信息与NIST17.L质谱库对照,确定各组分成分。以质谱峰面积代表各组分含量,用峰面积归一化法计算挥发性成分相对含量。
2 结果与分析
2.1 基因组概况及组分分析
对菌株JXNUWL7进行全基因组的测序,结果如表1所示,菌株全长为4 194 676 bp,GC含量为46.07%,包含45个重叠群(contigs),长度>9 000 bp的重叠群有31个。基因元件预测结果显示共有4 684个CDS,77个tRNA和8个rRNA,由基因长度分布图(图1)可得预测基因长度在150~299 bp的基因数量最多(667个,14.24%)。
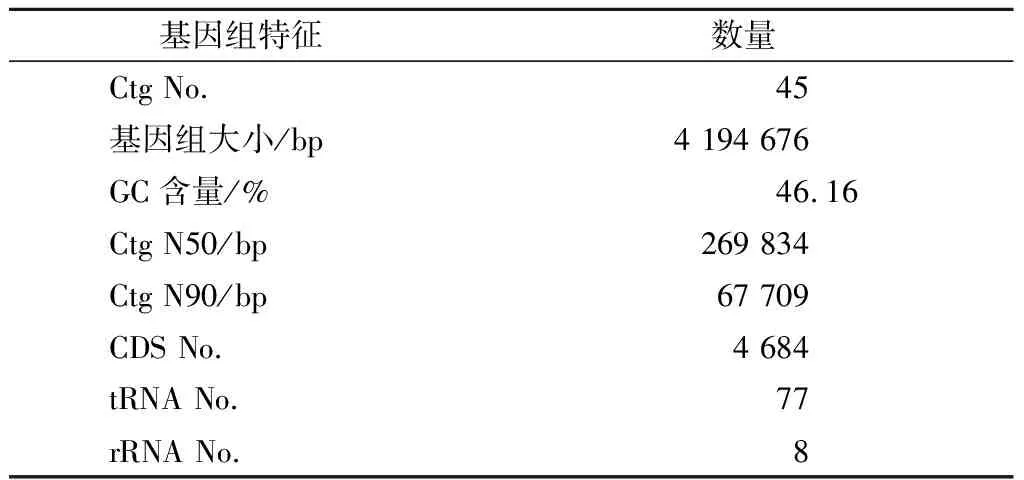
表1 菌株JXNUWL7基因组组装结果统计Table 1 Genome assembly results of strain JXNUWL7

图1 菌株JXNUWL7编码基因长度分布Fig.1 CDS length distribution of strain JXNUWL7
16S rRNA和gyrB基因构建系统发育树结果显示(图2),菌株JXNUWL7为地衣芽孢杆菌。将菌株JXNUWL7的全基因组序列提交至NCBI,获得登录号为:JAJAU000000000。
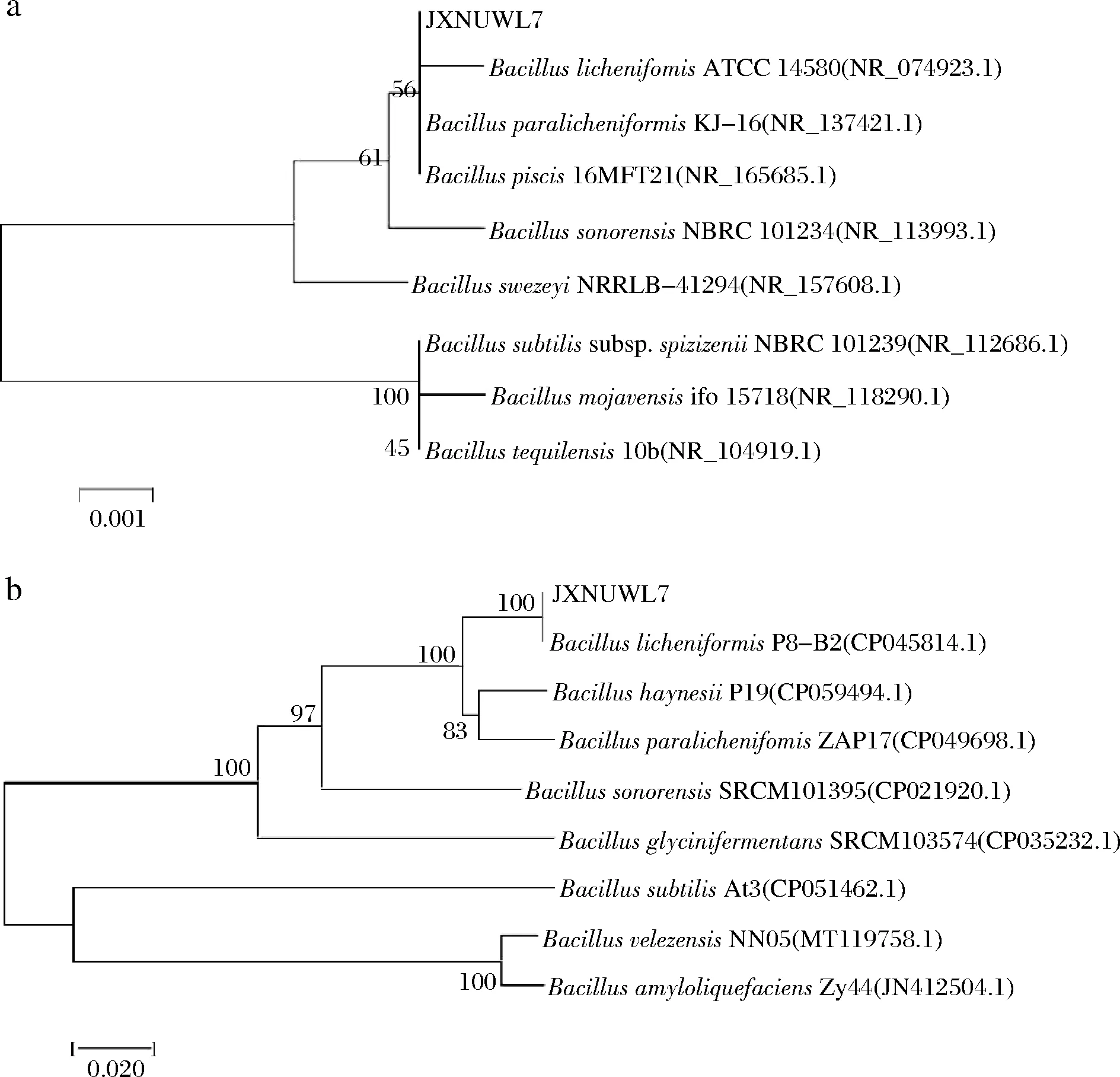
a-基于16S rRNA序列;b-基于gyrB序列图2 菌株JXNUWL7的系统发育树Fig.2 Phylogenetic tree of strain JXNUWL7
2.2 基因功能注释
2.2.1 基础注释
通过比对EggNOG数据库中的信息对菌株JXNU7基因组进行COG注释结果表明,共有3 379个CDS具有COG功能注释结果(图3),占基因总数的72.14%。其中碳水化合物代谢和氨基酸代谢是主要代谢过程,相关CDS分别为309个(9.14%)和300个(8.88%),主要的COG功能分别注释为磷酸转移酶系统(phosphotransferase system, PTS)组成成分和ABC转运蛋白。ABC转运蛋白参与物质运输,维持细胞渗透压平衡,可能和该菌的耐盐能力相关[15]。
GO注释结果(图4)显示有3 197个CDS注释归到3大类43个条目中,在生物学过程分类中,参与氧化还原过程和转录调控的CDS最多,分别为310个(6.62%)和265个(8.29%);在细胞成分分类中,膜的组成部分注释到的CDS数量最多,为1 024个(32.03%);在分子功能分类中,注释到与ATP结合和DNA结合功能相关的CDS最多,分别为355个(11.10%)和307个(9.60%)。

A-RA加工和修饰;B-染色质结构和动力学;C-能源生产和转化;D-细胞周期控制、细胞分裂、染色体分裂;E-氨基酸转运和代谢;F-核苷酸转运和代谢;G-碳水化合物运输和代谢;H-辅酶运输和代谢;I-脂质运输和代谢;J-翻译、核糖体结构和生物发生;K-转录;L-复制、重组和修复;M-细胞壁/膜/包膜生物发生;N-细胞运动;O-翻译后修饰、蛋白质周转、伴侣;P-无机离子转运和代谢;Q-次生代谢物的生物合成、运输和分解代谢;R-仅一般功能预测;S-功能未知;T-信号转导机制;U-细胞内运输、分泌和囊泡 运输;-防御机制;W-细胞外结构;Y-核结构;Z-细胞骨架图3 GOG注释分类统计Fig.3 COG annotation classification statistics chart
KEGG通路注释结果显示(图5),共2 276个基因注释到6个信号通路层次202条通路中。在代谢层次,与碳水化合物代谢(290个)和氨基酸代谢(209个)相关的基因占比最多。此外,KEGG共注释到了41个PTS系统,其中和纤维素代谢相关的PTS系统数最多(18个)。

图4 GO注释分类统计Fig.4 GO annotation classification statistics chart
如图6所示,JXNUWL7基因组中包含15条碳水化合物代谢通路信息,且淀粉和蔗糖代谢的基因数量最多(59个)。碳代谢注释结果表明菌株JXNUWL7可能代谢多种糖类,其中间产物将进入其他代谢通路进一步参与挥发性风味物质和功能性物质的合成。此外,地衣芽孢杆菌JXNUWL7含有129个与双组分系统相关的基因和128个ABC转运蛋白相关的基因,这些系统对于细菌感应环境刺激和适应环境胁迫有重要作用[16-17]。
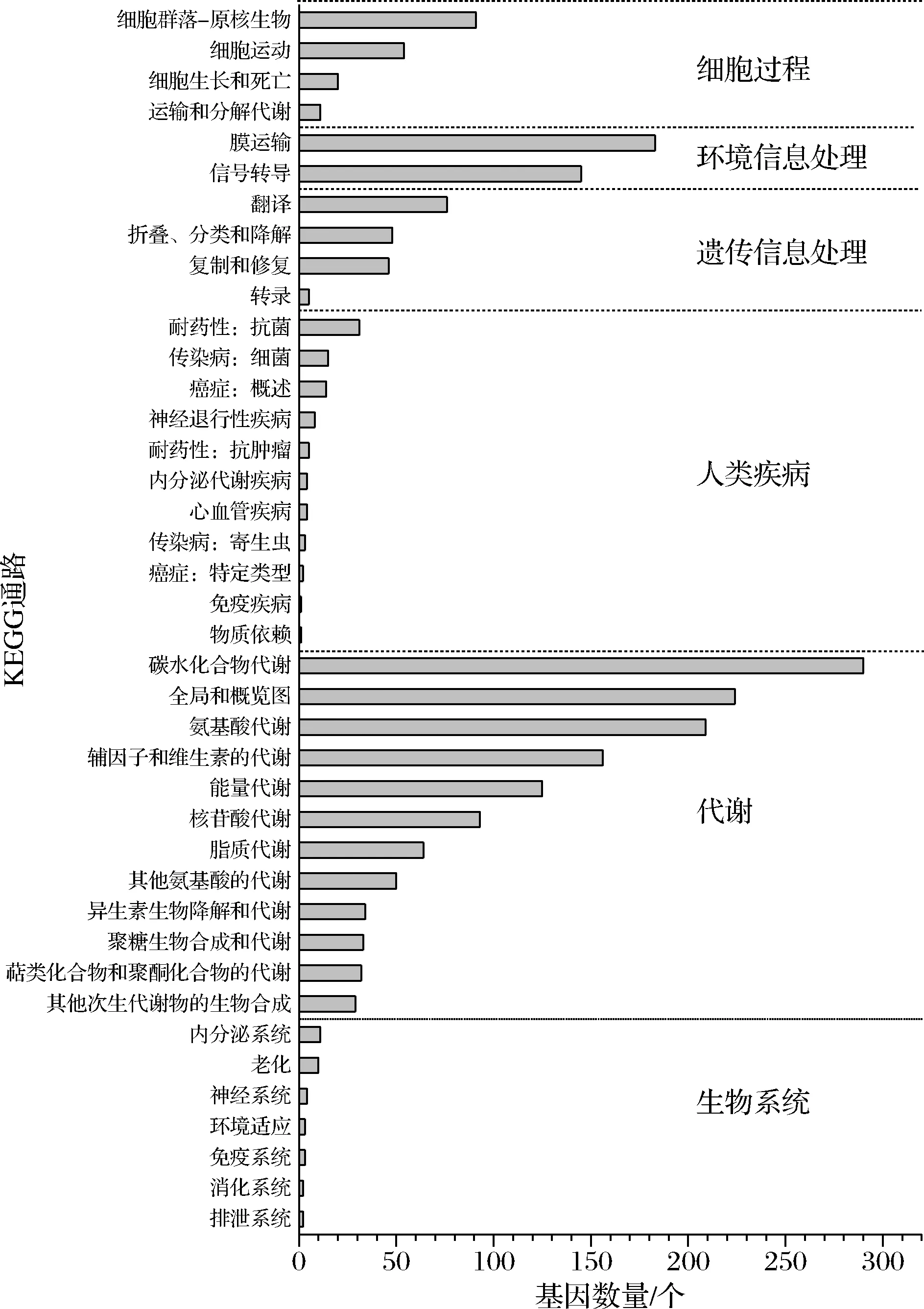
图5 菌株JXNUWL7的KEGG注释通路分类统计Fig.5 Classification statistics of KEGG annotation pathway about strain JXNUWL7
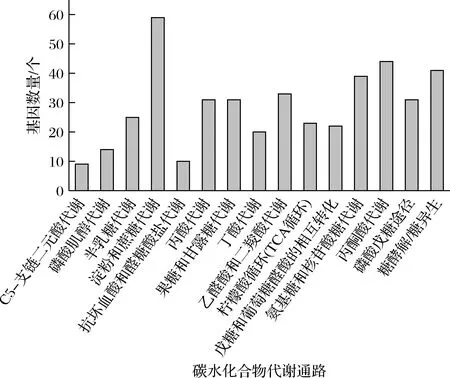
图6 JXNUXL7碳水化合物代谢通路统计图Fig.6 Statistical diagram of JXNUXL7 carbohydrate metabolism metabolic pathways
2.2.2 碳水化合物活性酶CAZy注释
将基因组序列与CAZy数据库进行比对,发现菌株JXNUWL7的基因组中共有116个基因编码的蛋白质结构域属于CAZy家族,包括30类糖苷水解酶家族(glycoside hydrolases, GHs)48个、10类糖苷转移酶家族(glycosyl transferases, GTs)34个、7类碳水化合物酯酶(carbohydrate esterases, CEs)14个、11类碳水化合物结合组件(carbohydrate-binding modules, CBMs)12个和7类多糖裂解酶(polysaccharide lyases, PLs)8个。其中注释为GT2、GT4、GT51、GH1、GH4、CE4和CE12的基因序列数量相对较多。研究表明,24%的原核生物中存在编码含有纤维素酶的GH家族的基因[18],其中GH1和GH3家族的基因编码β-葡萄糖苷酶,而其他GH家族存在编码内切和外切葡聚糖酶的基因。本研究在KEGG通路中,该菌株注释到的β-葡萄糖苷酶bglB和bglX分别属于GH1和GH3家族,它们在纤维素降解中有重要作用。
2.2.3 纤维素代谢
KEGG通路注释结果分析表明,共有290个基因和碳水化合物代谢相关,其中内切葡聚糖酶(EC:3.2.1.4)和葡萄糖苷酶(EC:3.2.1.20、EC:3.2.1.21)参与纤维素降解。为进一步确定菌株JXNUWL7是否可以降解纤维素,通过纤维素培养基结合刚果红染色法进行验证,结果表明该菌株能降解纤维素(图7)。通过KEGG注释结果分析菌株JXNUWL7的纤维素降解代谢相关基因并绘制出其主要的代谢通路(图8),对通路进行分析可知菌株JXNUWL7可以降解纤维素,并通过糖酵解和三羧酸(tricarboxylic acid,TCA)循环代谢生成的各类中间产物进一步参与氨基酸代谢和脂质代谢生成其他风味和功能性物质。根据对KEGG通路注释结果中风味物质合成相关途径分析的结果表明,该菌株可能通过莽草酸途径生成各类芳香族化合物,也可能通过丁酸代谢生成乙偶姻和丁二醇。与此同时,对脂肪酸代谢通路进行分析发现,该菌株因缺少关键酶CPT1的编码基因而无法代谢棕榈酰辅酶A,但包含从乙酰辅酶A合成不饱和酰基-[acp]的途径。这说明该菌株虽然对豆豉中粗脂肪的降解贡献不大,但可能代谢生成中长链风味物质,胡会萍等[19]所报道的和本研究一致。

图7 JXNUXL7 对纤维素的利用Fig.7 Utilization of JXNUXL7 for cellulose

CelC-PTS系统,特定于纤维二糖的ⅡA组件;CelF-6-磷酸-β-葡萄糖苷酶;Pgm-磷酸葡萄糖变位酶;GalU-UTP-葡萄糖-l-磷酸尿苷基转移酶;BglB/X-β-葡萄糖苷酶;AroH-分支酸变位酶;EntC-异分支酸合酶;EntB-双功能异分支酸裂解酶;EntA-2,3-二氢-2,3-二羟基苯甲酸脱氢酶;DhbE-2,3-二羟基苯甲酸酯-AMP连接酶;DhbB-芳基载体蛋白;DhbF-非核糖体肽合成酶;TrpE-邻氨基苯甲酸合酶组分Ⅰ;BioF-8-氨基-7-氧杂壬酸合酶;BioA-赖氨酸-8-氨基-7-氧杂壬酸氨基转移酶;BioD-脱硫生物素合成酶;BioB-生物素合酶;IlB-乙酰 乳酸合酶Ⅰ/Ⅱ/Ⅲ大亚基;AlsD-乙酰乳酸脱羧酶;ButA-丁二醇脱氢酶;Ldh-L-乳酸脱氢酶图8 JXNUXL7纤维素代谢通路和相关风味及功能性物质形成通路Fig.8 Cellulose metabolism pathway and the related flaors and functional substances formation pathways in JXNUXL7
2.3 次级代谢产物合成基因簇分析
利用antismash软件对样本的次级代谢产物合成基因簇进行预测分析,一共有9个次级代谢产物合成基因簇,具体的预测分析结果见表2。将菌株JXNUWL7所有基因簇与已知的次级代谢产物基因簇做BLAST比对后发现,有4个功能已知的合成基因簇和5个功能未知的合成基因簇,这说明菌株JXNUWL7基因组序列中可能存在新的活性物质合成基因簇。羊毛硫抗生素是一类作用于革兰氏阳性菌细胞膜上的热稳定抗菌肽[20],可以作为抗菌药物的替代品,地衣芽孢杆菌JXNUWL7基因组中羊毛硫抗菌肽基因簇(图9)与抗生素基因簇K21 A1/K21 A2(lichenicidin K21 A1/K21 A2)相似性达到100%,可能作为羊毛硫抗生素合成开发的目标菌种。该菌种含有由13个基因组成的铁载体基因簇,这些基因和芽孢杆菌类微生物的耐盐性能有关[21]。
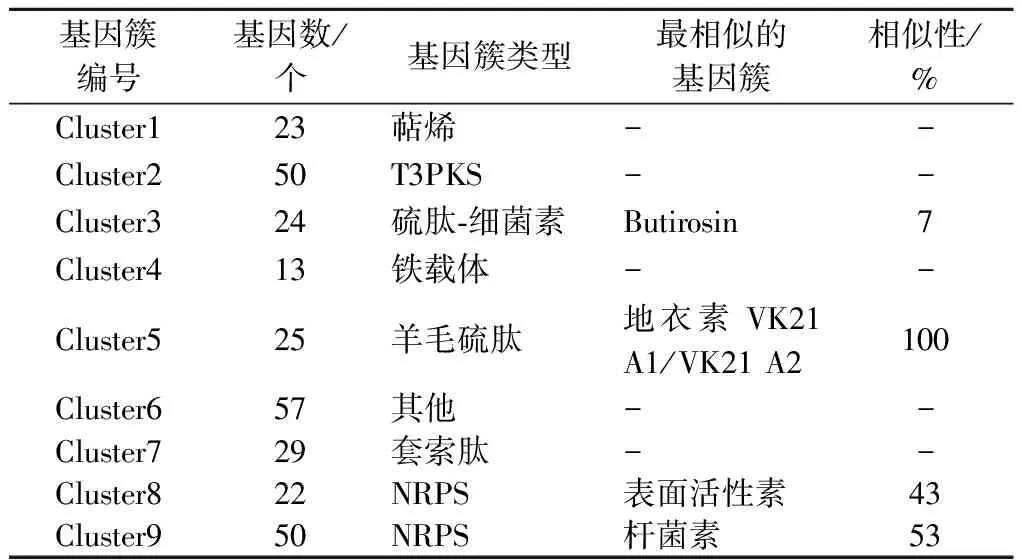
表2 JXNUWL7次级代谢产物合成基因簇预测分析结果Table 2 Prediction and analysis results of secondary metabolite synthesis gene cluster of JXNUWL7

图9 羊毛硫肽合成基因簇线性图谱Fig.9 Linear map of lantipeptide synthesis gene cluster 注:图谱展示预测基因簇中所有的基因,其中不同注释基因的颜色采用该基因在COG分类中的颜色的进行显示,不同颜色 代表的功能详见COG分析界面,灰色代表该基因没有注释到COG
2.4 挥发性风味物质测定
对该菌株发酵大豆的挥发性风味物质进行GC-MS测定,主要挥发性风味物质及相对含量见电子增强出版附件(https://doi.org/10.13995/j.cnki.11-1802/ts.030716),地衣芽孢杆菌JXNUWL7发酵黑豆样品中共鉴定出86种香气成分,主要包含9种酯类、6种醛类、15种酮类、7种吡嗪类。以往研究表明,地衣芽孢杆菌在大曲中主要产生吡嗪类、丁酸和丁酮类风味物质[22],但本研究中地衣芽孢杆菌发酵豆豉还可以产生大量芳香酯和苯乙酮类等对甜香和果香有重要贡献的挥发性风味物质,这可能是由于豆豉发酵环境中富含蛋白质。本研究对地衣芽孢杆菌JXNUWL7纯种发酵豆豉进行GC-MS分析结果表明,相对蒸煮黑豆的挥发性风味物质成分[23-24]而言,纯种发酵黑豆吡嗪类、芳香酯类、苯乙酮、长链酮类和丁酸类物质增加,而正己醇和甲基麦芽酚含量大幅下降,且苯甲醛含量几乎不变,这说明黑豆中的正己醇和甲基麦芽酚可能被地衣芽孢杆菌代谢利用。
3 结论
本研究探讨了菌株JXNUWL7基因组中可能涉及的糖苷酶种类,地衣芽孢杆菌JXNUWL7可能产生包括β-葡萄糖苷酶在内的66种碳水化合物活性酶,并含有丰富的纤维素代谢相关的磷酸转移酶系统,具备完整的纤维素降解通路,这对于降解纤维素等糖苷类物质生成相应的单糖,进而通过糖酵解和三羧酸循环等途径生成乙醇、乳酸、二磷酸尿苷葡萄糖等小分子物质作为风味物质和功能性物质前体具有重要作用,从而促进风味物质的产生。同时,通过GC-MS和全基因组测序对地衣芽孢杆菌JXNUWL7单菌发酵大豆的挥发性风味物质进行测定和分析,揭示出该菌株主要对豆豉芳香类、吡嗪类和中长链酮类风味物质有重要贡献,同时,解析出该菌株含有分支酸、乙偶姻、中长链酰基-[acp]等与风味物质合成相关的代谢途径。从基因层次上解读了该菌株在豆豉发酵过程中的耐盐和抗菌能力的原因。本研究通过全基因组学分析和GC-MS技术对地衣芽孢杆菌潜在的功能性基因进行挖掘以及挥发性风味物质形成进行分析,为研究曲霉型豆豉发酵过程中功能微生物的代谢机理奠定了基础。
