谷子GRAS转录因子家族的全基因组鉴定、表达分析及标记开发
2022-09-21王智兰韩康妮杜晓芬李禹欣连世超
王智兰 韩康妮 杜晓芬 李禹欣 连世超 王 军
(山西农业大学谷子研究所/杂粮种质资源发掘与遗传改良山西省重点实验室/杂粮种质创新与分子育种山西省重点实验室,山西 长治 046011)
GRAS基因家族编码一类植物特有的转录调控因子,参与信号转导、胁迫应答、分生组织形成等众多生物学过程,在植物组织的生长发育中发挥重要作用[1]。谷子作为世界性主要杂粮作物之一,基因组小,具有营养价值高、耐逆和高光效等特点,已经成为遗传和分子生物学研究的C4模式作物之一[2]。因此,鉴定GRAS家族基因并研究其在不同激素和逆境胁迫下的表达特性,对利用该家族基因进行谷子株高、产量和耐逆等性状的遗传改良具有重要意义。
GRAS转录因子家族命名来源于最初发现的3个家族成员GAI[3]、RGA[4]和SCR[5]。GRAS蛋白一般由400~700个氨基酸组成,其C端序列高度保守,一般由5个典型的结构域组成,即LHRI、VHIID、LHRII、PFYRE和SAW基序[6]。相比而言,GRAS蛋白N端含有不同的固有无序区域,使得该类蛋白可以通过改变N端结构来灵活地特异性识别配体,因此,GRAS家族表现出功能多样性[7],包括参与赤霉素[8]、光信号[9-11]和油菜素内酯[12]等多种信号转导通路,调控腋芽[13]、根[14-15]、分生组织生长[16-17]、分蘖[18]以及维持顶端优势[19-21]等,响应植物对盐、干旱[22]、遮蔽[23]和重金属[24]等非生物胁迫,并参与雄配子的形成过程[25]。根据N端结构域的差异,GRAS家族蛋白一般分为SCL3、SHR、PAT1、LISCL、DELLA、SCR、LS、DLT、SCL4/7和HAM等10个亚家族[26-28]。SCR亚家族蛋白N端包含与其他蛋白互作所必需的结构域,在抑制不对称细胞分裂中起作用[29];DELLA亚家族蛋白N端存在GA信号的感知结构域,水稻的SLR1[30],拟南芥的GID1[31]、RGA[32]、GAI[33]、以及RGL1、RGL2和RGL[34]等都属于DELLA蛋白。目前,已经在拟南芥[26]、水稻[26,35]、大豆[36]、小麦[37]、大麦[38]、藜麦[39]、大白菜[40]、人参[41]和胡桃[42]等30余种植物中对GRAS基因家族进行了鉴定和分析。
随着谷子基因组的公布[43-44],bZIP、DREB、TCP和MYB等转录因子基因家族已被相继鉴定[45-46],但谷子GRAS转录因子基因家族相关研究仍鲜有报道。鉴于此,本研究通过生物信息学方法对谷子全基因组GRAS转录因子基因家族成员进行鉴定,对所有成员的理化信息、结构功能、表达模式等进行分析。同时,利用实时荧光定量(quantitative real-time PCR, qRT-PCR)分析33个GRAS基因在4种植物激素和2种非生物胁迫条件下的表达模式,旨在为进一步解析GRAS基因家族的功能奠定基础。另外,根据DELLA亚家族基因SiGRAS23序列差异开发分子标记,并在遗传群体中进行检测,以期为今后谷子株高分子标记辅助选择提供参考。
1 材料与方法
1.1 谷子全基因组GRAS蛋白的鉴定及理化性质分析
从Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)数据库下载谷子蛋白数据,从Pfam数据库(http://pfam.xfam.org/)中下载GRAS家族的隐马氏模型文件(Pfam号码:PF03514)[47],通过HMMER软件对谷子蛋白进行相似性搜索,找到GRAS家族蛋白序列,使用SMART(http://smart.embl-heidelberg.de/)和CDD(http://www.Ncbi.Nlm.Nih.Gov/Structure/cdd/wrpsb.cgi)检测蛋白结构域[48],过滤结构不完整的序列。最后,使用 ProtParam(https://web.expasy.org/protparam/)查找谷子GRAS蛋白的相关信息,包括氨基酸数目、分子量、等电点和亲水性平均值(grand average of hydropathicity,GRAVY)。
1.2 谷子GRAS家族系统发育关系分析
用ClustalW对52个谷子(Setariaitalia)SiGRAS、34个拟南芥(Arabidopsisthaliana)AtGRAS[26]、56个水稻(Oryzesativa)OsGRAS[26]、84个玉米(Zeamays)ZmGRAS[27]、80个高粱(Sorghumbicolor)SbGRAS[27]和48个短柄草(Brachypodiumdistachyon)BdGRAS[28]等GRAS家族基因氨基酸序列进行多重序列比对,利用MEGA-X软件中邻接法(neighbor-joining)构建系统发育树,自展值(Bootstrap)设为1 000。参考AtGRAS和OsGRAS蛋白的分类信息[35]对系统发育树进行聚类分析,利用网站Evolview v3(https://www.evolgenius.info/evolview/#login)进行进化树美化[49]。
1.3 谷子GRAS基因结构和家族成员保守结构域分析
从Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)数据库下载谷子基因组GRAS序列信息。根据谷子GRAS家族成员的基因组DNA序列及编码区序列(coding sequence, CDS),利用GSDS 2.0软件[50](http://gsds.cbi.pku.edu.cn)分析其基因结构。利用在线预测软件MEME[51](http://eme.nbcr.net/meme/tools/meme)预测蛋白结构域,结构域查找的最大数值设定为15。
1.4 SiGRAS的组织表达分析
从谷子全生育期基因表达图谱和多组学数据库(http://sky.sxau.edu.cn/MDSi.htm)下载晋谷21号生长发育过程中各组织器官的RNA-Seq数据[44],包括19个不同的组织(T1~T19):萌发3 d种子(T1)、两叶一心龄整株苗(T2)、抽穗后2 d的顶2和3叶(T3)、灌浆期的穗下节间(T4)、灌浆期的旗叶(T5)、灌浆期的旗叶叶鞘(T6)、灌浆期的茎(T7)、灌浆期的顶部第4叶(T8)、灌浆期的顶部第4叶(T9)、灌浆期的根(T10)、分化初始阶段穗(T11)、分化第3阶段穗(T12)、未成熟的圆锥花序S2(T13)、未成熟的圆锥花序S4(T14)、灌浆期种子S1(T15)、灌浆期种子S2(T16)、灌浆期种子S3(T17)、灌浆期种子S4(T18)和灌浆期种子S5(T19),以此分析谷子GRAS基因的组织表达特性。利用SPSS软件包中的Zscore方法,对各成员基因在各组织中表达的FPKM值进行数据标准化处理,用TBtools[52]绘制SiGRASs的组织表达热图。
1.5 SiGRAS启动子区域顺式作用元件预测
下载SiGRASs启动子序列(起始密码子ATG上游1.5 kb)(https://phytozome.jgi.doe.gov/pz/portal.html)。将获得的启动子序列提交到PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),进行顺式作用元件预测。
1.6 qRT-PCR表达分析
根据GRAS基因家族启动子顺式作用元件预测结果,采用qRT-PCR方法,选取33个谷子GRAS基因(涵盖了该家族的10个亚类)用于分析这些基因对生长素(吲哚乙酸,indole-3-acetic acid, IAA)、赤霉素(gibberellin A3, GA3)、茉莉酸甲酯(methyl jasmonate, MeJA)、脱落酸(abscisic acid, ABA)、干旱(PEG6000)和冷(4℃)处理下的表达规律。选择饱满的长农35号种子播种于营养钵(营养土∶蛭石=1∶1),放置于光照培养箱(RTOP-268Y,托普云农,浙江)中,生长条件为光照14 h 30℃/黑暗10 h 25℃,待幼苗长至两叶一心时,分别用5 μmol·L-1IAA、100 μmol·L-1GA3、100 μmol·L-1MeJA、100 μmol·L-1ABA、20% PEG-6000和4℃分别处理幼苗,分别采集对照植株和处理后1、3、6和12 h的植株叶片。按照RNAiso Plus试剂盒(TaKaRa,日本)和步骤说明提取总RNA,采用PrimeScript Ⅱ 1ststrand cDNA synthesis kit(TaKaRa,日本)反转录为cDNA,qRT-PCR引物见附表S1。以谷子ACTIN作为内参基因。通过CFX96 Real-time PCR仪(Bio-Rad,美国)采集荧光实时定量数据,反应体系为10 μL:cDNA template(反转录第一链)1 μL、TB Green Premix Ex Taq Ⅱ(TAKARA,日本)5 μL、2 μmol·L-1特异引物1 μL,ddH2O 3 μL。扩增程序为:94℃预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸30 s,35个循环;72℃延伸5 min。65~98℃绘制溶解曲线。采用比较阈值法进行定量分析。程序结束后,根据Ct值计算C值,公式如下:
C=2-ΔCt(ΔCt=Ct目标基因-Ct内参基因)
3次重复的C值平均值为目的基因的相对表达量。采用SPSS 19软件中单因素方差分析方法进行相对表达量显著性差异检测(P<0.05)。
1.7 DELLA蛋白基因SiGRAS23分子标记开发
前期研究利用谷子矮宁黄×晋谷21号的F2分离群体,定位了7个与谷子株高相关的QTL,其中第5号染色体上存在3个株高QTL,分别为qPH5-1、qPH5-2、qPH5-3,qPH5-2的遗传效应值较大[53]。因此,本研究在上述定位区间内搜索相关GRAS基因家族成员,通过克隆测序,检测目标基因在矮宁黄和晋谷21号中的等位变异,开发分子标记,并在矮宁黄×晋谷21号的F5群体(AJF5)中192个株系中进行检测,结合株高表型值,进行相关性分析。同时,从谷子种质资源中随机选取株高表型变异从75.0~173.7 cm材料37份(附表S2),利用开发的分子标记检测上述材料的基因型。
2 结果与分析
2.1 谷子GRAS家族成员的鉴定及分析
利用HMMER软件共获得62个GRAS候选蛋白,使用SMART和CDD工具检测保守结构域,删除结构不完整的序列,最终保留52条具有完整谷子GRAS结构域序列。以SiGRAS命名谷子GRAS基因,将所对应的52条DNA序列作为谷子GRAS转录因子家族的候选基因。染色体定位结果显示,除第Ⅵ染色体上无GRAS外,52个SiGRASs呈不均匀状态分布在谷子8条染色体上(表1)。不同GRAS转录因子蛋白序列存在较大差异,氨基酸长度362~734 aa,蛋白分子量39.81~100.09 kDa,等电点为4.85~9.53,其中等电点小于7的GRAS蛋白有43个,偏酸性蛋白居多。除Seita.2G374000、Seita、3G137200和Seita.9G576800外,其余蛋白均具有亲水性(表1)。
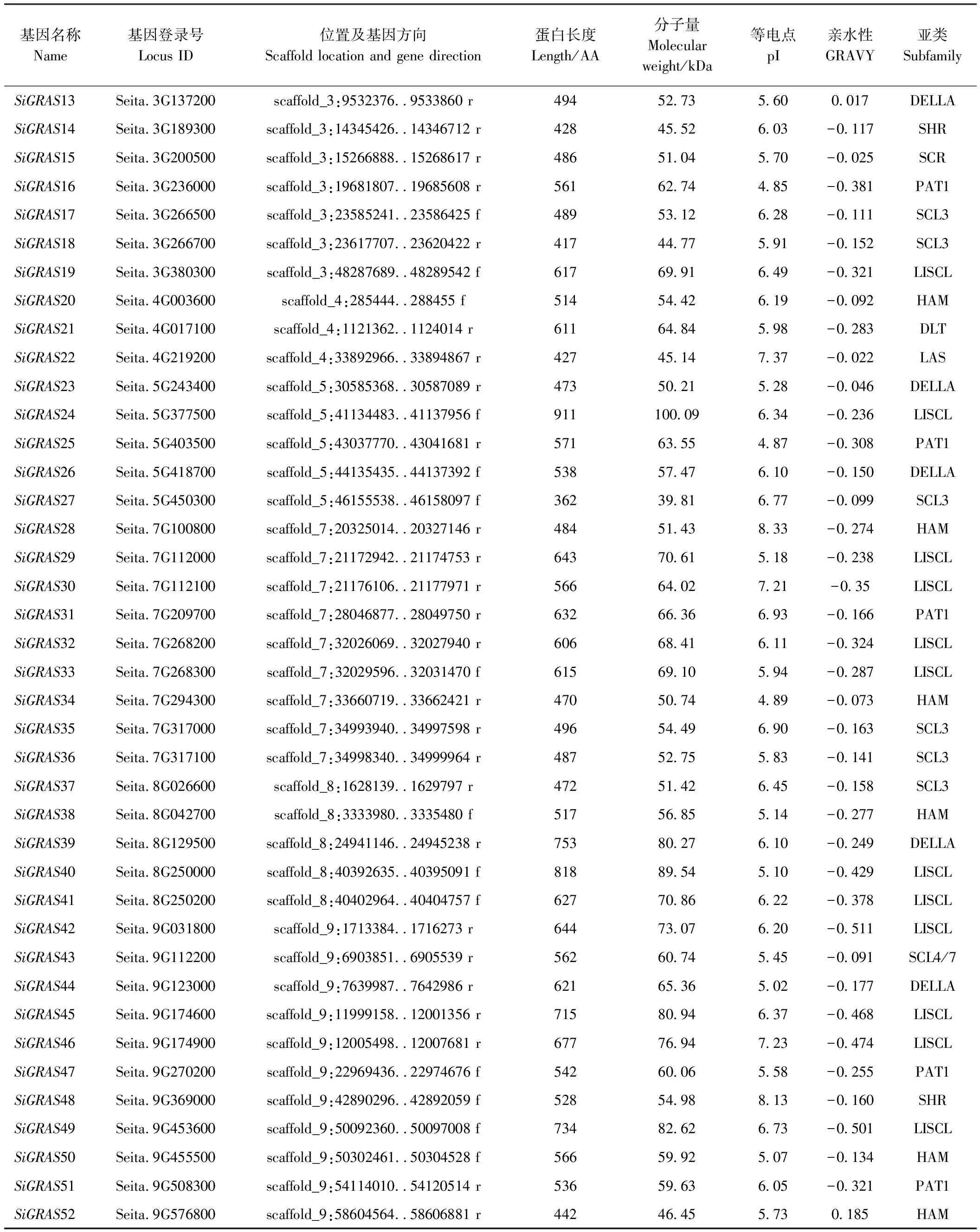
表1(续)
2.2 谷子GRAS转录因子家族系统发育分析
为深入理解谷子GRAS的进化关系,通过构建谷子与拟南芥(34个)、水稻(56个)、玉米(84个)、高粱(80个)和短柄草(48个)GRAS蛋白进化树,对系统发育树进行聚类分析(图1)。结果表明,谷子中52个GRAS转录因子家族可分成LISCL、PAT1、SCR、DELLA、DLT、SCL3、SHR、LAS、SCL4/7和HAM等10个亚家族(图1),其中LISCL亚家族成员最多,含15个蛋白;DLT和SCL4/7亚家族成员最少,各含1个蛋白。
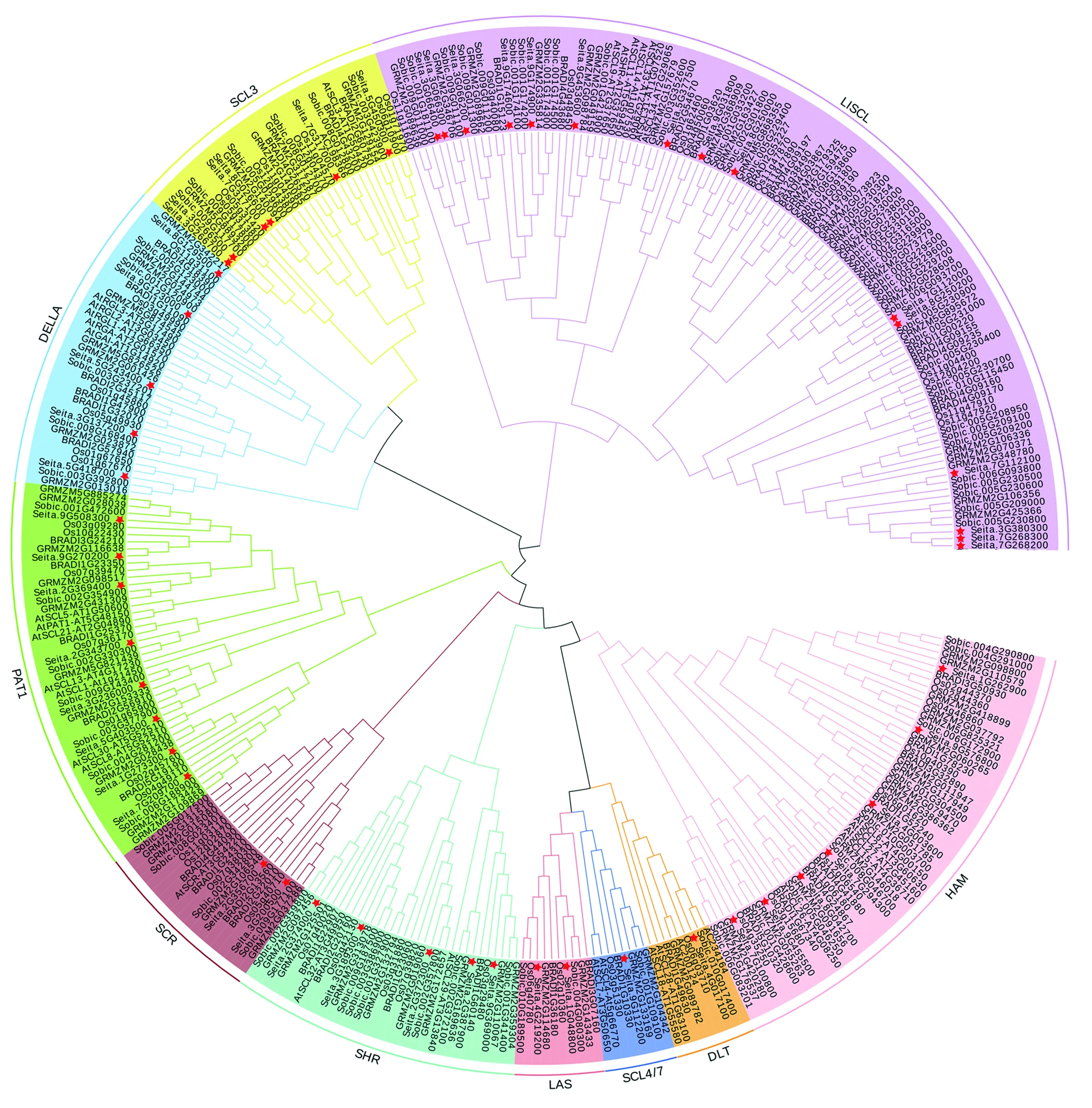
图1 谷子、拟南芥、水稻、玉米、短柄草和高粱的GRAS家族系统进化树Fig.1 Phylogenetic tree of GRAS gene family in Setaria italica, Arabidopsis thaliana, Oryza sativa, Zea mays, Sorghum bicolor and Brachypodium distachyon
2.3 GRAS基因结构和蛋白保守结构域分析
基因结构和蛋白保守域分析表明,SiGRASs多数无内含子,同一亚家族内,基因结构基本一致,保守基序具有相似性,暗示同一个亚家族的基因具有相似功能。所有SiGRAS蛋白都拥有由LHRI、VHIID、LHRII、PFYRE和SAW在C末端组成的GRAS结构域,与保守的C末端相比,SiGRAS蛋白的N末端序列在各亚族间差异较大。在同一亚家族中,SiGRAS蛋白质的氨基酸序列表现出高度一致性(图2)。
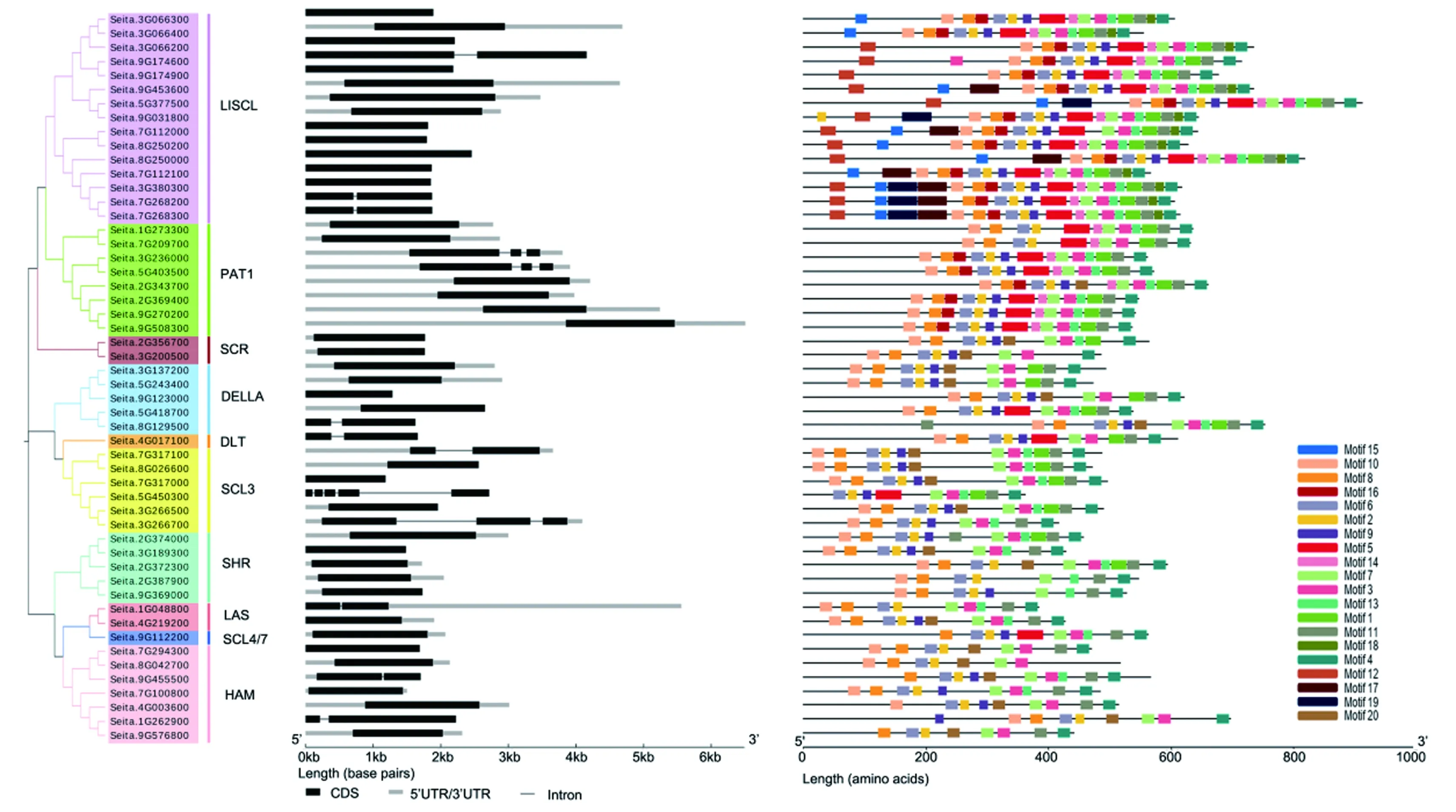
注:外显子和内含子分别用黑色方框和黑色线条表示,UTR用浅灰色方框表示。不同颜色代表不同的结构域。Note: Exons and introns were indicated by dark boxes and dark lines respectively, UTRs were indicated by light grey boxes. Different motifs were displayed in different colors.图2 谷子GRAS基因家族的进化树、基因结构和保守结构域Fig.2 Phylogenetic tree, gene structure and conserved domain of GRAS gene family in foxtail millet
2.4 SiGRAS的组织表达分析
为初步分析SiGRASs的功能,根据MDSi数据库中不同组织转录组数据的每千个碱基的转录每百万映射读取的片段(fragments per kilobase, FPKM),对52个SiGRASs组织表达特性进行了分析。结果表明,不同GRAS在谷子不同组织器官中的表达量存在明显差异,SHR亚族中各基因均在根中有较高的表达量,DELLA亚族中的Seita.5G243400和Seita.9G123000在茎中有较高的表达量,LISCL亚族中的Seita.9G453600和Seita.5G377500、PAT1亚族的Seita.2G369400、HAM亚族中的Seita.4G003600和Seita.1G262900在叶中有较高的表达量。另外,本研究发现一类特殊现象,即42.3%的基因在各组织器官中表达量很低(图3),研究共检测到22个这样的基因,包括LISCL亚族中的Seita.3G066300、Seita.3G066400、Seita.3G066200、Seita.9G174600、Seita.9G174900、Seita.7G112100、Seita.3G380300、Seita.7G268200和Seita.7G268300;SCR亚族中的Seita.3G200500;DELLA亚族中的Seita.3G137200和Seita.8G129500;SCL3亚族中的Seita.7G317100、Seita.7G317000、Seita.8G026600、Seita.3G266500和Seita.3G266700;SHR亚族中的Seita.3G189300;HAM亚族中的Seita.7G294300、Seita.8G042700、Seita.9G455500和Seita.7G100800。
2.5 SiGRAS启动子顺式作用元件预测与分析
SiGRAS含有多种与植物生长发育相关的顺式作用元件,如光反应、胚乳表达、分生组织表达、昼夜节律、细胞周期、多种植物激素以及与逆境相关的顺式作用元件,表明SiGRAS广泛参与谷子的生长发育。对激素以及与逆境相关的顺式作用元件进行分析发现,其中43个SiGRAS含有响应脱落酸的ABRE元件、46个SiGRAS含有响应茉莉酸甲酯的CGTCA-motif或TGACG-motif元件、27个SiGRAS含有响应生长素的TGA-element或AuxRR-core元件、27个SiGRAS含有响应赤霉素的P-box、TATC-box或GARE-motif元件以及19个SiGRAS含有响应冷胁迫的LTR元件。
2.6 SiGRAS在不同激素和逆境下表达模式分析
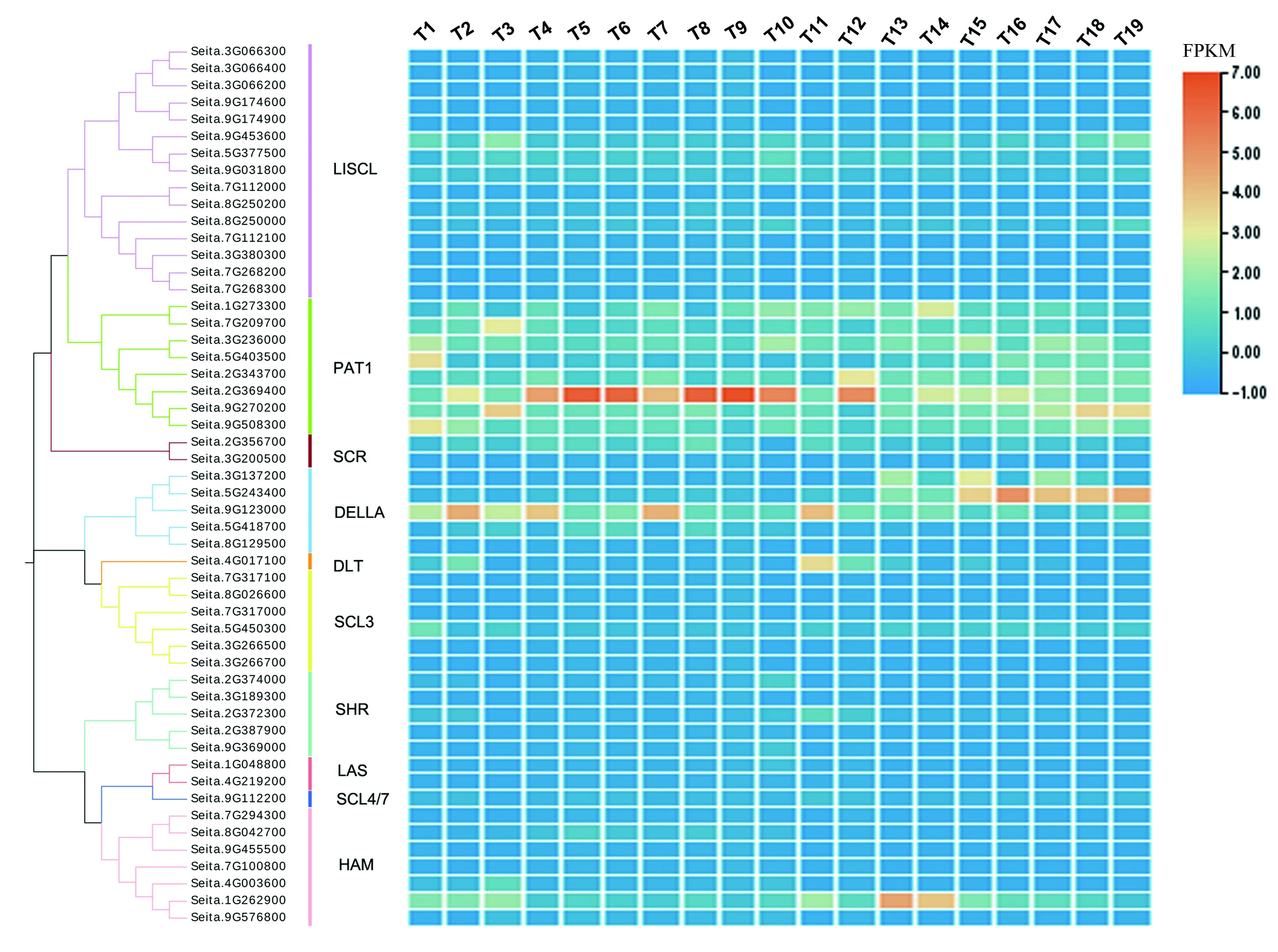
图3 GRAS基因在谷子不同组织和不同发育时期的表达模式分析Fig.3 Expression profile analysis of GRAS genes in different tissues and different developmental stages of foxtail millet
根据上述顺式作用元件预测结果,选取33个SiGRAS,分别对其在IAA、MeJA、GA3、ABA、4℃和PEG-6000处理1、3、6和12 h后的表达模式进行了分析。结果表明,33个SiGRASs均响应激素和非生物胁迫,但表达水平不同。18个基因(包括Seita.3G066400、Seita.3G066200、Seita.8G250200、Seita.7G112100、Seita.3G380300、Seita.7G268300、Seita.1G273300、Seita.3G200500、Seita.5G418700、Seita.4G017100、Seita.8G026600、Seita.7G317000、Seita.3G266700、Seita.2G374000、Seita.3G189300、Seita.1G048800、Seita.4G219200和Seita.7G294300)虽然对各种激素和逆境有不同程度的响应,但是表达仍处于较低水平。因此,本试验选取在不同激素和逆境处理下叶片中相对表达量较高的15个SiGRAS进行重点分析,PAT1亚族中Seita.2G369400对6种不同的处理响应最为敏感,而LISCL亚族Seita.9G453600 和HAM亚族Seita.4G003600对各种处理响应不明显;在4℃胁迫下,大部分基因在处理1、3 h 后表达逐渐上调,但在6 h后表达量逐渐下降;在IAA处理下,大部分基因在处理1 h后表达量急剧下降,而在 6 h 后又有小幅回升,但仍低于对照的表达水平;在MeJA、GA3、ABA、4℃和PEG-6000处理下,除Seita.2G369400外,大部分基因在1 h的表达量轻微上调或维持不变,在3 h后表达量逐渐下降(图4)。
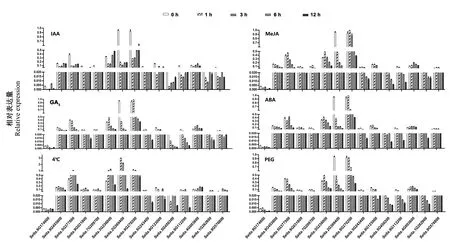
图4 不同激素和逆境胁迫下SiGRAS的相对表达量Fig.4 Relative expression of SiGRASs under different hormone and abiotic stress
对6种处理后6 h的基因相对表达量进行了分析,结果表明,与CK相比,在IAA处理下,Seita.9G174600显著上调表达,大多数基因显著下调表达,5个表达无显著差异;在GA3、MeJA、ABA和PEG处理下,除Seita.4G003600外,其余均显著下调表达;而在低温胁迫下,Seita.5G377500、Seita.3G236000和Seita.9G576800显著上调表达,而Seita.5G377500、Seita.5G243400和Seita.9G112200显著下调表达。Seita.7G209700、Seita.2G369400、Seita.5G243400和Seita.5G450300在4种激素和2种胁迫下相比对照均显著下调表达(图5)。
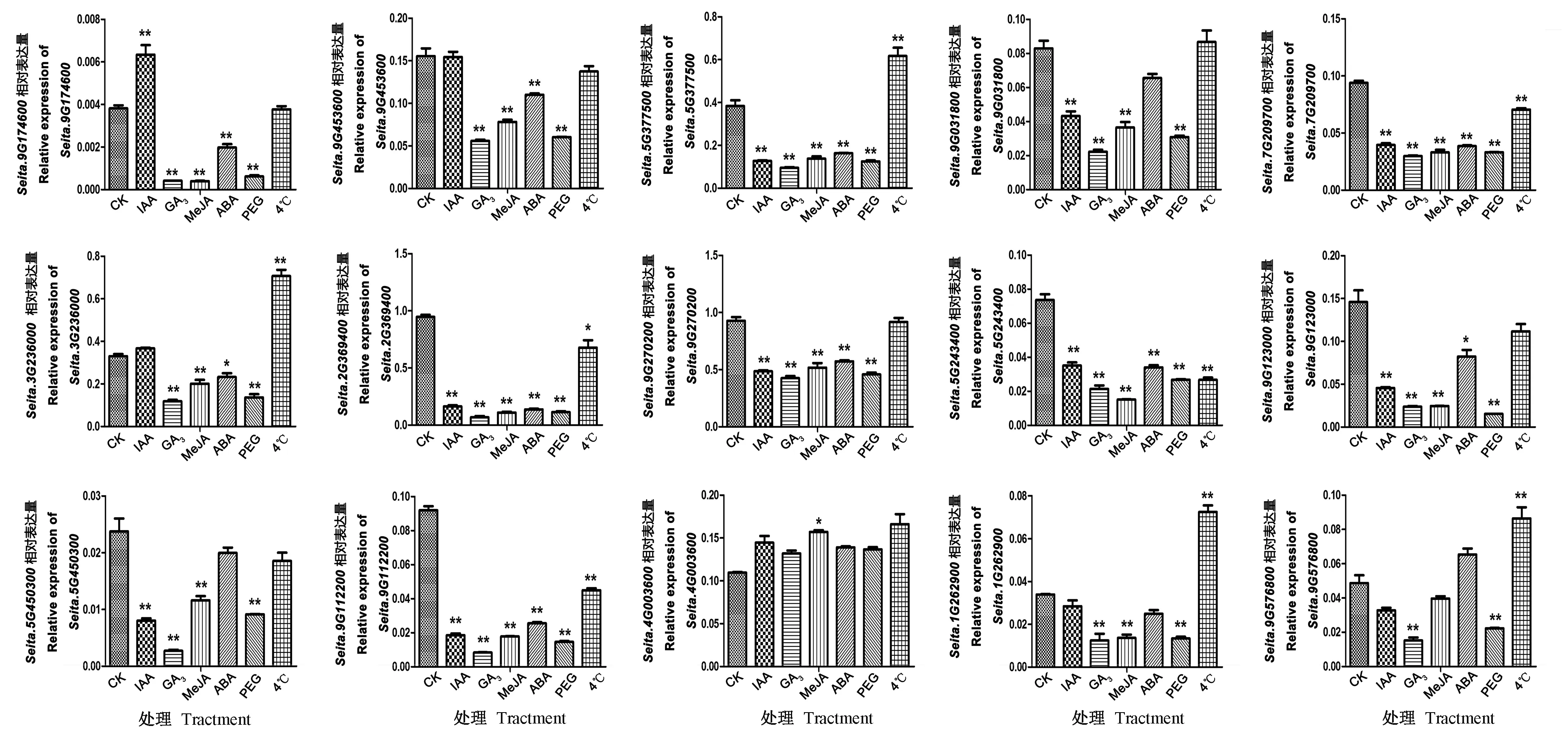
注:*和**分别表示在P<0.05和P<0.01水平差异显著。Note: * and ** mean significant difference at 0.05 and 0.01 level, respectively.图5 15个GRAS基因在谷子激素和逆境胁迫处理6 h后的表达模式分析Fig.5 Expression profile analysis of 15 SiGRAS genes at 6 h after different hormones and abiotic stresses treatment
2.7 DELLA蛋白基因SiGRAS23分子标记开发
在前期矮宁黄×晋谷21号的F2群体定位的株高QTL区间基础上,发现2个DELLA蛋白编码基因SiGRAS23(Seita.5G243400)和SiGRAS26(Seita.5G418700)分别位于qPH5-2和qPH5-1区间内或附近,分别扩增上述基因的序列(表2)。比对分析后发现,SiGRAS26在双亲中无差异位点,而SiGRAS23在双亲间存在7个差异位点,其中CDS区存在2个SNP,启动子区存在3个SNP和2处InDel。根据这些差异位点,本研究开发了D8-1、D8-2和D8-3等3个分子标记,分别位于启动子区-871 bp、启动子区-349 bp、CDS区627 bp(图6-A,表2)。利用开发的标记D8-1对190份AJF5(矮宁黄×晋谷21号)群体株系和37份株高差异较大的谷子材料进行基因型检测,将矮宁黄基因型命名为D8-1-A,晋谷21号基因型命名为D8-1-J,杂合基因型命名为D8-1-A/J,结果表明,分子标记D8-1与AJF5(矮宁黄×晋谷21号)群体中株高紧密连锁(P<0.01)(图6-B);在37份谷子亲本材料中,矮88、矮97、石矮大-1等矮杆材料与矮宁黄基因型一致,D8-1-A基因型材料的株高与另外2种基因型材料的株高存在显著差异,但D8-1-J基因型和D8-1-A/J基因型的材料间株高差异不显著(图6-C,附表S2),表明该基因可能与株高遗传相关,D8-1-A可能是谷子矮杆变异相关的基因型之一。
3 讨论
在植物中GRAS基因家族编码的转录因子数量众多,功能多样,参与调控许多重要生长发育过程。目前,该基因家族已经在多个物种中被鉴定,如在拟南芥[26,54]、水稻[26]、大豆[36]、小麦[37]、高粱[27]、玉米[27]、短柄草[28]、大麦[38]、藜麦[39]、大白菜[40]、人参[41]、大麻[22]和胡桃[42]分别鉴定出34、56、117、153、80、86、48、59、54和52个GRAS家族成员。本研究从谷子全基因组数据库中获得了52个GRAS家族成员,与水稻、短柄草和藜麦等作物中的数量相似,96%蛋白均具有亲水性,与前人结果一致[55]。根据进化树分支,结合拟南芥、水稻、玉米、高粱和短柄草同源基因的分类方法,将谷子GRAS家族同样分为10个亚类。

表2 DELLA蛋白基因SiGRAS23和SiGRAS26分段扩增引物及标记开发引物Table 2 Amplified primers and marker development primers of DELLA protein encoding gene SiGRAS23 and SiGRAS26
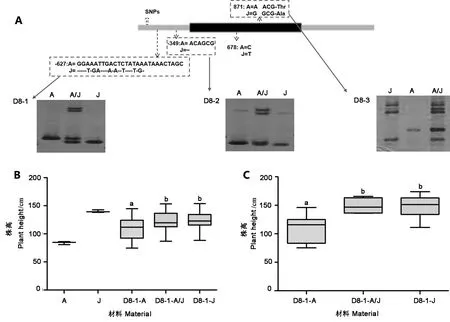
注:A图:SiGRAS23在矮宁黄和晋谷21号间的序列差异和分子标记开发,不同数字代表距基因起始密码子的位置,D8-1、D8-2、D8-3分别为开发的分子标记,A:矮宁黄,J:晋谷21号,A/J:杂合基因型;B图:D8-1在AJF5群体株系中的基因型检测;C图:D8-1在37份材料中的基因型检测。字母a和b表示不同等位变异间的显著差异(P<0.01)。Note: Figure A: Sequence difference and molecular markers development between Aininghuang and Jingu 21. Different numbers represent the position from the start codon of the gene. D8-1, D8-2 and D8-3 are the developed molecular markers, respectively. A: Aininghuang. J: Jingu 21. A/J: the heterozygous genotype line. Figure B: Genotyping in lines of AJF5 with D8-1. Figure C: Genotyping in 367materials with D8-1. a and b indicate significant differences at 0.01 level.图6 SiGRAS23在矮宁黄和晋谷21号间的序列差异和分子标记开发以及在不同材料中的基因型检测Fig.6 Sequence difference and molecular markers development between Aininghuang and Jingu 21, and genotyping in materials of SiGRAS23
SiGRAS各亚家族基因的组织器官表达特性分析表明,谷子各亚族基因与拟南芥中已报道同类亚族基因的表达特性和相应功能基本一致。大多数谷子PAT亚家族基因在叶中表达量较高,而叶是感受光信号的主要部位,推测该类基因在谷子phyA的信号转导中发挥作用;SHR蛋白参与根系径向形态的维持和根系生长[56-57]。谷子SHR亚族中各基因均在根中有较高的表达量,其对根系发育的调控作用有待进一步研究;DELLA蛋白通过GA应答反应调控植物茎的伸长[1,3-4]。本研究中,Seita.5G243400和Seita.9G123000等DELLA亚族中的基因在茎中有较高的表达量,推测这些基因可能通过茎的生长发育来调控谷子株高等性状。另外,谷子GRAS各亚族基因的组织表达特异性与水稻、短柄草、大豆中各亚族的组织表达特异性一致[26,28,36]。GRAS各亚类基因在不同物种中的保守表达谱表明,同源基因可能在这些特定的组织器官发育中发挥相同或相似的作用。
植物对各种激素和环境胁迫的应答中需要GRAS蛋白的参与[1-4,20-22]。本研究中,SiGRASs启动子区域存在多个与激素或逆境响应相关的顺式作用元件,经生长素、赤霉素、茉莉酸甲酯、脱落酸、干旱和冷等6种不同的处理后发现,33个SiGRAS表达水平存在较大差异,在同一亚族成员之间也存在较明显差异。在短柄草[28]、杨树[35]、梅花[55]和番茄[58]GRAS同一亚族不同基因中也出现类似的结果,表明同一亚族各基因在非生物胁迫反应和激素介导的信号通路中的作用可能不同。另外,本研究中,谷子PAT1亚族中Seita.2G369400对6种不同的处理响应最为敏感,暗示该基因是谷子在激素响应和逆境胁迫中的一个重要基因。GRAS家族PAT1分支基因不仅在光敏色素信号转导中发挥重要作用,也直接影响植物抗逆过程,该亚族中已报道的SlGRAS4[58]、AtGAI[59]和BdGRAS[60]等基因能积极响应低温胁迫。DELLA蛋白是GA信号关键负向调节因子,本研究中的3个DELLA亚族基因Seita.5G243400、Seita.9G123000和Seita.5G418700在GA3处理后的3、6、12 h均表现为下降趋势。基因家族通常在基因组中具有多个拷贝,这些基因表现出功能冗余性和特异性,5个MIR172基因在调控植物分生组织大小、叶片下表皮毛起始、茎的伸长、侧枝发生以及开花时间上具有冗余性[60],推测本研究中表达量极低的基因也存在基因冗余,但还需大量试验验证。
DELLA蛋白是GAs代谢通路中的负调控因子,属于植物特有的GRAS家族,调控植物生长发育,但其结构域相对复杂[1,3-4,34-38]。在拟南芥GRAS家族中共有5个DELLA蛋白,其N端均含有DELLA和VHYNP典型的结构域[26,54],在水稻中,DELLA亚家族成员中仅有OsSLR1含有DELLA和VHYNP结构域,而与其同聚为DELLA亚族的OsSLRL1和OsSLRL2并不含有这2个结构域[61],短柄草DELLA亚家族成员BdSLR1含有DELLA和VHYNP结构域而BdSLRL1不存在该结构域[28],在玉米中也出现了类似现象,但这些蛋白均作为GA信号的阻遏蛋白调控植株的生长发育,过表达这些基因的植株均表现为矮化。在本研究中,谷子DELLA亚家族有5个基因,除Seita.9G123000中发现DELLA结构域,其余4个蛋白均未发现DELLA结构域,这与水稻[26,61]、短柄草[28]、藜麦[39]中GRAS家族DELLA亚族结果相似。目前,对该现象的解释可能是,GRAS基因通过复制增加基因拷贝数,不同拷贝数导致基因序列发生改变,这些序列差异决定了功能特异的各种蛋白质。在这种情况下,DELLA蛋白复制DELLA、TVHYNP和polyS/T/V等功能结构域序列。研究人员推测GRAS序列复制可能发生在单子叶植物和双子叶植物分化之前,双子叶植物中存在DELLA基因复制事件,而单子叶植物中不存在DELLA基因复制[61]。DELLA结构域的有无是否会影响谷子DELLA蛋白的功能,还有待下一步研究。
SiDWARF1(Seita.9G123000)编码的DELLA蛋白,是目前谷子中唯一报道的GRAS家族基因,其突变体84133植株表现矮化[62]。本研究结合先前本课题组对谷子矮宁黄×晋谷21号F2分离群体株高性状的QTL定位[53],发现第V染色体与株高相关的QTL区间中包含2个编码DELLA蛋白的基因(SiGRAS23和SiGRAS26)。目前已知,根据SiGRAS23变异位点开发的分子标记D8-1与AJF5群体株高性状紧密连锁,下一步本研究小组拟从株高性状QTL的精细定位、克隆以及SiGRAS23基因功能验证两方面开展相关研究,以探明编码DELLA蛋白的SiGRAS23基因与株高的关系。此外,谷子中常见的矮杆材料,如矮88、矮97和石矮大-1等基因型均为D8-1-A,但也有少数材料的基因型和株高表型不相符,如晋谷20号、东山谷、合光3号、六十天还家(见附表S2)。鉴于株高为数量性状控制,推测这些材料中存在其他控制株高的主效基因或位点,有待于进一步发掘和利用。
4 结论
本研究对谷子GRAS基因家族进行了全基因组鉴定,共发现52个SiGRAS家族成员,可分为10个亚类。除第Ⅵ染色体上无GRAS基因外,其余8条染色体上均有SiGRASs分布,且具有典型的C端GRAS保守域和N端高度可变特异结构域。不同亚族基因表达具有明显的组织表达特异性,不同程度地响应生长素、赤霉素、茉莉酸甲酯、脱落酸等激素以及干旱和冷等胁迫。进一步研究发现,DELLA亚族中的SiGRAS23在遗传群体AJF5的双亲矮宁黄和晋谷21号间存在序列差异,据此开发的标记D8-1与该群体株高性状紧密连锁,D8-1-A可能是谷子矮杆的优异等位变异之一。
表S1 qRT-PCR所用引物信息Table S1 Primers used for qRT-PCR
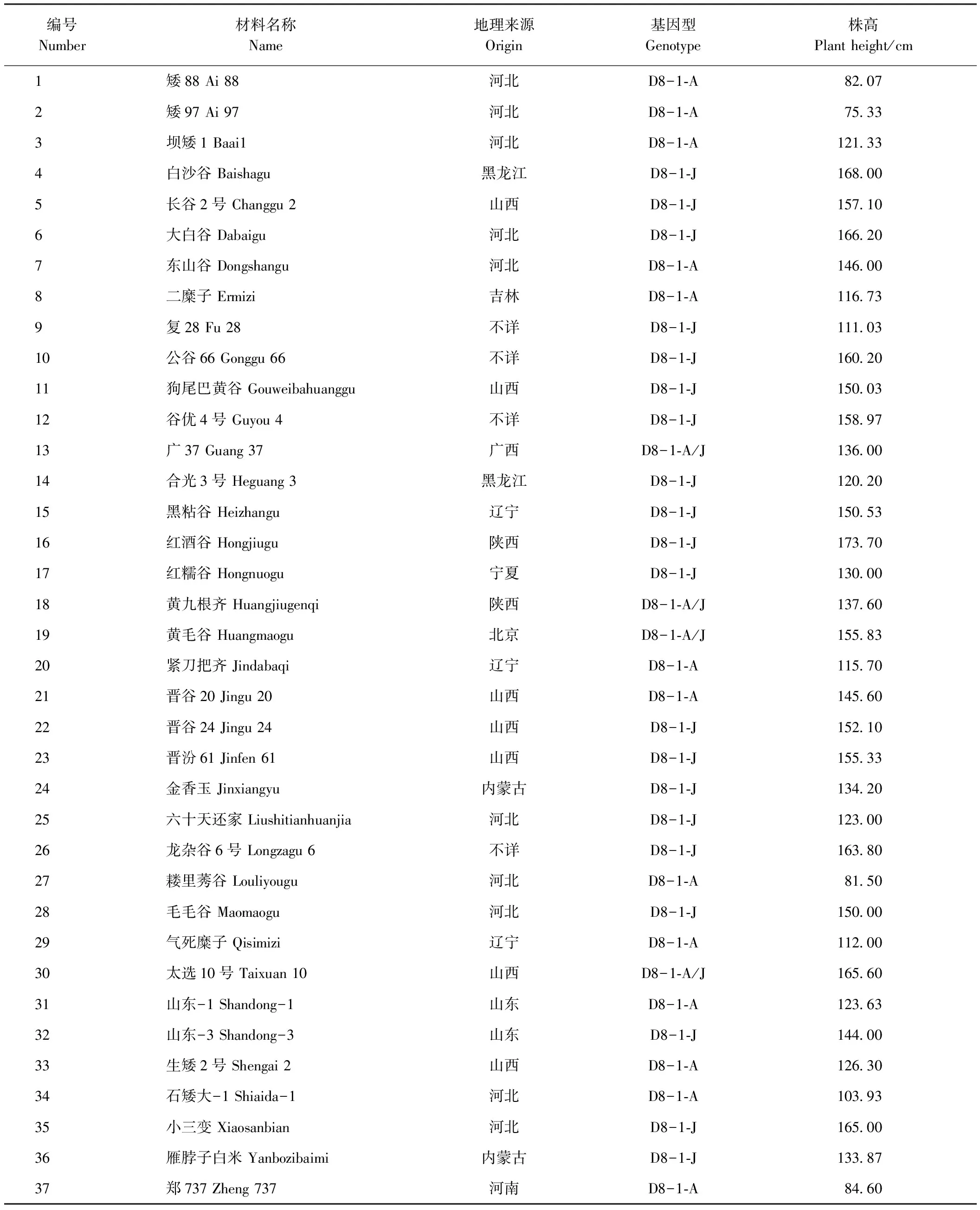
表S2 37份谷子材料信息Table S2 Profiles of 37 foxtail millet
