红花光调控信号途径关键基因CtHY5的克隆及表达分析
2022-09-20谭政委鲁丹丹余永亮许兰杰杨红旗李春明安素妨芦海灵梁慧珍
谭政委,鲁丹丹,李 磊,余永亮,许兰杰,杨红旗,杨 青,董 薇,李春明,安素妨,芦海灵,梁慧珍
红花光调控信号途径关键基因的克隆及表达分析
谭政委,鲁丹丹,李 磊,余永亮,许兰杰,杨红旗,杨 青,董 薇,李春明,安素妨,芦海灵,梁慧珍*
河南省农业科学院 芝麻研究中心,河南 郑州 450002
以药用植物红花为研究对象,克隆光信号途径关键转录因子基因,并对其进行生物信息学和表达模式分析,为红花基因的功能研究提供参考。以红花转录数据为参考,设计引物,采用PCR扩增方法从红花中克隆得到HY5的全长cDNA和DNA序列。运用生物信息学方法对该基因进行分析,预测编码蛋白的结构与功能,并通过荧光定量PCR方法检测基因在红花不同组织及花发育不同时期的表达情况。基因的cDNA全长为462 bp,编码153个氨基酸,DNA全长为1941 bp,包含4个外显子和3个内含子。生物信息学分析表明,CtHY5为亲水性蛋白,定位于细胞核中。系统进化树及模体结构分析结果表明CtHY5与来自菊科的刺菜蓟、黄花蒿、薇甘菊、向日葵、莴苣中的HY5进化关系较近。定量分析表明,在白色红花和红色红花中,基因均在花中表达量最高,其次为茎和苞片,在根中表达量最低。此外,除了根外,基因在白色红花各组织中的表达量要明显高于红色红花。首次从红花中克隆得到了光信号途径关键转录因子基因,并研究了该基因在不同花色红花品系中的表达模式,为进一步研究其生物学功能奠定了基础。
红花;光信号途径;HY5;基因克隆;表达分析
转录因子HY5(ELONGATED HYPOCOTYL5)是基本亮氨酸拉链类(basic leucine zipper,bZIP)转录因子类的转录因子,最早是在拟南芥中克隆得到[1]。编码的蛋白为光敏色素下游的核心光信号调节因子,在光信号通路中具有十分重要的作用,HY5的C末端具有特殊的bZIP结构域,能够直接与基因启动子作用元件G-box(CACGTG)相结合,从而能调控基因的表达它能通过不同的感受光信号接收光信号,并将光信号传递给下游作用元件,进而调控植物的生长发育[2-5]。
HY5广泛参与植物生长发育各个生理过程的转录或转录后调控。HY5通过结合基因上游的CUF-1元件与之协同调控拟南芥幼苗叶绿素积累和光形态建成[6],此外,HY5结合并正调控昼夜节律相关基因[7],促进矿物质营养元素转运基因的表达[8-9]。最新研究表明,黄花蒿中AaHY5通过与AaWRKY9的启动子结合来激活其表达,协同参与调控光和茉莉酸酯介导的青蒿素的生物合成[10];番茄中SlHY5在转录和翻译水平上直接调控果实成熟、花青素和类胡萝卜素合成相关基因的转录水平和蛋白翻译效率[11]。在不同的光照条件下,HY5也可以直接结合花青素生物合成基因查耳酮合成酶()基因、查耳酮异构酶()基因等,调节花青素的积累。光敏色素互作因子PIF3和HY5在光照下正调控花青素合成[12-13]。。
基因已经从拟南芥(L.) Heynh[1]、苹果Borkh.[14]、油菜Linn.[15]、梨Rehd.[16]、黄花蒿Linn.[10]、番茄Linn.[17]、苦荞(L.) Gaertn[18]等物种中完成了分离和鉴定。但是,目前在药用植物红花L.中尚未见基因的相关报道。
红花,别名草红花、红蓝花、刺红花,为菊科红花属一年生双子叶草本植物,其干燥花入药,具有活血通经、散瘀止痛的功效[19],红花花中主要的活性化合物是黄酮类化合物,目前从红花中分离得到类黄酮化合物超过60种[20]。其花颜色多样,如红色、白色、黄色、橙色等,其花色差异推测可能是由类黄酮组成成分和含量不同引起的,然而相关分子机制还未见报道。
本研究根据前期红花花期转录组测序数据,通过PCR扩增,获得了红花HY5同源基因的cDNA序列,并以红花叶片DNA为模板获得基因的全长DNA序列。通过生物信息学的分析方法对CtHY5蛋白的理化性质、保守结构域,疏水性亲水性,二级结构及三级结构进行了分析,并对CtHY5进行了系统进化分析。另外,检测了该基因在不同花色的红花品系、不同组织及不同花期的表达量,以期为进一步阐明光调控红花花青素及类黄酮生物合成调控的机制奠定基础,并为利用该基因改善红花中药材品质提供理论参考。
1 材料
供试材料为河南省农业科学院芝麻研究中心药用植物研究室收集的管状花为红色和白色红花品系,经河南省农业科学院芝麻研究中心梁慧珍研究员鉴定为菊科红花属植物红花L.,种植于河南农业科学院现代农业研究开发基地,自然条件生长,待植株生长至开花期,取根、茎、叶和苞片,并在管状花从花冠总苞片露出的第1、2、3、4、5和6天取管状花,分别标为S1、S2、S3、S4、S5和S6期,所有组织部位和样品取3个生物学重复,将采集的样品立即放入液氮中保存,并转移至−80 ℃超低温冰箱中保存备RNA提取。
2 方法
2.1 总RNA提取及cDNA第一链合成
按照北京华越洋生物公司的Quick RNA Isolation Kit试剂盒说明书提取样品的总RNA,通过1.2%琼脂糖凝胶电泳对提取的总RNA的质量和完整性进行检测,用NanoDrop 2000分光光度计对总RNA浓度进行测定,根据反转录试剂盒(TaKaRa公司,PrimeScriptTMRT reagent Kit with gDNA Eraser)说明书操作步骤(Code No. RR047A),将总RNA反转录为第一链cDNA。
2.2 DNA提取
按照北京华越洋DNA提取试剂盒提取红花幼叶DNA样品,通过0.8%琼脂糖凝胶电泳对提取的DNA进行检测,用NanoDrop 2000分光光度计对DNA浓度进行测定。
2.3 CtHY5基因的克隆
根据转录组测序的Unigene基因序列设计引物,上游引物:5’-ATGCAAGAGCAAGCTGCAA-3’,下游引物:5’-CTATTTCCTTTCTTGCATACCG-3’,分别以管状花基本伸出苞片时的红花管状花cDNA和红花幼叶DNA为模板,利用KOD酶进行PCR扩增得到基因的全长cDNA和DNA序列,反应程序如下:94 ℃、2 min(98 ℃、10 s,56 ℃、30 s,68 ℃、2 min)×35 循环68 ℃、5 min。PCR扩增产物经琼脂糖凝胶电泳检测,并利用生工Sangon Biotech SanPrep柱式DNA胶回收试剂盒(Lot.:AA24KA4369)回收目的片段,连接到TaKaRa pMD19-T载体上,转入TaKaRa DH5α感受态细胞,涂布于含氨苄青霉素抗性的LB培养基上,37 ℃培养16 h。挑取单菌落,PCR检测后,将阳性菌落送往公司测序。通过DNAMAN软件拼接出基因全长cDNA序列。
2.4 CtHY5基因序列的生物信息学分析
通过MEGA 7.0软件比对基因全长cDNA和DNA序列,找出基因内含子序列。利用ORF finder在线软件(https://www.ncbi.nlm.nih. gov/orffinder/)确认的CDS序列,CtHY5蛋白的氨基酸组成、蛋白质相对分子质量、理论等电点及稳定性等参数进行分析,通过在线软件ProtParam(https://web.expasy.org/protparam/)完成;使用NCBI中的CD-Search(https://www.ncbi.nlm. nih. gov/ Structure/cdd/wrpsb. cgi)对氨基酸序列的保守结构域进行鉴定。CtHY5蛋白的二级结构使用在线工具SOPMA(https://npsa-prabi.ibcp. fr/cgi- bin/npsa_ automat. pl?page=/NPSA/npsa_ sopma. html),三级结构通过SWISSMODLE(https:// swissmodel. expasy. org/ interactive)进行。通过TMHMM Server v2.0(http://www.cbs.dtu.dk/services/ TMHMM/)对CtHY5蛋白的跨膜结构域进行分析;选用SignalP-5.0Server在线软件(http://www.cbs. dtu.dk/services /SignalP/ index.php)以及PSORT在线软件(https://www. genscript.com/psort.html)分别进行信号肽预测和亚细胞定位分析。以CtHY5蛋白序列为模板,通过NCBI数据库中进行Blast P模块进行比对分析,找出其它物种中CtHY5蛋白的同源序列,利用DNAMAN对CtHY5蛋白与其它物种的HY5同源蛋白的同源性进行分析;通过MEGA7.0软件中的Neighbor-joining构建CtHY5蛋白系统化进化树,并通过Bootstrap方法对进化树进行检测,Bootstrap值设置为100。系统发育树中序列的模体结构采用MEME(https://meme-suite.org/meme/)在线网站进行分析。
2.5 实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)
通过qRT-PCR技术检测基因在管状花为红色和白色2个红花品系中根、茎、叶和苞片以及花不同发育时期1、S2、S3、S4、S5和S6期中表达量,利用Premier 5设计基因的荧光定量PCR引物,上游引物:5’-AAAGCCCGGCTG- ACAAAGA-3’,下游引物:5’-CGCACCTCCAACT- CCAACA-3’,内标参比基因为基因,上游引物:5’-CATCCATTATCCAACAATC-3’,下游引物:5’-AAGAGTAATCAGTCTCCA-3’,使用TaKaRa TB Green®Premix Ex TaqTMII(Tli RNaseH Plus)进行qRT-PCR反应,反应条件:95 ℃、3min,随后进行45个循环的95 ℃、10 s;55 ℃、30 s;72 ℃、28 s。通过熔解曲线分析监测PCR扩增的特异性,反应程序从55 ℃到94 ℃ 0.1 ℃/s速度。每个荧光定量PCR反应重复3次,通过2-ΔΔCt相对定量法对基因进行表达水平分析。以Student's检验(Student's-test)方法对基因在红色和白色两个红花品系中不同组织和花发育不同时期的表达量进行统计分析,<0.05表示差异具有显著水平。
3 结果与分析
3.1 CtHY5基因的克隆
利用RT-PCR方法,从管状花为红色的红花品系中得到了1个cDNA全长为462 bp的基因同源基因(图1-A),命名为。为了进一步对红花基因的内含子和外显子结构进行分析,以红花幼苗DNA为模板,进行PCR扩增,经测序、比对分析发现红花HY5基因DNA全长为1941 bp(图1-B),包含4个外显子,长度分别为97 bp、166 bp、157 bp和42 bp,3个内含子,分别为1083 bp、320 bp、和76 bp(图1-C)。
3.2 CtHY5基因的序列分析
红花基因cDNA序列经ORF finder在线软件分析,显示其具有1个长度为XX bp的完整读码框,编码153个氨基酸(图2-A),通过在线软件Prot Param对CtHY5蛋白的氨基酸组成及比例进行分析,统计结果表明Glu数量最多,占11.8%,trp和phe数量最少,占0.7%(表1)。其分子式为C711H1204N234O239S5,包含2393个原子,相对分子质量为17 015,理论等电点为9.73,半衰期为30 h,带正电荷氨基酸残基和带负电荷氨基酸残基数目分别为29、23,脂肪系数为66.99,亲水性指数为−1.014,不稳定指数为58.82,表明CtHY5为不稳定的亲水性蛋白。
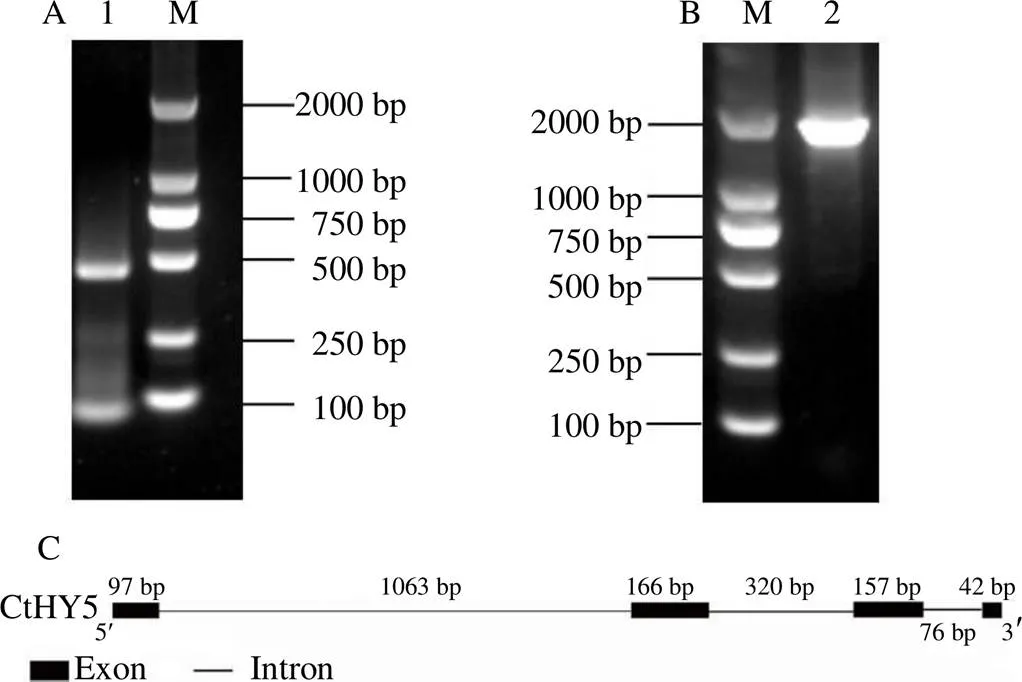
A-红花HY5 cDNA序列扩增 B-红花HY5 DNA序列扩增 C-红花HY5基因内含子、外显子结构示意图
用NCBI的CD-Search分析CtHY5蛋白的功能结构域,结果显示该蛋白属于bZIP转录因子超家族中的一员,含有3个保守结构域,分别为bZIP-HY5-like、BRLZ和bZIP-1(图2-A、B)。其中bZIP-HY5-like结构域包括86位到136位共51个氨基酸,其序列RLKRLLRNRVSAQQARERKKAYLLELEVRVKELE KK NSEVEERFSTLQNENQ;BRLZ结构域包括81位到143位共65个氨基酸,其序列KENKR LKRLLRNRVSAQQARERKKAYLLELEVRVKELEK KNSEVEERFSTLQNEN Q MLRHILK;bZIP-bZIP-1结构域包括84位到143位共60个氨基酸,其序列KRLKRLLRNRVSAQQARERKKAYLLELEVRVKELEKKNSEVEERFSTLQNENQMLRHILK。
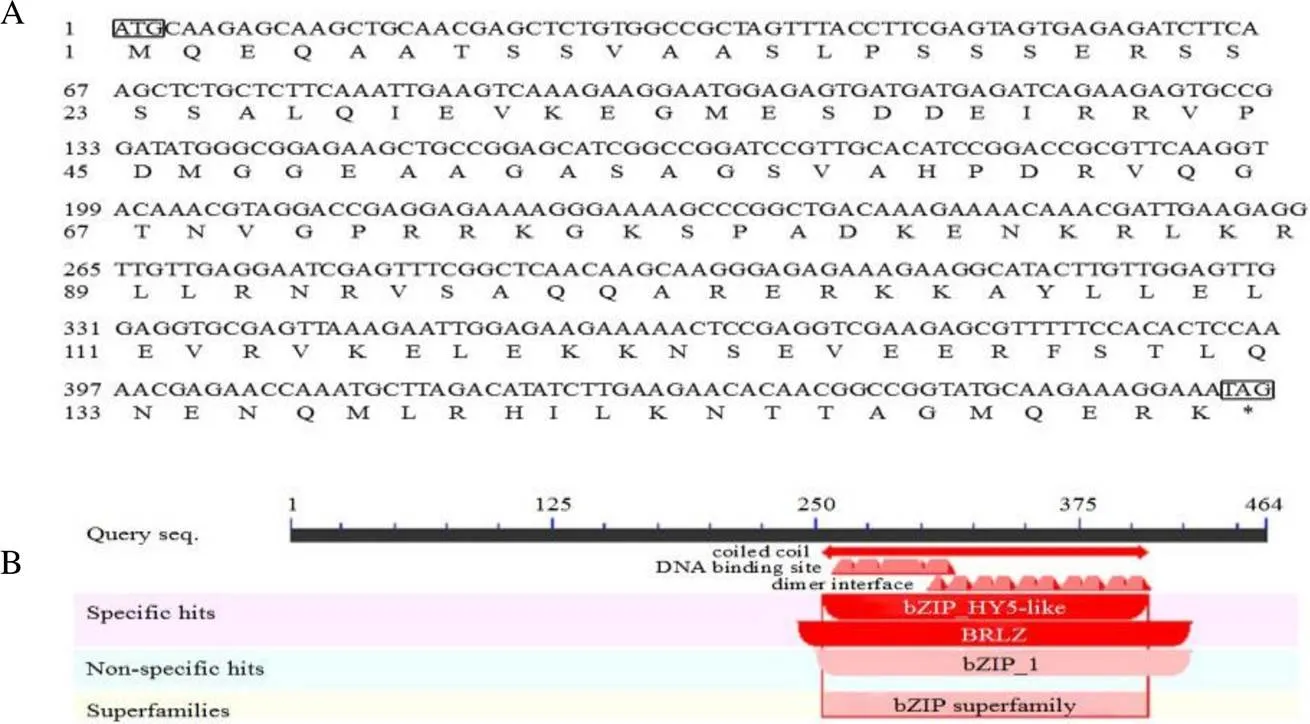
A-CtHY5基因的cDNA序列及其推导的氨基酸序列,黑色方框代表起始密码子和和终止密码子 B-CtHY5蛋白保守结构域预测
表1 CtHY5氨基酸成分
Table 1 Amino acid composition of CtHY5
氨基酸数量占比/%氨基酸数量占比/% Ala (A)15 9.8 Leu (L)12 7.8 Arg (R)1610.5 Lys (K)13 8.5 Asn (N) 7 4.6 Met (M) 5 3.3 Asp (D) 5 3.3Phe (F) 1 0.7 Gln (Q) 9 5.9Pro (P) 5 3.3 Glu (E)1811.8Ser (S)1711.1 Gly (G) 9 5.9Thr (T) 5 3.3 His (H) 2 1.3Tyr (Y) 1 0.7 Ile (I) 3 2.0Val (V)10 6.5
3.3 CtHY5基因的生物信息学分析
利用ExPASy-Prot Scale在线工具对CtHY5蛋白的亲水/疏水性分析表明,其中正值表示疏水性,负值表示亲水性,通过分析表明,CtHY5蛋白负值数量多于正值数量,且第10位缬氨酸(Val,V)具有最高分值1.144,疏水性最强,第84位赖氨酸(Lys,K)具有最低分值−3.044,亲水性最强(图3)。
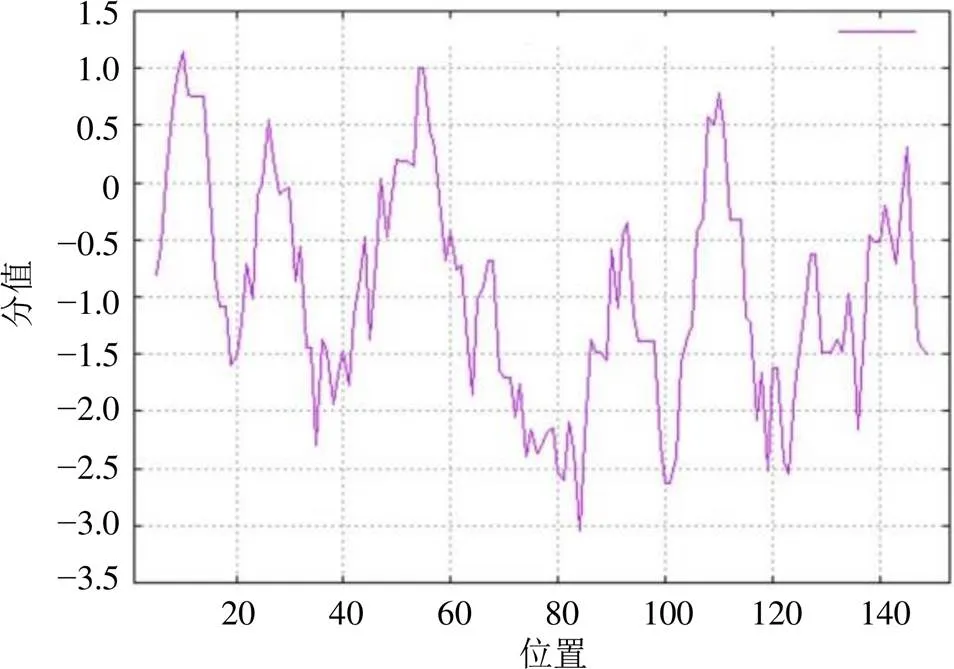
图3 CtHY5蛋白疏水性/亲水性预测
通过ExPASY中的SOPMA tool预测CtHY5蛋白的二级结构,结果显示CtHY5蛋白的二级结构中α-螺旋(α-helices)占58.17%,无规则卷曲(random coil)占36.60%,β-折叠(β-turn)和延伸链(extended strand)较少,分别为3.92%和1.31%(图4-A)。为了进一步了解红花HY5蛋白的结构,利用SWISS-MODEL对CtHY5蛋白进行同源建模,得到红花HY5蛋白的三维空间模型(图4-B),ExPAsy structure assessment程序评测推导,CtHY5蛋白模型6iak.1.A得分为0.74,与来自鸡中CREB3蛋白序列的相似性为34.78%,并且,该蛋白可能以同源二聚体的形式发挥生物学功能。
利用SignalP-5.0 Server在线软件对CtHY5蛋白的氨基酸序列进行信号肽分析,结果表明,该蛋白中无信号肽存在,为非分泌蛋白。为了进一步对CtHY5的功能进行预测,通过PSORT在线软件对CtHY5蛋白亚细胞定位进行分析,结果表明CtHY5定位于细胞核的概率为95.7%,定位与细胞质中的概率仅为4.3%,这与CtHY5作为转录因子在细胞核中调控基因表达的功能预测相一致。
3.4 CtHY5蛋白的系统进化分析
通过NCBI Blastp查找CtHY5在其他物种中的同源氨基酸序列,比对分析发现,CtHY5与刺菜蓟s Linn. HY5的相似性最高,为96.08%,与向日葵Linn. HY5的相似性为88.05%,与薇甘菊Kunth HY5的相似性为86.71%,与黄花蒿Linn. HY5的相似性为84.71%,与莴苣Linn. HY5的相似性为82.91%。与其他物种中HY5类似,CtHY5具有此类蛋白所特有的典型的碱性亮氨酸拉链结构域和DNA结合域,这些结构在不同物种中保守性较高,也是其发挥转录因子功能所必须的(图5)。
通过在NCBI、UniProt数据库及文献报道中搜索已克隆研究的其物种中HY5,与CtHY5进行系统进化树构建,分析CtHY5与其他物种中HY5的进化关系,从进化树分析发现,CtHY5与菊科中的刺菜蓟、黄花蒿、薇甘菊、向日葵、莴苣中的HY5亲缘关系较近,在进化树种聚为一支,而与来自其它科植物如芝麻、烟草、玉米、风信子中的HY5亲缘关系相对较远(图6)。
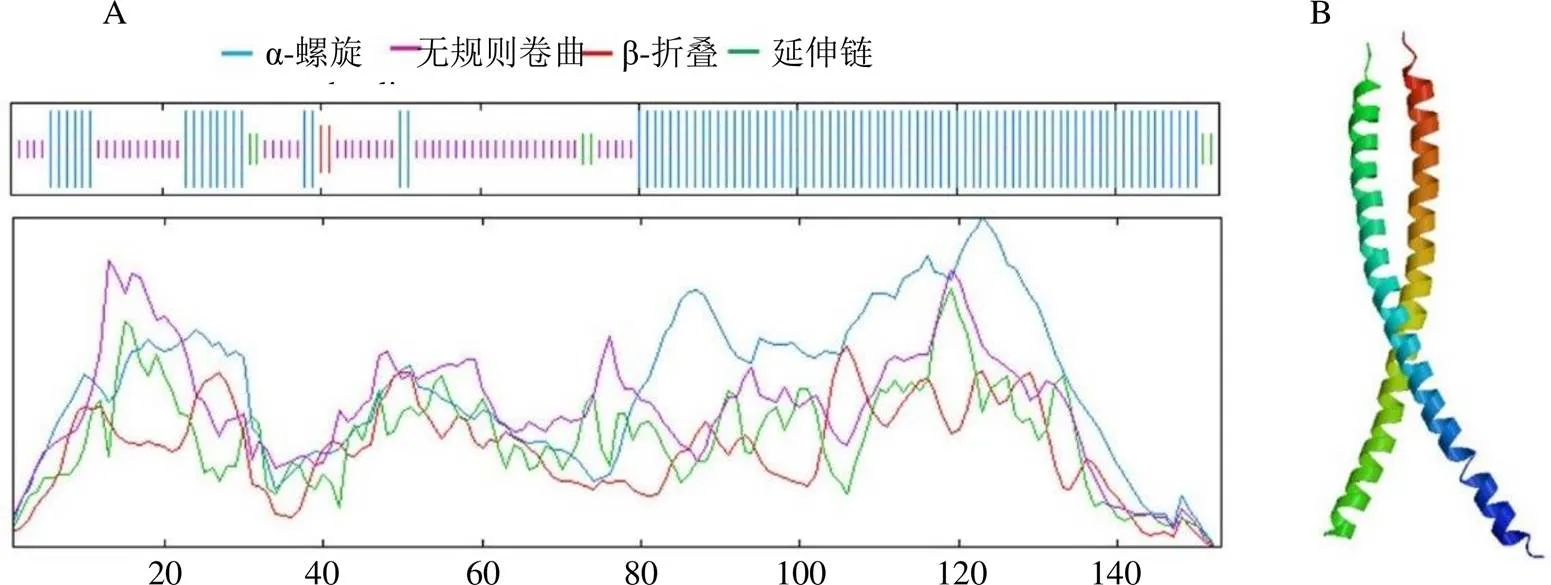
A-CtHY5二级结构 B-CtHY5三级结构

●表示被酪蛋白激酶II(CKII)磷酸化的丝氨酸位点; 横线代表蛋白的碱性区域,*标志为代表亮氨酸重复序列,方框为亮氨酸拉链结构域
通过MEME对系统进化树中17个HY5序列进行模块结构分析,分析表明基序列1和3最保守,在所有分析序列中都存在,基序5和6较特异,基序5只在油菜、玉米和风信子中存在,而基序6只在红花和刺菜蓟中存在。从模块结构特征可以看出,亲缘关系越近的物种中模块基序结构越相似。进一步比较分析发现,在所有分析序列中,只有刺菜蓟的模体基序与红花HY5一样,均含有4个模块基序,分别为基序1、2、3和6,这预示着它们可能具有相似的功能(图6)。
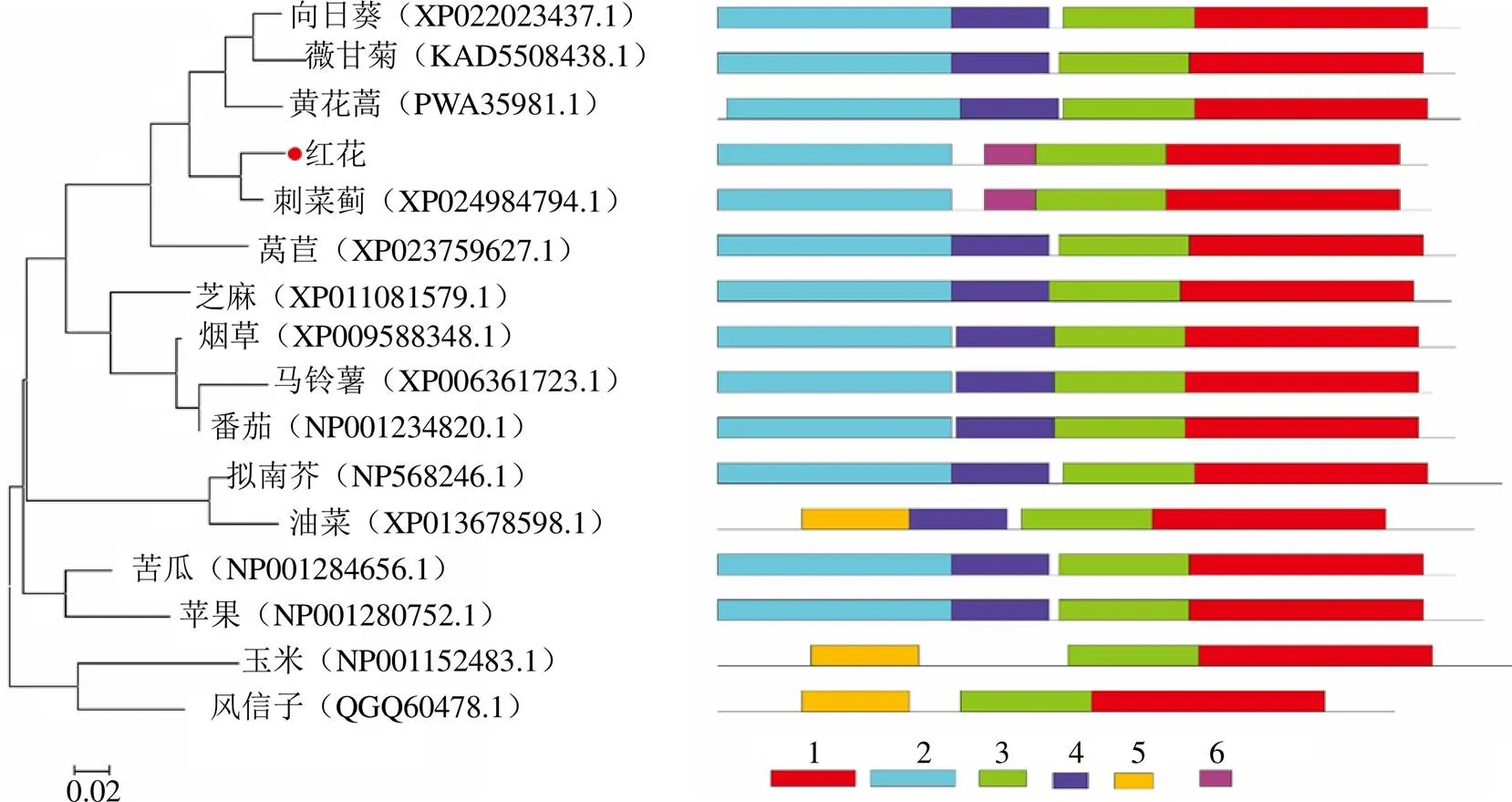
图6 不同植物基于HY5氨基酸序列的邻接法系统进化树及模体分析
3.5 CtHY5基因表达分析
利用RT-PCR检测基因的各组织中表达,结果显示基因在红花所有组织中均有表达,在花中表达量最高,其次是苞片和茎,在根中表达量最低,并且白色红花的苞片和花中基因表达量都明显高于红色红花(<0.01)(图7-A)。
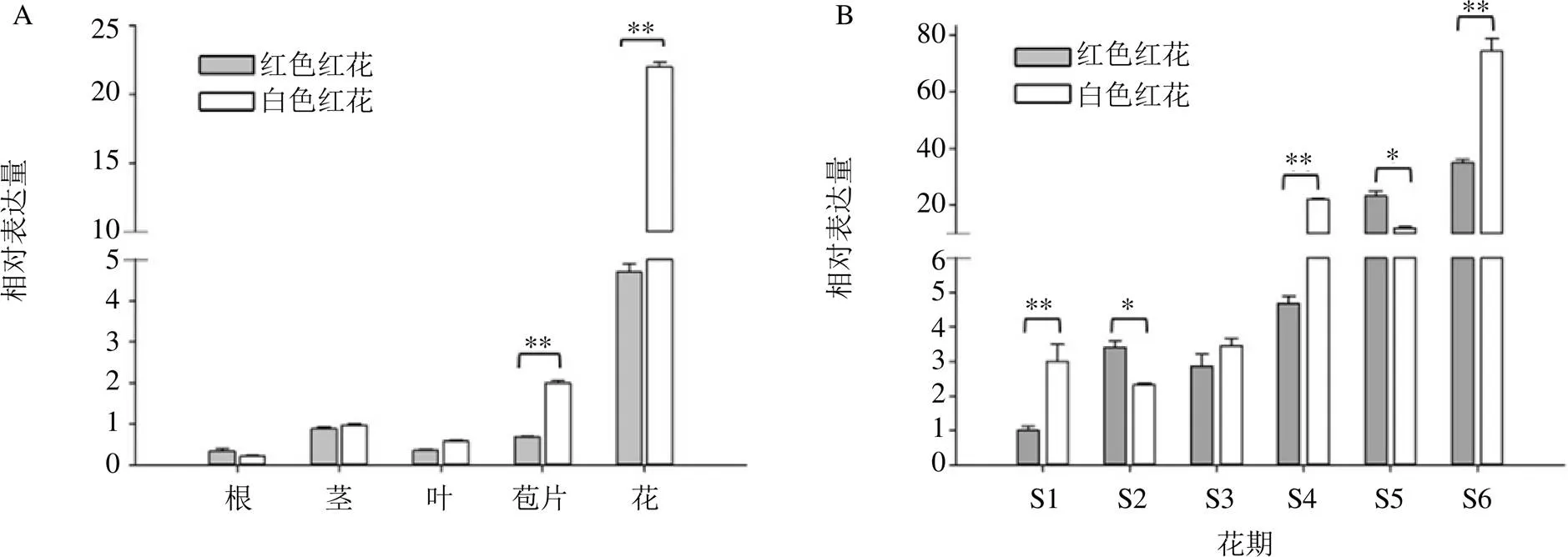
*表示差异显著,P<0.05,**表示差异极显著,P<0.01
对基因在花发育的不同时期定量分析结果表明,红色红花品系中基因的表达量呈现先增加后降低又增加的表达趋势,而白色红花品系中基因的表达量则呈现先降低后增加的表达趋势,并表现出白色红花品系中基因的表达量要整体高于红色红花,统计结果显示,白色和红色红花品系中基因的表达量在花发育的S1、S4和S6时期差异极显著(<0.01),S2和S5期差异显著(<0.05),S3期差异不明显(图7-B)。
4 讨论
HY5属于bZIP类转录因子家族,最早是在拟南芥中克隆得到[1]。HY5作为光敏色素下游的核心光信号调节因子,在光信号途径中发挥着重要的作用[21-22]。依据红花转录组测序数据,本研究克隆得到一个基因的cDNA和DNA全长序列,其DNA序列具有4个外显子和3个内含子。生物信息学分析显示,CtHY5与为亲水性蛋白,二级结构主要由α-螺旋组成和无规则卷曲组成,仅含有少量β-折叠和延伸链,三级结构预测显示CtHY5蛋白呈拉链形状,并且以同源二聚体的形式存在。亚细胞定位分析表明,该蛋白定位于细胞核中,这与其转录因子作用角色相一致,这些结果与已报道的云梨、苦荞中的HY5分析结果相一致[16, 22]。
序列分析表明,红花基因编码153个氨基酸,与拟南芥、苦荞和番茄中HY5相比氨基酸序列相对较短[11, 16, 22]。蛋白结构域预测分析发现,与黄花蒿中HY5蛋白类似[10],CtHY5蛋白也具有高度灵活、无序的N末端,这可能是造成CtHY5蛋白比其它植物中HY5同源蛋白相对较短的原因之一。并且,HY5蛋白N端含有一个酪蛋白激酶(casein kinase II)的磷酸化位点,该位点可以与具有E3泛素连接酶活性的COP1(constitutively photomorphogenic 1)蛋白直接相互作用。COP1与HY5在植物光信号转导中的作用相反,在黑暗条件下,COP1进入细胞核与HY5互作并将其泛素化,并促进26S蛋白酶对它的降解。而光照条件下,HY5被活化并相应光照参与其他生理反应调节过程[23-24]。本研究中CtHY5蛋白具有COP1蛋白磷酸化位点,这暗示着在红花中HY5极大可能通过与COP1蛋白互作参与植物光信号生理调节过程。
前人研究发现,HY5可以直接影响约3 000个基因的表达,基因可以通过其DNA结合结构域直接结合启动子区域的顺式元件调控基因其表达,这些顺式元件包括T/G box(CACGTT),E-box(CAATTG),GATA-box(GATGATA),ACE-box(ACGT),Z-box(ATACGGT),C-box(GTCANN),hybrid C/G(G)和C/A boxes,通过结合于含有这些顺式元件的基因参与植物光信号、生物节律、开花时间、营养元素、植物激素响应等各个生理过程[25-26]。最近的研究表明,基因在植物逆境胁迫响应中也起着非常重要的作用,在番茄中,HY5还可以通过结合于冷胁迫信号途径关键因子CBF(C-repeat binding factors)基因启动子区域激活其表达,从而增强番茄的抗冷性[27]。在拟南芥的研究中发现,HY5可以结合于基因启动子的一个新发现的顺式元件B(GGCCACGCCA)上并促进该基因在紫外、强光照和低温胁迫下的表达,从而提高植物的抗胁迫反应[28]。本研究克隆的CtHY5也具有其典型的DNA结合结构域,与拟南芥、番茄等植物一样,该结构域也位于CtHY5蛋白的碱性区域内,并且氨基酸序列具有高度的保守性,通过荧光定量PCR分析发现,基因在红花的根、茎、叶、苞片和花等组织中呈现组成型表达,在拟南芥中,在根、下胚轴、茎和花器官中也呈现组成型表达,大量的研究表明,基因参与拟南芥光形态建成、侧根发育、生物及非生物胁迫的多个生理过程[22],这暗示着基因也可能参与红花生长发育的多个生理过程中。
HY5在植物色素生物合成调控中也发挥着重要作用,HY5可以直接结合于植物的花青素生物合成途径相关基因、基因的启动子区域ACE元件上,调控这些基因的表达[12]。此外,HY5基因还调控类胡萝卜素合成基因及叶绿素合成基因、、的表达[29]。为了探索基因在红花色素合成调控中的作用,本实验对基因在2个不同花色红花品系的不同组织、花发育不同时期中的表达模式进行了分析,结果表明基因在红花管状花中表达量比其它组织部位都高,并且随着花发育进程呈现表达量增加的趋势,并且在管状花为白色的红花品系中的表达量要明显高于管状花花为红色红花品系中,这说明CtHY5可能参与了红花花色素的合成或积累调控过程,但其调控机制可能不同于其它植物,这为后续CtHY5调控红花花色素的机制提供了思路。
利益冲突 所有作者均声明不存在利益冲突
[1] Oyama T, Shimura Y, Okada K. TheHY5 gene encodes a bZIP protein that regulates stimulus-induced development of root and hypocotyl [J]., 1997, 11(22): 2983-2995.
[2] Ang L H, Chattopadhyay S, Wei N,. Molecular interaction between COP1 and HY5 defines a regulatory switch for light control ofdevelopment [J]., 1998, 1(2): 213-222.
[3] Hardtke C S, Gohda K, Osterlund M T,. HY5 stability and activity inis regulated by phosphorylation in its COP1 binding domain [J]., 2000, 19(18): 4997-5006.
[4] Jakoby M, Weisshaar B, Dröge-Laser W,. bZIP transcription factors in[J]., 2002, 7(3): 106-111.
[5] Cluis C P, Mouchel C F, Hardtke C S. Thetranscription factor HY5 integrates light and hormone signaling pathways [J]., 2004, 38(2): 332-347.
[6] Maxwell B B, Andersson C R, Poole D S,. HY5, circadian clock-associated 1, and a-element, DET1 dark response element, mediate DET1 regulation of chlorophyll a/b-binding protein 2 expression [J]., 2003, 133(4): 1565-1577.
[7] Lee J, He K, Stolc V,. Analysis of transcription factor HY5 genomic binding sites revealed its hierarchical role in light regulation of development [J]., 2007, 19(3): 731-749.
[8] Huang L F, Zhang H C, Zhang H Y,. HY5 regulates nitrite reductase 1 (NIR1) and ammonium transporter1;2 (AMT1;2) inseedlings [J]., 2015, 238: 330-339.
[9] Chen X B, Yao Q F, Gao X H,. Shoot-to-root mobile transcription factor HY5 coordinates plant carbon and nitrogen acquisition [J]., 2016, 26(5): 640-646.
[10] Fu X Q, Peng B W, Hassani D,. AaWRKY9contributes to light- and jasmonate-mediated to regulate the biosynthesis of artemisinin in[J]., 2021, 231(5): 1858-1874.
[11] Wang W H, Wang P W, Li X J,. The transcription factor SlHY5 regulates the ripening of tomato fruit at both the transcriptional and translational levels [J]., 2021, 8(1): 83.
[12] Shin J, Park E, Choi G. PIF3 regulates anthocyanin biosynthesis in an-dependent manner with both factors directly binding anthocyanin biosynthetic gene promoters in[J]., 2007, 49(6): 981-994.
[13] Shin D H, Choi M, Kim K,. HY5 regulates anthocyanin biosynthesis by inducing the transcriptional activation of the MYB75/PAP1 transcription factor in[J]., 2013, 587(10): 1543-1547.
[14] Liu W J, Wang Y C, Yu L,. MdWRKY11 participates in anthocyanin accumulation in red-fleshed apples by affecting MYB transcription factors and the photoresponse factor MdHY5 [J]., 2019, 67(32): 8783-8793.
[15] 郭继平. 油菜HY5基因的电子克隆及生物信息学分析 [J]. 北方园艺, 2013(23): 116-118.
[16] 苏俊, 陈璐, 张晓东, 等. “云红梨1号”HY5基因的克隆及生物信息学分析 [J]. 北方园艺, 2020(10): 39-44.
[17] Liu C C, Chi C, Jin L J,. The bZip transcription factor HY5 mediates CRY1a-induced anthocyanin biosynthesis in tomato [J]., 2018, 41(8): 1762-1775.
[18] 邓娇, 赵佳利, 张丽洁, 等. 苦荞FtHY5基因克隆、生物信息学及参与苦荞花青素合成分析[J]. 分子植物育种, 2021, 5: 12.
[19] Chen Z W, Liu L, Liu Y Y,. Hydroxysafflor yellow A induces autophagy in human liver cancer cells by regulating Beclin 1 and ERK expression [J]., 2020, 19(4): 2989-2996.
[20] Zhang L L, Tian K, Tang Z H,. Phytochemistry and pharmacology ofL [J]., 2016, 44(2): 197-226.
[21] Jing Y J, Zhang D, Wang X,.chromatin remodeling factor PICKLE interacts with transcription factor HY5 to regulate hypocotyl cell elongation [J]., 2013, 25(1): 242-256.
[22] Gangappa S N, Botto J F. The multifaceted roles of HY5 in plant growth and development [J]., 2016, 9(10): 1353-1365.
[23] Osterlund M T, Hardtke C S, Wei N,. Targeted destabilization of HY5 during light-regulated development of[J]., 2000, 405(6785): 462-466.
[24] Yu Y W, Wang J, Zhang Z J,. Ethylene promotes hypocotyl growth and HY5 degradation by enhancing the movement of COP1 to the nucleus in the light [J]., 2013, 9(12): e1004025.
[25] Zhang H Y, He H, Wang X C,. Genome-wide mapping of the-mediated gene networks inthat involve both transcriptional and post-transcriptional regulation [J]., 2011, 65(3): 346-358.
[26] Casal J J. Photoreceptor signaling networks in plant responses to shade [J]., 2013, 64: 403-427.
[27] Zhang L Y, Jiang X C, Liu Q Y,. The HY5 and MYB15 transcription factors positively regulate cold tolerance in tomato via the CBF pathway [J]., 2020, 43(11): 2712-2726.
[28] Hayami N, Sakai Y, Kimura M,. The responses ofearly light-induced Protein2 to ultraviolet B, high light, and cold stress are regulated by a transcriptional regulatory unit composed of two elements [J]., 2015, 169(1): 840-855.
[29] Toledo-Ortiz G, Johansson H, Lee K P,. The HY5-PIF regulatory module coordinates light and temperature control of photosynthetic gene transcription [J]., 2014, 10(6): e1004416.
Cloning and expression analysis of, a key gene involved in light signaling pathway in
TAN Zheng-wei, LU Dan-dan, LI Lei, YU Yong-liang, XU Lan-jie, YANG Hong-qi, YANG Qing, DONG Wei, LI Chun-ming, AN Su-fang, LU Hai-ling, LIANG Hui-zhen
Henan Sesame Research Center, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China
HY5 (ELONGATED HYPOCOTYL5) is a key transcription factor in light signaling pathway. In this study, thegene was cloned from, and the bioinformatics and expression profile analysis ofwere performed with bioinformatics and quantitative Real-time PCR (qRT-PCR) methods to provide a reference for the functional study of thegene.Based on the transcription data of safflower, primers were designed and the full-length cDNA ofwere cloned from safflower to obtain DNA sequences. The characteristics of physiochemical properties, structures and function of the deduced CtHY5protein were determined using a series of bioinformatics tools. The expression ofin different tissues and flower development stages of safflower was detected by fluorescence quantitative PCR.The full length cDNA sequence ofgene was 462 bp, encoding 153 amino acids, while the full length DNA sequences ofgene were 1 941 bp, containing four exons and three introns. Bioinformatics analysis showed that CtHY5 was hydrophilic protein and was located in the nucleus. Phylogenetic tree and motif structure analysis revealed that CtHY5 was more closely related to HY5 homologous protein in,,,and, which all of them belonged to Asteraceae. The results of qRT-PCR analysis showed that the expression ofgene was the highest in the flowers, followed by stems and bracts, and the lowest in roots. And the expression ofgene in all tissues of safflower was significantly higher in white safflower line than that in red safflower line.CtHY5, the key transcription factor of light signaling pathway, was first isolated from safflower in this study. The expression pattern ofin different lines of safflower was studied, which laid a foundation for further study on its biological function.
L.; light signaling pathway; HY5; gene clone; expression analysis
R286.12
A
0253 - 2670(2022)18 - 5825 - 09
10.7501/j.issn.0253-2670.2022.18.026
2022-02-11
财政部和农业农村部:国家现代农业产业技术体系资助(CARS-21);河南省中央引导地方科技发展专项自由探索类项目(YDZX20214100001804);河南省农科院新兴学科发展专项(2022XK03);河南省科技攻关项目(222102110379、222102110466);河南省农科院自主创新专项基金(2022ZC64)
谭政委(1983—),男,助理研究员,从事分子生药学研究。Tel: (0371)65738565 E-mail: zhwtan@126.com
梁慧珍(1968—),女,研究员,从事药用植物遗传育种及品质改良工作。Tel: (0371)65751589 E-mail: lhzh66666@163.com
[责任编辑 时圣明]
