拟南芥HWS是模拟靶标介导miRNA降解途径的核心因子
2022-08-30郭梦玮任国栋
杨 林,李 宁,郭梦玮,梅 俊,任国栋
(复旦大学 生命科学学院,植物科学研究所,上海 200438)
miRNA是一类长度在20~24个核苷酸的小分子非编码RNA,参与动植物生殖、发育以及环境应答等几乎所有生物学过程。miRNA组装到ARGONAUTE(AGO)蛋白上形成沉默复合体(RNA Induced Silencing Complex,RISC),并通过序列配对引导AGO蛋白到靶标RNA上,进而引起转录后水平的基因沉默[1]。体内miRNA表达水平主要受生成及降解两大代谢通路调控,miRNA表达水平异常可引起生物体一系列发育及生理缺陷[2-3]。过去20年的研究形成了较为完善的miRNA合成代谢通路,但关于miRNA降解的生化途径尚未完全建立[1]。
在植物中,甲基转移酶HUA ENHANCER 1(HEN1)介导的2′-O-甲基化修饰对于维持miRNA的稳定性至关重要[4]。HEN1缺失导致miRNA 3′末端受尿苷化加尾和(或)截短,进而失稳降解[5]。末端尿苷转移酶HEN1 SUPPRESSOR 1(HESO1)与UTP:RNA URIDYLYLTRANSFERASE 1(URT1)协同催化AGO1结合的miRNA尿苷化,促进非甲基化miRNA降解[6-10]。此外,拟南芥3′-5′核酸外切酶ATRIMMER 2(ATRM2)通过降解非甲基化miRNA/miRNA*,对进入沉默复合体的miRNA进行质量监控[11]。鉴于植物miRNA整体上呈高甲基化状态,因此非甲基化miRNA降解的生物学意义主要在于质量控制。
miRNA在环境响应中扮演着十分重要的角色,那么当细胞发育命运转变或外界环境变化时,必然有一部分miRNA需要被及时清除。2007年,Franco-Zorrilla等[12]发现磷饥饿可诱导长链非编码RNAInducedbyPhosphateStarvation1(IPS1)高表达。IPS1可被miR399靶向识别,但因其配对中间区存在3个碱基的凸起而不能被AGO1切割,因此IPS1与miR399的结合可以释放miR399对内源靶标PHO2的抑制,从而调节植物的低磷响应。研究人员将此类中间区域含有3个碱基凸起的靶标RNA称之为模拟靶标。根据此规则设计的人工模拟靶标如模拟靶基因MIM和短串联靶基因模拟(Short Tandem Target Mimic,STTM)被广泛应用于特定miRNA或miRNA家族的功能抑制[13-14]。继植物中报道模拟靶标后,研究人员发现模拟靶标同样存在于动物中,并在此基础上提出竞争性内源靶标的假说[15]。有意思的是,Yan等[14]发现STTM能诱导miRNA的降解,核酸外切酶SDN(Small RNA Degrading Nuclease)被报道参与该过程的调控,但具体工作机制不清。
本课题组及Detlef Weigel组各自通过正向遗传学手段鉴定到F-box蛋白HWS(HAWAIIAN SKIRT)功能缺失后可恢复STTM160和MIM156(target mimicry of miR156)引起的植株发育缺陷表型[16-17]。作为E3泛素连接酶成员,HWS行使功能依赖于其F-box结构域[16-17]。当HWS突变后,内、外源的模拟靶标及其对应miRNA因无法正常降解而在沉默复合体中堆积,提示HWS可能通过泛素-蛋白酶体途径介导结合模拟靶标的沉默复合体的选择性清除[16]。与植物中使用严谨的互补配对规则不同[18],动物miRNA种子区域(第2到8位)与靶基因的互补配对即可完成对靶标的识别[2]。靶标RNA与miRNA 3′区的额外互补配对会导致miRNA发生加尾或截短,并诱导miRNA降解,这种现象称为靶标介导的miRNA降解(Target Directed miRNA Degradation,TDMD)[19]。然而,敲除尿苷化加尾酶TUT4/7并不能阻碍miRNA降解,表明尿苷化并非TDMD的主要机制[20]。最新研究发现Cullin-Ring型E3泛素连接酶ZSWIM8通过识别结合3′区域存在额外配对的靶标的沉默复合体,启动泛素 蛋白酶体系统降解AGO,进而导致靶向miRNA释放并最终降解[21-22]。
为进一步研究模拟靶标介导miRNA降解的分子遗传机制,我们对STTM160转基因诱变群体进行了深度筛选,鉴定到7个莲座叶表型几乎完全恢复的突变体株。对这些突变体的基因型进行考察,发现它们均携带HWS基因的等位突变,表明HWS是模拟靶标介导miRNA降解途径最核心的成员。另外,我们还对HWS的组织表达模式、亚细胞定位以及保守性进行了分析,这些结果为后续深入研究HWS参与模拟靶标介导的沉默复合体选择性清除机制及生物学意义奠定了基础。
1 材料与方法
1.1 材料
拟南芥材料均为Col-0背景。突变体STTM160、STTM160hws-6、hws-6见已发表论文[16]。培养条件:光照16 h/黑暗8 h,光照强度为120μmol·m-2·s-1,温度(22±2)℃,湿度约75%。土壤配方为V蛭石∶V营养土∶V珍珠岩=9∶3∶0.5。
1.2 方法
1.2.1 EMS(甲磺酸乙酯)诱变
用超纯水将1 m LSTTM160转基因种子清洗干净,加水至25 m L,添加62.5μL的EMS(终浓度为0.25%,在通风橱中操作)。拧紧管盖后,用封口膜将管口封好,放置于摇床上处理15 h左右。尽可能吸走EMS处理液(废液加NaOH至终浓度1 mol/L去除诱变毒性),用40 m L左右的超纯水清洗种子8次,最后一次清洗时将种子浸泡1 h,以便让残留EMS从种子中扩散出去。将种子悬浮于0.1%琼脂糖溶液中,用移液器播于土壤中,收取M1代植株种子用于后续筛选。
1.2.2 总RNA的提取
取0.1 g花序组织,使用TRIzol法抽提总RNA并溶于20μL DEPC处理水中,用分光光度计(Biochrom,BioDrop)将同一批样品的RNA浓度调一致(~1μg/μL)。
1.2.3 小RNA Northern blot杂交
小RNA Northern blot杂交参考文献[16]。简要步骤如下:总RNA用50%PEG8000和5 mol/L NaCl处理后去除高分子量RNA,上清经乙醇、3 mol/L醋酸钠、糖原沉淀后得到低分子量RNA。低分子量RNA经变性丙烯酰胺凝胶电泳(8 mol/L尿素,16%丙烯酰胺)后,转移至尼龙膜上,紫外交联,用生物素或同位素标记探针杂交,最后化学显影。
1.2.4 实时荧光定量PCR
提取拟南芥花序的总RNA后,利用反转录试剂盒(Ta KaRa,PrimeScriptTMRT reagent Kit with gDNA Eraser)进行反转录反应。组合使用SYBR®Green master mix(BIO-RAD)进行定量PCR反应(反应程序:95℃,1 min;95℃,15 s;60℃,30 s;72℃,30 s;45个循环,65℃,5 s;95℃,5 s),反应结束后运用2-ΔΔCT法计算基因的相对表达量。
1.2.5 茎环-RT-PCR
2μg花序总RNA采用SuperScript Ⅲ反转录酶(ThermoFisher)进行反转录反应,具体实验操作步骤参考文献[23]。
1.2.6HWS基因的生物信息学分析
从Phytozome(https://phytozome-next.jgi.doe.gov/)、TreeGenes(https://treegenesdb.org/)、SGN(https://solgenomics.net/)、Ensembl(https://plants.ensembl.org/)下载不同植物的HWS同源蛋白质序列。随后提取出每个基因最长isoform所对应的蛋白质序列,使用OrthoFinder(https://github.com/davidemms/OrthoFinder)鉴定直系同源基因和基因重复事件[24]。运用MEGA-X(Molecular Evolutionary Genetics Analysis)软件进行序列比对,选择Neighbor-Joining算法(Bootstrap,设定初始值1 000次),构建系统发育树[25]。使用msa包绘制Logo-format图[26]。
1.2.7 引物信息
实验所用到的引物序列见表1。
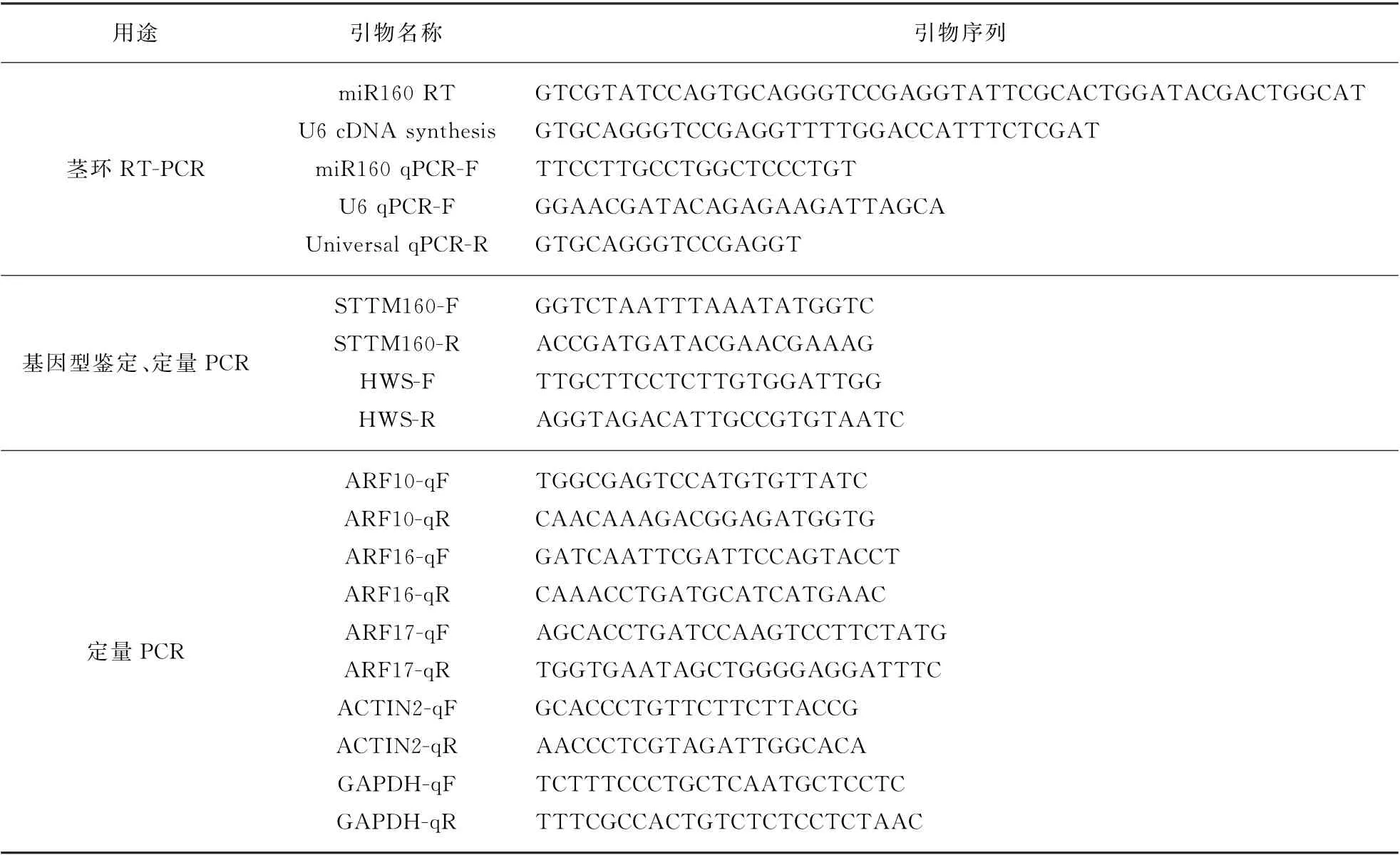
表1 引物清单Tab.1 List of primers
2 结 果
2.1 多个新的HWS等位突变恢复STTM160植株表型
STTM160单拷贝纯合转基因株系L3-9呈现叶片狭长,真叶锯齿状,育性下降等表型。实验室前期通过EMS诱变筛选L3-9的表型回复突变体,鉴定到两个HWS的等位突变体(STTM160hws-6,STTM160hws-7)可完全恢复L3-9的发育缺陷[16]。为进一步筛选参与模拟靶标介导miRNA降解的新成员,我们对诱变M2代突变体库进行深度遗传筛选,获得了7株表型恢复良好的突变体(图1(a),见第378页)。对上述回复突变体进行STTM160基因型检测,排除了野生型污染。为考察上述回复突变体是否携带HWS基因突变,我们用高保真热稳定DNA聚合酶扩增出HWS全长基因序列,对PCR产物进行测序后发现均含有HWS的突变,且突变位点均为G到A(或C到T),与EMS引起的突变类型一致(图1(b,c),见第378页)。由于正向遗传学是非偏向型的研究手段,因此该结果表明HWS是模拟靶标介导miRNA降解途径的核心成员。此外,我们还鉴定到两株表型部分恢复的突变体(数据未展示),基因型检测发现HWS位点为野生型,后续将构建作图群体对其进行基因定位。

图1 STTM160表型抑制子筛选鉴定到多个HWS等位突变Fig.1 Genetic screening of STTM160 suppressors identified multiple HWS allelic mutations
拟南芥HWS蛋白共411个氨基酸,包含3个区域,分别为N端的F-box区(第40~85位氨基酸),预测的跨膜区TMR(transmembrane-spanning region)(第120~140位氨基酸),以及近C端的Kelch-2结构域(第290~338位氨基酸)[27]。我们发现鉴定到的突变位点散布于HWS的各个区段。其中hws-9是F-box区域的突变,提示HWS参与miRNA降解依赖于泛素化途径。有意思的是,多个C末端非同义突变(hws-8,hws-12,hws-13)均能恢复STTM160的表型,表明C末端对于HWS功能发挥至关重要(图1(b,c))。
2.2 HWS突变显著恢复STTM160背景下miR160的表达水平
为排除STTM160转基因沉默造成突变体表型恢复的可能性,我们选取其中两个等位回复突变体(STTM160hws-8、STTM160hws-14,图2(a)),并利用qPCR检测其STTM160转基因的表达水平。结果表明,STTM160hws-8、STTM160hws-14中STTM160的表达量与亲本STTM160相当(图2(b)),证明STTM160hws回复突变体未发生转基因沉默。
随后,我们通过Northern blot和茎环-RT-PCR实验检测了STTM160hws-8、STTM160hws-14中miR160的表达水平。结果表明,STTM160hws-8、STTM160hws-14中miR160的丰度相较于STTM160得到了显著恢复(图2(c,d))。有意思的是,3个独立hws等位突变均不能将miR160完全恢复至野生型状态,提示可能存在其他分支途径。相应地,miR160靶标基因ARF10/ARF16/ARF17的表达在STTM160hws-8、STTM160hws-14中重新受到了不同程度的抑制(图2(e)),这与发育缺陷表型的恢复是一致的。
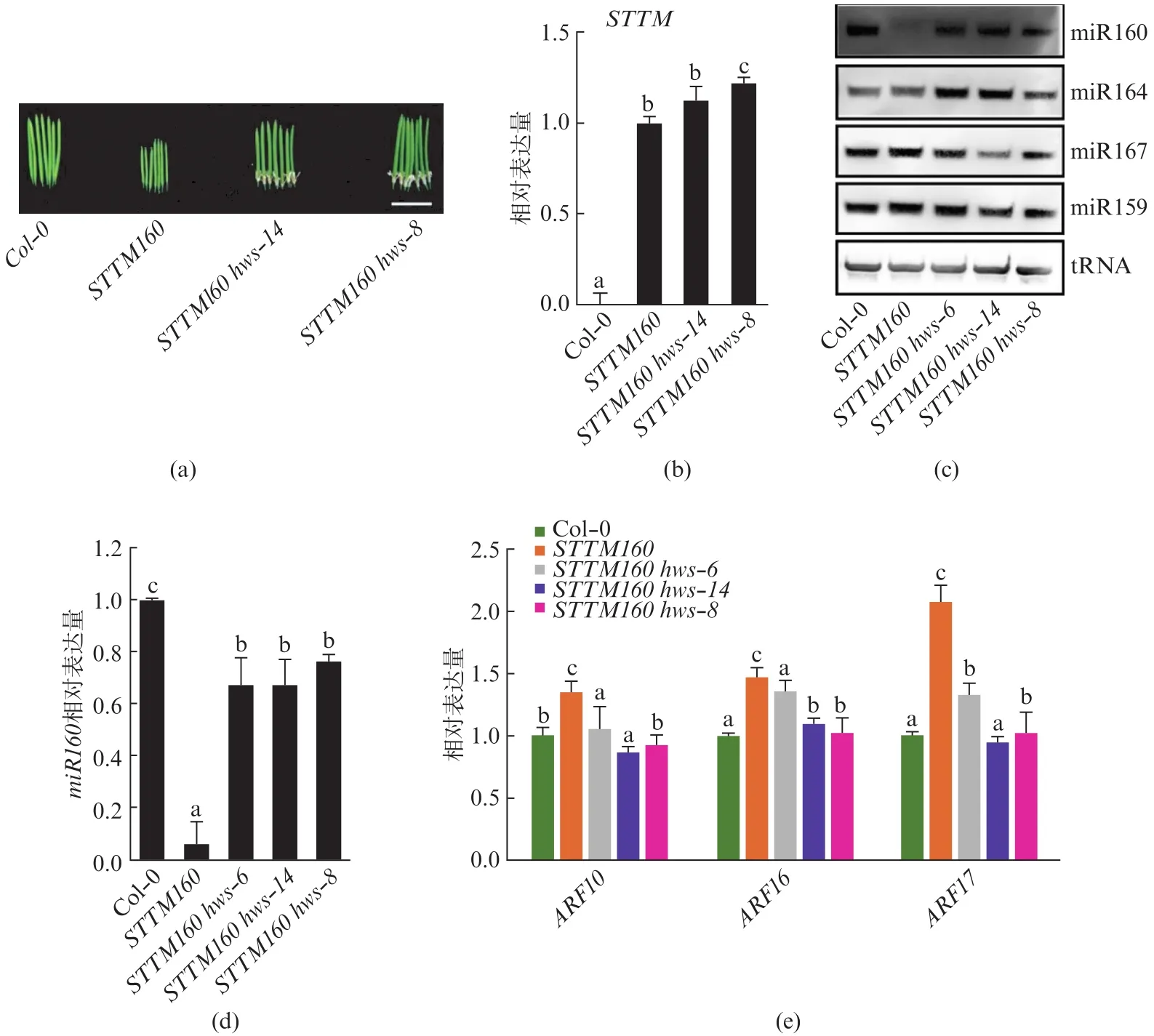
图2 HWS突变恢复miR160及其靶基因表达水平Fig.2 The HWS mutations restored the expression of miR160 and its target genes
2.3 HWS进化保守性分析
为考察HWS功能在不同物种中是否具有保守性,我们获取了不同植物中HWS的潜在同源序列。进化树分析表明HWS存在于蕨类植物江南卷柏,裸子植物火炬松,以及被子植物中,每个物种中含1~3个拷贝(图3见第380页)。我们发现在双子叶植物中,基因的分化晚于物种分化,提示功能上可能是冗余的;而在单子叶植物中,基因的分化早于物种的分化,表明功能上可能存在分化(图3)。小立碗藓、绿色鞭毛藻、及杜氏盐藻中与HWS相似性最高的序列并不和HWS处于同一进化分枝,提示HWS可能起源于蕨类植物(图3(a))。紧接着,我们考察了上述遗传学鉴定到的关键氨基酸位点是否保守,除E347(hws-13)在江南卷柏中对应位点为甘氨酸(Glycine,G)外,其他氨基酸位点在不同物种中均高度保守,提示HWS在不同的植物中行使相同或相近的功能(图3(b))。这些保守的氨基酸位点也为后续深入研究HWS不同结构域或基序的功能奠定了基础。
2.4 HWS组织表达模式与亚细胞定位
我们收集了拟南芥野生型Col-0不同部位的组织,运用qPCR检测HWS基因表达。结果表明,HWS基因在不同组织中均有表达,但表达水平存在较大差异,以花器官中的表达量为最高(图4(a)),该实验结果与文献[27]报道的组织表达模式及拟南芥公共RNA-seq文库数据[29]一致(图4(b))。
已有的研究表明HWS可能识别并通过泛素 蛋白酶体途径降解结合有模拟靶标的沉默复合体[16],因此我们考察了HWS与AGO1是否存在共定位。我们构建了YFP-HWS及RFP-AGO1表达载体。利用烟草瞬时转化系统,发现HWS在细胞核与细胞质中均有表达,且HWS与AGO1在细胞质中存在点状共定位(图4(c),白色三角)。

图4 HWS的组织表达模式及亚细胞定位Fig.4 Expression patterns of HWS in different tissues and subcellular location of HWS
3 讨 论
此前,两个独立的实验室分别通过筛选MIM156和STTM160的表型抑制子鉴定到了HWS[16-17]。在本研究中,对表型恢复程度好的STTM160抑制子的鉴定发现均为HWS的等位突变,表明HWS是模拟靶标介导的miRNA降解代谢通路的核心成员。
对HWS的进化分析表明HWS直系同源基因可能起源于蕨类植物。关于HWS直系同源基因的功能在其他植物中也有若干报道。番茄Micro-Tom中HWS突变会引起植株变高,叶片卷曲融合,叶片锯齿缺刻减少,单性结实,果实可溶性固形物的含量(Brix指数)增加等一系列表型[30]。此外,miR156、miR159、miR164等多个miRNA在Slhws-1突变体中均有积累,表明番茄Sl HWS负调控部分miRNA表达[30],但尚不清楚这种调控是发生在转录还是转录后水平。杨树PtaHWS在根中高表达,在低氮条件下,它能促进PtaRAP2.11与PtaNAC1转录,通过直接或间接影响miR164e前体分子的表达,从而调节侧根增殖过程[31]。水稻含有两个HWS同源基因,分别为OsLP/OsEP3(LARGERPANICLE/ERECT PANICLE3)和OsFBK1(F-boxKelch1)。其中OsLP/OsEP3调控水稻穗型和保卫细胞发育,进而影响叶片光合作用,并且可以互补拟南芥hws-1突变体的萼片融合表型[32-33]。而OsFBK1经生长素诱导表达,通过泛素/26S蛋白酶体途径降解肉桂酰辅酶A还原酶(Cinnamoyl-Co A Reductase,CCR)进而调控花药和根部的次生细胞壁加厚[34]。上述研究表明HWS在植物发育或胁迫应答中起重要调控作用,且HWS可能存在依赖和不依赖于模拟靶标介导的miRNA降解两种途径调控不同生物学过程。
HWS蛋白F-box区的保守氨基酸突变P56L可以恢复STTM160的表型(图1,图3(b)),而前期研究表明HWS可以与E3泛素连接酶组分ASK家族多个成员互作[27,35],且过表达野生型而非F-box删除的HWS可以导致严重的发育缺陷[16-17]。此外,HWS突变导致miRNA和模拟靶标RNA在沉默复合体中滞留[16]。综上,HWS很可能通过泛素/26S蛋白酶体途径选择性靶向降解结合有模拟靶标的沉默复合体,进而引起对应miRNA降解。与上述模型一致,烟草共定位实验表明YFP-HWS和RFP-AGO1存在共定位(图4)。尽管HWS与动物ZSWIM8均参与模拟靶标介导的miRNA降解[16,21-22],但两者并非同源基因,表明动植物中存在类似但不完全相同的生化途径。
在STTM160hws突变体中,STTM160依然表达(图2(b)),但植株发育缺陷以及ARF10/16/17的表达均得到了显著恢复(图1(a),图2(e)),提示在HWS突变情况下STTM160不能很好地作为分子海绵竞争miR160对ARF10/16/17的靶向抑制,但具体机制仍有待挖掘。我们在前期已发表的论文[16]中提出了miR160-AGO1、miR160-AGO1-STTM160、以及miR160-AGO1-ARF10/16/17之间存在动态平衡,当HWS突变时,miR160-AGO1-STTM160因不能正常降解而逐渐向miR160-AGO1及miR160-AGO1-ARF10/16/17转变,从而实现对ARF10/16/17的再抑制。此外,考虑到miR160与STTM160之间存在凸出泡而与ARF10/16/17不存在,推测miR160与ARF10/16/17的亲和力会强于STTM160。
IPS1是首个在植物中被证实有功能的保守内源性模拟靶标(endogenous Target Mimicry,e TM)[12]。研究人员开发了不同软件在多个物种中预测了大量潜在的eTM[36-39],但只有少数进行了严格的生物学功能验证[12,39-40]。借助hws突变体材料系统鉴定内源模拟靶标并研究其在发育和环境响应中的工作机制,将有助于揭示miRNA选择性降解的生理意义。
