基于DNA自组装级联反应的纳米开关的构建
2022-08-30吴沛柯温燕勤张金脉李晓柒师咏勇
吴沛柯,温燕勤,周 娟,张金脉,李晓柒,张 含,潘 敦,师咏勇,2
(1.上海交通大学Bio-X研究院,教育部发育和神经精神疾病遗传学重点实验室,上海 200030;2.上海交通大学医学院附属上海精神卫生中心 上海市精神疾病重点实验室,上海 200030)
DNA自组装技术(DNA纳米技术)是近几年的研究热点,其中的自组装指的是基本结构单元(如DNA)利用非共价键的作用而自发结合成有序结构的过程[1]。DNA分子具有尺寸可控制,组装操作简易,成本低廉的独特优势,这为纳米技术的进一步发展提供了可能。生物学家Holliday在研究酵母细胞染色体同源重组过程中时,意外观察到两条同源染色体的双链DNA间形成了十字交叉结构,经研究发现这是因为双链DNA发生了部分链交换。而后“结构DNA纳米技术”的开创者,美国科学家Seeman教授利用几何学和计算学再次研究了Holliday结构模型,发现Holliday结构具有新的空间取向,并认为以其作为基本单元可以拼接成二维甚至三维的有序复杂网状结构[2]。之后Seeman教授对这种结构的再发现和利用标志着DNA自组装技术的产生。经历了无数次尝试后,Seeman教授最终在探索实验中构建了一系列DNA纳米结构,如经典的四臂结结构[2]。有了Seeman教授开创性的研究成果[3],后续生物学家相继设计构建了由两股双螺旋并排而成,内部具有交叉(crossover)结构且更加稳定坚固的DX结构,以及利用两种tile(四臂结结构单元)组装出的条纹DNA平面网络等,这为DNA纳米材料的深入研究提供了多样的模板[4-5]。
DNA自组装实际上是热力学驱动下的DNA分子互补链之间自发互补配对的动力学过程,依靠的是非共价键(如氢键、静电作用等)。然而,如果想要使用tile构建复杂结构,大量的边界条件和多种单元组件会使得DNA序列设计难度的上升和保真度的下降。2006年Rothemund开发了一种从未报道过的DNA自组装技术——DNA折纸术[6],该技术的诞生标志着DNA纳米技术新时代的来临。DNA折纸术的基本原理是通过向一条长的DNA脚手架链上加入一系列经过特殊设计的短的DNA片段(订书钉链),依据碱基互补原则,可以构建出各种各样的纳米二维三维结构。相较于传统tile拼接自组装技术,DNA折纸术反应条件更加简易,退火时间通常少于2 h,同时具有高精准的可寻址性,该技术也可以组装更为复杂的结构,折纸产物很容易实现MDa级别分子量。2007年,Douglas实验室[7]开创了DNA三维纳米DNA结构组装的先河,首次成功运用DNA折纸术制备出了410 nm的六螺旋纳米管,并通过对该tile长度和浓度的调整,构建出了丰富的三维结构。之后还有更为复杂的六面体、纳米笼等结构的出现。2009年,Douglas等[8]首次获得蜂窝形状的三维结构单元,再通过分级组装成更大尺寸,更为复杂的三维结构。同年Dietz等[9]的弧线立体DNA纳米结构取得了成功。Han等[10]于2011年阐述了DNA折纸术用于构造任意三维纳米结构的可能性,由于该三维结构的腔体内部可用于放置特定的药物分子,这也为疾病的靶向治疗提供了可能。
DNA折纸术为分子结构的设计提供了前所未有的复杂性和纳米级分辨率。DNA固有的纳米尺度特性和分子识别特性使其有助于构建各种复杂精细的纳米结构,这也让DNA在生物学[11]、医学[12]、材料科学[13]和信息处理和存储[14-15]等领域有着广泛的应用。有学者通过构建5位纳米开关操作系统,实现了数据的写入、擦除和重写的功能,并能实现“或”“与”逻辑门。由于DNA纳米结构的独特优良特性,目前已经有大量研究通过DNA分子生化处理后完成相应的计算操作,进而模拟数学运算过程对问题通过DNA反应进行求解。然而对于DNA纳米开关的研究以及在DNA纳米开关上进行相应操作的研究并不充分。如何实现DNA纳米开关特定数据的输入以及数据的连续输入成为了研究热点。本研究通过在骨架链中依次加入不同的数据链(订书钉链),借助可以形成不同数目的环状结构来映射反应的步骤,再通过凝胶电泳筛选出级联反应的具体步骤,进而可以证明此开关的级联放大作用,具体过程如图1所示。本研究所构建的纳米开关级联反应结构将有望在数据存储释放方面有所应用。
1 材料和方法
1.1 材料
噬菌体M13mp18单链DNA和BtsCⅠ反应酶均购于New England Biolabs(NEB)公司,PEG购于上海Bioscience公司,寡核苷酸Oligos若干(骨架链,填充链,数据链,引发链,寻址链)以及BtsCI限制性片段互补寡核苷酸均由杰李公司合成(PAGE法纯化),PBS购于GE生命科学HyClone公司,磁珠购于Invitrogen公司,MgCl2、琼脂糖均购于Fisher Bio Reagents公司,TBE购于上海Bioscienc公司,gel red购于Biotium公司,MarkerⅣ和D15000 DNA Marker均购于上海天根生化科技有限公司。
1.2 方法
1.2.1 设计寡核苷酸序列
寡核苷酸的设计参考了文献[16]的序列并采用NUPACK软件进行结构模拟和稳定性验证。DNA纳米开关的制备也参考了文献[16]的报道,这种纳米开关具有能够同时输入多个数据链的特点,我们对此纳米开关进行了改进以实现纳米开关的级联反应。在本研究中,所采用的DNA纳米开关是一段长的DNA二聚物,它包含了一条长的单链DNA支架(采用了M13病毒)和若干短的互补寡核苷酸链,而短的寡核苷酸将影响整个DNA纳米结构的形成以及纳米开关的功能。本研究中短的寡核苷酸链中包含3条均匀分布的邮局链(post strand),可通过寻址链(address strand)替换,通过选择不同的寻址链与数据链结合进而形成数量不同的环状结构。寻址链核苷酸包含可以绑定到特定数据链(data strand)的单链延伸序列。同时本研究还创造性地设计了能够与寻址链特异结合并含有toehold序列的引发链(trigger strand),通过数据链的加入可以释放引发链进而引发级联反应,具体反应过程如图1(c)所示。当加入数据链1后,数据链1与A0,A1互补配对,中间的骨架链而会形成环状结构,同时会释放引发链1,引发链1与A2,A3互补配对,A2,A3中间的骨架链形成环状结构,同时释放引发链2,最终纳米开关会形成两个环状结构。若只加入数据链2,数据链2可与A2,A3互补配对,A0,A1不成环,A2,A3成环,最终形成一个环状结构,数据链2作用与引发链1一样,即使得A2和A3形成环状结构。具体的寡核苷酸序列见表1(第437~439页)。

表1 本文所用到的寡核苷酸序列Tab.1 Oligonucleotide sequences used in this study

(续表)

(续表)
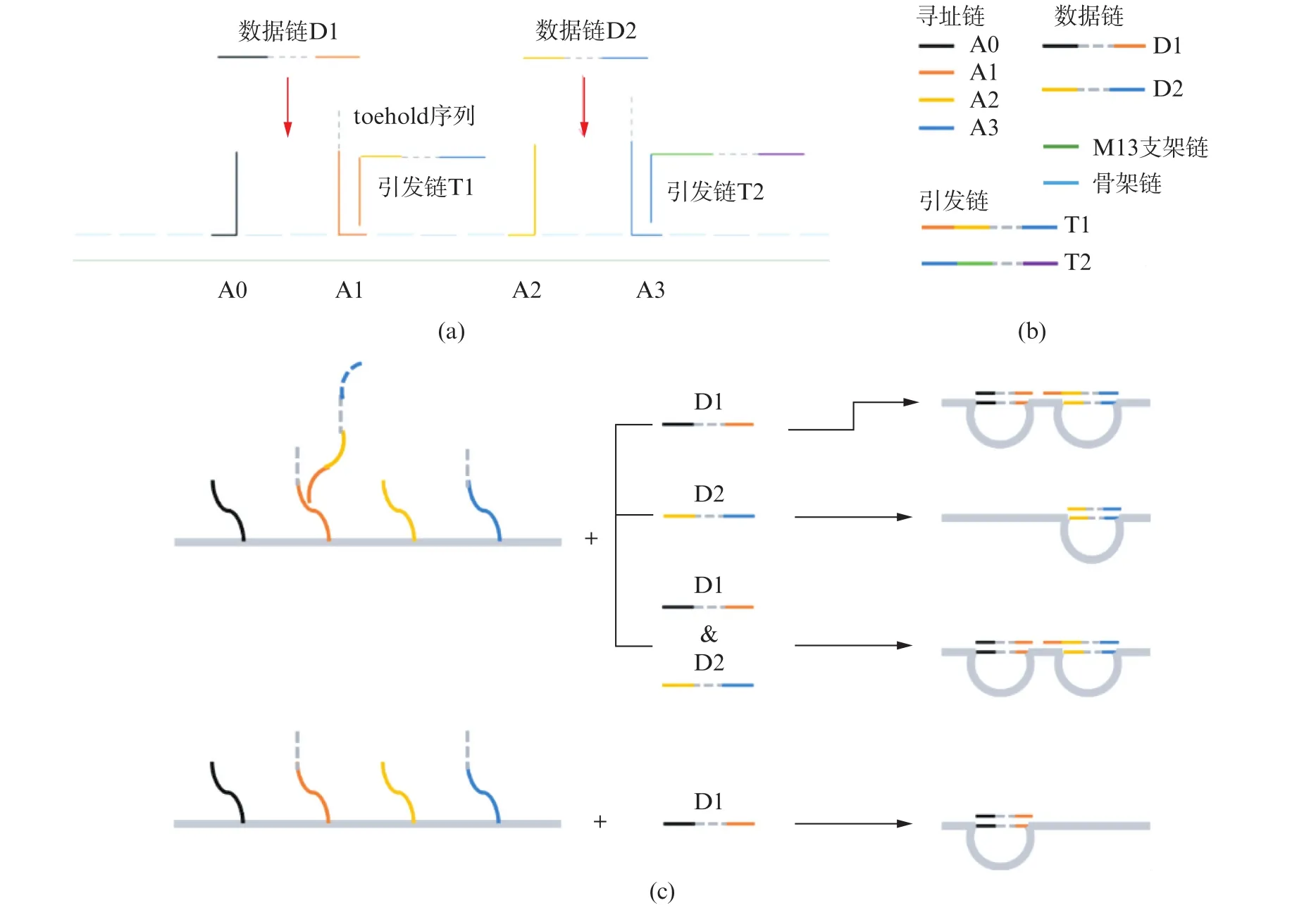
图1 纳米开关的级联反应示意图Fig.1 Sketch map of cascade reaction of nanoswitches
1.2.2 M13mp18噬菌体单链DNA的线性化
在PCR管(或96孔板)中加入5μL 100 nmol/L单链M13,再加入2.5μL 10×Cut Smart buffer,0.5μL 100μmol/LBtsCⅠ反应酶(用于与BtsCⅠ酶配对的寡核苷酸序列为CTACTAATAGTAGTAGCATTAACA TCCAATAAATCATACA)和16μL去离子水,吹吸混匀后短暂离心,PCR仪加热到95℃(30 s),仪器自然降温到50℃温度。退火完成后再加入1μLBtsCⅠ酶50℃孵育15 min,将混合物升温至95℃1 min以灭活,最后快速降温至4℃,通过电泳检测线性化结果。
1.2.3 纳米开关的构建
在线性化后的20 nmol/L的M13链中加入至少10倍的骨架Oligos(提前将引发链与A1链混合)。混合物退火,退火条件为从90℃到20℃并且每分钟下降1℃。退火后的产物用聚乙二醇(Polyethylene Glycol,PEG)沉降纯化或磁珠纯化均可以除去多余的未配对的寡核苷酸链,配对结果通过电泳检测。然后混合物在1×PBS中稀释以达到下一步操作所要求的浓度范围。
1.2.4 纯化纳米开关
为了去除会影响级联反应的寡核苷酸(引发链),本实验需要在纳米开关操作之前进行纳米开关的纯化操作,本实验主要采用PEG沉淀去除过量低聚物[17]和磁珠纯化这两个方法进行纯化。
1.2.5 纳米开关的操作和数据输入
将上述构建好的纯化过后的纳米开关稀释至400 pmol/L以下,并分为4份5μL的初始溶液,并将数据链寡核苷酸的最终浓度控制在2.5 nmol/L。第1组加入一定量的数据链1,第2组加入等量的数据链2,同时第3组在未加入引发链的纳米开关中加入数据链1,第4组同时加入数据链1和2(总量与前三组相同)。所有样品均放置在25℃孵育40 min。
1.2.6 纳米开关操作结果的检测与鉴定
参考文献[16]的方法,样品在0.8%琼脂糖凝胶中电泳,条件为180 V(恒定电压)室温下。用1×gel red(Biotium公司)与凝胶溶液混合,对凝胶进行预染色。最后通过Bio-Rad Gel Doc XR+gel成像仪观察胶图,并对结果进行分析。
2 结果与分析
2.1 M13线性化结果及纳米开关的构建
M13具有EcoRⅠ,SacⅠ和BamHⅠ等许多限制性酶切位点,参考之前文章[16]的报道,实验最终选择了限制性内切酶BtsCⅠ的酶切位点进行切割。由于M13为环状单链DNA,结合的上样缓冲液染料较少且不够牢固,故颜色较浅,当环状DNA被切割成单链DNA后,与染料结合的能力不会有显著提高,但是在琼脂糖凝胶的迁移速率会有明显的降低,此时可以通过电泳图直观地观察。如图2(a)(见第 440页)所示,线性化后的M13迁移速率(泳道2)小于单链环状DNA(泳道3,4)。图2(b)(见第 440页)展示了在线性化后的M13中加入骨架寡核苷酸后成功构建了纳米开关元件,泳道1为纳米开关(M13mp18+Oligos)的条带,泳道2为线性化M13条带,由于纳米开关为双链结构,因而在电泳道上的迁移速率明显低于线性化M13的迁移速率。
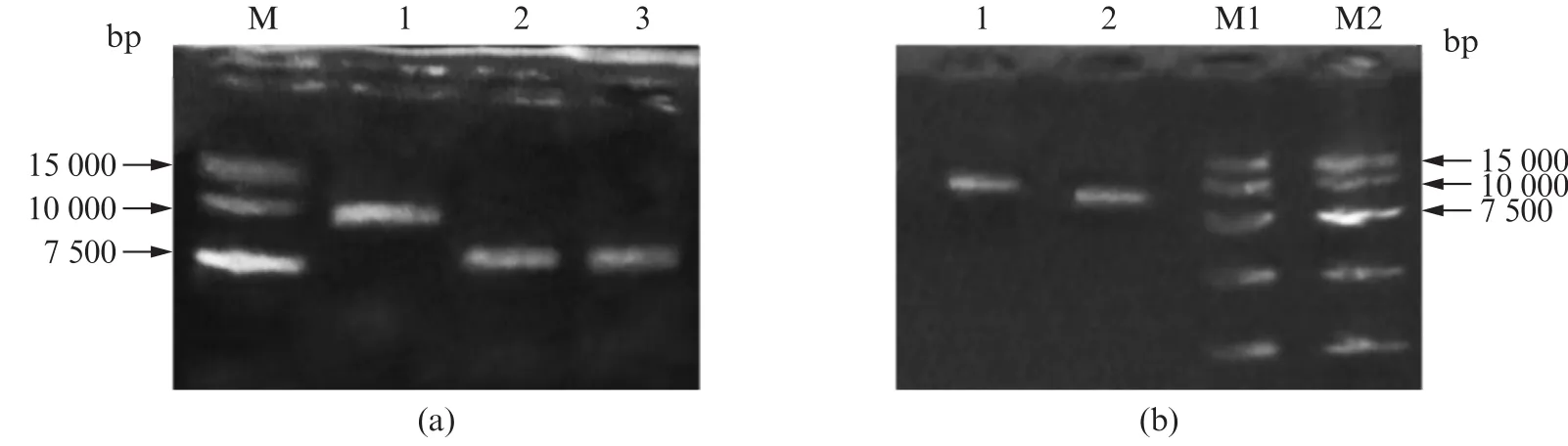
图2 (a)M13的线性化和(b)纳米开关的构建电泳分析Fig.2 Electrophoretic analysis for(a)linearization of M13 and(b)construction of nanoswitches
2.2 纳米开关的运行和数据输入
为了验证纳米开关的运行和数据输入的效果,我们在构建好的纳米开关上进行数据的输入和检测工作,在纳米开关中加入不同数据链(实验组1~4分别加入数据链1,数据链2,不含引发链(trigger-free)的数据链1,数据链1+2),结果如图3(a)所示。在纳米开关中加入数据链1之后会引发该开关的级联反应(泳道1),最终会形成两个环状结构,这与同时加入数据链1和数据链2的作用一致(图3(a)泳道4)。图3(a)中泳道1和泳道4的条带均在7 500~15 000 bp之间,且大体在同一位置,从而验证了该设想。在只有数据链2存在的条件下(图3(a)泳道2),纳米开关只能形成第二个环,而在没有引发链的条件下(图3(a)泳道3)能且只能形成第一个环,因此,实验组2和实验组3都只能形成一个环,两者电泳条带位置相当,并且条带位置低于实验组1和4。线性化的M13,构建好的纳米开关以及输入不同数据链后的纳米开关完整电泳结果如图3(b)所示。图3(b)结果表明线性化的M13迁移速率(泳道5)>纳米开关(泳道4)的迁移速率>加入数据链2的纳米开关的迁移速率(泳道1)>加入数据链1的迁移速率(泳道3)=加入数据链1和2(泳道2)的迁移速率。最终得出结论,单链结构的迁移速率>双链结构的迁移速率>双链上有环状结构的速率,且环状结构越多,迁移速率越慢。
图3 纳米开关的(a)数据输入和(b)运行的电泳分析Fig.3 Electrophoretic analysis for(a)data input and(b)the whole process of nanoswitch
3 讨 论
本次实验的设计主要参考了文献[16]关于DNA纳米结构的可重写记忆系统的实验设计。在参考实验基础上本研究创造性地构建了一种可以引发级联反应的新型纳米开关结构,并且能够通过电泳的方法快速简便地检测出结果。这种级联反应式的纳米开关可以精确地控制纳米结构形成不同数目的环状结构,根据碱基互补原则可以设计不同的开关序列(数据链)控制级联反应发生的位置和形成环状结构的数目。本实验采用的电泳方法与其他的检测方法相比(包括荧光共振能量转移(Fluorescence Resonance Energy Transfer,FRET)、光学输出、电化学检测或原子力显微镜(Atomic Force Microscope,AFM)相比,电泳检测更加简单并且便宜,在实际应用中可操作性更强。由于该电泳方法检测时间短并且方法的操作难度较低,因此该方法适合于使用点检测,特别是可以考虑到使用手持式无缓冲凝胶系统(如Invitrogen e-gel系统)。
在我们所构建的纳米开关结构中,除了用于本实验简单的级联反应之外,由于DNA经过组装后的结构具有独特的纳米尺度,可寻址性和力学刚性,且物理化学稳定性等优势,使得本实验构建的纳米开关结构在数据读取与存储、生物检测以及药物运输等方面都有极大的潜在应用价值。
DNA纳米开关级联反应的基本原理是,特定DNA序列结合DNA纳米开关后,会引起DNA构象变化。级联反应的不同状态可以通过不同数量的环状结构得到体现,并通过电泳图进行展示。通过纳米开关两种不同状态之间的环状数量来报告特定数据链的识别。数据链1和数据链2的加入使得DNA纳米开关在琼脂糖凝胶上的迁移不同,表明它们在两种可能状态下的结构不同。在数据存储方面,大多基于DNA的逻辑系统都是二进制的,因此也需要二进制输入格式,针对DNA纳米开关可通过不同的DNA链分别代表0和1。之前学者通过构造纳米开关,实现了不同数据输入的快速检测[16]。本研究所构造的纳米开关可以规定特定的数据链分别代表0,1,通过对纳米开关的操作实现数据的快速存储和读取。随着以寡核苷酸为基础的技术(如合成生物学中的生物传感和调节系统)的复杂性增加,此类系统的复杂性和多路输出信息变得可以利用。在生物标志物检测方面,本研究也有一定的实际应用价值。例如,生物来源的两个疾病标记物寡核苷酸输入(分别代表数据链1和数据链2),可以通过制造能够引发和不能引发级联反应的纳米开关对标志物1(形成2个环)和标志物2(形成1个环)进行检测。在药物运输方面,一方面由于DNA的无毒性,另一方面目前研究表明由于病毒颗粒和DNA折纸结构具有极为相似的形态,这使得DNA折纸构造的结构更易被细胞主动获取[12]。同时DNA折纸结构相较于简单的线性单、双链DNA具有更强的刚性和稳定性,因而在生物体环境中可持续相对较长的时间不被降解,可以设计结构来保护药物分子,由于DNA折纸结构还具有可寻址性,据此特性可以使药物到达体内的目标靶点后才被降解发挥作用。因而未来可以通过构造设计特定的纳米开关结构实现药物的靶向运输。例如通过在引发链上搭载药物,通过引发链的寻址功能实现药物的靶向运输,实现药物功能。
4 结 论
总之,本次研究已经证明可以使用DNA纳米开关以纳摩尔水平的灵敏度有效地检测不同数据链输入下的纳米开关级联反应,通过不同数据链的输入进行数据的存储和读取。这些纳米开关可以为各种目标预先制作,并且可以作为混合读取检测分析的“智能试剂”。甚至有望可以从随机寡核苷酸池中检测到目标序列(特定的数据链),或从胎牛血清中检测到目标序列,这可以说明其在生物传感应用中的潜在用途。这些DNA纳米开关价格低廉,与传统的复杂检测手段相比,材料成本很低。由于电泳操作技术要求性不高,同时材料只需要普通的凝胶电泳材料,因此这项技术有望为许多研究人员所接受。针对本实验所构建的DNA纳米结构,我们未来可以应用该结构于单倍体的精准分型之中,即先选取某一人类基因组DNA某一性状易感基因的目标片段,在λ核酸外切酶作用下实现单链DNA模版的获取,在根据该片段的SNP设计相应的寻址链,通过将不同SNP整合到纳米开关结构之中,未来有望可以实现人类疾病基因的快速高效检测。此外,我们的纳米开关操作系统以核酸序列的分子识别为基础,通过整合适体或共轭体。记忆操作和简单的决策可以通过生物输入或输出,这个内存类型有望应用于整合利用分子机器人技术,特别是DNA折纸结构,将数据存储或计算与传感或驱动相结合。同时由于DNA特殊的生物学地位在,药物靶向运输和靶向治疗方面也有着潜在的巨大应用价值。已经有许多关于利用DNA折纸术得到的结构作为模板组装功能纳米材料或分子,能够获得光学、电磁学等性能可控的纳米器件[18-20]或药物载体[21-23]。
