hsa-miR-34a在肿瘤中的表达及生物信息学分析
2022-08-26文舒展付子乐陈淑英
文舒展, 付子乐, 陈淑英
(1.复旦大学附属华山医院骨科,上海 200040;2.复旦大学附属中山医院肝外科,上海 200032;2.复旦大学附属华山医院检验医学科,上海 200040)
微小RNA(microRNA,miRNA)是长度约22 nt的非编码RNA,在后生动物和植物基因的转录后调节中起关键作用[1]。miRNA由非外显子区域基因组转录,成熟后可与目标信使RNA(messenger RNA,mRNA)结合,与靶mRNA不完全互补的miRNA在蛋白质翻译水平上可抑制其靶mRNA表达(在哺乳动物中比较普遍),或影响mRNA的稳定性,引起靶mRNA的降解(在植物中比较常见)[2]。在许多重要的生物学过程中都有miRNA的参与,如细胞增殖和凋亡、血细胞发育、炎症细胞激活、神经发生、胎儿发育[2-3]。人微小RNA-34a(homo sapiens microRNA-34a,hsa-miR-34a)被认为是肿瘤抑制的主要调节因子,在许多肿瘤中表达缺失或降低[4],通过miR-34a再表达治疗肿瘤已成为一个重要的研究方向。本研究通过生物信息学方法预测hsa-miR-34a的靶基因,采用功能富集、信号通路富集、蛋白质相互作用分析等方法对其进行分析,结合miR-34a在肿瘤中表达的变化,以期能进一步分析hsa-miR-34a在肿瘤发生、发展中的作用,为后续的临床和基础研究提供参考。
1 材料和方法
1.1 资料收集
在Pubmed(https://pubmed.ncbi.nlm.nih.gov/)、Web of science(https://www.webofscience.com/wos/alldb/basic-search)、OVID(https://ovidsp.dc2.ovid.com/ovid-a/ovidweb.cgi)数据库进行文献搜索,文献检索主题词:“hsa-miR-34a”“microRNA”“cancer”“bioinformatics analysis”“GO”“enrichment analysis”,对检索到的文献进行归纳整理,分析hsa-miR-34a在不同类型肿瘤中的表达变化。
1.2 miRNA同源性分析
采用UCSC Genome Browser(http://genome-asia.ucsc.edu/index.html)对hsa-miR-34a对应基因组在人染色体上的位置进行定位,使用miRBase在线工具(http://www.miRbase.org)检索获得不同物种中miR-34a的染色体定位和碱基序列,通过比对获得同源保守序列。
1.3 靶基因预测
分别通过TargetScan(http://www.targetscan.org/)、miRDB(http://www.miRdb.org/)、miRWalk(http://miRwalk.umm.uni-heidelberg.de/)和mirDIP(http://ophid.utoronto.ca/mirDIP/index.jsp)4个数据库获得hsamiR-34a的靶基因预测结果,取4个预测结果的交集,以mirDIP数据库的数据为参照标准,采用Target Score功能进行排序,筛选前30个基因作为预测靶基因集。
1.4 靶基因表达分析
利用TiGER数据库(http://bioinfo.wilmer.jhu.edu/tiger/)获得靶基因集合在正常人体不同组织和器官中的表达情况。采用Graphpad Prism 8.0软件对数据进行可视化分析,采用单因素方差分析进行统计分析。
1.5 靶基因功能与通路富集分析
利用PANTHER(http://pantherdb.org/)和DAVID(https://david.ncifcrf.gov/home.jsp)在线工具对靶基因集进行基因本体论(Gene Ontology,GO)富集分析和REACTOME通路富集分析。
1.6 蛋白质互作分析
将预测靶基因对应的蛋白质名称录入STRING数据库(https://string-db.org/),得出靶基因对应蛋白质的相互关系网络图。
1.7 统计学方法
采用Graphpad Prism 8.0软件对靶基因表达数据进行可视化分析,采用单因素方差分析进行统计分析。采用Fisher精确概率法统计通路富集分析数据。以P<0.05为差异有统计学意义。
2 结果
2.1 hsa-miR-34a在不同肿瘤中的表达情况
文献检索结果显示,乳腺癌、前列腺癌、膀胱癌等肿瘤中hsa-miR-34a表达显著降低,见表1。
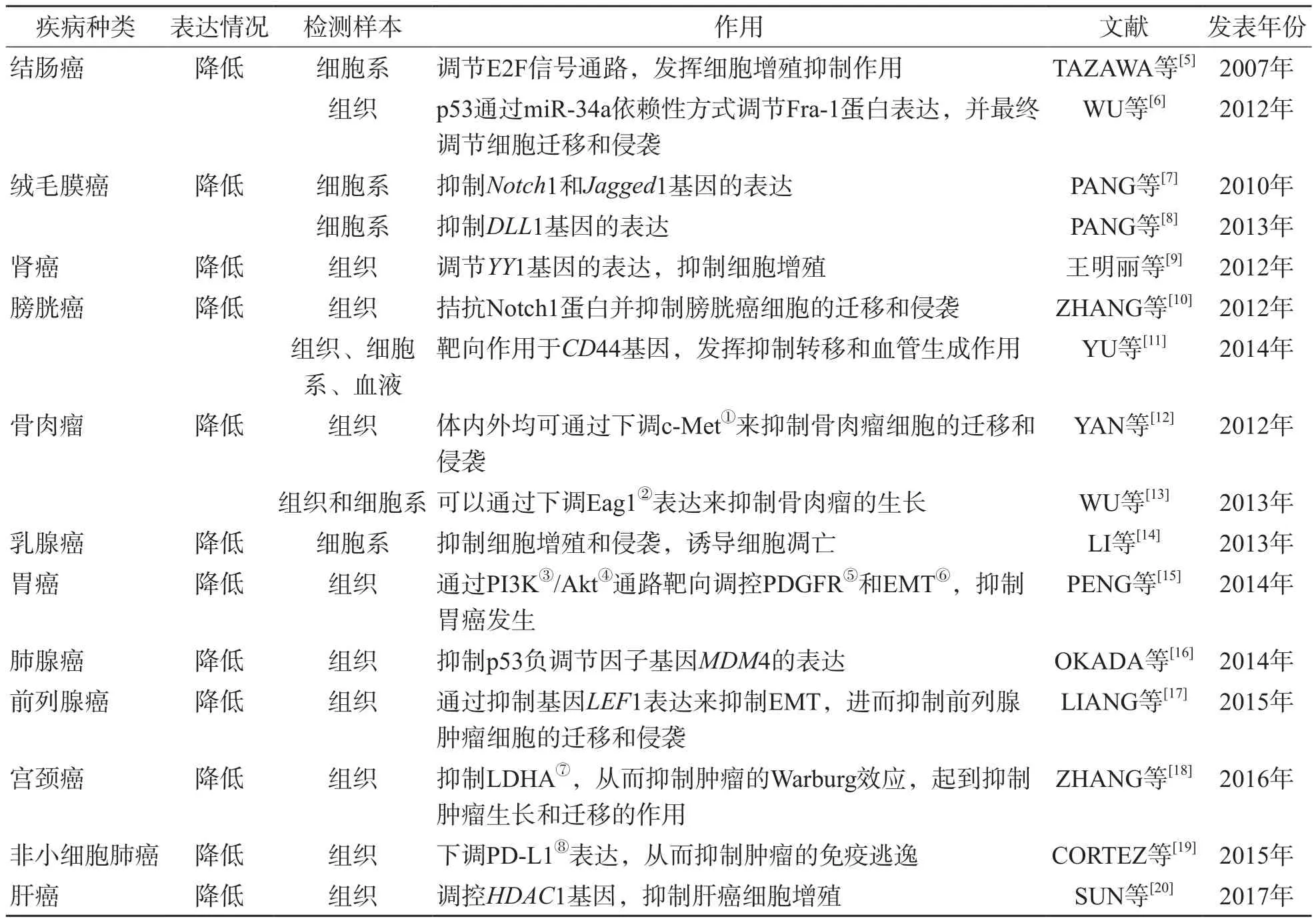
表1 不同肿瘤中hsa-miR-34a的表达情况及作用
2.2 hsa-miR-34a同源性分析结果
通过miRBase、UCSC Genome Browser分析得到hsa-miR-34a对应基因组定位于人染色体1p36.22[9151668-9151777],长度为110 bp,序列号为MI0000268。分析小鼠、猕猴等不同物种的miR-34a,发现“UGGCAGUGU”序列在不同物种间高度保守。见表2。

表2 不同物种中miR-34a的保守序列
2.3 hsa-miR-34a的靶基因预测结果
采用TargetScan、miRDB、miRWalk和mirDIP数据库预测hsa-miR-34a的靶基因,分别得到靶基因数量为754、899、7 345、1 399个,取交集后得到225个靶基因,然后采用Target Score功能排序,选取前30个靶基因作为后续分析的靶基因集合,分别为:MDM4、DLL1、RAP1GDS1、FAM167A、HCN3、SDK2、FAM76A、E2F5、FKBP1B、PPP1R11、SCN2B、MGAT4A、NAV3、SYT1、SATB2、CELF3、XYLT1、MET、MYCN、PACS1、NAV1、CACNA1E、LGR4、TGIF2、VAMP2、FUT9、PKP4、RRAS、ABR、MSR1。
2.4 hsa-miR-34a靶基因的表达
采用TiGER数据平台分析靶基因集在人体各组织器官中的表达,未查询到FAM167A、CELF3、CACNA1E、PPP1R11、MGAT4A、PACS1、TGIF2、FUT9基因相关信息,22个靶基因在人体不同器官组织的特异性表达谱,见图1。单因素方差分析结果显示,不同器官和组织之间各靶基因的相对表达量差异均无统计学意义(P>0.05)。

图1 22个靶基因在正常人体组织中的表达
2.5 hsa-miR-34a的靶基因GO富集分析结果
利用PANTHER在线工具对30个靶基因进行细胞组分和分子功能的GO富集分析,结果显示,靶基因SYT1和VAMP2集中表达在网格蛋白囊泡及囊泡膜(P<0.000 1),转运物质包括谷氨酸、单胺、γ-氨基丁酸;获得HCN3、SCN2B、SYT1、VAMP2基因GO分子功能注释信息,主要与电压门控钠通道活性及与syntaxin-1蛋白有关(P<0.05);成功获得26个基因GO生物学过程注释信息(P<0.05),主要集中于多细胞生物过程和发育、神经系统发生和发育、解剖结构发育等。见表3、表4和图2。
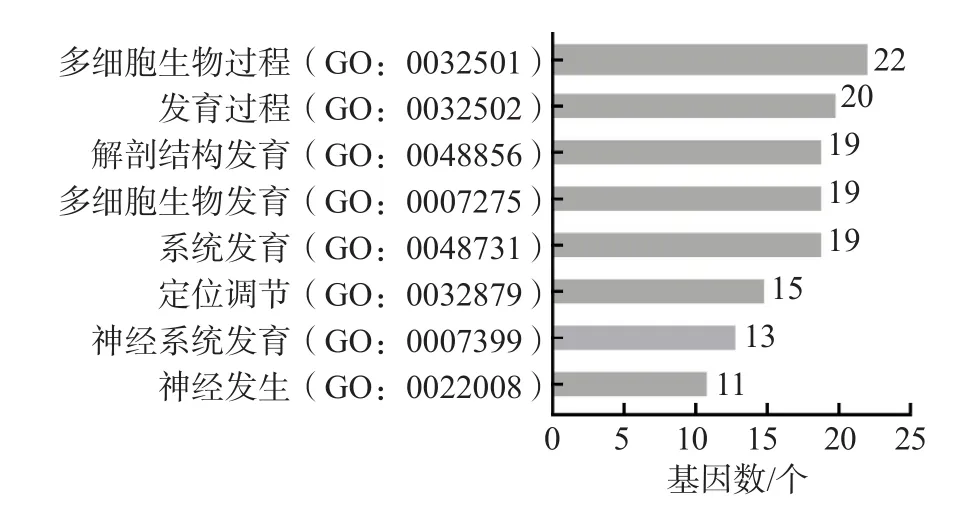
图2 hsa-miR-34a预测靶基因的GO生物学过程分析结果

表3 hsa-miR-34a预测靶基因的GO细胞组分分析结果

表4 hsa-miR-34a预测靶基因的GO分子功能分析结果
2.6 hsa-miR-34a的靶基因通路富集分析
在GO富集分析的基础上,利用已有的生物通路数据,通过DAVID在线工具对靶基因集进行REACTOME生物通路富集分析。结果显示,hsa-miR-34a靶基因集中富集在B型和G型肉毒杆菌毒素毒性(富集倍数>300),同时还富集在人信号素4D(semaphorin 4D,Sema4D)介导的细胞黏附和迁移抑制,γ-氨基丁酸的合成、释放、再吸收和降解,乙酰胆碱等神经递质的释放循环等生物信号通路中。见表5。
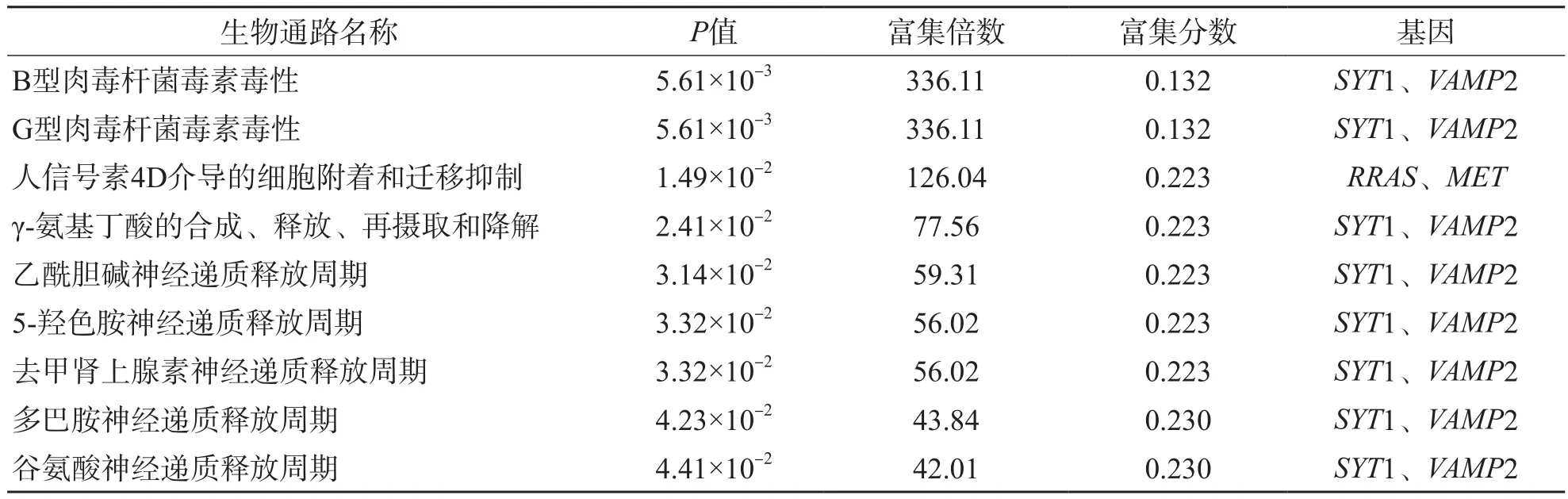
表5 hsa-miR-34a预测靶基因的通路分析结果
2.7 hsa-miR-34a的靶蛋白相互作用网络分析
利用STRING数据库构建hsa-miR-34a的30个靶基因对应蛋白的互作网络,见图3。根据靶基因对应蛋白在该网络中的连接性,对靶基因重新进行排序,排在前5位的靶基因分别为SYT1、VAMP2、MET、RRAS、CACNA1E,这5个基因对应蛋白的关联值均>10。

图3 hsa-miR-34a的30个靶基因对应蛋白的互作网络
3 讨论
miRNA属于高度保守的非编码RNA分子,可特异性识别靶基因3'非翻译区(3' untranslated region,3' UTR)的相应靶位点,抑制特定靶基因的表达或降解靶基因。本研究结果显示,hsamiR-34a在乳腺癌、膀胱癌等多种肿瘤中的表达显著降低。有研究结果显示,hsa-miR-34a可通过抑制700多个与细胞增殖、存活和可塑性有关的转录物来抑制肿瘤的发生、发展[21],但具体作用机制尚不清楚。因此,对hsa-miR-34a进行多层面的生物信息学分析,对后续研究肿瘤的发生、发展及治疗有一定的指导意义。
目前已有一些研究提示了hsa-miR-34a抑癌的机制,在生理条件下,miR-34a在转录中沉默其原癌靶基因的表达,miR-34a的上调会导致细胞凋亡、细胞周期停滞、细胞衰老,进而可以抑制肿瘤细胞繁殖和迁移[4,22],目前证实其涉及的靶基因、靶蛋白或信号通路有MET、CD44、Notch1、LEF1、E2F、PI3K/Akt等。miR-34a的低表达与其受到CpG甲基化修饰有关[23-24],有研究证明,人乳头瘤病毒(human papillomavirus,HPV)通过HPV E6/p53信号通路使miR-34a甲基化,抑制其表达,调节宫颈癌的Warburg效应,促进肿瘤的生长和侵袭[18]。
本研究选取进化程度不同的物种进行同源性分析,miR-34a对应基因序列在不同物种中的相似性较高,其中“UGGCAGUGU”序列为不同物种共同具有的保守区,人与倭黑猩猩、猕猴、婆罗洲猩猩等同源性较高的物种在基因上的位置也高度相似。本研究分析了22个靶基因在人体各器官和组织中的表达情况,发现大部分靶基因在人体各器官组织中均有表达,且在不同器官和组织中的表达量差异均无统计学意义(P>0.05),可见hsa-miR-34a的靶基因大部分具有广谱表达性;miR-34a对应基因的高度保守性和靶基因的广谱表达性提示miR-34对应基因可能有多个识别位点或多个靶标,发挥着重要的生物学功能。
本研究通过4个数据库(TargetScan、miRDB、miRWalk和mirDIP)预测hsa-miR-34a的靶基因,结果不尽相同,且基因数差距较大,取交集后基因数仍有225个。这一方面提示miR-34a生物学功能的广泛性和复杂性,另一方面也说明可能存在较高的假阳性率。因此,本研究采用Target Score功能对获得的基因进行排序,选取前30个靶基因进行分析,以增加预测的精确度,但在精确度增加的同时可能会对结果的全面性产生一定的影响。
在30个靶基因中,MDM4、Notch1、LEF1、Jagged1等基因已被证明在miR-34a抑制肿瘤中发挥重要的作用,进一步验证了miR-34a的抑癌作用。这些靶基因可作为分析miR-34a抑制肿瘤作用机制的重点方向。Target Score排序最高的基因为MDM4,是p53上游重要的调控因子。在生理条件下,miR-34a的转录主要由关键抑癌基因p53调控,通过与miR-34a区域附近多个典型的p53结合位点结合产生影响,但几个反馈回路,如p53/miR-34a/MDM4反馈环提示miR-34a参与了p53的调节[21,25-26]。
相对于对单个靶基因的研究和分析,多个靶基因的整合富集分析有助于更直接和全面地了解miR-34a的生物学功能。本研究通过GO富集分析发现hsa-miR-34a生物学功能广泛,其靶基因集中表达于网格蛋白囊泡和囊泡膜及其物质转运、细胞黏附和迁移、神经递质的合成和释放、多细胞生物过程等,这些都可能对肿瘤发生的各阶段产生影响,如miR-34a的靶基因富集于Sema4D介导的细胞黏附和迁移抑制,这很可能是miR-34a抑制肿瘤细胞黏附和迁移的重要机制,但目前尚缺乏直接的实验证据。值得注意的是,本研究通路分析及组织表达分布的结果均具有较强的指向性,提示miR-34a在神经系统的发育、形成及功能中具有重要的意义,而肿瘤的发生与神经因素也有着密不可分的关系[27],由此推测miR-34a抑制肿瘤发生的机制可能与神经因素调节有关。
综上所述,本研究通过REACTOME通路分析和GO分析,初步探讨了miR-34a的靶基因及其生物学功能,可为后续的临床和基础研究提供参考依据。
