新型冠状病毒Omicron变异株密码子偏爱性及其进化分析
2022-08-11李亚飞罗春雨石哲芳
李亚飞,罗春雨,石哲芳,刘 奇
2019年12月,由新型冠状病毒(Severe Acute Respiratory Syndrome Coronavirus 2,SARS-CoV-2)引起的新型冠状病毒肺炎(Corona Virus Disease 2019,COVID-19)暴发,截至2022年2月,SARS-CoV-2大流行已在全球造成超过4亿人感染和超过600万人死亡,对全球健康和经济发展构成了严重威胁。SARS-CoV-2为单股正链RNA病毒,主要编码核衣壳蛋白(nucleocapsid protein,N蛋白)、刺突糖蛋白(spike glycoprotein,S蛋白)、膜蛋白(membrane protein,M蛋白)和高度疏水的包膜蛋白(envelope protein,E蛋白)4种结构蛋白和ORF1ab、ORF1a、ORF3a、ORF6、ORF7a、ORF7b、ORF8、ORF10共8种非结构蛋白(nonstructural protein,NSP)[1]。
冠状病毒的突变率高,复制容易出错[2]。随着感染人口基数的增多,SARS-CoV-2变体也随之出现。世界卫生组织(World Health Organization,WHO)将能引起大范围流行的变异株归类为关切变异株(Variants of Concern,VOC)[3],目前主要包括Alpha 变体(B.1.1.7 首次发现于英国),Beta变体(B.1.351 首次发现于南非),Gamma变体(P.1 首次发现于巴西)、 Delta变体(B.1.617.2 首次发现于印度)及2021年11月发现的Omicron最新变异体(B.1.1.529/BA.1 首次发现于南非)[4]。Omicron自发现以来传播迅速,目前在非洲已经成为SARS-CoV-2感染的主导病毒株[5]。
不同种生物在蛋白质编码过程中同义密码子的使用频率是非均衡的,存在特定倾向使用的现象,即其具有密码子使用的偏爱性[6],与其基因表达或者其他选择压力如自然选择或者突变压力等有关。研究证明,病毒关键蛋白编码基因中特异性的密码子使用偏倚与其复制能力、适应性免疫逃避能力和致病性等都有着密切关系[7]。目前Omicron的来源和现有疫苗抗性等尚不明确,进行Omicron与其他VOC毒株密码子使用偏爱性分析可为探究其复制能力、适应性免疫逃避能力和致病性等方面的差异研究提供分子机制基础。因此,我们分析了Omicron不同蛋白编码基因密码子使用特性的改变及影响因素,并与标准株Wuhan-Hu-1及其他4种VOC(Alpha、Beta、Gamma、Delta)进行比较分析,研究其密码子偏爱性改变及方向性变化并探索之间的进化关系。
1 材料与方法
1.1 材 料
1.1.1 目的序列来源 收集截至2022年2月底NCBI SARS-CoV-2数据库(https://www.ncbi.nlm.nih.gov/sars-cov-2/)中VOC所有高质量完整序列:Alpha(B.1.1.7)65 143条、Beta(B.1.351)295条、Gamma(P.1)4 010条、 Delta(B.1.617.2)6 018条、Omicron 34 396条(B.1.1.529/BA.1)及SARS-CoV-2标准株Wuhan-Hu-1(NC_45512.2)序列。
1.1.2 计算分析软件 EMBOSS子程序CUSP(https://www.bioinformatics.nl/cgi-bin/emboss/cusp)用于计算目的基因密码子GC3值和各蛋白密码子使用频率(Frequency)等数据;Codon W软件计算分析有效密码子数和同义密码子相对使用频率等;Sigmaplot 14.0软件绘图分析;MEGA 11.0软件构建系统进化树;SPSS 22.0软件进行蛋白聚类分析等。
1.2 方 法
1.2.1 有效密码子数分析 利用Codon W软件计算目的病毒株各蛋白的有效密码子数(Effective Number of Codon,ENC)。ENC取值范围为20~61,用于描述密码子使用偏离随机选择的程度,能反应同义密码子非均衡使用的偏好程度[8]。ENC值越小使用偏性越强(每个氨基酸只使用一个密码子时值为20;各密码子使用概率相等时值为61)。ENC<35说明具有较强烈的密码子使用偏性,ENC>55表明使用偏性弱。
1.2.2 相对同义密码子使用度 相对同义密码子使用度(Relative Synonymous Codon Usage,RSCU)表示某一特定的密码子在编码对应氨基酸的同义密码子间的相对概率[9]。若某一密码子的RSCU=1,表示没有使用偏性;RSCU>1,表示该密码子为使用相对较多的密码子;RSCU>1.5,说明该密码子为使用高频密码子。
1.2.3 ENC-plot分析 ENC-plot是以ENC值为纵坐标,GC3(密码子第3位为G或C的密码子含量)为横坐标绘制图形,用于反应突变压力对密码子使用偏倚的影响程度[10]。标准曲线显示的是密码子使用偏性仅受突变压力影响的情况。当对应点落在标准曲线附近时,提示突变是影响观察到的密码子偏性的主要因素;远离曲线时,提示密码子偏倚还受自然选择等因素的影响。
1.2.4 中性分析 以GC3为横坐标,GC12为纵坐标作中性绘图分析(Neutrality Plot)和回归曲线分析,二者的相关性分析结果可反映突变压力和自然选择对密码子使用模式的相对影响[11]。当回归系数为1或接近1时,则该蛋白编码基因GC3和GC12相关性显著,其密码子3个位置上的碱基组成无明显差异,突变压力为影响密码子偏性的主要因素。若回归系数为0或者远离1,则说明自然选择为影响密码子使用的主要因素。
1.2.5 奇偶偏好分析 奇偶规则(Parity Rule 2,PR2)用于评估自然环境选择或突变压力对基因密码子使用模式的影响。若2个互补链DNA之间不存在任何突变或选择效应上的偏倚,则预测其密码子A=T、C=G[12]。此规则以目的基因的GC偏向值[G3/(G3+C3)]为横坐标,AT偏向值[A3/(A3+T3)]为纵坐标进行PR2-plot绘图。中心坐标点(0.5,0.5)代表A=T、C=G(PR2)的位置,表示此处目的基因内链密码子使用没有偏差,不受突变和翻译选择影响。若点在四个象限分布均匀,则表示密码子使用偏好由突变引起,分布不均则表示还受自然选择等其他因素影响。
1.2.6 基于密码子偏性的聚类分析和发育树分析
利用MEGA 11.0软件对比分析6种SARS-CoV-2病毒株全基因组序列,采用最大似然法(Maximum Likelihood Method,MLM)构建其系统进化树,描述其之间的相关关系和可能的进化过程[13]。采用EMBOSS 子程序CUSP计算不同病毒株各蛋白CDS基因序列的密码子使用频数,再利用SPSS 22.0软件系统进行聚类分析。
2 结 果
2.1 有效密码子数(ENC)分析 Wuhan-Hu-1及5种VOC(Alpha、Beta、Gamma、Delta、Omicron)全基因组结果(图1)显示,各蛋白ENC分布范围为26.38~57.89。Omicron蛋白ENC均值为46.05±7.80,较Wuhan-Hu-1(46.02±8.03)略高,其总体密码子偏性较弱。进一步对12种蛋白编码基因的ENC值分别进行分析发现,Omicron的S(t=6 327.577,P<0.01)、E(t=17 358.633,P<0.01)、ORF1ab(t=3 859.225,P<0.01)、ORF1a(t=37 046.417,P<0.01) 4种蛋白的ENC较Wuhan-Hu-1出现了增高,表明其密码子偏性减弱; M(t=-10 313.811,P<0.01)、N(t=-2 209.230,P<0.01)、ORF3a(t=-4487.525,P<0.01)、ORF7b(t=-24 736.158,P<0.01) 4种蛋白的ENC较Wuhan-Hu-1密码子偏性增强。对比其他4种VOC,Omicron变异株 S、ORF1ab、ORF1a蛋白ENC值最大,M、ORF3a、ORF7b蛋白ENC值最小。
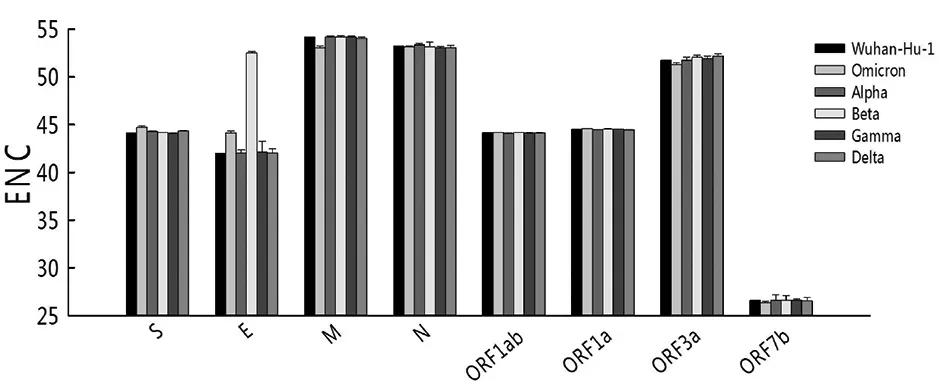
图1 SARS-CoV-2 及其VOC各蛋白ENC分布Fig.1 ENC values of SARS-CoV-2 and VOC
2.2 RSCU分析 为进一步探索Omicron各蛋白基因组密码子使用情况的具体变化情况,参考Gun Li[14]等人计算方法,分析比较了Omicron和Wuhan-Hu-1各蛋白RSCU与人基因组RSCU 差异的变化(结果见表1)。与Wuhan-Hu-1相比,Omicron RSCU更接近人基因组密码子的,以“+”表示;更远离人基因组的以“-”表示。结果显示Omicron病毒株的S(靠近密码子28个;远离18个)、ORF1ab(靠近密码子14个;远离9个)、ORF1a(靠近密码子15个;远离13个)等主要蛋白均有更多数量的密码子与人基因组密码子偏爱性趋于一致,尤其以S蛋白最为明显。S和ORF1ab编码基因覆盖了整个基因组的近4/5,说明Omicron密码子使用模式相较Wuhan-Hu-1更靠近人类基因组。
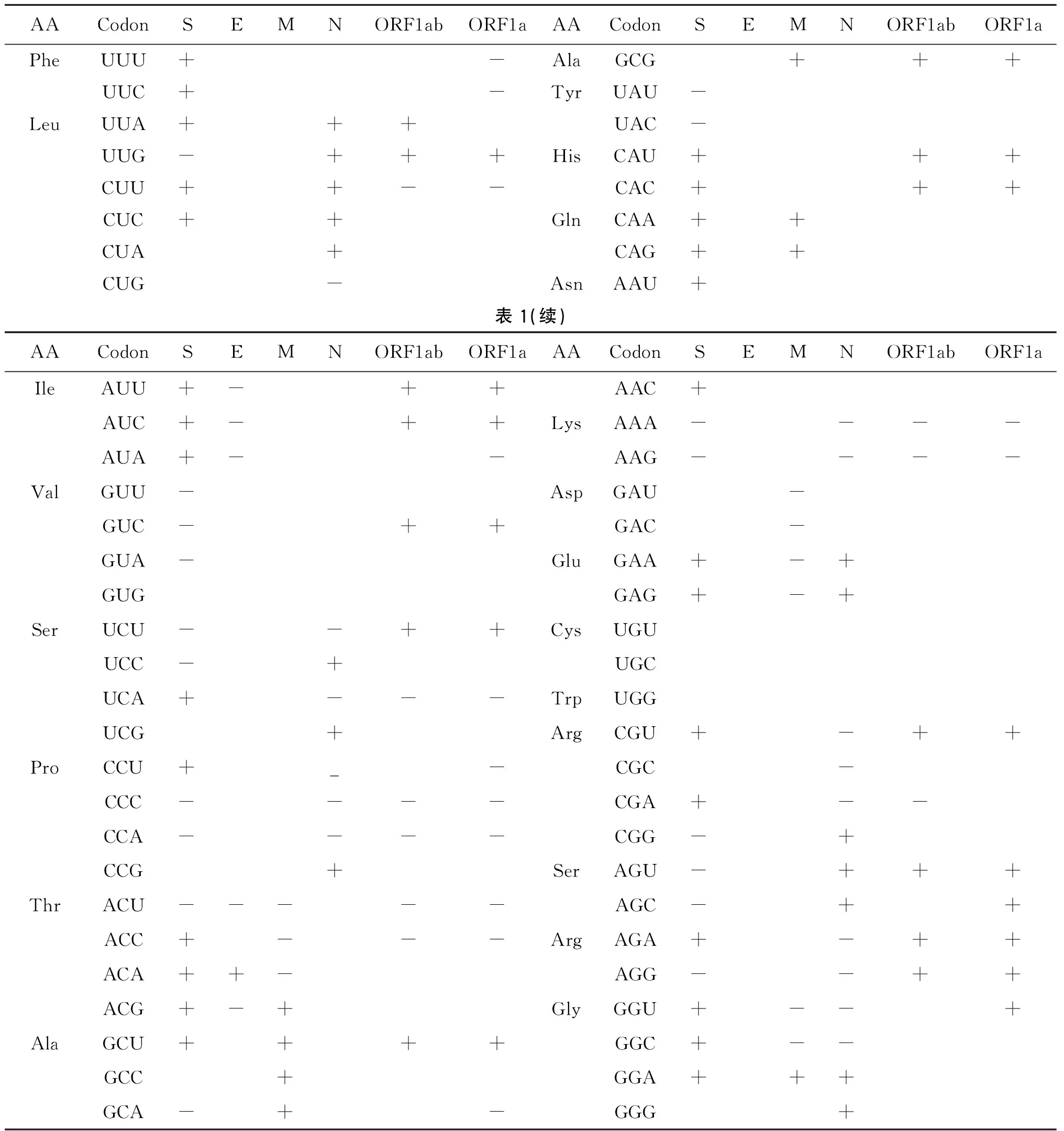
表1 Omicron突变株RSCU与人基因组RSCU的关系Tab.1 Relationship of RSCU between Omicron variant and human genome
2.3 ENC-plot分析 为了探究Omicron与Wuhan-Hu-1及其他VOC病毒株密码子使用偏性改变的影响因素,对其12种蛋白编码基因CDS进行ENC-plot绘图分析。总体结果(图2A)显示Omicron总体相较其他VOC和Wuhan-Hu-1更接近标准曲线,说明Omicron的总体密码子使用偏倚改变受突变压力的影响更多。各蛋白分析结果可以看出,Omicron的 S蛋白和E蛋白相较于其他VOC更接近于标准曲线,表明这两种蛋白较其他VOC受突变压力的影响更大。而M蛋白和ORF3a相较其他VOC距离标准曲线更远,表明相较其他VOC,这些蛋白受突变压力的影响小。
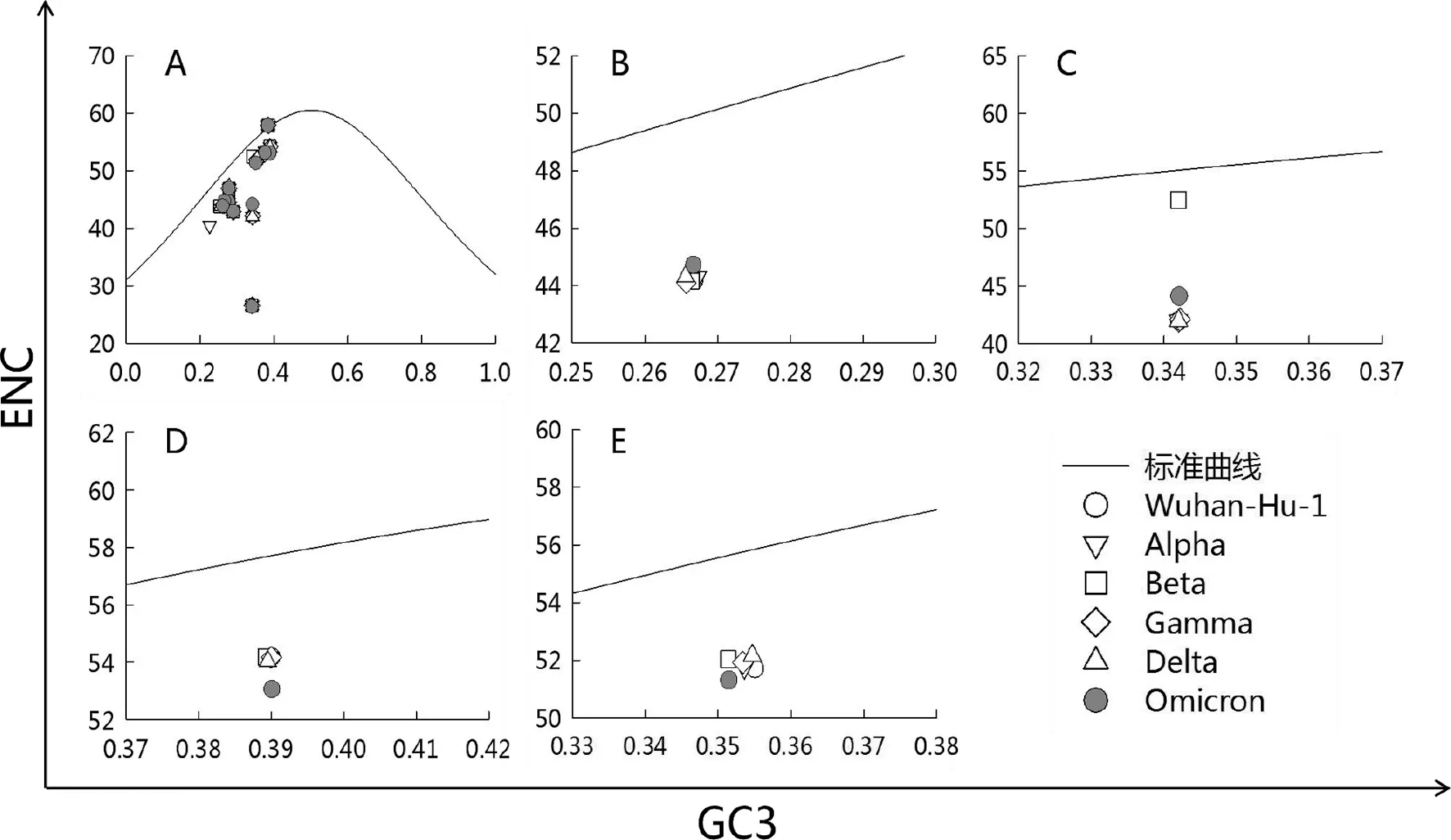
注:A为SARS-CoV-2 Wuhan-Hu-1及其5种VOC 12种蛋白CDS总图;B、C、D、E分别S、E、M、ORF3a蛋白ENC-plot图。图2 Omicron 和其他VOC病毒株ENC-Plot分析Fig.2 ENC-Plot analysis of Omicron variant and other VOC
2.4 中性绘图分析 利用中性分析探究5种VOC之间密码子使用偏倚差异的影响因素,各蛋白中性绘图分析结果(图3)显示S、E、ORF1ab、ORF1a、ORF3a、ORF6、ORF7b等大多数蛋白斜率b均远离1,回归模型均具有统计学意义。GC3和GC12之间的弱相关性表明,这几种蛋白之间密码子使用偏倚之间的差异则更多受自然选择压力的影响,突变压力起次要作用。该结果提示,尽管冠状病毒基因具有高突变性,但在SARS-CoV-2不断变异进化过程中,各蛋白经受的选择压力是不一致的,自然选择作用导致了不同VOC之间的S、E、ORF1ab等蛋白密码子使用偏爱性差异的变化,这可能与世界范围的疫苗接种有关。
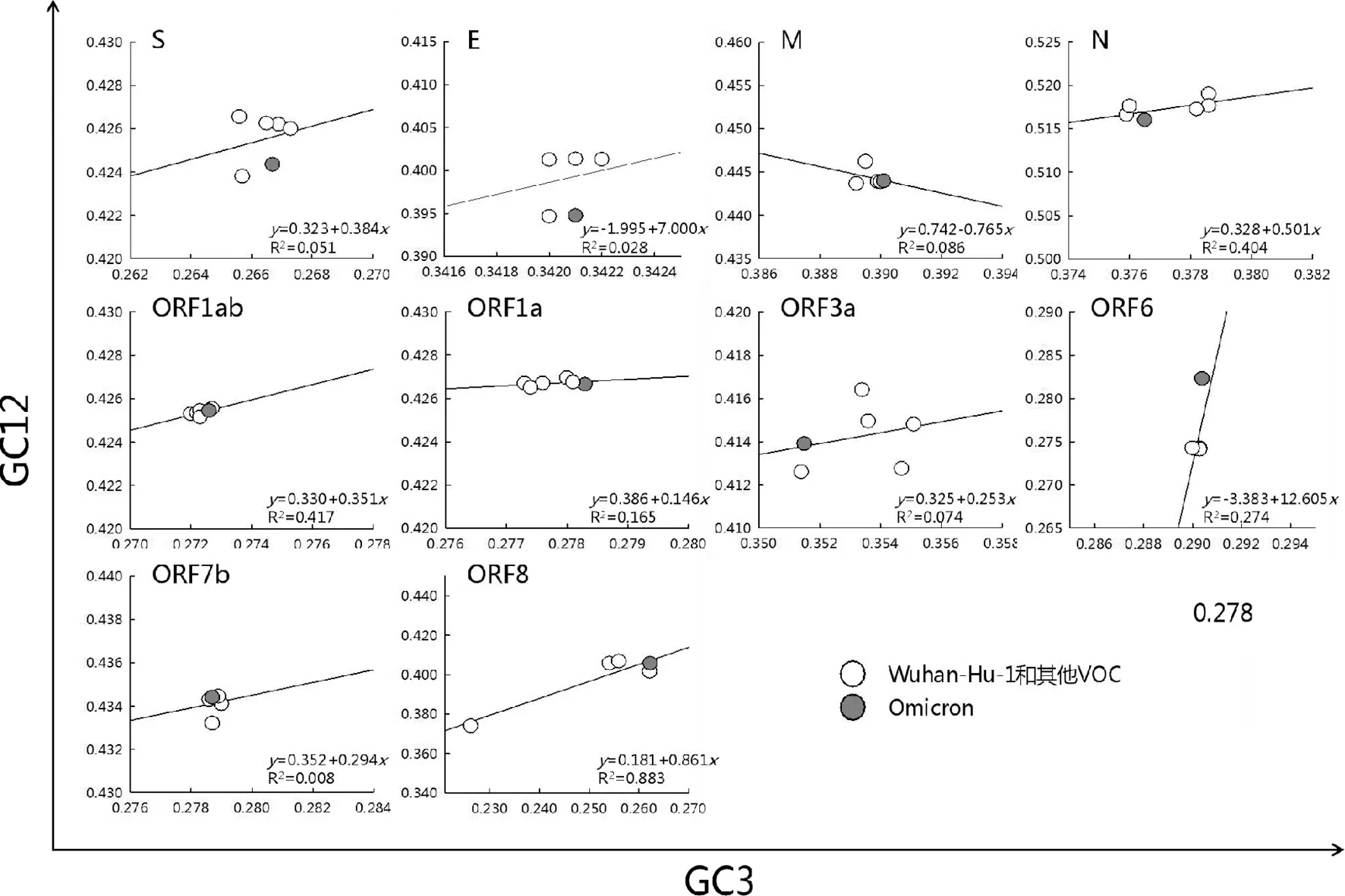
图3 Omicron 和其他VOC病毒株部分蛋白中性分析Fig.3 Neutral plot analysis of partial protein of Omicron and other VOC
2.5 奇偶偏好分析 PR2分析结果显示(图4),Omicron与SARS-CoV-2 Wuhan-Hu-1及其他VOC各CDS主要集中在左下象限,密码子使用不平等。多数点位于y<0.5以下,提示各VOC基因组第3位密码子C/T的使用频率仍旧较高,与前期对SARS-CoV-2的研究[16]一致。该结果还进一步证明了除突变压力外,自然选择等其他因素也在影响VOC密码子使用偏好性。
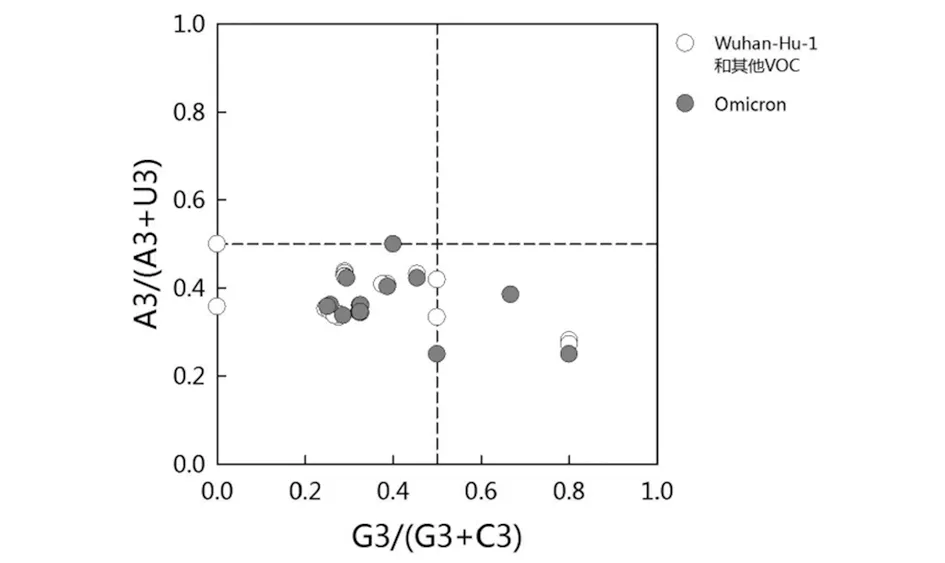
图4 SARS-CoV-2及VOC病毒株的所有CDS的PR2分析Fig.4 PR2 analysis of all CDS of SARS-CoV-2 and VOC
2.6 系统发育分析和聚类分析 Mahmoud Kandeel等[13]采用邻位归并法(Neighbor-joining Method,NJ)方法分析出Omicron与Alpha关系密切,同为一个分支。我们利用MEGA 11.0分析软件,基于最大似然法(Maximum Likelihood Method,MLM)分析系统发育,结果显示Omimon已形成了一个新的分支群(图5A)。而基于密码子偏爱性的聚类分析结果显示,Omicron的S、ORF1a蛋白(图5B、C)与其他VOC差异最大,单独聚为一类;E、N、ORF3a等(图5D、E、F)大多数蛋白与其他VOC关系密切。
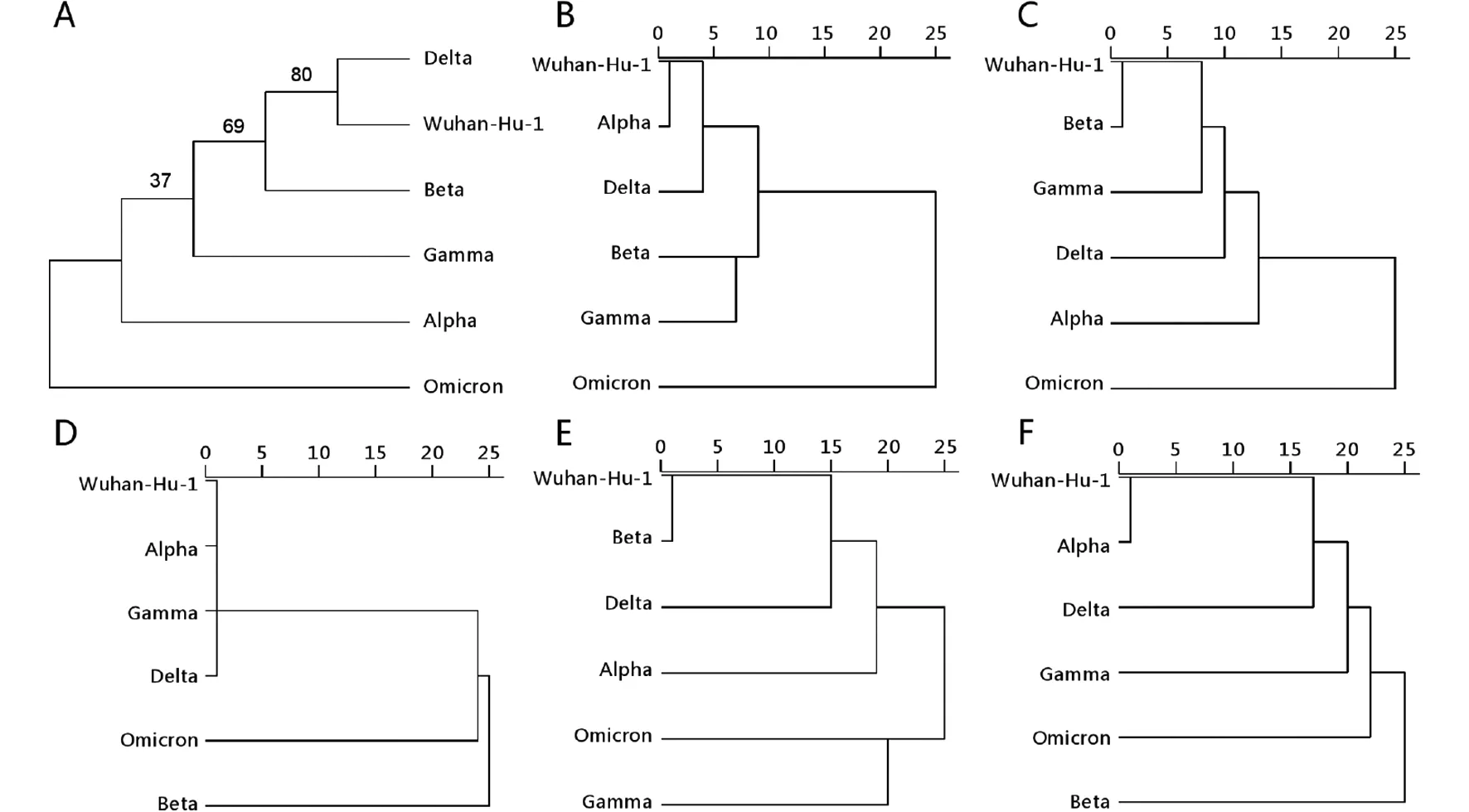
注:A为VOC及Wuhan-Hu-1 系统发育分析;B、C、D、E、F分别为基于Wuhan-Hu-1及VOC病毒株的S、ORF1a、E、N、ORF3a蛋白编码序列密码子偏爱性的聚类分析。图5 SARS-CoV-2 VOC病毒株的系统发育分析和部分蛋白聚类分析Fig.5 Phylogenetic analysisand of partial protein's cluster analysisof SARS-CoV-2 VOC virus strain
3 讨 论
2021年11月9日,南非首次报道了SARS-CoV-2 Omicron的变异株(B.1.1.529/BA.1),11月25日感染Omicron病例数的占比就高达新增病例的75%,其传播速度比Delta毒株更快[17],传染性比原始毒株高500%以上[18]。全球共享流感数据倡议组织(Global influenza data sharing initiative,GISAID)显示Omicron 目前已在163个国家传播,成为全球优势流行毒株。2022年1月8日,中国内陆也出现了Omicron首例病例[18]。前人研究发现,SARS-CoV-2的突变主要发生在N、S以及ORF1a、ORF3a和ORF8等蛋白中[19]。Omicron 50多处突变位点同样集中在S、ORF1ab蛋白[18],其中S蛋白高达 32 处及以上,是 Delta 的两倍[20]。Omicron氨基酸突变位点的增多,被认为是其传播性增加的主要原因[21]。本研究则主要以Omicron各蛋白的密码子偏爱性改变作为研究对象,探究其与流行病学之间的联系,为Omicron提供基因水平上的相关研究基础。
研究发现,Omicron 关键蛋白如S、E、ORF1ab蛋白等密码子使用偏爱性较其他VOC进一步减弱,这种使用偏性的减弱能使其在不竞争有限的tRNA资源的情况下保持有效复制,从而更易于在宿主细胞中表达[22]。这可能与Omicron具有更强的传播速度快[5]有关。N、M、ORF3a等蛋白使用偏性增强则可能会降低该蛋白在宿主体内的表达量,降低病毒对宿主的损伤,逃避宿主免疫防御,这是与宿主协同进化的结果。该结果从基因水平上解释了Omicron出现传播速率快,症状较轻,多数为轻型患者或者无症状感染者[5]这一流行病学特点的原因。
此外,为适应宿主,病毒密码子使用偏性往往会朝宿主方向进化[23-24]。RSCU结果显示,Omicron毒株S、ORF1ab等关键蛋白的密码子使用模式较早期的Wuhan-Hu-1更靠近人类基因密码子,两者使用模式更趋于一致。这意味着较Wuhan-Hu-1相比,Omicron在宿主体内基因利用和翻译效率增加,导致S、ORF1ab编码表达的蛋白量的提高。鉴于S蛋白主要负责介导病毒进入,ORF1ab编码产生的一系列非结构蛋白主要参与病毒的复制和释放[24],因此,这种与人类基因组密码子使用模式一致性的提升,可能是Omicron感染性增强的另外一个原因。
我们进一步研究了Omicron 密码子偏爱性的影响因素。其中,中性分析结果提示自然选择是导致SARS-CoV-2密码子使用偏性改变的主要动力,这可能与世界范围的疫苗接种有关。而ENC-plot 分析进一步显示Omicron毒株S、E蛋白受到了比其他VOC更大的突变压力,推测这是Omicron为逃避免疫而在其他VOC毒株突变基础上进一步突变积累的结果。此外,系统发育分析显示,Omicron已形成了一个新的分支群。而基于密码子偏爱性的聚类分析发现,Omicron毒株N、E、ORF3a蛋白分别与其他VOC聚为一类,而S、ORF1a蛋白的密码子偏爱性已经发生较大改变并单独聚为一类。
综上可见,关键蛋白密码子偏爱性的改变可能是SARS-CoV-2突变株,尤其是Omicron感染性增强的重要原因。这提示我们在进行SARS-CoV-2流行监测及研究中,除了关注氨基酸的突变外,也应进一步加强对其密码子偏爱性变化的关注。
利益冲突:无
引用本文格式:李亚飞,罗春雨,石哲芳,等. 新型冠状病毒Omicron变异株密码子偏爱性及其进化分析[J].中国人兽共患病学报,2022,38(7):559-565,576. DOI:10.3969/j.issn.1002-2694.2022.00.085
