耐药基因blaTEM-1、blaIMP-3、fosA3多重重组聚合酶扩增(RPA)快速检测方法的建立和应用
2022-07-23朱凝瑜梁倩蓉郑晓叶丁雪燕曲道峰浙江省水产技术推广总站浙江杭州300浙江工商大学食品与生物工程学院浙江省食品安全重点实验室浙江杭州3008
朱凝瑜,贺 泽,梁倩蓉,郑晓叶,丁雪燕*,曲道峰* (.浙江省水产技术推广总站,浙江 杭州 300;.浙江工商大学 食品与生物工程学院 浙江省食品安全重点实验室,浙江 杭州 3008)
β-内酰胺类抗生素因其抗菌作用范围较广,近年来一直被广泛应用于临床医疗,但也因此使得各类细菌对其抗性大大增加[1]。磷霉素对革兰阴性和阳性需氧菌具有广谱抗菌作用,对于临床重要的耐药菌,包括产超广谱β-内酰胺酶(ESBL)的肠杆菌科细菌、碳青霉烯类耐药的肠杆菌科细菌(carbapenem-resistantEnterobacteriaceae,CRE)等细菌,仍有较高的抗菌活性[2]。而超广谱β-内酰胺抗生素中的碳青霉烯类抗生素更是因其较其他β-内酰胺具有更强的抗菌效果和较低的耐药率[3],曾一度被认为是对抗细菌感染的最后一道防线[4]。但近年来随着大量超广谱β-内酰胺类耐药基因(如blaTEM)[5]和部分碳青霉烯类耐药基因(如blaIMP)[6-7]的发现和检出,使得人们不得不重视抗生素的滥用导致的耐药性增加问题,急需寻求新的有效抗生素。磷霉素的细菌耐药率也有所增长,其主要的耐药基因fosA3检出率在10%以下[8-9]。耐药基因的存在与菌株产生耐药性有关[10-11],在耐药性产生中起到一定的作用,需引起关注。
目前检测耐药基因的方法有传统PCR、荧光PCR、全基因组测序等,重组酶聚合酶扩增(recombinase polymerase amplification,RPA)技术作为一种新兴的恒温核酸扩增技术[12],相对于传统方法来说,具有耗时短,操作简单,无需特定设备等优点。国内尚无利用RPA技术同时检测3种耐药基因的报道。本研究选择3类抗生素的典型耐药基因,即超广谱β-内酰胺类耐药基因blaTEM-1、碳青霉烯类耐药基因blaIMP-3和磷霉素耐药基因fosA3,设计3组独立的RPA引物,建立并优化对应3种耐药基因的多重RPA快速检测方法。同时对从水产养殖环境及水生动物体内分离到细菌进行检测,并与传统PCR检测方法进行比较。本研究旨在解决养殖产业链各环节现场快速检测耐药基因难题,对养殖环境进行耐药风险评估,为保障水产品的食用安全提供技术支撑。
1 材料与方法
1.1 菌种收集以E.coliTOP10(BNCC353819,购自国家兽医微生物菌种保藏中心)为阴性对照;将3株分别携带耐药基因blaTEM-1、blaIMP-3、fosA3 大肠杆菌的耐药基因分别通过细菌接合试验转移到大肠杆菌TOP10中,使其分别携带1个、3个基因(命名为TOP10-blaTEM-1、TOP10-blaIMP-3、TOP10-fosA3和TOP10-all),用作阳性对照。100株从水产养殖环境及300株从省内主养水产品种(大口黑鲈、黄颡鱼、中华鳖、南美白对虾等)体内分离到的细菌菌株(编号为T2011001~T2011400)为本实验室保存。
1.2 主要试剂TwistAmp®DNA Basic Kit 购自英国TwistDx公司;2×Taq PCR Master Mix、dNTPs(10 mmol/L)、 DL2000 DNA Marker均购自生工生物工程(上海)股份有限公司;TIANamp Bacteria DNA Kit细菌基因组DNA提取试剂盒购自天根生化科技(北京)有限公司。
1.3 引物设计根据NCBI上发布的blaTEM-1(NG_050145.1)、blaIMP-3(NG_049194.1)、fosA3(NG_050407.1)基因序列,选择各基因的特异性保守区域,根据RPA技术要求利用Primer 6.0软件设计RPA引物[13-15],同时设计常规PCR引物。引物序列见表1,均由生工生物工程(上海)股份有限公司合成。

表1 引物序列及相关信息
1.4 多重RPA体系的建立根据TwistAmp®DNA Basic Kit试剂盒的操作说明,建立多重 RPA 的反应体系,总反应体系50 μL:Primer Free Rehydration Buffer 29.5 μL,MgAc(280 mmol/L) 2.5 μL,blaTEM-1、blaIMP-3、fosA3 三对基因上、下游引物各1 μL,样品DNA模板5 μL,ddH2O补足体积。37℃恒温反应30 min,将反应产物电泳后通过凝胶成像仪进行检测。
1.5 多重RPA反应条件的优化分别对多重RPA法的反应温度( 37,38,39,40,41,42℃)、反应时间(5,10,15,20,25,30,35,40 min)、引物浓度(200,270,340,410,480 nmol/L)进行优化,确定最佳反应条件。
1.6 多重RPA反应特异性和灵敏度检测
1.6.1多重RPA反应的特异性 提取TOP10-blaTEM-1、TOP10-blaIMP-3、TOP10-fosA3、TOP10-all以及TOP10的细菌DNA作为模板,采用上述最佳反应条件的RPA体系进行检测,评估所建立的检测方法的特异性。
1.6.2多重RPA反应的敏感性 提取TOP10-all菌株DNA作为模板,用ddH2O 对其进行10倍倍比稀释(100~105),用最佳反应条件进行 RPA 扩增,确定耐药基因的检测限。
1.7 多重RPA方法的应用从养殖用水进水口水样中分离得到的100株菌株及水产动物体内分离到的300株菌株,共400株菌株,提取DNA作为模板,用多重RPA方法和普通PCR方法检测上述3种耐药基因,对比检测结果。
2 结果
2.1 多重RPA反应条件的优化
2.1.1多重RPA反应时间的优化 以携带有全部3种耐药基因载体菌(TOP10-all)的DNA为模板,对多重RPA法的反应温度、反应时间、引物浓度进行优化。反应结果如图1~3所示,37~42℃条件下blaTEM-1、blaIMP-3均有清晰且明亮的条带出现,但在37℃条件下fosA3条带较亮;5~40 min反应时间均能检测到3种耐药基因,但20 min时条带最亮且最为清晰;blaTEM-1和blaIMP-3耐药基因引物浓度在270 nmol/L以上时条带较亮并且清晰,fosA3耐药基因在引物浓度为480 nmol/L时条带较亮。因此,确定多重RPA的最佳反应温度为37℃,最佳反应时间为20 min,最佳引物浓度为480 nmol/L。

M.DL2000 DNA Marker;1~8.5,10,15,20,25,30,35,40 min;9.阴性对照图1 耐药基因blaTEM-1、blaIMP-3 和fosA3多重RPA最适反应时间优化结果

M.DL2000 DNA Marker;1~6.37,38,39,40,41,42℃;7.阴性对照图2 耐药基因blaTEM-1、blaIMP-3 和 fosA3多重RPA最适反应温度优化结果
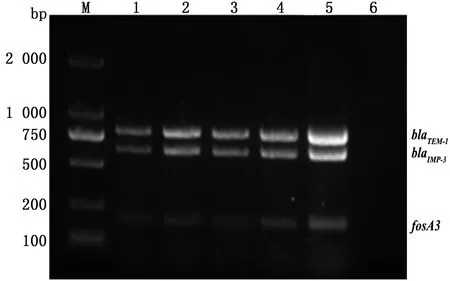
M.DL2000 DNA Marker;1~5.200,270,340,410,480 nmol/L;6.阴性对照图3 耐药基因blaTEM-1、blaIMP-3和fosA3多重RPA最适引物浓度的优化结果
2.2 多重RPA反应的特异性分别对携带1个、3个耐药基因的TOP10-blaTEM-1、TOP10-blaIMP-3、TOP10-fosA3和TOP10-all菌株以及阴性对照TOP10菌株的细菌DNA模板进行多重RPA检测。结果如图4所示,均可扩增出相应的目的条带,而阴性对照则没有目的条带出现,说明建立的多重RPA特异性较强。

M.DL2000 DNA Marker;1.TOP10-fosA3 DNA;2.TOP10-blaIMP-3 DNA;3.TOP10-blaTEM-1 DNA;4.TOP10-all DNA;5.TOP10 阴性对照DNA图4 耐药基因blaTEM-1、blaIMP-3 和fosA3多重RPA的特异性检测结果
2.3 多重RPA反应的敏感性经测定及计算,提取的TOP10-all菌株DNA浓度为1.8×107拷贝/μL,用ddH2O对其进行10倍倍比稀释后进行多重RPA检测。检测结果如图5所示,体系对blaTEM-1、blaIMP-3基因的最低检出限为18拷贝/μL,对fosA3基因的最低检出限为1.8×102拷贝/μL。

M.DL2000 DNA Marker;1~8.100,10-1,10-2,10-3,10-4,10-5,10-6,10-7模板浓度图5 耐药基因blaTEM-1、blaIMP-3 和fosA3多重RPA的敏感性检测结果
2.4 多重RPA快速检测方法应用及效果评价对400株菌株进行多重RPA检测(表2),其中养殖用水进水口水样分离菌株的blaTEM-1的RPA检出率为81.00%(81/100),blaIMP-3和fosA3的检出率分别为11.00%(11/300)和29.00%(29/100);常规PCR对3者的检出率分别为81.00%(81/100),11.00%(11/300)和29.00%(29/100)。水产动物体内分离菌株的blaTEM-1的RPA检出率为73.67%(221/300),blaIMP-3和fosA3的检出率分别为1.33%(4/300)和23.33%(70/300);常规PCR对3者的检出率分别为73.33%(220/300),1.33%(4/300)和23.33%(70/300)。RPA方法的检出率与常规PCR基本一致。可以用于临床样品细菌中耐药基因的检测。根据检测结果,推断是由养殖用水等上游环境的耐药基因污染导致了水产动物体内分离菌株耐药基因的出现。

表2 多重RPA与普通PCR检测符合率 %
3 讨论
RPA技术是一种新兴的恒温核酸扩增技术,其在单链DNA结合蛋白和DNA聚合酶的复合作用下,先将模板DNA解链,进而把引物与匹配模板形成复合体,再延伸生成新的配对链,如此循环往复以指数增长方式达到检测阈值[16]。RPA 技术在近几年已经趋于成熟,很多学者都将其应用在研究中,但仅有少数学者使用RPA技术来检测细菌中的耐药基因,如马骉等[17]使用RPA技术对磺胺类耐药基因进行检测,屠博文等[18]使用RPA技术对金属β内酰胺酶基因blaNDM进行检测等。本研究通过对blaTEM-1、blaIMP-3、fosA3 三种耐药基因序列设计引物,成功建立了一套特异性强,灵敏度高的多重RPA检测方法。相较传统PCR检测而言,既缩短了检测时间(从约3 h缩短至约1 h),只需要在恒温条件(37℃)下操作,设备依赖低,并且能单次检测3种耐药基因,大大地提升了检测效果和检测效率。可为多种类大批量细菌的耐药基因快速检测提供技术支撑,为养殖产业链各环节快速摸清耐药基因情况,减缓耐药基因的传播提供了极大便利。
超广谱β-内酰胺酶中的碳青霉烯类抗生素作为较新型的抗生素在近几年内不断被投入使用,但细菌也因此进化出新的耐药基因,产生了一定的耐药性。根据中国细菌耐药监测网公布的数据显示,2020年临床上大肠埃希菌对头孢西丁和头孢他啶2种β-内酰胺类抗生素的耐药占比分别为6.1%和11.2%,对头孢吡肟的耐药占比已达25.7%,而对其他β-内酰胺类抗生素的耐药占比均已超过50%;对碳青霉烯类抗生素亚胺培南、厄他培南和美罗培南的耐药占比分别为2.0%,2.1%和2.2%;对磷霉素的耐药占比为5.8%,耐药形势不容乐观。
细菌耐药性与其本身特性、耐药基因、药物的使用情况等因素有关[19],其中耐药基因起到了很大作用。耐药基因可通过纵向或横向转移,纵向转移即携带耐药基因的生物通过分裂等方式增殖,其耐药基因随着遗传物质的复制而增加;横向转移是指细胞内部或不同生物个体之间遗传物质的交流,它打破了耐药基因传播时种属的界限,更应引起关注。主要途径有质粒、转座子等可移动基因元件的接合转移,噬菌体介导的转导和转化等[10-11],其中可移动耐药基因元件在耐药性的产生和传播中发挥了重要作用[20]。如本研究中菌株的磷霉素耐药基因fosA3检出率已达到24.75%,大大高于其他文献报道的10%,所检测耐药基因的对应抗生素均在水产养殖中不可使用。并且养殖用水分离菌株中3种耐药基因的检出率均高于水产动物体内分离菌株,存在从养殖用水传播至水产动物的可能。这些结果一定程度上意味着浙江省内水产养殖环境的耐药形势已十分严峻。虽然水产上人畜共患的细菌较为少见,大多数条件致病菌对人致病性较弱,但是通过横向转移,携带的耐药基因伴随着含有耐药菌的废水等其他媒介流动、生物体间相互接触,而转至其他人类易感的细菌体内,加快耐药性的蔓延速度。人接触被耐药菌污染的水或养殖生物、食品等时,一旦被感染,将会给治疗带来困难。因此追踪调查耐药基因谱,对养殖环境进行风险评估,更科学地控制细菌的耐药情况,减少环境污染风险迫在眉睫。
本研究建立了同时检测3种耐药基因的多重RPA快速检测方法,为多种类大批量细菌的耐药基因快速检测提供技术支撑,为快速摸清养殖产业链各环节耐药基因情况、减缓耐药基因的传播提供了极大便利,同时也为进行养殖环境耐药风险评估,保障水产品的食用安全奠定了基础。
