油棕SAUR基因家族的全基因组鉴定及生物信息学分析
2022-07-14周丽霞杨蒙迪曹红星杨耀东
周丽霞 杨蒙迪 曹红星 杨耀东





摘要:【目的】挖掘并鑒定油棕生长素上调小RNA(SAUR)基因家族成员,并对其进行生物信息学分析,为油棕生长素信号转导机制的研究提供参考。【方法】根据拟南芥SAUR基因序列,在油棕全基因组数据库中同源序列搜索,并利用CDD和Pfam数据库进行SAUR蛋白结构域分析,剔除无保守结构域的序列,最后利用生物信息学软件对油棕SAUR基因家族成员进行可视化分析。【结果】从油棕全基因组中共鉴定出40个SAUR基因家族成员(EgSAUR1~EgSAUR40),有31个基因在油棕14条染色体上呈不均匀分布,其中4号染色体上分布的基因最多为7个;除EgSAUR14和EgSAUR24基因含有2个外显子外,其余38个基因均仅含1个外显子,无内含子。40个EgSAURs蛋白亚细胞定位于细胞核,其二级结构均由α-螺旋、β-转角、无规卷曲和延伸链组成,以无规卷曲和α-螺旋所占比例较高,其中有11个EgSAURs蛋白呈酸性,29个EgSAURs蛋白呈碱性。40个油棕SAUR蛋白被划分为四大类,其中第I(除EgSAUR27蛋白外)、II(除EgSAUR3和EgSAUR22外)、III、IV类蛋白均有Auxin_inducible保守结构域。40个EgSAURs蛋白共含10种保守基序(Motif),其中Motif 2存在于所有基因成员中。在花中特异表达的EgSAURs基因(EgSAUR4、EgSAUR9、EgSAUR10、EgSAUR24、EgSAUR29、EgSAUR30、EgSAUR32和EgSAUR33)数量最多,其次为根、茎和叶,在中果皮中特异表达的基因数最少。【结论】基因重复和组织表达特异性是油棕SAUR家族基因的两大特点,推测该家族基因对油棕花的生长发育及授粉受精过程发挥调控作用。
关键字: 油棕;SAUR;全基因组;生长素;生物信息分析;鉴定
中图分类号: S565.9 文献标志码: A 文章编号:2095-1191(2022)04-1011-10
Genome-wide identification and bioinformatics analysis of the oil palm SAUR gene family
ZHOU Li-xia, YANG Meng-di, CAO Hong-xing, YANG Yao-dong*
(Coconut Research Institute, Chinese Academy of Tropical Agricultural Science/Hainan Provincial Key
Laboratory of Tropical Oil Crops Biology, Wenchang, Hainan 571339)
Abstract:【Objective】To identify members of the small auxin-up RNA(SAUR) gene family in oil palm and analyze them by bioinformatics, so as to provide a foundation for the study of auxin the signal transduction mechanism in oil palm. 【Method】Homologs of Arabidopsis thaliana SAUR genes were identified in the whole genome database of oil palm, their protein sequences analyzed by CDD and Pfam database and sequences without conserved SAUR sequence motifs were eliminated. The SAUR gene family of oil palm was then analyzed by bioinformatics. 【Result】A total of 40 oil palm SAUR genes(EgSAUR1-EgSAUR40) were obtained from the whole genome database, and 31 genes were unevenly distributed on the 14 chromosomes of oil palm, with 7 genes on chromosome 4, which contained the most number of genes. The two EgSAURs genes, EgSAUR14 and EgSAUR24, contained two exons, while the other 38 genes contained only one exon without an intron. 40 EgSAURs proteins were all localized in the nucleus. From predictions of the secondary structure of EgSAURs proteins, it was found that 40 EgSAUR proteins had four structural characteristics: α - helix, β-turn, random coil and extended chain. The proportions of random coil and α-helix were higher. 11 EgSAURs proteins were acidic and 29 EgSAURs proteins were alkaline. The 40 EgSAURs proteins were divided into four categories,of which the I (except for EgSAUR27 protein), II (except for EgSAUR3 and EgSAUR22), III and IV proteins all have Auxin_inducible conservative domain. The oil palm SAUR gene family contained 10 conserved Motifs and Motif 2 existed in all gene members. The number of EgSAUR genes specifically expressed in flowers was the most(EgSAUR4, EgSAUR9, EgSAUR10, EgSAUR24, EgSAUR29, EgSAUR30, EgSAUR32 and EgSAUR33), followed by roots, stems and leaves. The number of genes specifically expressed in the mesocarp was the least. 【Conclusion】Gene duplication and tissue expression specificity are the two characteristics of oil palm SAUR gene family members. It is speculated that SAUR gene plays a regulatory role in flower growth and development as well as pollination and fertilization in oil palm.
Key words: oil palm; SAUR; genome-wide; auxin; bioinformatics analysis; identifacation
Foundation items: National Natural Science Foundation of China (31870670)
0 引言
【研究意义】生长素是植物体生长发育所必需的调节因子,其参与植物细胞的分裂和分化、顶端优势、向光性、果实发育及器官衰老等诸多方面,在植物体整个生命过程中均发挥着重要的调节作用(方佳等,2012)。生长素早期应答基因主要包含3个家族,分别为生长素酰胺合成酶基因(GH3)、生长素/吲哚-3-乙酸合成酶基因(Aux/IAA)和生长素上调小RNA基因(SAUR),其中SAUR基因对生长素的响应最快、最强烈(李亚男等,2008)。SAUR基因能在2~5 min内响应生长素,并编码特异性小分子蛋白,用以促进生长素介导的细胞伸长(Knauss et al.,2003)、调节生长素的合成及转运(Chae et al.,2012)及传递乙烯受体信号等(Kong et al.,2013)。由此可见,SAUR基因家族在植物生长发育的诸多方面均发挥着关键作用,但目前只有一小部分功能被鉴定,还有很大挖掘及鉴定空间。油棕(Elaeis guineensis Jacq.)属棕榈科多年生木本乔木,是热带地区重要的油料作物之一,也是目前世界上产油效率最高的作物,平均产油量是花生的7~8倍、大豆的9~10倍,享有“世界油王”之美誉(Basri et al.,2005)。因此,通过生物信息学方法从油棕全基因组中鉴定挖掘SAUR家族成员,并对其理化性质及表达特异性进行分析,以期揭示油棕SAUR基因家族的生物学功能,对探究其在油棕生长发育过程中的调控机理具有重要的意义。【前人研究进展】目前对不同植物SAUR基因家族已有研究报道。赵敬会等(2012)利用生物信息学方法对白菜SAUR基因家族进行鉴定分析,结果鉴定出143个SAUR基因家族成员,其编码的蛋白中,有111个蛋白偏碱性,31个蛋白偏酸性,1个蛋白呈中性;该家族分化为2个亚类,有51个同源对,基因重复是该家族最大的特点。李小玉等(2014)从桑树中克隆得到1个SAUR基因,全长为1186 bp,开放阅读框(ORF)长度为549 bp,编码182个氨基酸残基,含有1个SAUR-specific保守结构域,通过实时荧光定量PCR检测发现其在桑树不定根长出皮层时的表达量显著上升,推测该基因参与了桑树扦插过程中不定根的形成。李锡花(2017)从4个棉花品种的全基因组中鉴定获得145个SAUR基因家族成员,可将其分为10个亚组,该家族均含有生长素诱导的SAUR-specific保守结构域,SAUR基因家族成员在棉花發育早期的纤维及胚珠中呈现多样化的表达模式。李傲等(2018)从葡萄全基因数据中鉴定出64个SAUR基因家族成员,其中分布在3号染色体上的基因数量最多(37个),且该家族基因长度较短,有59个基因只有1个外显子,通过功能预测发现该家族主要具有生长因子、结构蛋白、转录、转录调控及胁迫和免疫应答功能。饶丽莎(2018)利用实时荧光定量PCR检测杉木SAUR基因(ClSAURs)在愈伤组织不同形成时期、不同组织及不同生长素处理时间的表达情况,结果发现ClSAUR2和ClSAUR24基因主要在诱导初期及分化期显著表达,ClSAUR25基因在愈伤组织诱导、分裂及分化整个过程中均显著表达,ClSAUR50、ClSAUR91和ClSAUR39基因主要在诱导初期表达。候春江(2018)研究发现拟南芥SAUR5基因定位在细胞核中,具有转录抑制活性,参与调控拟南芥对脱落酸(ABA)和生长素的响应,其表达受ABA抑制,但受生长素诱导。王红飞和尚庆茂(2019)从黄瓜全基因组中鉴定出73个SAUR基因家族成员,其在干旱、高温及机械损伤等胁迫下呈不同的表达模式,其中CsaSAUR15基因在高温和干旱胁迫下表达上调。郭栋等(2019)从玉米全基因组鉴定出91个SAUR基因家族成员,其中有22个分布在2号染色体上,基因扩增分别为分散复制和片段复制模式,该家族具有1个SAUR-specific保守结构域,其编码的蛋白均含有3个α-螺旋和3个β-折叠结构,3个β-折叠分别位于α1、α2和α3-螺旋之间,通过β-折叠与连接形状的结构,说明玉米SAUR蛋白结构较稳定。刘懿瑶等(2020)克隆获得2个油茶SAUR基因(CoSUAR32和CoSUAR50),二者在自交授粉后子房中36 h 时的表达量显著高于异交子房,在自交授粉后花柱中24 h 时的表达量显著高于异交花柱,推测CoSAUR32和CoSAUR50基因参与油茶自交不亲和过程。王福生等(2020)从柑橘全基因组中鉴定出70个SAUR基因家族成员,大多基因不含内含子,均含有4个保守Motif,顺式作用元件分析结果发现该家族成员上游序列含有光响应、转录因子结合、激素响应及低温响应等元件,且大多SAUR基因均可响应外源吲哚乙酸(IAA)和低温胁迫处理。【本研究切入点】近年来,随着油棕基因组测序和转录测序工作的相继完成,为油棕基因组学的研究提供了更加全面的思路和条件。目前,关于油棕生长素的研究鲜有报道,仅杨蒙迪等(2020)利用生物信息学方法对油棕全基因组Aux/IAA基因家族成员进行鉴定分析,但未见有关油棕SAUR基因家族鉴定分析的研究报道。【拟解决的关键问题】利用生物信息学方法从油棕全基因组中挖掘和鉴定油棕SAUR基因家族成员,并对该家族成员进行基因结构、蛋白理化性质等分析,为油棕生长素相关基因的功能分析及分子调控机理研究提供参考。
1 材料与方法
1. 1 油棕SAUR家族基因序列获取及鉴定
首先从TAIR数据库(http://www.arabidopsis.org)中下载获得拟南芥SAUR基因序列,以拟南芥SAUR基因序列为探针,阈值设定为e-10,在油棕基因组数据库中(GCA_000442705.1)进行BLASTp检索同源序列,从而获取油棕SAUR基因序列,将其在美国麻省理工学院在线基因扫描服务器(The GENSCAN Web Server at MIT)(http://hollywood.mit.edu/GENSCAN.html)搜索以获得SAUR蛋白序列,通过NCBI在线工具CDD(http://www.ncbi.nlm.nih.gov/cdd)和Pfam数据库(http://pfam.xfam.org/)进行蛋白结构域分析,剔除无保守结构域的蛋白序列。
1. 2 染色体定位、基因结构及聚类分析
应用TBtools对油棕和拟南芥SAUR基因成员进行染色体定位、基因结构分析及保守结构域的预测;利用MEME在线软件(https://meme-suite.org/meme/tools/meme)分析油棕SAUR蛋白保守基序(Motif),检索的最大Motif数设置为10,利用MEGA 6.0进行聚类分析。
1. 3 蛋白理化性质分析
利用ProtParam(https://web.expasy.org/protparam/)在线分析SAUR蛋白的理化性质,利用Prot-Comp(http://linux1.softberry.com/berry.phtml?topic=prot-comppl&group=programs&subgroup=proloc)在线预测蛋白的亚细胞定位,利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白二级结构。
1. 4 基因启动子序列分析
提取转录起始位点上游2000 bp序列,将其提交到PlantCARE(http://bioinformatics.psb.ugent.be/we-btools/plantcare/html/)在线服务网站进行序列预测及分析。
1. 5 表达模式分析
从NCBI网站分别下载油棕不同组织的转录组数据,包括授粉后中果皮15周(SRR190698)、17周(SRR190699)、21周(SRR190701)、23周(SRR190702)、果仁(SRR851067)、根(SRR851071)、叶(SRR851096)、茎(SRR851103)和花(SRR851108),从中提取SAUR基因的FPKM值并对其进行log2FPKM转换,利用TBtools绘制基因在不同组织中的表达热图。
2 结果与分析
2. 1 油棕SAUR基因家族成员鉴定结果
以拟南芥SAUR基因序列为探针,在油棕全基因组中搜索SAUR同源序列,对其编码的蛋白保守结构域进行筛选及剔除,共鉴定出40个SAUR基因家族成员(EgSAUR1~EgSAUR40)具体信息如表1所示。40个EgSAURs基因长度为305~8568 bp,且均为正义链。
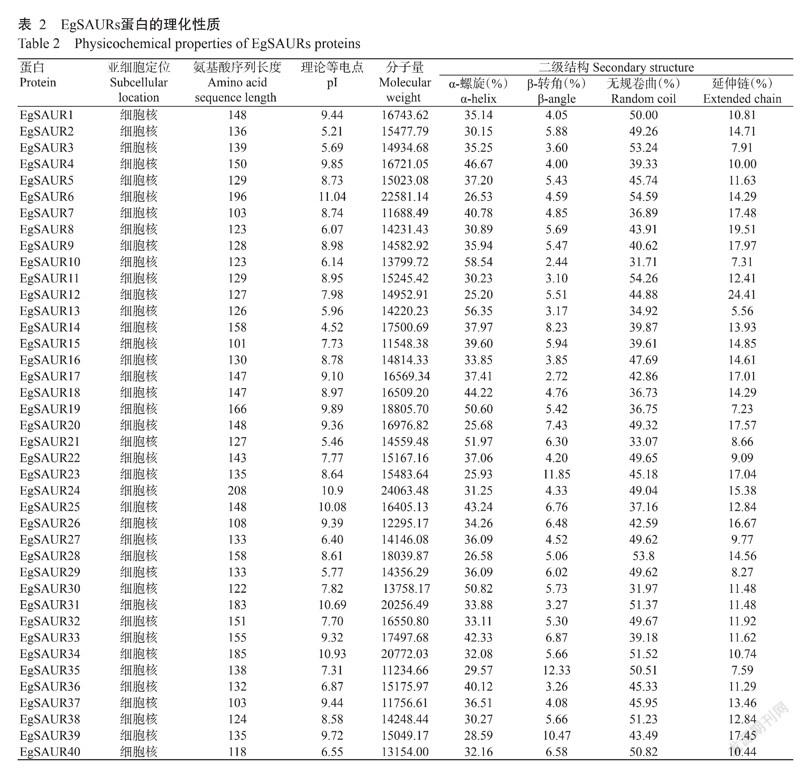
2. 2 油棕SAUR蛋白理化性质分析结果
40个EgSAURs蛋白的理化性质如表2所示。40个蛋白均定位于细胞核中;11个EgSAURs蛋白的理论等电点(pI)<7,呈酸性,29个EgSAURs蛋白的pI>7,呈碱性,其中EgSAUR14蛋白的pI最小,为4.52,EgSAUR6蛋白的pI最大,为11.04。40个EgSAURs蛋白的二级结构均由α-螺旋、β-转角、无规卷曲和延伸链组成,其中无规卷曲和α-螺旋所占比例均较高,β-转角和延伸链所占比例均较少。
2. 3 油棕SAUR基因家族成员染色体定位结果
由图1可知,40个EgSAURs基因中,有31个不均匀分布14条染色体上,其余9个基因未定位在染色体上。其中,4号染色体上分布的基因最多,为7个;6号染色体上有3个基因,3号、5号、7号、10号、11号、12号、15号和16号染色体上均为2个,2号、9号和13号染色体分布最少,均只有1个基因。同一染色体上位于同一个或相邻的基因间区域的基因,如4号染色体上的EgSAUR4和EgSAUR7、EgSAUR5和EgSAUR6、EgSAUR9和EgSAUR10;5号染色体上的EgSAUR11和EgSAUR12;6号染色体上的EgSAUR13、EgSAUR14和EgSAUR15;11号染色体上的EgSAUR23和Eg-SAUR24,可能存在串联重复关系。
2. 4 油棕SAUR家族基因结构分析结果
由表1和图2可知,EgSAURs基因结构较简单,基因长度均较短,EgSAUR14和EgSAUR24含有2个外显子和1个内含子,其余38个基因均仅含有1个外显子,无内含子。
2. 5 系统发育进化树构建及保守结构和保守Motif分析结果
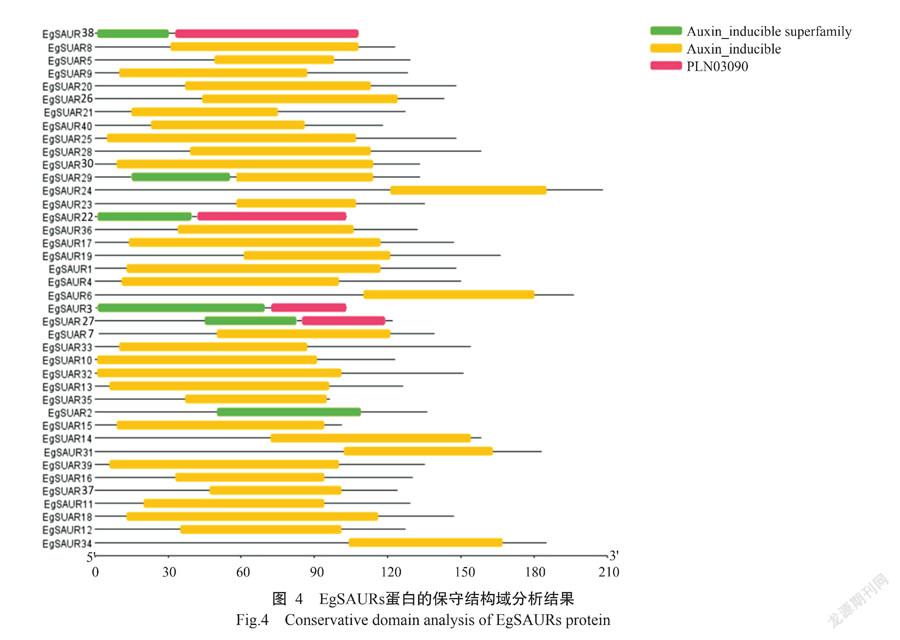
基于油棕和拟南芥SAUR蛋白序列构建系统发育进化树,如图3所示。40个油棕SAUR蛋白被划分为四大类,其中第I类含有15个EgSAURs蛋白,除EgSAUR27蛋白含Auxin_inducible super family和PLN03090结构域之外,其他蛋白均有Auxin_induci-ble保守结构域(图4);第II亚类有5个EgSAURs蛋白,其中EgSAUR3和EgSAUR22含有Auxin_inducible super family和PLN03090结构域,EgSAUR19、Eg-SAUR20和EgSAUR28仅含Auxin_inducible 保守结构域;第III类有11个EgSAURs蛋白,均含Auxin_ inducible 保守结构域,第IV类含有9个基因,其中EgSAUR38含Auxin_inducible super family和PLN-03090,其余均為Auxin_inducible 结构域(图4)。Auxin_inducible super family和Auxin_inducible保守结构域能调控生长素响应基因的表达,PLN03090可通过结合特异性DNA发挥生长调控作用。由于不同亚类的EgSAURs蛋白含有的保守结构域不同,说明这些EgSAURs蛋白具有不同的功能。
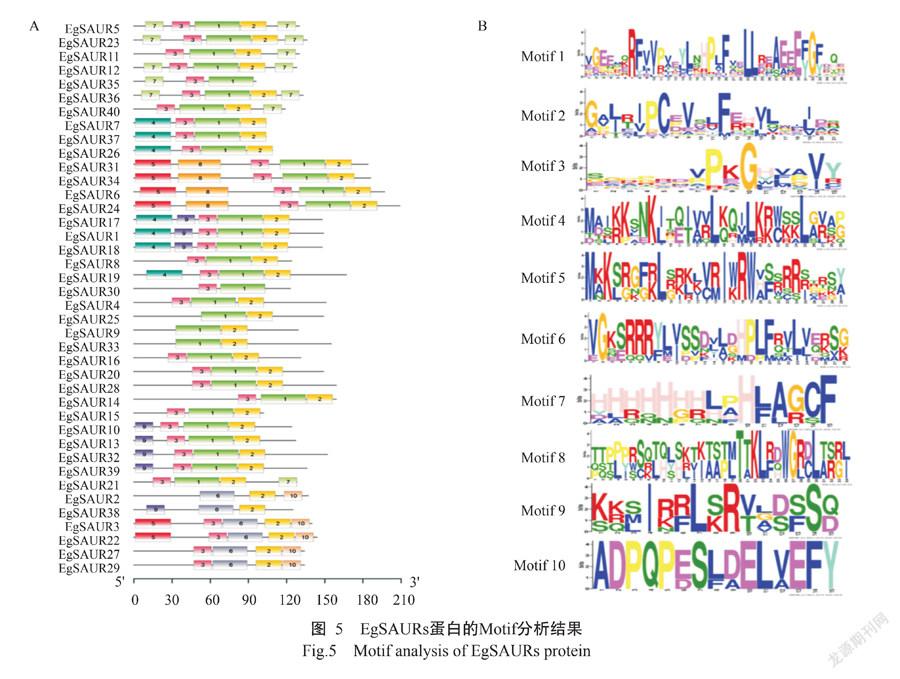
40个EgSAURs蛋白含10种Motif类型(图5-A和图5-B),第I类蛋白除 EgSAUR29和EgSAUR30外,均含有Motif 1和Motif 2。第II类蛋白均有Motif 2 、Motif 6和Motif 10,第III类蛋白均含有Motif 2 和Motif 6,此外EgSAUR38 蛋白有Motif 9,其他4个蛋白均含有Motif 10,此外EgSAUR3和EgSAUR22还含有Motif 5。第IV类蛋白中EgSAUR4、EgSAUR13、EgSAUR14、EgSAUR15、EgSAUR26和EgSAUR39含有Motif 1、Motif 2和Motif 3,此外EgSAUR13和EgSAUR39还含有Motif 9,EgSAUR26含有Motif 4、EgSAUR2含有Motif 2、Motif 6和Motif 10,EgSAUR25含有Motif 1和Motif 2。
2. 6 油棕SAUR基因家族成员启动子的顺式作用元件分析结果
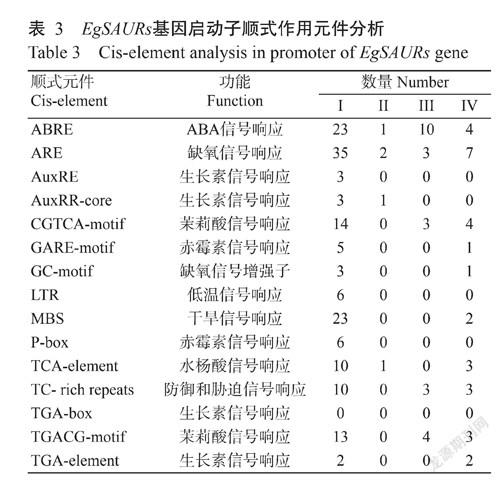
为深度了解EgSAURs基因的表达调控模式,运用PlantCARE预测启动子顺式作用元件,结果鉴定出多种响应环境和激素信号的顺式作用元件,表明EgSAURs基因表达调控机制较复杂,可能受内、外因子的多重调控。由表3可知,与生长素(IAA)相关的作用元件类型最多,其次为赤霉素(GA)和茉莉酸(JA)。除这3类顺式作用元件外,还鉴定到ABA、干旱、低温等信号响应相关的顺式作用元件。同时分析了系统发育进化树中4类蛋白编码基因启动子的顺式作用元件类型,结果发现第I类蛋白的基因启动子区顺式作用元件类型最丰富,包括ABA、缺氧、干旱、激素等响应元件,表明该类群家族成员对抵御非生物胁迫发挥重要作用,其次为第III和IV亚类,包括防御、胁迫响应元件和激素响应元件,推测该类群基因可能对植物生长发育具有重要调控作用。
2. 7 油棕SAUR基因家族成员表达模式分析结果
根据不同组织转录组数据得到的FPKM值,对EgSAURs基因的表达量进行分析,结果如图6所示,EgSAUR7基因在中果皮15周、17周、21周、23周及茎中的表达量较高,在根、叶和花中不表达;EgSAUR9基因在果仁、茎和花中表达较高,在其他几个组织中不表达或表达量极低;EgSAUR24、EgSAUR29和EgSAUR33基因在花中的表达量较高;EgSAUR36基因在根中表达量最大;其他34个基因的表达量较低或不表达。结果表明部分EgSAURs基因具有组织表达特异性。
3 讨论
SAUR作为生长素早期响应基因中最大的一个家族,其在调控植物细胞分裂和分化、器官伸长等方面均具有重要的调控作用,诸多作物的SAUR基因相继被挖掘及报道,如水稻(Kant et al.,2009)、棉花(Abbas et al.,2013)、小叶杨(Chen et al.,2013)和西瓜(Zhang et al.,2017),说明SAUR基因家族成员在植物中普遍存在。随着高通量测序技术的不断发展,诸多作物的全基因组测序数据相继公布,目前已基于测序数据开展SAUR基因家族成员鉴定及挖掘研究,如白菜中有61个(赵敬会等,2012),玉米中有91个(郭栋等,2019),黄瓜中有73个(王红飞和尚庆茂,2019)。本研究基于油棕全基因组数据,共鉴定出40个SAUR基因家族成员,通过染色体定位分析发现这些基因不均匀地分布在14条染色体上,且部分基因处于同一位置或相邻位置,呈簇状分布,推测其与家族的重复基因有关,基因重复也是油棕SAUR基因家族成员的重要特点,在油棕演化过程中发挥非常重要的作用,且该特点在西瓜(Zhang et al.,2017)和玉米(郭栋等,2019)等作物的SAUR基因中也被发现。
本研究通过基因结构分析发现,油棕SAUR基因高度保守,长度均较短,大多数基因只含有1个外显子,不含内含子,与已报道的水稻(Jain et al.,2006)、马铃薯(Ren and Gray,2015)和杨树(Hu et al.,2018)SAUR基因无内含子结果相一致。通过MEME在线分析发现,油棕SAUR基因家族含有10个保守Motif,其中Motif 2存在于所有基因成员中,可作为鉴定SAUR基因家族成员的重要依据。此外,聚类分析处于同一分支上的油棕SAUR基因具有相似数量的保守Motif和排列顺序,不同分枝间的保守Motif数存在一定的差别,推测该现象是由于SAUR基因在进化过程中丢失或新增保守Motif引起的,因此聚类分析中遗传关系较近的基因可能功能也相似。
本研究通过基因表达分析发现,油棕SAUR基因家族成员具有不同的组织表达模式,其中在花中特异表达的EgSAURs基因数量最多,如EgSAUR4、EgSAUR9、EgSAUR10、EgSAUR24、EgSAUR29、Eg-SAUR30、EgSAUR32和EgSAUR33在花中均表达量较高,其次为根、茎和叶,在中果皮中特异表达的基因数最少,与Guo等(2016)在黄瓜中的研究结果较一致。推测这些基因可能参与了油棕花的生长及授粉受精过程,该结果为研究油棕授粉受精相关基因的功能打下基础。
4 结论
基因重复和组织表达特异性是油棕SAUR家族基因的两大特点,推测该家族基因对油棕花的生长发育及授粉受精过程发挥调控作用。
参考文献:
方佳,何勇清,余敏芬,郑炳松. 2012. 植物生长素响应因子基因的研究进展[J]. 浙江农林大学学报,29(4):611-616. [Fang J,He Y Q,Yu M F,Zheng B S. 2012. Recent advan-ces with auxin response factors(ARFs):A review[J]. Journal of Zhejiang A & F University,29(4):611-616.] doi:10.3969/j.issn.2095-0756.2012.04.020.
郭棟,杜媚,周宝元,刘颖慧,赵明. 2019. 玉米SAUR基因家族的鉴定与生物信息学分析[J]. 植物遗传资源学报,20(1):90-99. [Guo D,Du M,Zhou B Y,Liu Y H,Zhao M. 2019. Identification and bioinformatics analysis of maize SAUR gene family[J]. Journal of Plant Genetic Resources,20(1):90-99.] doi:10.134308.cnkijpgr.2018070 7001.
候春江. 2018. SAUR5在脱落酸和生长素信号转导中的作用[D]. 长春:东北师范大学. [Hou C J. 2018. Roles of SAUR5 in ABA and auxin signaling[D]. Changchun:Northeast Normal University.]
李傲,崔梦杰,陈珂,许瀛之,贾海峰,房经贵. 2018. 葡萄SAUR基因家族鉴定与生物信息学分析[J]. 植物遗传资源学报,19(2):326-337. [Li A,Cui M J,Chen K,Xu Y Z,Jia H F,Fang J G. 2018. Identification and bioinformatics analysis of the SAUR gene family in grape[J]. Journal of Plant Genetic Resources,19(2):326-337.] doi:10. 13430/j.cnki.jpgr.2018.02.016.
李锡花. 2017. 棉花SAUR基因家族分析与功能验证[D]. 杨凌:西北农林科技大学. [Li X H. 2017. Identification and functional analysis of the SMALL AUXIN UP RNA (SAUR) gene family in cotton[D]. Yangling:Northwest A & F University.]
李小玉,張晓峰,杜伟,聂浩,唐壮,班月圆,杜小龙,程嘉翎. 2014. 桑树MaSAUR2基因的克隆、序列特征及时间特异性表达[J]. 江苏农业科学,42(12):34-39. [Li X Y,Zhang X F,Du W,Nie H,Tang Z,Ban Y Y,Du X L,Cheng J L. 2014. Cloning,sequence characterization and time-specific expression of MaSAUR2 gene from mulberry[J]. Jiangsu Agricultural Sciences,42(12):34-39.] doi:10.15889/j.issn.1002-1302.2014.12.009.
李亚男,冯霞,陈大清. 2008. ARF、Aux/IAA和生长素受体对基因表达的调控[J]. 安徽农学通报,14(7):36-39. [Li Y N,Feng X,Chen D Q. 2008. Regulation of gene expression by ARF,Aux/IAA and auxin receptors[J]. Anhui Agricultural Science Bulletin,14(7):36-39.] doi:10. 3969/j.issn.1007-7731.2008.07.013.
刘懿瑶,周俊琴,卢梦琪,余姝姝,杨进,谭晓风. 2020. 油茶CoSAUR32 和CoSAUR50基因的克隆及表达分析[J]. 分子植物育种,19(10):3208-3215. [Liu Y Y,Zhou J Q,Lu M Q,Yu S S,Yang J,Tan X F. 2020. Cloning and expression analysis of CoSAUR32 and CoSAUR50 genes in Camellia oleifera[J]. Molecular Plant Breeding,19(10):3208-3215.] doi:10.13271/j.mpb.019.003208.
饶丽莎. 2018. 杉木生长素早期应答基因SAUR的克隆与功能分析[D]. 福州:福建农林大学. [Rao L S. 2018. Clo-ning and function analysis of the auxin early response gene SAUR of Cunninghamia lanceolate[D]. Fuzhou:Fujian Agriculture and Forestry University.]
王福生,余洪,胡洲,管德龙,张盼,朱世平,赵晓春. 2020. 柑橘属SAUR基因家族的全基因组鉴定及表达分析[J]. 园艺学报,47(1):23-40. [Wang F S,Yu H,Hu Z,Guan D L,Zhang P,Zhu S P,Zhao X C. 2020. Genome-wide analysis of SAUR gene family in Citrus[J]. Acta Horticulturae Sinica,47(1):23-40.] doi:10.16420/j.issn.0513-353x. 2019-0581.
王红飞,尚庆茂. 2019. 黄瓜SAUR基因家族的鉴定与表达分析[J]. 园艺学报,46(6):1093-1111. [Wang H F,Shang Q M. 2019. Genome-wide identification and expression analysis of the SAUR gene family in Cucumis sativus[J]. Acta Horticulturae Sinica,46(6):1093-1111.] doi:10. 16420/j.issn.0513-353x.2018-0873.
杨蒙迪,金龙飞,周丽霞,窦雅静,曹红星,李新国. 2020. 油棕Aux/IAA基因家族鉴定与生物信息学分析[J]. 分子植物育种,19(5):1451-1465. [Yang M D,Jin L F,Zhou L X,Dou Y J,Cao H X,Li X G. 2020. Identification and bioinformatics analysis of Aux/IAA family in oil palm (Elaeis guineensis Jacq.)[J]. Molecular Plant Breeding,19(5):1451-1465.] doi:10.13271/j.mpb.019.001451.
赵敬会,王瑞雪,李荣冲,梁晶龙,张涛. 2012. 白菜SAUR基因家族的生物信息学分析[J]. 中国农学通报,28(22):130-137. [Zhao J H,Wang R X,Li R C,Liang J L,Zhang T. 2012. Bioinformatics analysis of SAUR gene family in Brassica rapa[J]. Chinese Agricultural Science Bulletin,28(22):130-137.] doi:10.3969/j.issn.1000-6850.2012.22.023.
Abbas M,Alabadi D,Blazquez M A. 2013. Differential growth at the apical hook:all roads lead to auxin[J]. Frontiers in Plant Science,4:441-449. doi:10.3389/fpls.2013. 00441.
Basri W M,Abdullah S N A,Henson I E. 2005. Oil palm-achievements and potential[J]. Plant Production Science,8(3):288-297. doi:10.3390/agronomy2040240.
Chae K,Isaacs C G,Reeves P H. 2012. Arabidopsis SMALL AUXIN UP RNA63 prometes hypocotyl and stamen filament elongation[J]. The Plant Journal,71(4):684-697. doi:10.1111/j.1365-313X.2012. 05024.x.
Chen J H,Song Y P,Zhang H,Zhang D Q. 2013. Genome-wide analysis of gene expression in response to drought stress in Populus simonii[J]. Plant Molecular Biology Reporter,31(4):946-962. doi:10.1007/s11105-013-0563-6.
Guo W,Tian P,Zhang F X,Qin H,Miao H,Chen Q W,Hu Z Y,Gao L,Wang M J,Gu X F,Huang S W,Chen M S,Wang G. 2016. Integrative analyses of nontargeted volatile profiling and transcriptome data provide molecular insight into VOC diversity in cucumber plants (Cucumis sativus)[J]. Plant Physiology,172(1):603-618. doi:10. 1104/pp.16.01051.
Hu W F,Yan H W,Luo S S,Pan F,Wang Y,Xiang Y. 2018. Genome-wide analysis of poplar SAUR gene family and expression profiles under cold,polyethylene glycol and indole-3-acetic acid treatments[J]. Plant Physiology Biochemistry,128:50-65. doi:10.1016/j.plaphy.2018.04.021.
Jain M,Tyagi A K,Khurana J P. 2006. Genome-wide analysis,evolutionary expansion,and expression of early auxin-responsive SAUR gene family in rice(Oryza sativa)[J]. Genomics,88(3):360-371. doi:10.1007/s11105-013-0563-6.
Kant S,Bi Y M,Zhu T,Rothstein S J. 2009. SAUR39,a small auxin-up RNA gene,acts as a negative regulator of auxin synthesis and transport in rice[J]. Plant Physiology,151 (2):691-701. doi:10.1104/pp.109.143875.
Knauss S,Rohrmeier T,Lehle L. 2003. The auxin-induced maize gene ZmSAUR2 encodes a short-lived nuclear protein expressed in elongating tissues[J]. Molecular Basis of Cell and Developmental Biology,278(26):23936-23943. doi:10.1104/pp.109.143875.
Kong Y Y,Zhu Y B,Gao C, She W J, Lin W Q, Chen Y, Han N, Bian H W, Zhu M Y, Wang J H. 2013. Tissue-specific expression of SMALL AUXIN UP RNA41 differen-tially regulates cell expansion and root meristem patter-ning in Arabidopsis[J]. Plant and Cell Physiology,54:609-621. doi:10.1093/pcp/pct028.
Ren H,Gray W M. 2015. SAUR proteins as effectors of hormonal and environmental signals in plant growth[J]. Molecular Plant,8(8):1153-1164. doi:10.1016/j.molp.2015. 05.003.
Zhang N,Huang X,Bao Y N,Wang B,Zeng H X,Cheng W S,Tang M,Li Y H,Ren J,Sun Y H. 2017. Genome-wide identification of SAUR genes in watermelon(Citrullus lanatus)[J]. Physiology and Molecular Biology of Plants,23 (3):619-628. doi:10.1007/s12298- 017-0442-y.
收稿日期:2021-03-01
基金項目:国家自然科学基金项目(31870670)
通讯作者:杨耀东(1966-),https://orcid.org/0000-0002-6599-5665,博士,研究员,主要从事于农业生物技术,E-mail:yyang@catas.cn
第一作者:周丽霞(1982-),https://orcid.org/0000-0001-5616-9452,主要从事于热带油料作物分子育种,E-mail:glzz_2009@163.com
