基于CRISPR/Cas9技术构建STING基因敲除猪肺泡巨噬细胞系及其对I型干扰素转录的影响
2022-07-14黄秋艳杨烨城张昆丽孟繁明李剑豪朱向星王塑天唐冬生
黄秋艳 杨烨城 张昆丽 孟繁明 李剑豪 朱向星 王塑天 唐冬生


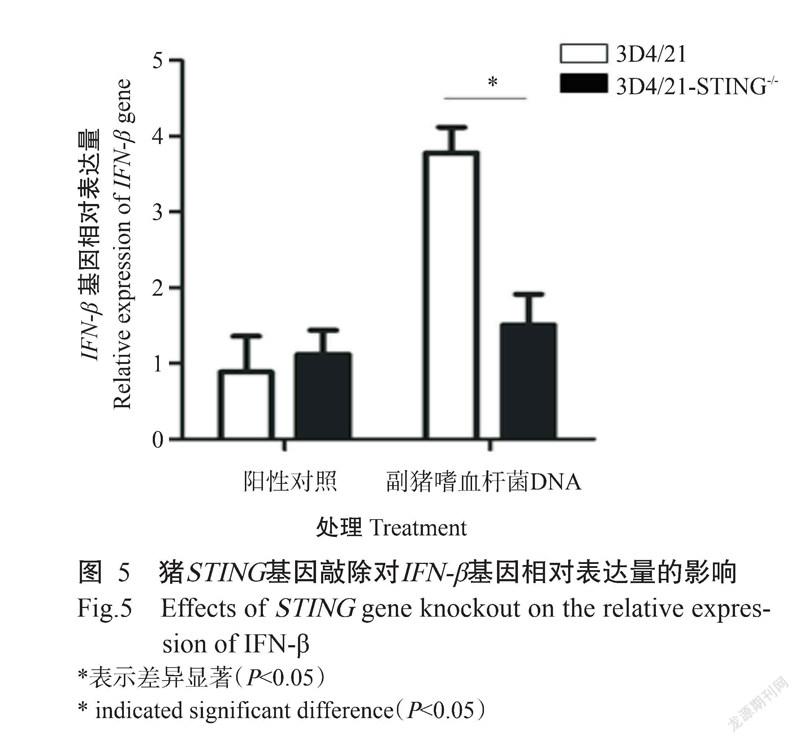
摘要:【目的】闡明干扰素基因刺激因子(STING)在猪抗病原微生物感染中的作用机制,为猪传染性胃肠炎、流行性腹泻和猪伪狂犬病等病毒性疾病的科学防控提供参考依据。【方法】基于CRISPR/Cas9技术,在STING基因第4、第8外显子中寻找高分靶点并设计sgRNA序列,将退火的sgRNA与酶切的LentiCRISPRv2载体用T4 DNA连接酶连接以获得LentiCRISPRv2-STING-sgRNA慢病毒载体(STING-sgRNA);以不同的STING-sgRNA慢病毒载体组合及包装质粒psPAX2和包膜质粒pMD2.G共同转染293T细胞,得到含sgRNA的慢病毒再转染3D4/21细胞;经嘌呤霉素筛选和有限稀释法获得单克隆细胞株,通过PCR、测序及Western blotting鉴定STING基因的敲除效果;并采用实时荧光定量PCR验证STING基因敲除对I型干扰素表达的影响。【结果】以不同的STING-sgRNA慢病毒载体组合与HA-STING过表达载体共同转染293T细胞,均能在细胞内对STING真核表达载体产生编辑效果,且以STING-sgRNA(1+5)慢病毒载体组合的编辑效率最高。以编辑效率最高的STING-sgRNA(1+5)慢病毒载体组合及包装质粒psPAX2和包膜质粒pMD2.G共同转染293T细胞包装出慢病毒,再用慢病毒感染3D4/21细胞,结果获得1株STING基因大片段(4989 bp)缺失的3D4/21细胞株,Western blotting检测未发现STING蛋白,说明STING基因敲除3D4/21细胞(3D4/21-STING-/-)构建成功。与野生型3D4/21细胞相比,在转染副猪嗜血杆菌DNA刺激下,3D4/21-STING-/-细胞中的IFN-β基因转录水平显著降低(P<0.05)。【结论】采用CRISPR/Cas9技术能成功大片段敲除3D4/21细胞中的STING基因,而导致STING基因功能丧失;STING基因敲除会导致细胞在病原微生物DNA刺激时I型干扰素转录障碍,也提示STING基因可能是猪抗病原微生物感染的关键因子。
关键词:CRISPR/Cas9;STING基因;猪肺泡巨噬细胞;基因敲除;I型干扰素
中图分类号:S852.43 文献标志码: A 文章编号:2095-1191(2022)04-0891-08
STING gene knockout pig alveolar macrophage cell model constructed by CRISPR/Cas9 technology and its effect on type I
interferon transcription
HUANG Qiu-yan1,2, YANG Ye-cheng1,2, ZHANG Kun-li3, MENG Fan-ming2,
LI Jian-hao2, ZHU Xiang-xing1, WANG Su-tian2*, TANG Dong-sheng1*
(1School of Life Science and Engineering, Foshan University/Guangdong Provincial Key Laboratory of Animal Molecular Design and Precise Breeding/Guangdong Provincial Research Center of Gene Editing Engineering Technology, Foshan,Guangdong 528225, China; 2Institute of Animal Science,Guangdong Academy of Agricultural Sciences/State Key Laboratory of Livestock and Poultry Breeding/Guangdong Key Laboratory of Animal Breeding and Nutrition,Guangzhou,Guangdong 510610, China; 3Institute of Animal Health, Guangdong Agricultural Science/Key Laboratory of Livestock Disease Prevention of Guangdong Province/Scientific Observation and Experiment Station of Veterinary Drugs and Diagnostic Techniques of Guangdong Province, Ministry of Agriculture and Rural Affairs,
Guangzhou, Guangdong 510640, China)
Abstract:【Objective】To elucidate the mechanism of interferon gene stimulating factor (STING) in the anti-pathogenic microbial infection of pigs, so as to further provide a reference for the scientific prevention and control of viral diseases such as porcine transmissible gastroenteritis, epidemic diarrhea and porcine pseudorabies. 【Method】High-scored targets were found in exons 4 and 8 of STING gene and corresponding sgRNA sequences were designed based on CRISPR/Cas9 technology. The annealed sgRNAs were linked with the enzyme digested LentiCRISPRV2 carrier with T4 DNA ligase to obtain LentiCRISPRV2-STING-sgRNA lentivirus carrier (STING-sgRNA); Different combinations of STING sgRNA lentivirus carriers, packaging plasmid psPAX2 and envelope plasmid pMD2.G were transfected into 293T cells to obtain lentivirus containing sgRNA and then transduced into 3D4/21 cells. Monoclonal cell lines were obtained by puromycin screening and limited dilution method. The knockout efficiencies of the STING gene were identified by PCR amplification, Sequencing and Western blotting; The effect of STING gene knockout on the expression of type I interferon was verified by real-time fluorescent quantitative PCR. 【Result】When 293T cells were transfected with different combinations of STING-sgRNA lentivirus carrier and HA-STING over expression vector, the editing effect of STING eukaryotic expression carrier could be detected in cells, and the combination of STING-sgRNA(1+5) lentivirus carrier showed the supreme editing efficiency. Thus, the STING-sgRNA(1+5) lentivirus carrier combined with the packaging plasmid psPAX2 and the envelope plasmid pMD2.G were transfected 293T cells to package lentivirus, and then infected 3D4/21 cells with lentivirus. The results showed that a 3D4/21 cell line with a large deletion of the STING gene (4989 bp) was obtained. The STING protein was not observed by Western blotting, indicating that the STING gene knockout 3D4/21 cells (3D4/21-STING-/-) were successfully constructed. The transcription level of IFN-β in 3D4/21-STING-/- cells decreased significantly (P<0.05) compared with parental cells when stimulated by transfection of Haemophilusparasuis DNA. 【Conclusion】By applying CRISPR/Cas9 technology, STING gene is successfully knock out in 3D4/21 cells, resulting in loss of function of STING gene; STING knockout leads to the transcription disorder of type I interferon when cells are stimulated by DNA, which also suggests that STING gene may be a key factor in the anti-pathogenic microbial infection of pigs.
Key words: CRISPR/Cas9; STING gene; pig alveolar macrophage cell 1cells; gene knockout; type I interferon transeniplion
Foundation items: National Natural Science Foundation of China (32002153,32002298);Guangzhou Basic and Application of Basic Research Project (202102020177);Guangdong Academy of Agricultural Sciences Scientific Innovation Strategy Special Fund (R2019YJ-YB2004)
0 引言
【研究意义】干扰素基因刺激因子(Stimulator of interferon genes,STING)又名TMEM173、MITA、ERIS或MPYS,是一种与内质网相关的信号分子,编码379个氨基酸残基,蛋白分子量约42 kD,N端包含4~5个跨膜结构域,C端含有1个球状的C端结构域(Ishikawa and Barber,2008)。STING是天然免疫通路cGAS-STING中的重要衔接蛋白,在各种组织器官及免疫相关巨噬细胞、树突状细胞和上皮细胞中以二聚体的形式进行表达(Sun et al.,2009;Xie et al.,2010)。I型干扰素介导的先天免疫反应是机体抵抗病原微生物入侵的第一道防线,在宿主感染炎症的早期阶段,巨噬细胞在炎症性疾病中会产生IL-6、IL-1β和TNF-α等细胞因子(Ma et al.,2019;Xu et al.,2019),而这些细胞因子在炎症过程中发挥着重要调节作用。当宿主受到严重感染时,免疫反应应答产生IL-6、IL-1β和TNF-α等细胞因子,有利于控制宿主自身免疫性疾病和炎症相关肿瘤发生的病理状况(Du et al.,2013)。鉴于IFN-β具有抗炎特性,可用于自身免疫疾病、炎症紊乱等疾病的治疗。因此,通过STING基因敲除研究其对IFN-β的影响,对揭示STING蛋白激活的信号转导在体内微生物清除中的作用机理具有重要意义。【前人研究进展】STING是2008年发现的一种可激活先天性免疫反应的胞内蛋白(Sun et al.,2009)。当病原微生物感染细胞后,病原微生物的基因组DNA释放进入细胞质,与细胞质中的环磷酸鸟苷—腺苷(cGAMP)合成酶cGAS结合,活化的cGAS催化ATP-GTP产生cGAMP,进而激活STING。STING能识别细菌自身合成的环二核苷酸(CDNs),使I型干扰素在细胞和个体水平上受到显著影响(Deng et al.,2014)。Ahn等(2012)研究发现,采用氧化偶氮甲烷(AOM)和耐氧化葡聚糖钠(DSS)处理STING缺陷的小鼠以模仿MYD88缺失,STING缺失小鼠对溃疡性结肠炎癌变(CAC)更敏感。West等(2015)研究表明,病毒诱导的线粒体DNA应激会导致mtDNA泄漏到细胞质中并激活STING途径和細胞因子的产生。此外,AOM诱导的DNA损伤能活化STING而诱导细胞因子产生(Irrazábal et al.,2014);而具有STING获得性功能变化的患者常伴有血管病变,STING识别病变产生的cGAMP从而激活干扰素(Warner et al.,2017)。脱氧核糖核酸酶II(DNase II)是一种存在于溶酶体中的生物酶,能清除死亡细胞和细胞核排出的DNA,DNase II缺失会增加STING信号的激活和I型干扰素的产生,从而引起系统性自身炎症(Yoshida et al.,2005;Ahn et al.,2012;Rodero et al.,2017)。近年来,CRISPR/Cas9技术已广泛应用于动物的基因组编辑,尤其是疾病治疗及疾病模型构建等方面(Lei et al.,2018;Bao et al.,2019)。基因编辑是精确操纵细胞内DNA序列以改变细胞命运和生物体特性的技术,有效提高了研究者对遗传学的认识,并为治疗遗传性疾病提供了重要工具。研究表明,利用CRISPR/Cas9介导的同源重组HR方法将小鼠解偶联蛋白1(UCP1)有效连接至猪内源性UCP1基因座中,能得到UCP1基因敲入猪(Zheng et al.,2017);以CRISPR/Cas9介导的基因编辑进行MSTN基因双敲除,然后进行体细胞核移植,成功获得MSTN基因双敲猪(Zhu et al.,2020)。【本研究切入点】STING基因在病原菌微生物感染过程中至关重要,其激活产生的信号转导会进一步诱导IFN-β产生,但至今鲜见利用CRISPR/Cas9技术探索STING基因发挥抗病功能的相关研究报道。【拟解决的关键问题】利用CRISPR/Cas9技术构建STING基因敲除猪肺泡巨噬细胞系,并在此基础上研究STING基因敲除对病原微生物DNA刺激的影响,旨在阐明猪抗病原微生物感染的分子机制,为猪传染性胃肠炎、流行性腹泻和猪伪狂犬病等病毒性疾病的科学防控提供参考依据。
1 材料与方法
1. 1 试验材料
猪肺泡巨噬细胞系(3D4/21)和HEK-293T细胞由畜禽育种国家重点实验室保存提供;LentiCRISPRv2载体购自Addgene公司;包装质粒psPAX2和包膜质粒pMD2.G购自Sigma公司;质粒中提(D6915)、大提(D6926)试剂盒购自OMEGA公司;DNA胶回收试剂盒(DP214-02)、细胞/细菌/酵母基因组DNA提取试剂盒(DP302-02)及大肠杆菌DH5α感受态细胞(CB101)购自天根生化科技(北京)有限公司;脂质体转染试剂盒(Lipo293)、BCA蛋白浓度测定试剂盒(P0010)及ECL化学发光液(P0018)购自碧云天生物技术有限公司;Lipofectamine 3000转染试剂(L3000015)购自Invitrogen公司;DMEM高糖培养基、澳洲胎牛血清及Opti-MEM培养基购自Gibco公司;限制性内切酶Bsmb I(#R0739L)和T4 DNA连接酶(#M0202L)购自NEB公司。
1. 2 试验方法
1. 2. 1 引物设计与合成 登录https://www.ncbi.nlm. nih.gov/,拷贝猪STING基因序列(GenBank登录号ACJ70708.1),在STING基因第4、第8号外显子中寻找高分靶点并设计sgRNA序列。根据sgRNA序列设计退火引物(表1),再根据sgRNA位置设计扩增该片段的引物(表2),所有引物均委托生工生物工程(上海)股份有限公司合成。
1. 2. 2 LentiCRISPRv2线性化载体制备 将设计好的STING基因5对sgRNA上、下游引物(表1)进行退火杂交,退火体系:Oligo-1 9.0 μL,Oligo-2 9.0 μL,T4 DNA Buffer 2.0 μL分别进行退火,退火条件:95 ℃ 10 min;95~45 ℃,每2 min下降1 ℃;45~16 ℃,每1 min下降1 ℃。退火后即获得双链DNA。同时,采用限制性内切酶Bsmb I对LentiCRISPRv2载体进行酶切,酶切反应体系:LentiCRISPRv2载体1.0 μg,Bsmb I 2.0 μL,Buffer 3-1 5.0 μL,ddH2O补足至50.0 μL;酶切条件:37 ℃孵育15 min。所有酶切产物以1.0%琼脂糖凝胶电泳进行检测,切割并回收目的条带,纯化的LentiCRISPRv2线性化载体-20 ℃保存备用。
1. 2. 3 LentiCRISPRv2-STING-sgRNA慢病毒载体构建及验证 以T4 DNA连接酶连接LentiCRISPRv2线性化载体与退火引物,转化DH5α感受态细胞后接种至含氨苄青霉素(Amp)的LB培养基上培养过夜,挑选单个菌落,接种至含Amp的LB液体培养基中继续培养。经菌液PCR鉴定,选取鉴定正确的菌液以去内毒素质粒提取试剂盒提取质粒,将PCR鉴定呈阳性的重组质粒送至生工生物工程(上海)股份有限公司测序。测序正确重组质粒对应的菌液扩大培养16 h后,采用去内毒素质粒试剂盒提取质粒,并按照顺序分别命名为LentiCRISPRv2-STING-sgRNA1(STING-sgRNA1)、LentiCRISPRv2-STING-sgRNA2(STING-sgRNA2)、LentiCRISPRv2-STING-sgRNA3(STING-sgRNA3)、LentiCRISPRv2-STING-sgRNA4(STING-sgRNA4)和LentiCRISPRv2-STING-sgRNA5(STING-sgRNA5),-20 ℃保存备用。
1. 2. 4 不同sgRNA对STING基因敲除效率检测 使用完全培养基(DMEM+10% FBS+1% PS)培养293T细胞,待细胞生长汇合度达60%~70%时进行瞬时转染,共设7个组:HA-STING+STING-sgRNA(1+4),HA-STING+STING-SgRNA(1+5),HA-STING+STING-sgRNA(2+4),HA-STING+STING-sgRNA(2+5),HA-STING+STING-sgRNA(3+4),HA-STING+STING-gRNA(3+5),HA-STING+LentiCRISPRv2(阳性对照),LentiCRISPRv2(阴性对照)。转染试剂A:125.0 μL Opti-MEM培养基与5.0 μL Lipo293™转染试剂混合;转染试剂B:125.0 μL Opti-MEM培养基中加入2.0 μg STING-sgRNA慢病毒载体和0.5 μg HA-STING质粒或空载体质粒。将转染试剂A与转染试剂B混合后在室温下孵育15 min,然后加入6孔培养板的培养基中,混匀,37 ℃继续培养24 h后加入RIPA裂解细胞收集蛋白。采用BCA法测定总蛋白水平,经聚丙烯酰胺凝胶电泳分离后转膜(250 mA,90 min),以检测HA-STING蛋白的表达水平。转膜结束后,用TBST缓冲液洗膜3次,每次10 min;5%脱脂奶粉常温摇床上封闭2 h,加入以1%脱脂奶粉配制的特异性HA抗体(1∶1000),4 ℃孵育过夜;收集一抗冻存,用TBST缓冲液洗膜3次,每次10 min;加入以1.0%脱脂奶粉配制的辣根过氧化物酶(HRP)标记山羊抗兔二抗(1∶5000),室温摇床上孵育1 h,再用TBST缓冲液洗膜3次,每次5 min;加入电化学发光液后曝光,以GAPDH为内参基因。
1. 2. 5 STING-sgRNA慢病毒包装 转染前1 d,接种293T细胞至10 cm培养皿中,以转染当天的细胞汇合度达90%为宜,转染前更换10 mL新鲜完全培养基(含10%血清的DMEM)。通过Lipo293将STING-sgRNA重组质粒与包装质粒psPAX2和包膜质粒pMD2.G共转染293T细胞,置于37 ℃的CO2培养箱中继续培养,分别于第36和48 h时收集细胞上清液,3000 r/min离心10 min,去除细胞碎片,收集病毒原液上清液,4 ℃下30000 r/min离心2 h;然后以3 mL PBS重悬病毒,分装,-80 ℃保存备用。
1. 2. 6 慢病毒感染3D4/21细胞及抗性筛选 复苏液氮冻存的3D4/21细胞,待生长稳定后以每孔1.5×105个细胞传代于6孔培养板中,恒温培养箱培养过夜;取出-80 ℃超低温保存的病毒液,4 ℃融化后加入DMEM培養基,混匀,按要求加入6孔培养板中,以纯的DMEM培养基为空白对照。病毒感染24 h后,去除细胞上清液,更换新鲜的DMEM培养基,继续培养6~8 h后更换为含嘌呤霉素的完全培养基进行筛选,每隔2 d更换含Puro的培养基,持续药筛7 d,待空白对照组的细胞全部死亡,试验组使用含Puro的培养基继续培养。采用有限稀释法筛选单克隆细胞系,单克隆细胞系经扩大培养后,选择部分细胞提取DNA,并以STING-F1/STING-R1引物进行PCR扩增,若1.0%琼脂糖凝胶电泳出现195 bp的目的条带即可判定该细胞存在STING基因大片段缺失,反之则不出现任何条带。为进一步确定出现PCR扩增阳性结果的细胞株为STING基因大片段缺失细胞株,将PCR扩增产物送至生工生物工程(上海)股份有限公司测序,测序结果与野生型参考序列进行对比,以确定是否在2个靶点间发生大片段缺失。同时,对阳性单克隆细胞继续培养并提取蛋白,以STING(1∶1000,CST)和GAPDH(1∶5000,Affinity)为一抗,通过Western blotting检测STING基因敲除效率,并以野生型3D4/21细胞为对照。
1. 2. 7 猪STING基因敲除对IFN-β基因转录的影响
将副猪嗜血杆菌H45接种至TSA培养基中进行扩大培养,采用细菌基因组DNA提取试剂盒提副猪嗜血杆菌DNA,取5 ng/mL的副猪嗜血杆菌DNA分别转染野生型3D4/21细胞和STING基因敲除3D4/21细胞(3D4/21-STING-/-)。转染16 h后提取细胞RNA,反转录合成cDNA,通过实时荧光定量PCR检测细胞中的IFN-β基因表达情况。实时荧光定量PCR的引物为IFN-β-F(5'-GTGGAACTTGATGGGCAGAT-3')和IFN-β-R(5'-TTCCTCCTCCATGATTTCCTC-3');扩增程序:95 ℃预变性30 s;95 ℃ 5 s,60 ℃ 30 s,进行40个循环。
1. 3 统计分析
试验数据采用SPSS 20.0进行Studentʼs t 检验及单因素方差分析(One-way ANOVA)。
2 结果与分析
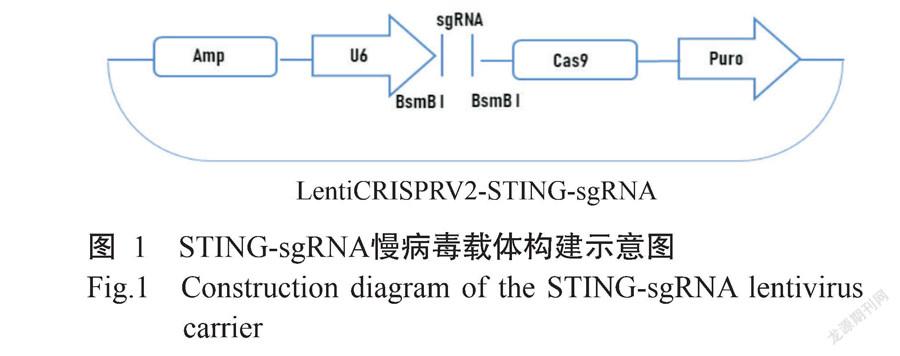
2. 1 STING-sgRNA慢病毒载体的构建及验证结果
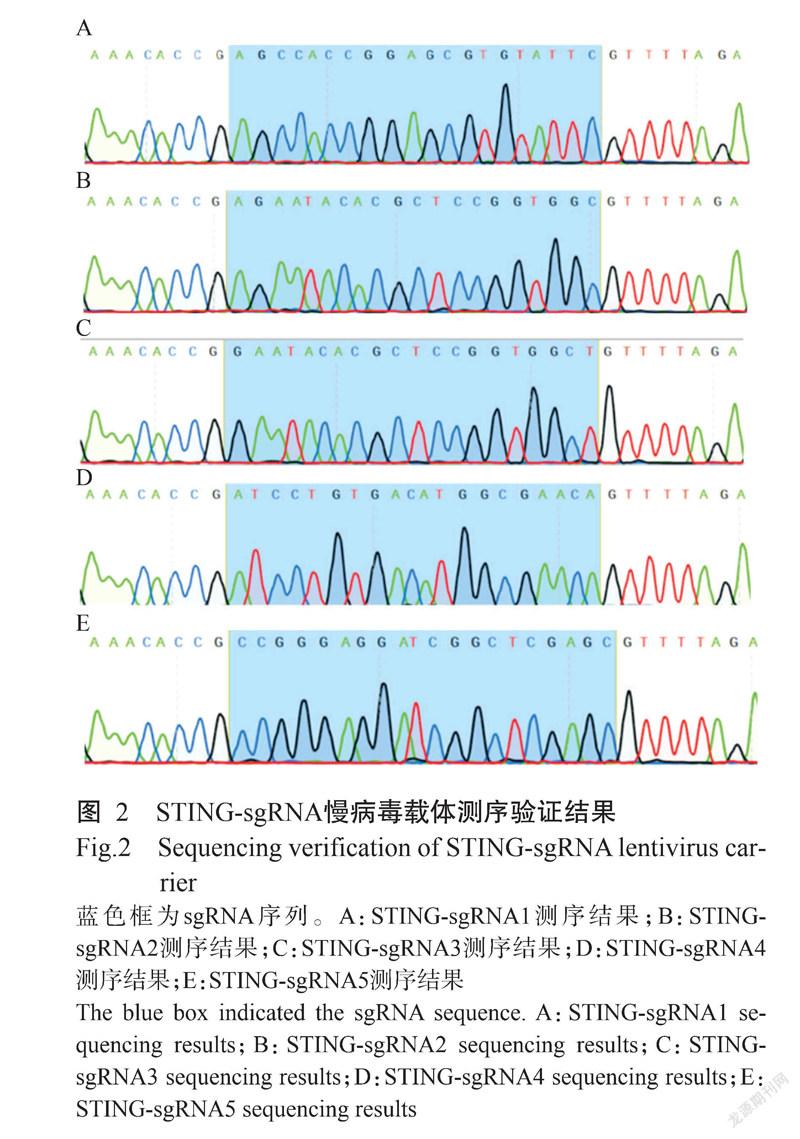
经限制性内切酶Bsmb I酶切后的LentiCRISPRv2载体与退火的sgRNA进行连接,以构建STING-sgRNA慢病毒载体(图1)。为确认STING-sgRNA慢病毒载体是否构建成功,将其送至生工生物工程(上海)股份有限公司测序,测序结果(图2)与插入的sgRNA序列信息符合,说明STING-sgRNA慢病毒载体构建成功。
2. 2 不同STING-sgRNA慢病毒载体组合对STING基因敲除效率的影响
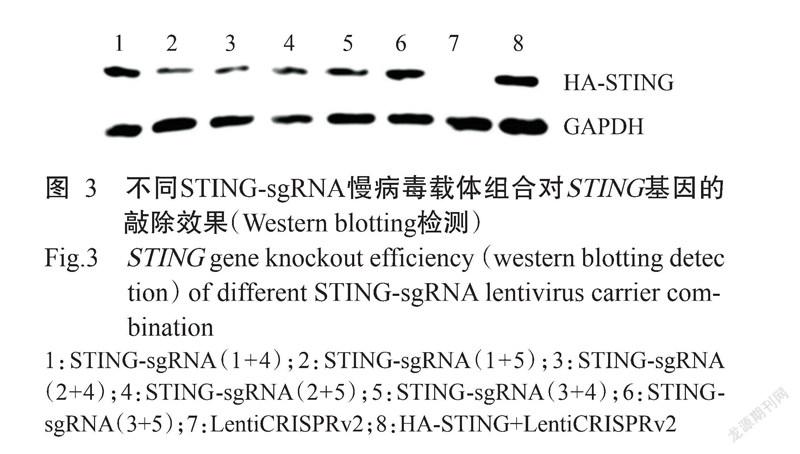
以不同STING-sgRNA慢病毒载体组合与HA-STING过表达载体共同转染293T细胞,采用HA抗体蛋白检测时,均能检测到携带有HA标签的外源STING条带。Western blotting检测结果(图3)显示,转染STING-sgRNA(1+4)、STING-sgRNA(1+5)、STING-sgRNA(2+4)、STING-sgRNA(2+5)、STING-sgRNA(3+4)及STING-sgRNA(3+5)慢病毒载体组合的293T细胞,能在细胞内对STING真核表达载体产生编辑效果,且以STING-sgRNA(1+5)慢病毒载体组合的编辑效率最高。
2. 3 STING基因敲除3D4/21细胞株的筛选及测序鉴定结果
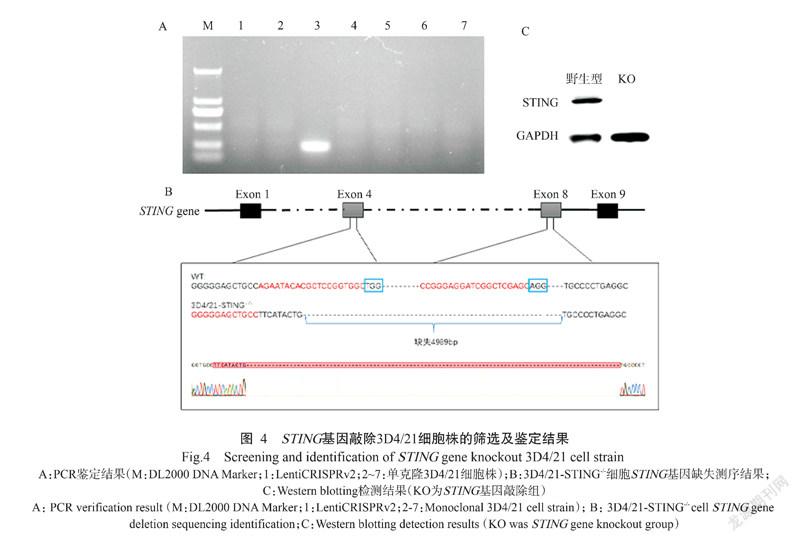
以编辑效率最高的STING-sgRNA(1+5)慢病毒载体组合及包装质粒psPAX2和包膜质粒pMD2.G共同转染293T细胞,成功包装出慢病毒,再用慢病毒感染3D4/21细胞,经嘌呤霉素筛选和有限稀释法获得6株可能存在STING基因敲除的单克隆3D4/21细胞株。对所有挑取的单核克隆细胞株进行鉴定,提取其基因组DNA进行PCR鉴定,1.0%琼脂糖凝胶电泳结果显示,仅有1株单克隆3D4/21细胞株扩增获得预期的目的条带(195 bp),即判定该细胞株存在STING基因大片段缺失,其他5株单克隆3D4/21细胞株均未出现任何条带(图4-A)。同时将扩增获得的目的片段送至生工生物工程(上海)股份有限公司测序,测序结果与野生型3D4/21细胞进行比对,表明成功获得1株STING基因大片段(4989 bp)缺失的3D4/21细胞株(图4-B)。提取野生型3D4/21细胞和3D4/21-STING-/-细胞的蛋白样品,Western blotting检测结果(图4-C)也表明3D4/21-STING-/-细胞中的STING蛋白未见表达,进一步证实STING基因敲除成功。
2. 4 猪STING基因敲除对IFN-β基因转录的影响
通过Lipofectamine 3000转染试剂将副猪嗜血杆菌DNA转染至3D4/21-STING-/-细胞中,同时以野生型3D4/21细胞为对照。转染16 h后,通过实时荧光定量PCR检测STING基因敲除对IFN-β基因转录的影响。结果(图5)显示,与野生型3D4/21细胞相比,在转染副猪嗜血杆菌DNA刺激下,3D4/21-STING-/-细胞中的IFN-β基因转录水平显著下降(P<0.05),表明敲除STING基因会影响IFN-β基因转录。
3 讨论
基因编辑是一种基因工程技术,具有操作简单、精确高效等特点,可在细胞和生物体的基因组中进行精准编辑,尤其是CRISPR/Cas9技术已在疾病治疗、基因检测、基因功能调节等方面得到广泛应用(刘雷雷等,2019;朱丽颖等,2020;冯江浩等,2021;史梦然等,2021)。已有研究表明,癌症基因EGFR可通过CRISPR/Cas9基因编辑被敲除,促使下游信号UBXN1表达上调,抑制NF-κB活化,进而抑制肿瘤的生长(Huang et al.,2017)。在猪模型中,由单基因缺失引起的杜氏肌营养不良症(DMD)可通过CRISPR/Cas9基因编辑来纠正(Moretti et al.,2020)。此外,Yang等(2018)應用CRISPR/CAS9基因编辑技术靶向成功构建CD163基因敲除猪,在攻毒试验中CD163基因敲除猪并未表现出病毒血症、抗体反应、高烧等与猪繁殖与呼吸综合征病相关的临床症状。可见,随着基因编辑技术的发展及人们对疾病新有效靶点的不断认识,临床上可通过纠正致病基因或有效突变以达到疾病治疗的效果。本研究基于CRISPR/Cas9技术成功构建获得STING基因敲除3D4/21细胞(3D4/21-STING-/-),为今后深入研究STING基因在猪源疾病感染中的作用机理提供了技术支撑。
近年来,针对STING基因的研究越来越广泛。STING是一种中央衔接蛋白,可介导DNA病毒感染的先天免疫反应(Liu and Wang,2016)。STING被激活后,招募下游效应分子TBK1和IRF3,通过磷酸化、泛素化和亚细胞易位等方式转导,最终诱导I型干扰素表达(Baldwin et al.,2004;Ferguson et al.,2012)。干扰素作为关键细胞因子,具有抗病毒和细胞调节的特性,与先天免疫反应密切相关。其中,IFN-β由许多类型的细胞分泌,可通过刺激巨噬细胞和NK细胞引发抗病毒反应,且具有抗肿瘤活性(Kal-liolias and Ivashkiv,2010;González-Navajas et al.,2012;Liu et al.,2014)。STING作为具有抗病原微生物感染的活性蛋白,在多种病原感染的内源性免疫激活及调节过程中发挥重要作用。至今,有关STING的结构、功能及其免疫调节作用在鼠及人类中已有深入研究,但针对猪的研究尚不明确(Chung et al.,2019)。最新研究发现,结核分枝杆菌编码蛋白MmsA会通过抑制STING-TBK1-IRF3信号通路,或通过促进P62介导STING的自噬和降解,而影响I型干扰素的产生(Sun et al.,2020)。此外,Wu等(2020)研究证实,具有消除干扰素依赖性活性能力的STING1S365A/S365A突变小鼠既保留了STING的干扰素非依赖活性,又能防止HSV-1感染或肿瘤免疫逃避等,即STING对RNA病毒和DNA病毒的免疫反应均有影响。本研究结果表明,与野生型3D4/21细胞相比,在副猪嗜血杆菌DNA的刺激下,3D4/21-STING-/-细胞中的IFN-β基因转录水平显著降低,表明STING基因缺失会导致I型干扰素转录障碍,也提示STING基因可能是猪抗病原微生物感染的关键因子。可见,STING可抵抗多种病原微生物的入侵,其研究可为癌症、肥胖和糖脂代谢等疾病治疗提供新的思路。
4 結论
采用CRISPR/Cas9技术能成功大片段敲除3D4/21细胞中的STING基因,而导致STING基因功能丧失;STING基因敲除会导致细胞在病原微生物DNA刺激时I型干扰素转录障碍,也提示STING基因可能是猪抗病原微生物感染的关键因子。
参考文献:
冯江浩,魏思昂,闫丽欢,崔洁,冯焱. 2021. CRISPR/Cas9基因编辑技术及应用研究概述[J]. 动物医学进展,42(3):123-126. [Feng J H,Wei S A,Yan L H,Cui J,Feng Y. 2021. Progress on gene editing technology and application of CRISPR/Cas9[J]. Progress in Veterinary Medicine,42(3):123-126.] doi:10.3969/j.issn.1007-5038. 2021.03.026.
刘雷雷,贾启鹏,李宗帅,杨洋,李海江,赵兴绪,张勇. 2019. 利用CRISPR/Cas9基因编辑技术靶向敲除奶山羊胎儿成纤维细胞SCD1基因[J]. 甘肃农业大学学报,54(6):30-38. [Liu L L,Jia Q P,Li Z S,Yang Y,Li H J,Zhao X X,Zhang Y. 2019. Targeted knockout of SCD1 gene in dairy goat fetal fibroblasts by CRISPR/Cas9 gene editing technology[J]. Journal of Gansu Agricultural University,54(6):30-38.]doi:10.13432/j.cnki.jgsau.2019.06.005.
史梦然,沈宗毅,张楠,王璐瑶,喻长远,杨昭. 2021. CRISPR/Cas9系统在疾病研究和治疗中的应用[J]. 生物工程学报,37(4):1205-1228. [Shi M R,Shen Z Y,Zhang N,Wang L Y,Yu C Y,Yang Z. 2021. CRISPR/Cas9 techno-logy in disease research and therapy:A review[J]. Chinese Journal of Biotechnology,37(4):1205-1228.] doi:10.13345/j.cjb.200401.
朱丽颖,郑月萍,徐雪珍,段芊芊,韩妮莎. 2020. 一种准确、简便测定CRISPR/Cas9基因编辑效率的方法[J]. 江苏农业学报,36(2):299-305. [Zhu L Y,Zheng Y P,Xu X Z,Duan Q Q,Han N S. 2020. A convenient and accurate method for determining the efficiency of CRISPR/Cas9-based gene editing[J]. Jiangsu Journal of Agricultural Sciences,36(2):299-305.]doi:10.3969/j.issn.1000-4440. 2020.02.007.
Ahn J,Gutman D,Saijo S,Barber G N. 2012. STING manifests self DNA-dependent inflammatory disease[J]. Proceedings of the National Academy of Sciences of the United States of America,109(47):19386-19391. doi:10.1073/pnas.1215006109.
Baldwin S L,Powell T D,Sellins K S,Radecki S V,Cohen J J,Milhausen M J. 2004. The biological effects of five feline IFN-α subtypes[J]. Veterinary Immunology and Immunopathology,99(3-4):153-167. doi:10.1016/j.vetimm. 2004.01.012.
Bao A L,Burritt D J,Chen H F,Zhou X N,Cao D,Tran L S P. 2019. The CRISPR/Cas9 system and its applications in crop genome editing[J]. Critical Reviews in Biotechnology,39(3):321-336. doi:10.1080/07388551.2018.1554621.
Chung K W,Dhillon P,Huang S Z,Sheng X,Shrestha R,Qiu C X,Kaufman B A,Park J,Pei L M,Baur J,Palmer M,Susztak K. 2019. Mitochondrial damage and activation of the STING pathway lead to renal inflammation and fibrosis[J]. Cell Metabolism,30(4):784-799. doi:10.1016/j.cmet.2019.08.003.
Deng L F,Liang H,Xu M,Yang X M,Burnette B,Arina A,Li X D,Mauceri H,Beckett M,Darga T,Huang X N,Gajewski T F,Chen Z J,Fu Y X,Weichselbaum R R. 2014. STING-dependent cytosolic DNA sensing promotes radiation-induced type I interferon-dependent antitumor immunity in immunogenic tumors[J]. Immunity,41(5):843-852. doi:10.1016/j.immuni.2014.10.019.
Du C Y Q,Choi R C Y,Zheng K Y Z,Dong T T X,Lau D T W,Tsim K W K. 2013. Yu Ping Feng San,an ancient Chinese herbal decoction containing Astragali Radix,Atractylodis macrocephalae Rhizoma and Saposhnikoviae Radix,regulates the release of cytokines in murine macrophages[J]. PLoS One,8(11):e78622. doi:10.1371/journal.pone.0078622.
Ferguson B J,Mansur D S,Peters N E,Ren H W,Smith G L. 2012. DNA-PK is a DNA sensor for IRF-3-dependent innate immunity[J]. eLife,1:e00047. doi:10.7554/eLife.00047.
González-Navajas J M,Lee J,David M,Raz E. 2012. Immunomodulatory functions of type I interferons[J]. Nature Reviews Immunology,12(2):125-135. doi:10.1038/nri3133.
Huang K,Yang C,Wang Q X,Li Y S,Fang C,Tan Y L,Wei J W,Wang Y F,Li X,Zhou J H,Zhou B C,Yi K K,Zhang K L,Li J,Kang C S. 2017. The CRISPR/Cas9 system targeting EGFR exon 17 abrogates NF-κB activation via epigenetic modulation of UBXN1 in EGFRwt/vIII glio-ma cells[J]. Cancer Letters,388:269-280. doi:10.1016/j.canlet.2016.12.011.
Irrazábal T,Belcheva A,Girardin S E,Martin A,Philpott D J. 2014. The multifaceted role of the intestinal microbiota in colon cancer[J]. Molecular Cell,54(2):309-320. doi:10.1016/j.molcel.2014.03.039.
Ishikawa H,Barber G N. 2008. STING is an endoplasmic reti-culum adaptor that facilitates innate immune signalling[J]. Nature, 455(7213):674-678. doi:10.1038/nature07317.
Kalliolias G D,Ivashkiv L B. 2010. Overview of the biology of type I interferons[J]. Arthritis Research & Therapy,12(S1):S1. doi:10.1186/ar2881.
Lei Z,Deng M H,Yi Z J,Sun Q,Shapiro R A,Xu H B,Li T L,Loughran P A,Griepentrog J E,Huang H,Scott M J,Huang F Z,Billiar T R. 2018. cGAS-mediated autophagy protects the liver from ischemia-reperfusion injury independently of STING[J]. American Journal of Physiology-Gastrointestinal and Liver Physiology,314(6):G655-G667. doi:10.1152/ajpgi.00326.2017.
Liu X,Wang C. 2016. The emerging roles of the STING adaptor protein in immunity and diseases[J]. Immunology, 147(3):285-291. doi:10.1111/imm.12561.
Liu Y,Jesus A A,Marrero B,Yang D,Ramsey S E,Sanchez G A M,Tenbrock K,Wittkowski H,Jones O Y,Kuehn H S,Lee C C R,DiMattia M A,Cowen E W,Gonzalez B,Palmer I,DiGiovanna J J,Biancotto A,Kim H,Tsai W L,Trier A M,Huang Y,Stone D L,Hill S,Kim H J,St Hilaire C,Gurprasad S,Plass N,Chapelle D,Horkayne-Szakaly I,Foell D,Barysenka A,Candotti F,Holland S M,Hughes J D,Mehmet H,Issekutz A C,Raffeld M,McElwee J,Fontana J R,Minniti C P,Moir S,Kastner D L,Gadina M,Steven A C,Wingfield P T,Brooks S R,Rosenzweig S D,Fleisher T A,Deng Z,Boehm M,Paller A S,Goldbach-Mansky R. 2014. Activated STING in a vascular and pulmonary syndrome[J]. The New England Journal of Medicine,371(6):507-518. doi:10.1056/NEJMoa1312625.
Ma X W,Jiang Z Y,Li N,Jiang W,Gao P,Yang M J,Yu X Y,Wang G F,Zhang Y. 2019. Ets2 suppresses inflammatory cytokines through MAPK/NF-κB signaling and directly binds to the IL-6 promoter in macrophages[J]. Aging(Albany NY),11(22):10610-10625. doi:10.18632/aging.102480.
Moretti A,Fonteyne L,Giesert F,Hoppmann P,Meier A B,Bozoglu T,Baehr A,Schneider C M,Sinnecker D,Klett K,Fröhlich T,Rahman F A,Haufe T,Sun S,Jurisch V,Kessler B,Hinkel R,Dirschinger R,Martens E,Jilek C,Graf A,Krebs S,Santamaria G,Kurome M,Zakhartchenko V,Campbell B,Voelse K,Wolf A,Ziegler T,Reichert S,Lee S,Flenkenthaler F,Dorn T,Jeremias I,Blum H,Dendorfer A,Schnieke A,Krause S,Walter M C,Klymiuk N,Laugwitz K L,Wolf E,Wurst W,Kupatt C. 2020. Somatic gene editing ameliorates skeletal and cardiac muscle failure in pig and human models of Duchenne muscular dystrophy[J]. Nature Medicine,26(2):207-214. doi:10.1038/s41591-019-0738-2.
Rodero M P,Tesser A,Bartok E,Rice G I,Della Mina E,Depp M,Beitz B,Bondet V,Cagnard N,Duffy D,Dussiot M,Frémond M L,Gattorno M,Guillem F,Kitabayashi N,Porcheray F,Rieux-Laucat F,Seabra L,Uggenti C,Volpi S,Zeef L A H,Alyanakian M A,Beltrand J,Bianco A M,Boddaert N,Brouzes C,Candon S,Caorsi R,Charbit M,Fabre M,Faletra F,Girard M,Harroche A,Hartmann E,Lasne D,Marcuzzi A,Neven B,Nitschke P,Pascreau T,Pastore S,Picard C,Picco P,Piscianz E,Polak M,Quartier P,Rabant M,Stocco G,Taddio A,Uettwiller F,Valencic E,Vozzi D,Hartmann G,Barchet W,Hermine O,Bader-Meunier B,Tommasini A,Crow Y J. 2017. Type I interferon-mediated autoinflammation due to DNase II deficiency[J]. Nature Communications,8(1):2176. doi:10.1038/s41467-017-01932-3.
Sun W X,YANG L,Chen L,Chen H H,You F P,Zhou X,Zhou Y,Zhai Z H,Chen D Y,Jiang Z F. 2009. ERIS,an endoplasmic reticulum IFN stimulator,activates innate immune signaling through dimerization[J]. Proceedings of the National Academy of Sciences of the United States of America,106(21):8653-8658. doi:10.1073/pnas.0900850106.
Sun Y F,Zhang W,Dong C S,Xiong S. 2020. Mycobacterium tuberculosis MmsA (Rv0753c) interacts with STING and blunts the type I interferon response[J]. mBio,11(6):e03254-19. doi:10.1128/mBio.03254-19.
Warner J D,Irizarry-Caro R A,Bennion B G,Ai T L,Smith A M,Miner C A,Sakai T,Gonugunta V K,Wu J J,Platt D J,Yan N,Miner J J. 2017. STING-associated vasculo-pathy develops independently of IRF3 in mice[J]. Journal of Experimental Medicine,214(11):3279-3292. doi:10. 1084/jem.20171351.
West A P,Khoury-Hanold W,Staron M,Tal M C,Pineda C M,Lang S M,Bestwick M,Duguay B A,Raimundo N,MacDuff D A,Kaech S M,Smiley J R,Means R E,Iwasaki A,Shadel G S. 2015. Mitochondrial DNA stress primes the antiviral innate immune response[J]. Nature,520(7548):553-557. doi:10.1038/nature14156.
Wu J J,Dobbs N,Yang K,Yan N. 2020. Interferon-independent activities of mammalian STING mediate antiviral response and tumor immune evasion[J]. Immunity,53(1):115-126. doi:10.1016/j.immuni.2020.06.009.
Xie L L,Liu M L,Fang L R,Su X C,Cai K M,Wang D,Chen H C,Xiao S B. 2010. Molecular cloning and functional characterization of porcine stimulator of interferon genes (STING)[J]. Developmental & Comparative Immunology,34(8):847-854. doi:10.1016/j.dci.2010.03.005.
Xu M D,Jiang Z Y,Wang C L,Li N,Bo L L,Zha Y P,Bian J J,Zhang Y,Deng X M. 2019. Acetate attenuates inflammasome activation through GPR43-mediated Ca2+-dependent NLRP3 ubiquitination[J]. Experimental & Molecular Medicine,51(7):1-13. doi:10.1038/s12276-019-0276-5.
Yang H Q,Zhang J,Zhang X W,Shi J S,Pan Y F,Zhou R,Li G L,Li Z,Cai G Y,Wu Z F. 2018. CD163 knockout pigs are fully resistant to highly pathogenic porcine reproductive and respiratory syndrome virus[J]. Antiviral Research,151:63-70. doi:10.1016/j.antiviral.2018.01.004.
Yoshida H,Okabe Y,Kawane K,Fukuyama H,Nagata S. 2005. Lethal anemia caused by interferon-beta produced in mouse embryos carrying undigested DNA[J]. Nature Immunology,6(1):49-56. doi:10.1038/ni1146.
Zheng Q T,Lin J,Huang J J,Zhang H Y,Zhang R,Zhang X Y,Cao C W,Hambly C,Qin G S,Yao J,Song R G,Jia Q T,Wang X,Li Y S,Zhang N,Piao Z Y,Ye R C,Speakman J R,Wang H M,Zhou Q,Wang Y F,Jin W Z,Zhao J G. 2017. Reconstitution of UCP1 using CRISPR/Cas9 in the white adipose tissue of pigs decreases fat deposition and improves thermogenic capacity[J]. Procee-dings of the National Academy of Sciences of the United States of America,114(45):E9474-E9482. doi:10.1073/pnas.1707853114.
Zhu X X,Zhan Q M,Wei Y Y,Yan A F,Feng J,Liu L,Lu S S,Tang D S. 2020. CRISPR/Cas9-mediated MSTN disruption accelerates the growth of Chinese Bama pigs[J]. Reproduction in Domestic Animals,55(10):1314-1327. doi:10.1111/rda.13775.
收稿日期:2021-06-17
基金項目:国家自然科学基金项目(32002153,32002298);广州市基础与应用基础研究项目(202102020177);广东省农业科学院科技创新战略专项(R2019YJ-YB2004)
通讯作者:王塑天(1989-),https://orcid.org/0000-0002-3554-5022,博士,副研究员,主要从事抗病基因功能研究与抗病育种工作,E-mail:wstlyt@126.com;唐冬生(1962-),https://orcid.org/0000-0001-9466-6687,博士,研究员,主要从事基因编辑与生物治疗技术研究工作,E-mail:tangdsh@163.com
第一作者:黄秋艳(1996-),https://orcid.org/0000-0002-2052-347X,主要从事抗病育种与基因编辑研究工作,E-mail:1305006274 @qq.com
