基于生物信息学方法分析软组织肉瘤中甲状腺激素受体相互作用蛋白13的表达及临床意义△
2022-07-09于得全邵秋菊刘锐梁军高宏屈焕敏
于得全,邵秋菊,刘锐,梁军,高宏,屈焕敏
空军军医大学唐都医院1放疗科,3政治部,西安 710038
2西安交通大学第一附属医院放疗科,西安 710038
软组织肉瘤是一类来源于间充质的异质性肿瘤,约占成年人恶性肿瘤的1%[1]。软组织肉瘤通常表现为无症状肿块,多分布于腹膜后、内脏和四肢。根据组织病理学的异质性特征、临床表现和分子特征,可将软组织肉瘤分为50种不同的组织学类型[2]。软组织肉瘤的治疗方法主要是手术联合放化疗,但由于软组织肉瘤亚型众多,化疗药物的治疗有效性很低,并且某些亚型对化疗药物的反应有限,因此软组织肉瘤患者的治疗效果不是很满意[3]。因此,寻找有价值的生物靶标尤为重要。甲状腺激素受体相互作用蛋白13(thyroid hormone receptor interactor 13,TRIP13)位于人类5号染色体,能够编码包含432个氨基酸残基的蛋白质,是抗激动蛋白抗体(anti-actin antibody,AAA)-腺苷三磷酸(adenosine triphosphate,ATP)家族成员之一。TRIP13主要参与纺锤体组装检查点(spindle assembly checkpoint,SAC)失活、染色体联会和DNA断裂或修复[4]。有研究表明,TRIP13在多种肿瘤中呈高表达,且与患者的不良预后有关[5]。然而,TRIP13在软组织肉瘤中的表达和作用还缺乏研究。本研究应用生物信息学方法分析TRIP13在软组织肉瘤中的表达、与预后的关系、突变情况及生物学功能,旨在为进一步分析软组织肉瘤发生的分子机制和靶向治疗提供基础理论。
1 资料与方法
1.1 Oncomine数据库
Oncomine数据库是一个公开访问的在线数据库,提供了多种肿瘤微阵列信息的全基因组转录分析[6]。本研究通过Oncomine数据库分析TRIP13在常见肿瘤和软组织肉瘤组织中的表达情况,检索条件如下。①gene:TRIP13;②analysis type:cancer vs normal analysis;③cancer type:sarcoma;④data style:mRNA;⑤阈值条件:P-Value﹤0.05,Fold Change﹤2,GENE Rank:Top 10%。
1.2 GEPIA数据库
GEPIA数据库是一个用于分析来自TCGA和GTEx的9376个肿瘤样本和8589个正常样本的RNA测序表达数据的平台[7]。本研究通过GEPIA数据库检索TRIP13在软组织肉瘤组织和正常组织中的表达量。
1.3 CCLE数据库
CCLE数据库是能够为研究遗传体变异、候选靶标、小分子和生物疗法提供可靠数据的在线网站[8]。本研究通过CCLE数据库检索TRIP13在软组织肉瘤细胞中的表达水平。
1.4 Kaplan-Meier Plotter数据库
Kaplan-Meier Plotter数据库是基于TCGA和GEO数据库中的癌症数据分析基因表达与患者预后关系的开放性网站[9]。在Kaplan-Meier Plotter数据库中每个分析的风险比(hazard rate,HR)、95%置信区间(confidence interval,CI)和P值均由网站自动生成,根据此参数患者被分为TRIP13高表达组和TRIP13低表达组,分析TRIP13表达水平对软组织肉瘤患者总生存时间(overall survival,OS)和无病生存时间(disease-free survival,DFS)的影响。P﹤0.05为差异有统计学意义。
1.5 cBioPortal数据库
cBioPortal是一个可以分析多维癌症基因组学的在线工具[10]。选取软组织肉瘤数据(来自TCGA,PanCaner Atlas),分析TRIP13基因突变情况及突变类型。此外,研究TRIP13基因突变与软组织肉瘤患者预后的关系。P﹤0.05为差异有统计学意义。
1.6 String数据库
String数据库是通过整合所有公开的蛋白质相互作用数据,预测蛋白质的潜在功能[11]。本研究通过String数据库探索与TRIP13相互作用的蛋白质网络,在数据库中输入“TRIP13”,物种选择“Homo sapiens”,置信度设置为0.400,相互作用最大数设置为10。
1.7 Metascape数据库
Metascape是一个基于40多个独立的知识库提供基因注释、富集分析的网站[12]。将TRIP13共表达基因导入Metascape数据库,进行基因本体(gene ontology,GO)功能富集分析和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,参数设置如下。“Min Overlap”:3;“P Value Cutoff”:0.01;“Min Enrichment”:1.5。
2 结果
2.1 TRIP13在常见恶性肿瘤中的表达情况
应用Oncomine数据库检索TRIP13在各种肿瘤中的表达情况,结果显示,共有354项研究结果,106项研究结果显示肿瘤组织和正常组织中TRIP13表达情况比较差异有统计学意义,其中TRIP13高表达的研究95项,TRIP13低表达的研究11项,TRIP13在大部分肿瘤中表达水平升高。(图1)

图1 Oncomine数据库中TRIP13在不同肿瘤中的表达情况
2.2 TRIP13在软组织肉瘤中的表达情况
本研究应用Oncomine数据库共检索出10个显示TRIP13在软组织肉瘤组织和正常组织中表达水平差异具有统计学意义的数据集,主要包括黏液纤维肉瘤、多形性脂肪肉瘤、去分化脂肪肉瘤、黏液样/圆形细胞脂肪肉瘤、平滑肌肉瘤、纤维肉瘤、恶性纤维组织细胞瘤等(表1)[13-14]。10个数据集的荟萃分析显示,与正常组织相比,TRIP13在软组织肉瘤中呈显著高表达(P=1.20×10-4),中位表达数值为346.5。应用GEPIA2数据库进行验证分析,结果显示,与正常组织相比,TRIP13的表达水平在软组织肉瘤组织中升高。基于CCLE数据库的结果,在39种不同肿瘤的细胞系中,TRIP13的表达水平仍以升高为主,其中软组织肉瘤细胞系中TRIP13的表达水平排第12位(图2)。TRIP13在软组织肉瘤组织和细胞中表达水平均升高。

表1 TRIP13在不同类型软组织肉瘤中的表达
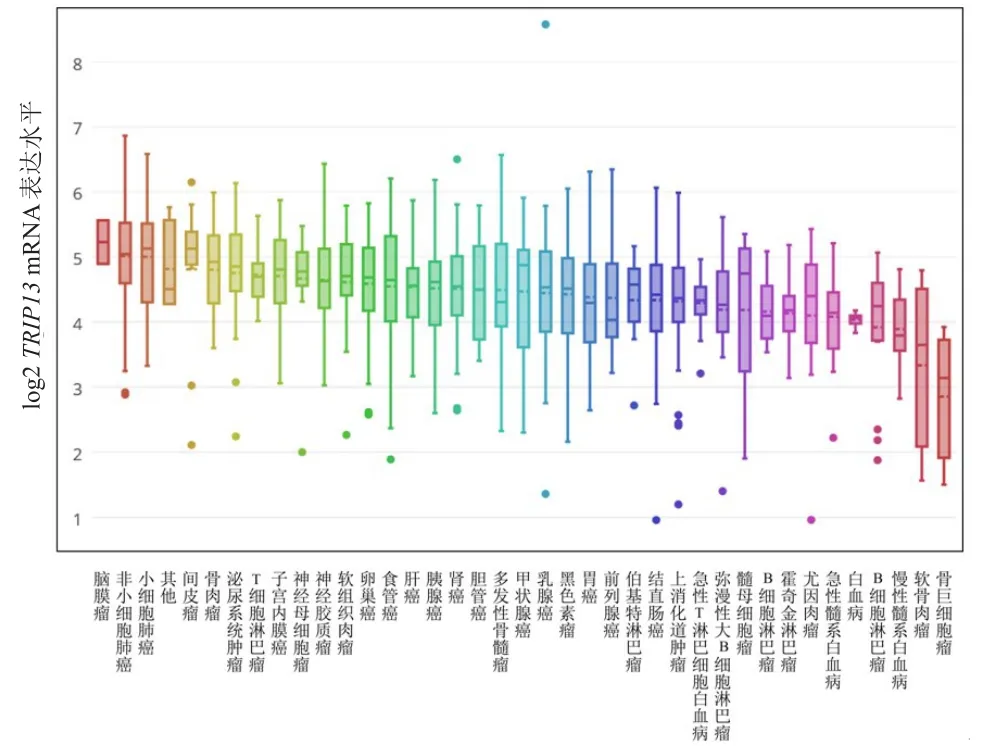
图2 CCLE数据库中TRIP13在不同肿瘤细胞系中的表达差异
2.3 TRIP13表达与软组织肉瘤患者预后的关系
应用Kaplan-Meier Plotter数据库对软组织肉瘤患者进行生存分析。在259例患者的OS数据中,TRIP13高表达组(n=139)患者的中位OS为60.83个月,短于TRIP13低表达组(n=120)患者的82.13个月,差异有统计学意义(HR=1.58,P=0.028)(图3A)。在152例患者的DFS数据中,TRIP13高表达组(n=115)患者的DFS上四分位数为11.00个月,短于TRIP13低表达组(n=37)患者的49.33个月,差异有统计学意义(HR=2.31,P=0.013)(图3B)。
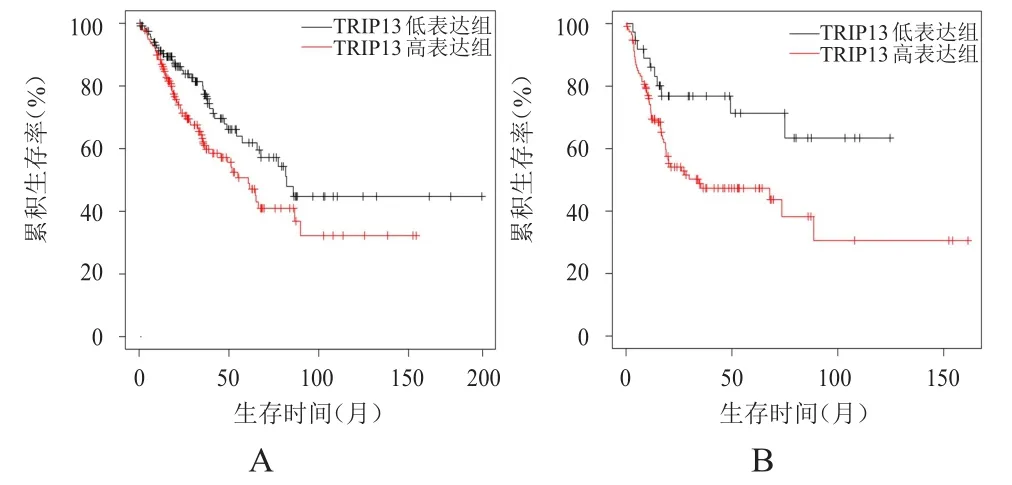
图3 Kaplan-MeierPlotter数据库分析TRIP13对软组织肉瘤患者OS和DFS的影响
2.4 TRIP13突变情况及与软组织肉瘤患者预后的关系
应用cBioPortal数据库探索TRIP13在软组织肉瘤患者中的突变情况,结果显示,51例(21%)患者发生TRIP13基因突变,包括扩增、缺失和mRNA升高,其中mRNA升高的比例最高(图4A、4B)。TRIP13突变组(n=51)患者的中位OS明显短于TRIP13未突变组(n=200)(P=8.158×10-5),TRIP13突变组患者的中位DFS明显短于TRIP13未突变组(P=4.4×10-6)(图4C、4D)。

图4 cBioPortal数据库分析软组织肉瘤患者中TRIP13突变情况及与预后的关系
2.5 TRIP13的蛋白质相互作用网络
应用String网站绘制TRIP13的蛋白质相互作用网络,共有11个节点,45条边,与TRIP13相互作用的蛋白包括:DNA修复蛋白RAD51,减数分裂重组蛋白SPO11,BUB1有丝分裂检查点丝氨酸/苏氨酸蛋白激酶B(BUB1 mitotic checkpoint serine/threonine kinase B,BUB1B),极光激酶B(aurora kinase B,AURKB),细胞分裂周期5样蛋白(cell division cycle 5 like,CDC5L),细胞分裂周期蛋白 20(cell division cycle 20,CDC20),驱动蛋白家族成员11(kinesin family member 11,KIF11),细胞周期蛋白依赖性激酶1(cyclin dependent kinase 1,CDK1),细胞周期蛋白A2(cyclin A2,CCNA2),有丝分裂阻滞缺陷 2样蛋白 1(mitotic arrest deficient 2 like 1,MAD2L1)。(图5)
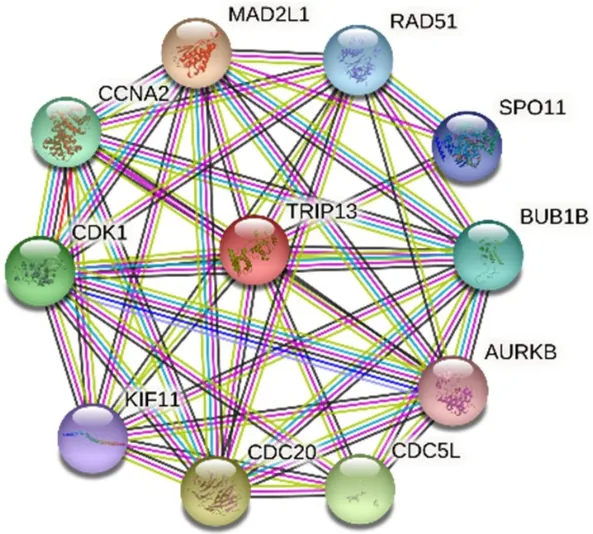
图5 String数据库中绘制TRIP13的蛋白质相互作用网络
2.6 TRIP13相关的GO功能富集分析和KEGG通路富集分析
为进一步探究TRIP13在软组织肉瘤中的潜在分子机制,应用Metascape数据库对与TRIP13相互作用最密切的蛋白进行GO功能富集分析和KEGG通路富集分析。GO功能富集分析结果显示,生物学过程(biological process,BP)包括细胞周期检查点、细胞分裂、蛋白质定位到染色体、组蛋白磷酸化、减数分裂、DNA修复,细胞成分(cell component,CC)主要包括纺锤体、染色体区、细胞质核周区、转移酶复合体,分子功能(molecular function,MF)主要包括激酶活性、ATP酶活性、蛋白激酶活性。KEGG通路主要包括细胞周期、人T细胞白血病病毒1感染、黄体酮介导的卵母细胞成熟、卵母细胞减数分裂、病毒致癌作用。(图6)
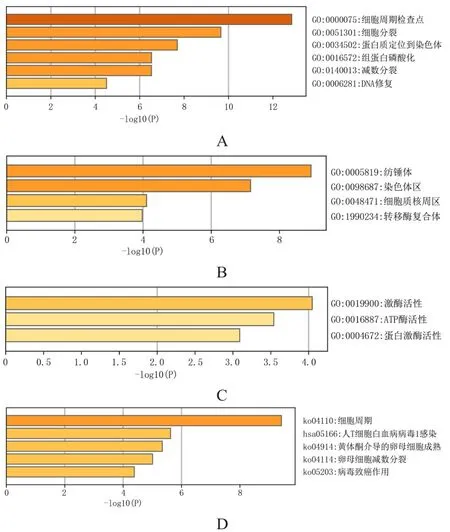
图6 Metascape数据库中GO功能富集分析和KEGG通路富集分析
3 讨论
软组织肉瘤的组织类型较多且具有异质性,目前缺乏有效的诊断和治疗手段,随着分子生物学的发展,一些与软组织肉瘤相关的分子如锌指蛋白1并列体(juxtaposed with another zinc finger protein 1,JAZF1)、多梳蛋白 SUZ12、14-3-3蛋白质ε(14-3-3 protein epsilon,YWHAE)、NUT家族成员2(NUT family member 2,NUTM2)、碳酸酐酶Ⅸ(carbonic anhydraseⅨ,CAⅨ)相继被发现[15-16]。通过研究这些分子与软组织肉瘤发生发展的关系,进一步寻找生物靶标,能够对软组织肉瘤的诊断和治疗提供帮助,有利于改善患者的预后。
TRIP13通过调节SAC的活性维持染色体基因组的稳定性,在有丝分裂染色体的分离中具有至关重要的作用[17]。而染色体的不稳定性可以使细胞具有更多种类的体细胞拷贝数改变(somatic copy number alteration,SCNA),促进肿瘤的发生发展[18]。研究发现,TRIP13的表达水平与多种肿瘤密切相关。Zhang等[19]研究表明,TRIP13能够与YWHAZ相互作用,通过调节G2/M期转化和上皮-间充质转化(epithelial-mesenchymal transition,EMT),促进结直肠癌细胞的增殖、侵袭和迁移。另一研究发现,微小RNA(microRNA,miRNA)-515-5p可能通过靶向TRIP13抑制前列腺癌细胞增殖、迁移和侵袭[20]。
然而,目前关于TRIP13在软组织肉瘤中表达情况的研究较少。本研究首先通过Oncomine数据库获得TRIP13在多种肿瘤中的表达数据,结果显示,TRIP13在多种肿瘤中扮演癌基因的角色,尤其是在软组织肉瘤中,有10个具有统计学意义的数据集。GEPIA和CCLE数据库验证了这一观点。然后应用Kaplan-Meier Plotter数据库分析了TRIP13表达水平与软组织肉瘤患者预后的关系,结果表明,TRIP13高表达的患者具有较短的OS和DFS,与TRIP13是癌基因的观点一致。由于基因组学的改变是软组织肉瘤的特征,本研究又通过cBioPortal数据库探讨了TRIP13突变对软组织肉瘤患者预后的影响,结果显示,TRIP13在软组织肉瘤患者中的突变率为21%,其中mRNA升高的比例最高,并且TRIP13突变患者的预后更差,提示TRIP13可能是判断软组织肉瘤预后的新靶标。为了进一步研究软组织肉瘤的发病机制,本研究应用String和Metescape数据库分析TRIP13的共表达基因以及功能富集和通路富集。与TRIP13关系最密切的是RAD51,RAD51是一种腺苷三磷酸酶,可在单链DNA上形成核蛋白丝。RAD51通过调节DNA双链断裂修复、复制应激和减数分裂维持DNA复制的保真度[21]。准确的DNA修复和复制对于基因组稳定性和肿瘤预防至关重要。有研究证实,在长期暴露于铬颗粒的肺癌细胞中,RAD51发生同源重组,导致DNA双链断裂,染色体不稳定,促进肺癌的发生[22]。说明TRIP13可能主要通过影响肿瘤细胞的减数分裂过程,改变细胞的增殖、分化和凋亡过程。
本研究也具有一定的局限性,首先,本研究通过生物信息学方法分析了TRIP13在软组织肉瘤中的表达水平及与预后的关系,但由于应用来自多个不同数据库的数据,很难保证不同数据库之间的统一性。其次,由于数据库中缺少软组织肉瘤临床分期和病理类型数据,未研究TRIP13在不同临床分期和病理类型软组织肉瘤中的作用,这需要收集标本进一步研究证实。最后,没有利用基础实验验证这些数据库中的数据,这将在未来的研究中完成。后续可以继续验证TRIP13的表达与组织亚型、转移、病理分级的关系,进行相应的基础实验,探究软组织肉瘤的发病机制。
综上所述,TRIP13可能在软组织肉瘤的发生发展中发挥关键作用,有望成为软组织肉瘤诊断和治疗的新靶标。
