水稻染色质重塑因子OsINO80通过调节植物激素水平参与避荫反应
2022-07-05王培亮尹刘翻
王培亮,尹刘翻,俞 瑜
(1. 复旦大学 生命科学学院,上海 200438; 2. 复旦大学 遗传工程国家重点实验室,上海 200438)
水稻作为世界最重要的粮食作物之一,是全世界超过半数人口的主食来源。在农业生产中为了提高产量往往会选择提高种植密度,但是高密度的种植会让植物之间相互遮挡并争夺阳光,上层植物可以吸收红光和蓝紫光,并将远红光反射回去[1],下层植物获取到的总光强降低,蓝光光强衰弱,光合作用有效辐射(Photosynthetically Active Radiation, PAR)值、蓝光/绿光比值和红光(red light, R)/远红光(far-red light, FR)比值(R/FR)都会有所降低,这种光环境我们称之为荫影环境(shade)[2-3]。植物在面对荫影条件时,进化出了一系列完整的机制来减小荫影的影响,该机制被称之为避荫反应。短期的荫影条件下,植物会出现叶柄伸长、侧枝减少、节间伸长和叶片夹角减小等表型。长期的荫影条件下,植物会出现开花提前和减少叶绿素合成等表型。除此以外,长期荫影条件还可能会影响植物的抗病性[4-7]。
植物通过精细的光受体系统来感知和响应光环境的变化。阳光中的红光和远红光(600~750 nm)主要是由phyA到phyE这5个光敏色素[8]感知,蓝光(350~500 nm)的受体主要是以下3种: 隐花色素(cry)、向光素(phototropins)和ZTL蛋白[9]。各种光受体在植物生长发育中或协同或拮抗,共同将光信号感知并转导至下游的光敏色素互作因子Phy-Interacting Factors(PIFs)和负调控因子Constitutive Photomorphogenic 1(COP1),进而调控植物内激素水平和相关基因的表达来改变植物的生长发育以便更好地适应环境[10]。PIFs是光信号转导途径中一种重要的组分,在光敏色素介导的光信号转导途径中起中枢作用。许多研究表明PIFs与植物激素信号有协同关系[11]。在避荫反应中,光受体phyB感知到R/FR比值低的光信号,然后其光的稳态平衡被打破,phyB活性减低,促使PIFs转导光信号调控植物激素合成基因或下游避荫反应相关基因的表达。在此过程中包括生长素(IAA)、赤霉素(GA)、乙烯(ETH)以及油菜素内酯(BR)等的植物内源激素都在代谢或运输途径上受到了调控,各类激素或协同或拮抗,共同调控植物表现为更适宜外部环境的表型: 如下胚轴和叶柄伸长、叶片夹角变小等,以获得更多的阳光。
在拟南芥中,表观遗传因子也参与植物的光信号转导。光敏色素互作因子PIF7招募H3K36me3识别蛋白morf-related gene 1/2(MRG1/2)到下游基因特异区域(G-box)在组蛋白乙酰转移酶的协助下通过提高相关基因组蛋白乙酰修饰水平,进而促进避荫反应应答基因的转录[12]。组蛋白乙酰转移酶TBP-associated factor1(TAF1)/HAF2和General Control Nonderepressible 5(GCN5)是拟南芥中光调控生长和相关基因表达所必需的,GCN5也与光响应启动子存在直接相关性[13]。而最近研究表明染色质重塑因子AtINO80通过调控HY5的转录抑制拟南芥光形态建成过程[14]。
染色质重塑因子(chromatin remodeling factor)是一类重要的表观遗传因子,它依赖水解ATP释放的能量,然后通过驱使核小体位置的改变,即改变核小体的间距和排列,从而改变染色质的结构完成与DNA相关的各种生理功能[15-17]。染色质重塑因子INO80最初在酵母中纯化并鉴定,后被证实广泛参与真核生物并影响多种生物学功能。在动物中可以调控基因转录和DNA复制,并存在依赖ATP进行DNA损伤修复和维持染色质稳定性的功能[18];在植物模式生物之一的拟南芥中,INO80被发现参与了其生长发育的各个阶段,从细胞增殖和复制到花期的调控,再到维持基因组的稳定性,都发挥了重要的作用[19];而在重要的粮食作物水稻中有关INO80的研究还较少,其作为单子叶植物的模式生物,极具研究价值。近年来,有文章报道OsINO80在水稻的不同组织中有着广泛的表达,可以通过影响GA的合成来调控株高等表型,推测在众多生物学过程中发挥十分重要的作用,而且水稻中无法获得纯合的OsINO80T-DNA插入突变体,其纯合突变会致死,这表明它在水稻中的功能可能更强[20]。
虽然双子叶植物拟南芥中表观遗传因子调控光响应基因的报道较多,但是在单子叶植物水稻中表观遗传因子是否参与光响应通路的过程还不是很清楚。在本研究中我们发现水稻染色质重塑因子OsINO80一个很有意思的功能: 它可以参与避荫反应。我们通过模拟自然环境中的荫影条件,发现OsINO80敲减植株对荫影环境不敏感,通过构建和分析高质量转录组数据我们探索了OsINO80参与避荫反应的机制。OsINO80是避荫反应的正调控因子,在荫影环境时,促进了暗形态建成相关基因的表达,同时抑制光形态建成相关基因的转录。进一步表明,OsINO80主要调控避荫反应中植物激素代谢相关基因的转录水平。
1 材料和方法
1.1 材料
本课题研究中所使用的水稻材料均来自于粳稻品种日本晴亚种(OryzasativasspjaponicacvNipponbare)。水稻转基因材料OsINO80敲减植株(RNAi-1/RNAi-2)和OsINO80过表达(OE-1/OE-2)植株来自本课题组[20]。转录组高通量测序(RNA-seq)由上海晶能生物有限公司完成。
1.2 方法
1.2.1 转基因植株荫影条件下的表型分析
将野生型(NIP)、OsINO80敲减植株(RNAi-1/RNAi-2)和其过表达植株(OE-1/OE-2)的种子铺排在在半固体培养基(0.4%琼脂)上,28 ℃连续白光下生长1 d,然后继续在白光下或者转移到模拟遮荫条件(红光: 16 μE·m-2·s-1;远红光: 48 μE·m-2·s-1;蓝光: 1.5 μE·m-2·s-1)下生长4 d,共5 d[21]。每组选取20株长势一致的幼苗,测量并统计幼苗的胚芽鞘长度、第一节长、全长,并进行两次重复实验。
1.2.2 RNA提取
取5棵模拟遮荫条件下生长5 d的NIP或OsINO80、RNAi-2的幼苗,迅速放入液氮中,用研钵研磨成粉末后,取用北京天根生化有限公司生产的RNAprep pure植物总RNA提取试剂盒提取RNA。每个基因型植株准备3个重复。
1.2.3 RNA-seq文库构建
提取好的RNA用KAPA stranded mRNA-seq Kit试剂盒进行链特异性RNA-seq建库。实验步骤按照试剂盒(Illumina®Platforms, KR0960-v5.17)说明书(见www.kapabiosystems.com)进行。每个基因型3个重复。先用贝克曼库尔特公司(Beckman Coulter, USA)生产的带有oligo(dT)的RNA clean XP纯化磁珠对10 μg总RNA进行mRNA富集。然后用加热的方式把mRNA打断成200 bp左右的片段,以此为模版用随机引物合成第一链cDNA,加入RNase H使其产生缺口,RNA会在缺口处不断延伸后生成第二链cDNA。之后对双链cDNA进行纯化、末端修复以及3′末端加尾,再连接上测序接头。
最后将得到的文库DNA进行PCR扩增和纯化,用琼脂糖凝胶电泳选取250~500 bp大小的片段进行回收。每个基因型选取两个重复,文库送到上海晶能生物有限公司用Illumina Hiseq X测序仪进行双端150 bp读长的高通量测序。
1.2.4 RNA-seq数据分析
首先用软件FastQC V0.11.7[22]检测原始数据的质量。然后用Cutadapt V2.9[23]去除测序接头和质量低于20的碱基,过滤短于20 bp的序列得到高质量的reads用于后续分析。而后我们用软件HISAT2 V2.1.0[24]将数据比对到国际水稻基因组(MUS7)上,用SAMtools V1.9[25]筛选出比对质量高于20的reads并计算reads的数量。FeatureCounts v2.0.0[26]用来计算基因上的原始reads数目。DEseq2 V1.22.2[27]将原始reads标准化并找到差异表达基因。每个基因的转录水平用FPKM(Fragment Per Kilometer Per Million Reads)值表示,我们以|log2(Fold Change)|≥log2(1.5)和Padj(调整后的P)≤0.05为联合标准鉴定差异表达基因。
DeepTools v1.3[28]软件被用来生成bigwig文件并形成reads相关性热图。Bigwig文件的可视化在IGV v2.4.0[29]软件上完成。
1.2.5 基因本体论(GO)富集分析
GO富集分析采用的是线上水稻功能注释工具CARMO(http: ∥bioinfo.sibs.ac.cn/carmo/Gene_Annotation.php)[30]。我们以P≤0.05为标准筛选出基因显著富集的生物学过程用于后续分析。
2 结果与分析
2.1 转基因植株荫影条件下的表型分析
比较白光和在模拟遮荫环境下生长的野生型(NIP)幼苗,发现遮荫生长使幼苗的胚芽鞘和第一节明显伸长,整株也表现为伸长。但是,OsINO80敲减植株(RNAi-1和RNAi-2)在遮荫条件下的胚芽鞘、第一节以及整株伸长和白光下相比都没有明显变化;与之相反,OsINO80过表达水稻(OE-1,OE-2)株系的胚芽鞘、第一节和整株伸长却更显著(图1)。这些结果说明染色质重塑因子基因OsINO80的转录表达能够促进幼苗在避荫条件下的下胚轴和第一节生长,暗示了OsINO80可能正向调控避荫反应。
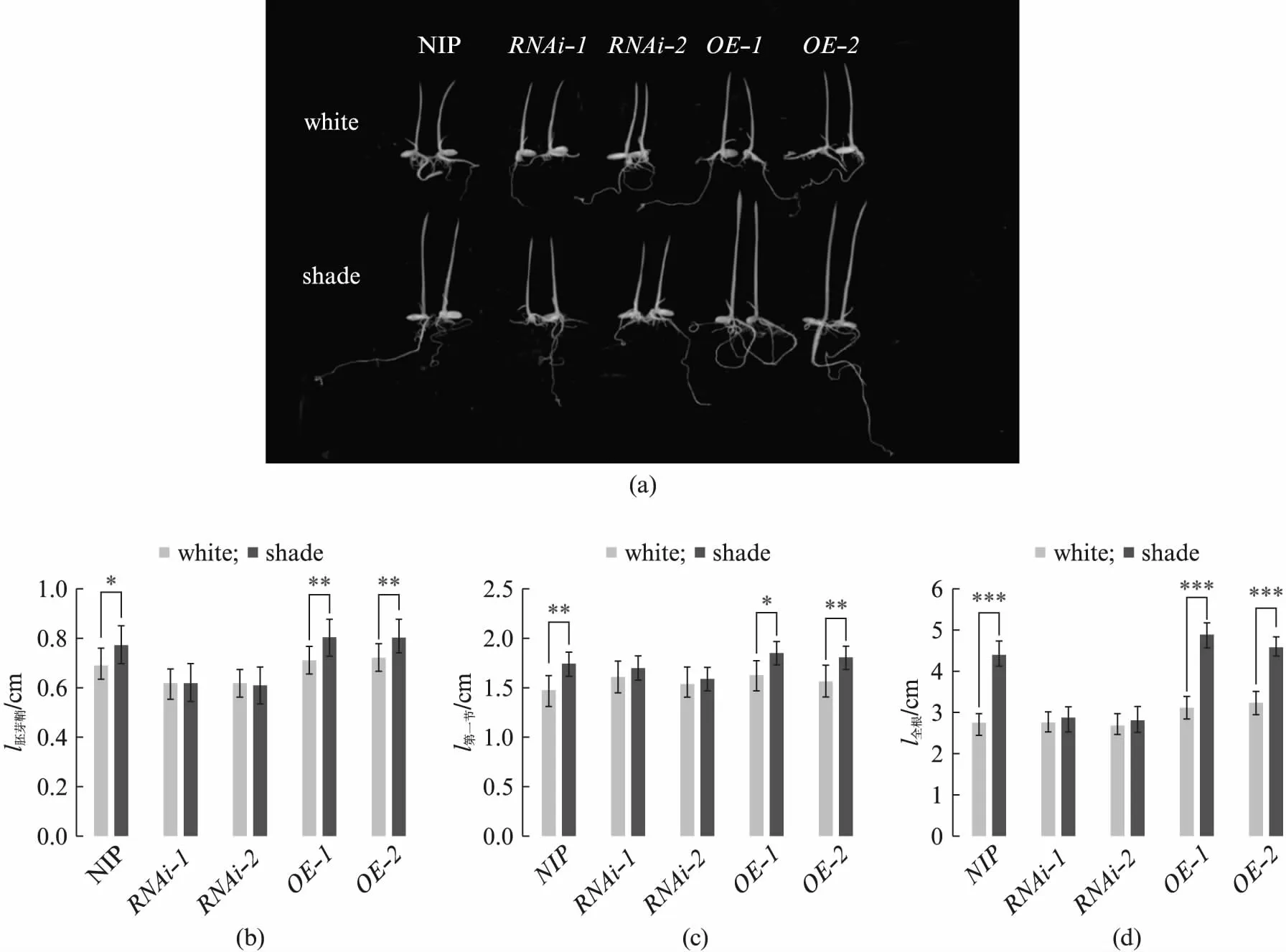
图1 OsINO80敲减植株和过表达的避荫反应表型分析Fig.1 Phenotypic analysis for shade avoidance of OsINO80 RNAi and OsINO80 overexpression(a) 野生型(NIP)、OsINO80敲减植株(RNAi-1/RNAi-2)和OsINO80过表达水稻(OE-1/OE-2)分别在白光条件和荫影条件生长5 d的幼苗的不同表型,Scale bar: 2 cm;(b) 胚芽鞘(coleoptile)长度;(c) 第一节(the first leaf)长;(d) 全长(每组20株幼苗),根据Student’s t检验,野生型与突变体上的星号*表示差异有统计学意义(*代表P<0.05;* *代表P<0.01;* * *代表P<0.001)。
2.2 高质量RNA-seq数据的获取
为进一步研究OsINO80在避荫条件下的功能,我们构建了转录组数据测序文库。文库的PCR扩增片段经电泳后发现,在250~500 bp处有清晰且明亮的条带,DNA片段大小吻合且浓度较高(图2(a)),初步证明建库成功。之后我们选取250~500 bp的DNA片段割胶回收并将前两个重复送样进行高通量二代测序。拿到原始数据后,我们筛选出碱基质量较高的reads,将其与国际水稻基因组(MUS7)转录组数据进行比对分析。4个样本的原始reads都有着超过3×107的reads,测序深度超过了10倍的基因组。而且所有样本比对到基因组上的reads率都超过85%(图2(b))。
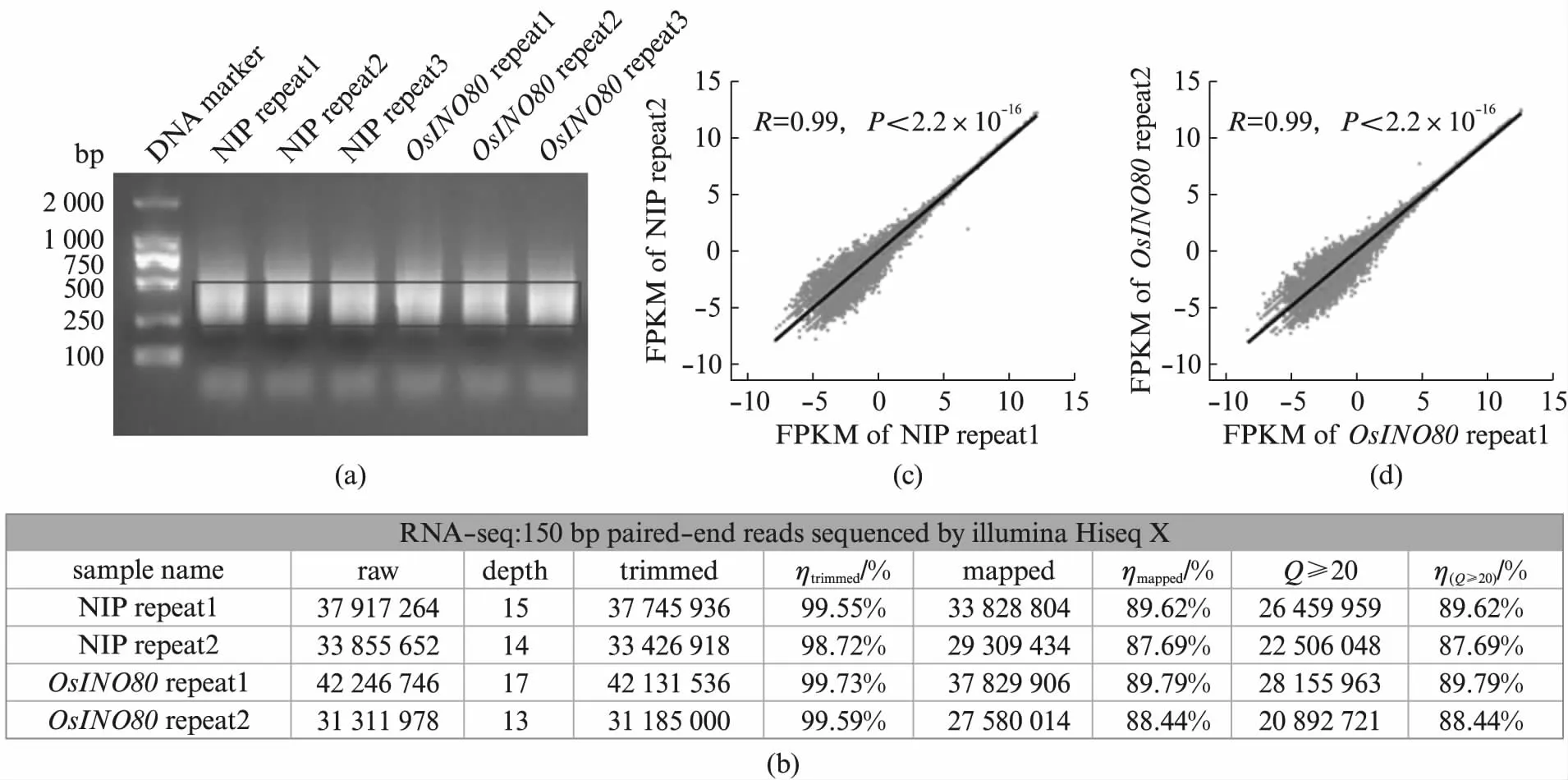
图2 OsINO80高质量建库数据的获取Fig.2 Obtaining of high-quality library of RNA-seq data for OsINO80(a) 文库扩增后的DNA琼脂糖凝胶图,红框内为进行后续回收的目的条带;(b) 测序数据的reads数目统计。raw是原始reads数目,depth是原始数据的测序深度,trimmed是去除低质量碱基和接头后的reads数目,ηtrimmed是去除低质量碱基和接头后的reads数目占原始reads数的百分比,mapped是比对到基因组上的reads数目,ηmapped是比对到基因组上的reads数目占原始reads数的百分比,Q≥20是比对到基因组上的质量高于20的reads的数目,η(Q≥20)是比对到基因组上的质量高于20的reads数占原始reads数的百分比;(c,d) NIP和OsINO80敲减植株的两个重复之间的相关性。横坐标为重复1中各基因的转录水平,纵坐标为重复2中各基因的转录水平,黑线的斜率为1,是两次重复中基因转录水平相同的位置,R代表斯皮尔曼相关性系数,P代表相关性的显著性。
之后我们计算了基因的表达水平,用FPKM值进行标准化。NIP和OsINO80敲减植株的两个重复中基因的表达量都有较高的相关性(NIP和OsINO80敲减植株的两个重复的斯皮尔曼相关性系数R=0.99,P<2.2×10-16)(图2(c,d))。这些数据表明该转录组数据的重复性较好,是稳定可靠的。
2.3 荫影条件下OsINO80敲减植株的差异表达基因分析
对RNA-seq数据进行进一步的分析。首先查看了OsINO80本身的转录水平,在OsINO80敲减植株中,基因OsINO80的表达量确实存在着显著的下降,而附近其他的基因的表达量并没有明显变化(图3(a),见第264页),验证了材料的准确性以及建库的稳定性。将OsINO80敲减植株与NIP的基因表达量进行了比对,以|log2(Fold Change)|≥log2(1.5)且Padj≤0.05为标准,鉴定到了5 881个差异表达基因(Differential Expression Genes, DEGs),其中有2 923个是表达量上调基因,有2 958个是表达量下调基因(图3(b),见第264页)。
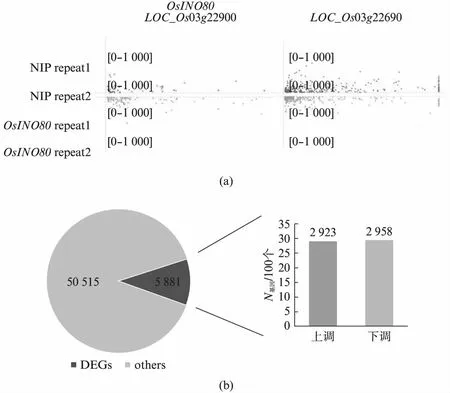
图3 NIP和OsINO80敲减植株中的基因表达水平Fig.3 Gene expression level of NIP and OsINO80 RNAi(a) OsINO80的表达量在IGV软件的展示图。左图为基因OsINO80的表达量,右图为附近随机基因的表达量,[0—1 000]是基因表达量的展示范围;(b) OsINO80敲减植株中差异表达基因情况。扇形图中DEGs是OsINO80敲减植株中的差异表达基因数目,others是其他基因数目;柱形图中红色代表表达量上调基因数目,红色代表表达量下调基因数目。
2.4 OsINO80参与调控避荫反应相关基因的表达
我们首先查阅已发表的影响避荫反应的基因[21],通过比较避荫条件和持续白光下的野生型的转录水平,找到了513个表达量上调的荫影诱导基因(shade-induced genes)和1 626个表达量下调的荫影抑制基因(shade-repressed genes)(图4(a))。我们通过GO分析研究了这两类基因参与的生物学过程。从结果中我们可以看出,荫影诱导基因参与了细胞生长、生长素激活信号通路、生长素响应、植物细胞壁组织、细胞壁塑造等和植物暗形态建成相关的生物学过程,而荫影抑制基因参与了逆境响应、生物刺激响应、脱落酸响应、高光强响应、乙烯响应和光形态建成等相关的生物学过程(图4(b))。

图4 OsINO80调控避荫反应相关基因Fig.4 OsINO80 regulates genes related to shade avoidance(a) 影响避荫反应的基因中,荫影诱导基因和荫影抑制基因的数量分布;(b) 荫影诱导基因和荫影抑制基因的GO分析: 红色代表上调基因,绿色代表下调基因,其对应的横坐标为参与相关生物学过程的基因的个数;(c) OsINO80敲减植株差异表达基因与荫影诱导基因维恩图;(d) OsINO80敲减植株差异表达基因与荫影抑制基因维恩图;重叠基因的显著性水平采用Fisher’s exact test(* * *代表P<0.001);(e,f) 图4(c,d)中重叠基因的火山图,x轴是-lg Padj,代表基因表达量变化的显著性,y轴是log2(Fold Change),代表基因表达量变化倍数。红色点为上调基因,绿色点为下调基因。
我们进一步研究OsINO80敲减植株中的差异表达基因和避荫反应相关基因[21]的关系,结果显示在OsINO80敲减植株中有159个差异表达基因与荫影诱导基因重合(图4(c)),其中114个基因下调,45个基因上调(图4(e));同时,有534个基因差异表达基因与避荫反应中荫影抑制基因重合(图4(d)),296个基因表现为上调,238个基因下调(图4(f))。这些结果进一步暗示OsINO80参与水稻避荫反应的调控。
2.5 差异表达基因中激素通路相关基因的分析
避荫反应在植物体内受到庞大的调控网络影响,调控网络包括各类激素、多种调节转录因子和修饰蛋白等的合成及其代谢和运输过程[31]。我们在OsINO80调控的基因中发现了许多在避荫反应中起到重要作用的内源性激素通路的相关基因。赤霉素合成通路中的关键酶OsGA20ox2、OsGA3ox2,乙烯合成通路中的关键酶OsACO1、OsACO3,油菜素内酯的关键合成基因OsBRD2都在OsINO80敲减植株中显著性地下调(图5(a),见第266页)。除了合成途径,植物激素的代谢途径也受到了OsINO80的调控,促进脱落酸分解代谢的关键基因OsABA8ox2在OsINO80敲减植株中的表达量显著上调(图5(b),见第266页)。另外乙烯应答转录因子OsBIERF3和脱落酸受体OsPYL6这些激素信号转导途径相关的关键因子也在OsINO80敲减植株中显著性地下调(图5(c),见第266页)。
参与避荫反应的植物激素的上游是一系列的光受体,光受体下游的光敏色素互作因子也与植物激素协同作用、互相影响[32],因此为了进一步探究OsINO80调控这些植物激素的机制,我们查看了参与到水稻避荫反应中的光敏色素因子和光敏色素互作因子的转录水平。水稻中有3个光敏色素因子phyA、phyB和phyC,其中phyB被证实参与避荫反应[33-34],但是在OsINO80敲减植株中phyB的编码基因OsPHYB的表达量变化并不明显。phyB的下游是一类光敏色素互作因子PIFs,在水稻的PIF同源基因中,OsPIL14与拟南芥PIF4和PIF5高度同源,而且被报道可以特异性地活性状态的OsPHYB结合,参与暗形态建成过程[35]。在OsINO80敲减植株中,OsPIL14的表达量也没有发生显著性变化;有意思的是phyB的下游参与光形态建成的关键转录因子HY5,其编码基因OsHY5L1显著上调(图5(b),见第266页),但是HY5在暗形态建成中被COP1降解,作用不大[36-37]。这些结果说明OsINO80在避荫反应过程中可能直接作用于植物激素通路的基因。
3 讨 论
大多数的植物是不可以移动的,所以在整个生命周期必须在固定的区域,适应不停改变的环境。植物的正常生长发育过程是由两部分来调控的: 一方面是自身的内源信号,比如激素等,另一方面是环境信号,比如阳光、水、温度等[38]。阳光不仅是植物能量的来源,还是调控植物生长发育的环境信号。外界光条件的改变会导致植物产生不同的性状,如开花时间、气孔开闭、向光性和避荫反应等[39-41]。在本文我们报道了水稻染色质重塑因子OsINO80参与调控避荫反应。我们通过模拟自然环境中的荫影条件,发现OsINO80敲减植株出现了失去应对避荫反应的表型,结合转录组数据表明OsINO80可以调控多种植物激素的合成代谢过程来参与避荫反应。
有报道已证实水稻OsINO80参与调控了赤霉素的合成,在OsINO80敲减植株中,赤霉素合成相关基因的表达量和赤霉素含量都有显著降低,并且表型在施加外源活性GA3后可以恢复至与野生型相同,这有效地回答了激素水平与OsINO80介导的避荫反应基因表达变化之间的因果关系,赤霉素的合成确实依赖OsINO80[20]。本研究发现,OsINO80还可以调控很多的植物激素的合成代谢关键酶或者应答因子,包括脱落酸,乙烯,油菜素内酯等。众所周知,这些植物激素或协同或拮抗,在植物的生命过程中发挥了非常重要的作用,其中不仅包括植物细胞伸长、细胞壁塑造等生长过程,还有开花、衰老等发育过程,抗旱、抗高温低温等非生物胁迫的抗逆过程,抗真菌抗虫害等生物胁迫过程。这说明OsINO80通过植物激素参与的过程可能非常广泛,很有可能还存在其他未知的重要功能,值得我们进一步的探索。
在拟南芥中,AtINO80也被报道了参与光响应过程[19],这暗示染色质重塑因子INO80在植物中的功能是比较保守的。但是二者的调控机制还是有较大差异,AtINO80调控光受体下游的正向调节转录因子HY5,促进了光形态建成过程,和拟南芥中不一样,水稻OsINO80并不调控光受体和光敏色素转录因子PIFs,而是直接影响了下游的众多参与避荫反应的植物激素的合成。这也暗示水稻中OsINO80调控暗形态建成的过程可能更加复杂。而且在水稻中研究光响应过程更具有现实意义,因为水稻是粮食作物,为了增加产量而密集种植必然会涉及到避荫反应,我们的工作可以为农业生产提供一定程度上的指导。
除了暗形态建成,水稻OsINO80可能也参与了植物光形态建成过程。OsINO80敲减植株中,光形态建成正向调节因子HY5其合成基因OsHY5L1有着显著性的上调。与这个结果一致的是,拟南芥中HY5也被报道在光形态建成中受到了AtINO80的抑制。因此OsINO80在水稻中可能也参与了光形态建成过程。对应至表型,光形态建成促进因子的表达量显著上调导致突变体提前光形态建成也可能是OsINO80敲减植株对避荫反应不敏感的一个原因。有报道认为HY5在避荫反应下会被降解,低程度荫影环境还会触发负调控回路,恢复至未遮荫状态[42]。因此,下一步HY5的蛋白量的检测或许可以帮助我们更深地认识这个问题。
