三角鲂MHCⅠα基因全长cDNA克隆与生物信息学分析
2022-06-29马恒甲
刘 凯,谢 楠,郭 炜,马恒甲
(杭州市农业科学研究院 水产研究所,浙江 杭州 310024)
主要组织相容复合体(major histocompatibility complex,MHC)是一类具有高度多态性的分子,其可调控脊椎动物的组织移植和免疫反应。如西域若花鳉()鱼鳞移植的成功率与其基因位点的匹配率紧密相关,基因表达水平在病毒感染的虹鳟()性腺细胞中显著上升。由于在调节先天免疫和适应性免疫中的重要作用,基因受到免疫学家的广泛关注。根据结构和功能不同,MHC可分为Ⅰ类、Ⅱ类和Ⅲ类。其中,MHCⅠ类蛋白质分布于几乎所有有核细胞上,具有向CD8T细胞呈递细胞内抗原(例如病毒)的功能;MHCⅠ首先结合被细胞内蛋白降解的小肽并转移至细胞膜,然后再将小肽呈递至CD8T细胞,激活细胞毒性T细胞。MHCⅡ类蛋白质主要分布在抗原呈递细胞的表面,如树突状细胞、巨噬细胞等,主要负责呈递外源性抗原,以激活特异性CD4T辅助细胞。而MHCⅢ类蛋白质主要是一类与免疫调节相关的蛋白质,负责编码一些与补体系统和炎症反应等相关的免疫蛋白,该类蛋白质不具备抗原提呈功能,因而研究较少。由于Ⅰ是脊椎动物适应性免疫的重要基因之一,该基因已先后从多种鱼类中被克隆出来。MHCⅠ首先在鲤()上被发现,随后第一条Ⅰ序列被克隆出来,然后在各种鱼类中克隆出了越来越多的Ⅰ基因,如细鳞鲑()、大西洋鲑()、斑马鱼()、虹鳟、草鱼()和团头鲂()等。
三角鲂()属于鲤科(Cyprinidae)鲂属()鱼类,历史上其广泛分布在黑龙江、黄河和长江等流域。近年来,由于大量过度捕捞,加上水域生态环境等因素的变化,上述流域的三角鲂野生资源量日益下降,全国仅钱塘江流域建有国家级的三角鲂原种场。三角鲂具备生长快、病害少、养殖效益高、成活率高、肉质好、经济价值高等优势,已成为浙江省主要特色名优淡水养殖品种之一,并已形成一定的养殖规模。目前,对于三角鲂繁育、养殖技术的研究较多,而相关功能基因研究缺乏。已有研究表明,鱼类Ⅰ基因在组织中广泛分布,在抗感染免疫中具有重要作用。前期三角鲂与团头鲂抗嗜水气单胞菌()感染的比较转录组学分析结果提示,Ⅰ基因在三角鲂抗嗜水气单胞菌感染中具有重要作用(相关数据未发表),但至今未见有关三角鲂Ⅰ基因相关研究报道。本研究基于同源序列比对,开展了三角鲂Ⅰ基因cDNA全长克隆,并基于生物信息学方法对其结构特征与理化性质,以及转录组表达进行了分析,以期为其生物学功能研究提供参考。
1 材料与方法
1.1 实验材料
实验鱼为钱塘江三角鲂,保存在国家级钱塘江三角鲂原种场。采用断尾法将麻醉后的三角鲂放血,用剪刀剪开腹部,在肝部位取约3 g组织,切成厚度<5mm的小块后浸泡于无液氮RNA样品保存液(北京百泰克生物技术有限公司)中,并置于-80 ℃冰箱中保存备用。
1.2 引物设计与合成
根据GenBank数据库中团头鲂(登录号为HM151020)、草鱼(登录号为AB109779)等相关物种的Ⅰ全长序列信息,利用Primer-BLAST软件(http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)进行引物设计并合成了2条简并引物(MHCⅠ-Z-NF1和MHCⅠ-Z-NR1)。用这2条引物进行Ⅰ核心片段扩增,并以此核心片段为基础进行3′ RACE和5′ RACE扩增,设计合成4条特异性引物(MHCⅠ-3-GSP1, MHCⅠ-3-GSP2, MHCⅠ-5-GSP1和MHCⅠ-5-GSP2)。同时,根据TAKARA公司3′和5′RACE扩增试剂盒使用说明,合成4条通用引物用于Ⅰ基因3′ RACE和5′ RAC扩增(3′ RACE: Outer Primer和Inner Primer; 5′ RACE: Univers Primer和Short Primer),引物序列见表1。

表1 三角鲂MHCⅠα基因cDNA全长克隆使用的引物序列
1.3 基因克隆
按照TRIzol试剂(Invitrogen公司,美国)说明书提取三角鲂肝总RNA;利用HiScript II 1st Strand cDNA Synthesis Kit(南京诺唯赞生物科技股份有限公司)进行cDNA第一链的合成。以反转录的cDNA为模板,利用Phanta Max Super-Fidelity DNA Polymerase(南京诺唯赞生物科技股份有限公司)进行核心片段扩增,利用3′-Full Race Core Set with PrimeScript RTase[宝生物工程(大连)有限公司]和SMARTer RACE 5′/3′ Kit[宝生物工程(大连)有限公司]进行二轮巢式3′ RACE和5′ RACE扩增。反应体系参照试剂盒说明书设置。核心片段扩增采用常规PCR方法,PCR反应条件为:95 ℃ 15 s,55 ℃ 15 s,72 ℃ 30 s,35个循环;72 ℃延伸5 min。RACE扩增采用巢式PCR方法,使用Ⅰ特异性扩增引物和试剂盒通用引物进行。PCR反应条件参照试剂盒说明进行,其中3′ RACE第1轮扩增退火温度(annealing temperature, Ta)设置为56 ℃,3′ RACE第2轮扩增Ta设置为59 ℃,5′ RACE第1轮扩增Ta设置为65 ℃,5′ RACE第2轮扩增Ta设置为62 ℃。扩增产物经1%琼脂糖凝胶电泳检测后,将目的条带切胶回收,连接到pMD19-T载体上,转化大肠埃希菌() DH5α菌株进行TA克隆并进行测序,测序工作由上海派森诺生物科技有限公司完成。使用UGENE v38.1软件的CAP3组件进行拼接,获得Ⅰ的cDNA全长。
1.4 生物信息学分析
利用UGENE v38.1软件分析Ⅰ基因开放阅读框(open reading frame,ORF)并将其翻译为相应的氨基酸序列;利用CDD在线工具(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析蛋白质的保守结构域;Ⅰ基因编码蛋白质的二级结构、三级结构、信号肽、磷酸化、-糖基化,-糖基化位点和跨膜区域预测参照张普等的方法进行;利用COILS在线工具(https://embnet.vital-it.ch/software/COILS_form.html)对蛋白质卷曲螺旋区域进行预测;利用Protcomp 9.0在线工具(http://www.softberry.com/berry.phtml?topic=protcompan&group=programs&subgroup=proloc)对蛋白质进行亚细胞定位;利用ProtParam和ProtScale在线工具(https://web.expasy.org/protparam/, https://web.expasy.org/protscale/)对蛋白质的基本理化性质进行预测和分析,如氨基酸数量、分子量、等电点等,以及蛋白质亲/疏水性。
1.5 BLAST比较与系统进化分析
利用NCBI网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)的BLAST模块进行同源比对。基于Ⅰ编码的氨基酸序列,利用PREQUAL软件对非同源位点遮盖后,使用UGENE v38.1软件的MAFFT组件进行序列比对,并使用Jalview软件进行可视化呈现,再利用Divvier软件对序列进行对齐,使用IQ-TREE软件基于最大似然法构建三角鲂与其他物种的系统进化树,替代模型选择JTT+F+R4,进化树的可靠性通过SH-aLRT检验和Ultrafast Bootstrap法进行检验(重复1000次)。
1.6 转录组表达分析
细菌攻毒实验参照方献平等的方法,仅取样时间和取样组织不同,选取了0、3、24 h 3个时间点,每个时间点取三角鲂5尾鱼,每尾鱼分别取肌肉、肝、血液3个样品等量混合后作为实验材料,浸泡于RNA保存液中,于-80 ℃冰箱中保存备用。RNA提取与建库测序委托杭州英睿生物科技有限公司完成。测序比对、表达量计算等步骤参照刘凯等的方法。利用BLAST软件,以测序获得的Unigens为模板,将克隆获得的三角鲂Ⅰ基因序列进行BLAST比对,从Unigens中鉴定出Ⅰ基因并进行表达量分析,表达量以FPKM (fragments per kilobase million)值表示。
2 结果与分析
2.1 MHCⅠα基因cDNA克隆与序列分析
利用简并引物,通过PCR扩增,获得Ⅰ核心扩增片段587 bp(图1-A)。根据此基因序列,重新设计特异性引物,联合RACE通用引物,分别进行3′ RACE和5′ RACE基因片段的扩增(图1-B、1-C),产物经琼脂糖凝胶电泳检测后进行TA克隆、测序,使用UGENE v38.1软件对测序片段进行拼接后获得Ⅰ基因cDNA全长。根据Klein等提出的MHC命名规则,该Ⅰ基因被命名为Mete-UAA,GenBank登录号为MW759850。Mete-UAA序列全长2 102 bp,包括72 bp的5′端非编码区(5′ untranslated region, 5′ UTR),986 bp的3′端非编码区(3′ untranslated region, 3′ UTR),1 044 bp的编码序列(coding DNA sequence, CDS),包含347个氨基酸密码子和1个终止密码子TAA。在Ⅰ基因的3′ UTR末端含有1个Poly(A)的典型信号序列(AAAAAAAAAAAA)。在MHCⅠα氨基酸序列中含有3个胞外结构域(alpha结构域)、1个跨膜区域(TM/CY区)和1个前导肽(信号肽)(图2)。

A,MHCⅠα基因核心片段;B,3′ RACE第二轮扩增后片断;C,5′ RACE第二轮扩增后片断。Marker,DNA标准物质。

图2 三角鲂MHCⅠα基因cDNA全长及其编码的氨基酸序列
2.2 MHCⅠα基因编码蛋白质的生物信息学分析
2.2.1 保守结构域、二级结构和卷曲螺旋区域
利用CDD在线工具对Mete-UAA氨基酸序列中保守结构域进行分析,结果表明,Mete-UAA氨基酸序列包含一个MHCⅠ超家族结构域(甘氨酸-亮氨酸,Gly-Leu)和1个免疫球蛋白(immunoglobulin, Ig)超家族结构域(缬氨酸-赖氨酸,Val-Lys),其中MHCⅠ超家族结构域由2个alpha结构域(alpha1和alpha2)折叠形成肽结合位,Ig超家族结构域则是MHCⅠα蛋白中具有Ig样结构的alpha结构域,即alpha3结构域(图3-A)。利用SOPMA在线工具对Mete-UAA氨基酸序列二级结构进行分析,在该二级结构中α-螺旋(α-helix)占25.94%,β-转角(β-turn)占9.22%,延伸链(extended strand)占26.80%,无规则卷曲(random coil)占38.04%(图3-B)。从整体结构来看,该二级结构中无规则卷曲和延伸链是Mete-UAA所编码蛋白的主要结构元件,α-螺旋和β-转角相对较少,序列两端分别以1个无规则卷曲和1个α-螺旋结构结束。COILS在线工具分析显示,该氨基酸序列可能存在卷曲螺旋区域(coiled coil regions),其概率值在窗口步长14条件下最高,窗口步长21条件下次之,窗口步长28条件下最低(图3-C)。
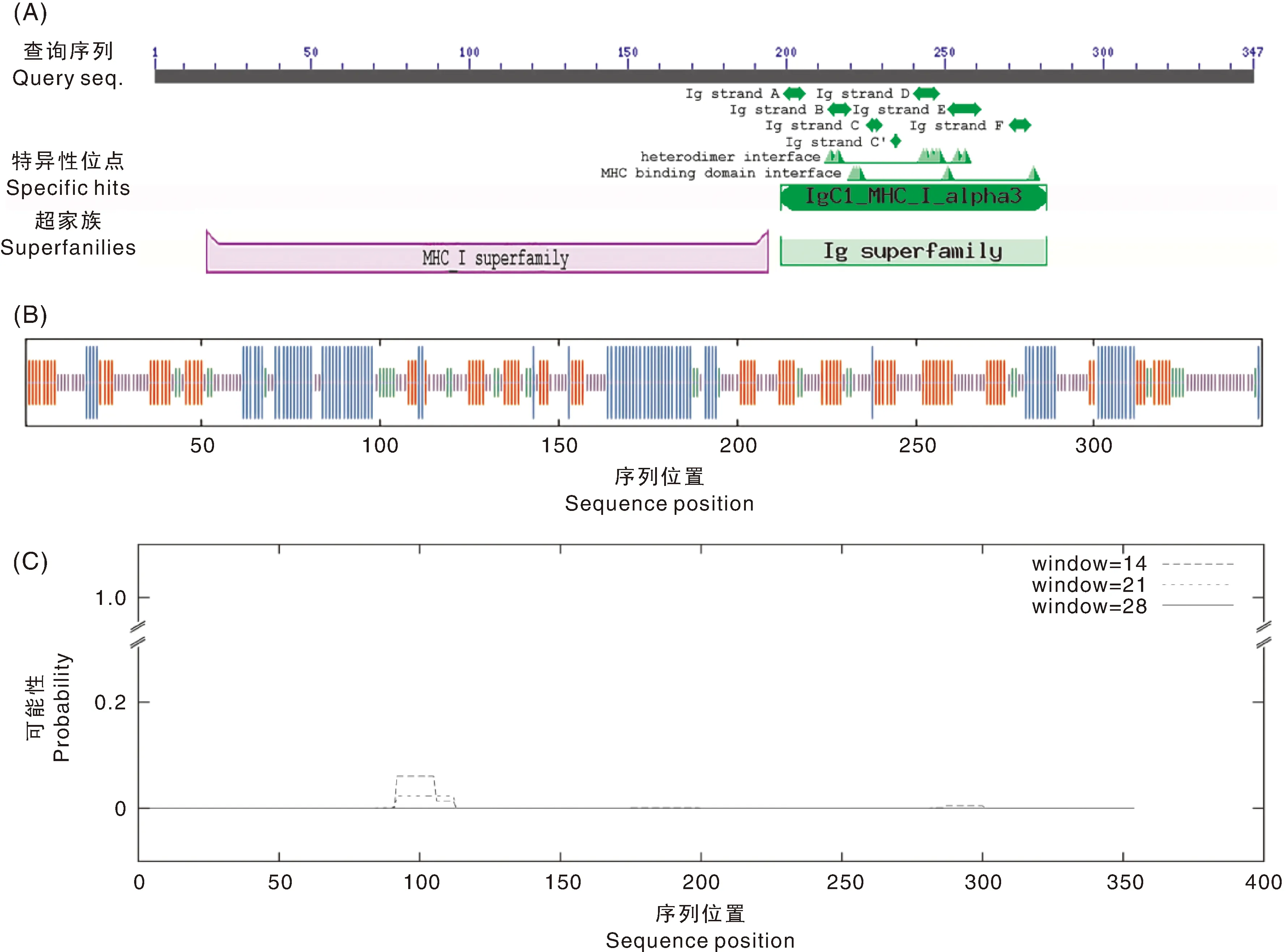
图B中蓝色代表α-螺旋,绿色代表β-转角,紫色代表延伸链,红色代表无规则卷曲。
2.2.2 三级结构
利用SWISS-MODEL在线工具对Mete-UAA氨基酸序列进行同源建模以预测其三级结构,以硬骨鱼类的MHCⅠ蛋白质(6lbe.1.A)为模板,在第17~289位氨基酸残基处进行建模,与模型序列的一致性为72.22%,相似性为0.53,覆盖度为62.71,蛋白模型的GMQE得分为0.71(图4-A)。而团头鲂Meam-UBA*F8-0102与模型序列的一致性为80.07%,相似性为0.56,覆盖度为62.79,蛋白模型的GMQE得分为0.74(图4-B),比三角鲂略高。
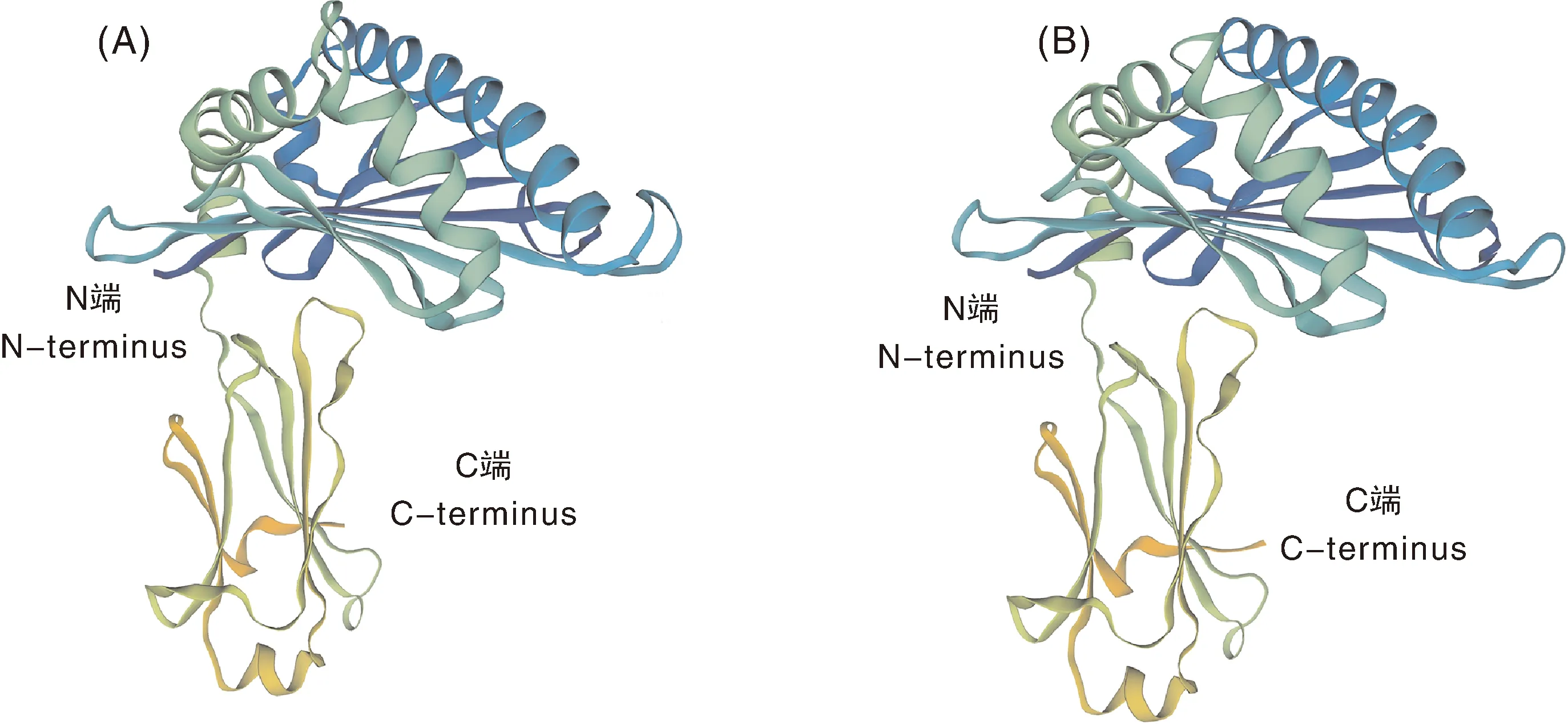
A,三角鲂Mete-UAA编码蛋白的三级结构预测模型;B,团头鲂Meam-UBA*F8-0102编码蛋白的三级结构预测模型。
2.2.3 信号肽、磷酸化位点、糖基化位点、跨膜结构域和亚细胞定位
利用SignalP-5.0在线工具预测Mete-UAA氨基酸序列的信号肽,结果如图5-A所示。信号肽的剪切位点位于丙氨酸-甘氨酸(Ala-Gly)之间,其概率为0.901 7,信号肽存在的似然概率为0.997 2,该信号肽由16个氨基酸残基(甲硫氨酸-丙氨酸,Met-Ala)组成。NetPhos 3.1 Server在线工具分析表明,Mete-UAA氨基酸序列含有39个磷酸化位点,包括15个苏氨酸(Threonine, Thr)位点、20个丝氨酸(Serine, Ser)位点、4个酪氨酸(Tyrosine, Tyr)位点(图5-B)。利用NetNGlyc 1.0 Server和NetOGlyc 4.0 Server在线工具分析Mete-UAA氨基酸序列的糖基化位点,结果表明,Mete-UAA在天冬酰胺(Asn)处含有-糖基化位点1个,其概率为0.553(图5-C),还含有3个-糖基化位点(Ser, Ser, Ser)。TMHMM Server v.2.0在线工具分析跨膜结构域显示,Mete-UAA氨基酸序列含有跨膜结构域,甲硫氨酸-甘氨酸(Met-Gly)处于膜外,异亮氨酸-酪氨酸(Ile-Tyr)跨越细胞膜,而赖氨酸-丙氨酸(Lys-Ala)则处于膜内(图5-D)。利用Protcomp 9.0在线工具进行亚细胞定位表明,Mete-UAA位于膜上。

图5 三角鲂MHCⅠα蛋白质信号肽(A)、磷酸化位点(B)、N-糖基化位点(C)和蛋白跨膜区(D)预测
2.2.4 理化性质分析
ProtParam在线工具分析结果显示,Mete-UAA的分子式为CHNOS,分子量为38 699.18,等电点为5.23;Mete-UAA编码的347个氨基酸残基中,Gly含量最高,为9.20%;其次是Thr,为8.10%;半胱氨酸(Cysteine, Cys)和Met含量最少,均为1.20%;包含带负电荷的酸性氨基酸[天冬氨酸(Aspartic acid, Asp)和谷氨酸(Glutamic acid, Glu)]42个,包含带正电荷的碱性氨基酸[精氨酸(Arginine, Arg)和Lys]32个;Mete-UAA编码蛋白质的不稳定指数为30.32,脂肪族指数为74.12。ProtScale在线工具分析结果表明,Mete-UAA编码氨基酸序列中,疏水性最强氨基酸位于Val处,其值为3.567;亲水性最强氨基酸位于Ser和脯氨酸(Proline, Pro)Pro处,其值均为-3.200;347个氨基酸残基中有249个氨基酸残基表现为负值,推测Mete-UAA是亲水性蛋白质。
2.3 BLAST比较与系统进化分析
2.3.1 BLAST比较
经BLAST检索比对发现,Mete-UAA氨基酸序列与团头鲂Meam-UBA*F8-0102 (NCBI蛋白质数据库编号:AEE87250)的MHCⅠα蛋白质同源性最高为75.79%,而与其他MHCⅠα蛋白质同源性较低(Meam-UBA*F8-0101,AEE87249,73.49%;Meam-UAA*F0-0101,AEE87247,67.05%;Meam-UAA*F0-0201,AEE87248,67.05%)。与草鱼MHCⅠα (BAD01521)同源性稍低于Meam-UBA*F8-0102,为75.25%。其次,与鲤(CAA62497)同源性为65.24%,与斑马鱼(AAH74095)同源性为56.77%,与虹鳟(AAG25199)同源性为51.18%,与大西洋鲑(AAN75113)同源性为51.03%,与细鳞鲑(BAA09553)同源性为50.65%。使用UGENE v38.1软件(Okonechnikovet al., 2012)的MAFFT组件对以上硬骨鱼类的MHCⅠα氨基酸序列进行多重比对后发现,Mete-UAA具有4个高度保守的半胱氨酸残基,其中2个位于alpha2结构域,另2个位于alpha3结构域(图6)。

每列上的氨基酸残基一致性越高则该列的颜色越深,圈中字符为高度保守的半胱氨酸残基。
2.3.2 系统发育分析
基于Ⅰ编码的氨基酸序列,将三角鲂与团头鲂、草鱼和鲤等其他硬骨鱼类一起构建最大似然(maximum likelihood,ML)系统发育树,以探讨三角鲂Ⅰα与其他硬骨鱼类的系统发育关系,结果如图7。由于银大麻哈鱼()和大鳞大麻哈鱼()的MHC基因为非典型结构,以这2个大麻哈鱼作为外类群构建进化树。在该系统进化树中,各物种均可按照相应的分类地位划分为鲤形目、鲑形目、鲈形目、鲽形目和慈鲷目等。

进化树节点上的数字分别表示SH-aLRT检验和Ultrafast Bootstrap校验的百分数。
2.4 转录组表达分析
将期望值设为1e-5进行BLAST比对后,从Unigenes中共鉴定出50条Ⅰ基因序列,序列一致性范围为73.77%~100%,比对序列长度从43 bp到1 721 bp不等。选取比对分值最高的前5个序列g.Unigene002682 (序列一致性为86.636%,长度为1 721 bp)、g.Unigene005230 (序列一致性为94.153%,长度为838 bp)、g.Unigene006339 (序列一致性为89.267%,长度为969 bp)、g.Unigene004553 (序列一致性为84.792%,长度为914 bp)、g.Unigene006993 (序列一致性为85.512%,长度为849 bp)为代表进行表达量分析,结果如图8所示。0 h时5个序列的平均表达量(FPKM值)为11.42,3 h时5个序列的平均表达量(FPKM值)为33.06,24 h时平均表达量降到3.68,表达量下降为3 h表达量的11.13%。

图8 三角鲂MHCⅠα基因表达的转录组分析
3 讨论
Ⅰ基因在脊椎动物的免疫反应中起重要作用。本研究从三角鲂肝中克隆得到一全长2 102 bp的Ⅰ基因Mete-UAA,其编码347个氨基酸残基。结构分析表明,Mete-UAA具有MHCⅠα的保守结构特征,包括3个alpha结构域和TM/CY区。在Mete-UAA中发现了多个糖基化位点,这在其他硬骨鱼中也有发现,例如舌齿鲈()和卵形鲳鲹()等。三角鲂Mete-UAA与草鱼Ⅰ基因(BAD01521)具有较高的序列一致性,但与其他草鱼、硬骨鱼类等Ⅰ同源基因的序列一致性较差,这也反映出Ⅰ基因的高度多态性。也正因为Ⅰ基因的高度多态性,使其可以与不同抗原肽相结合,进而递呈不同抗原,清除侵入体内的异物。
MHCⅠ是由重链(MHCⅠα)和β2-微球蛋白(β2m)以非共价键构成的二聚体,MHCⅠα包含1个前导肽、3个胞外结构域(alpha1, alpha2和alpha3)、1个跨膜区和1个胞质区。MHCⅠα的alpha1和alpha2结构域是高度可变且多态的,其可以在肽结合区(peptidebindingregion,PBR)位点形成肽结合槽,而alpha3结构域因与CD8和β2m共进化而相对保守。基于4个团头鲂Ⅰα基因的序列比对分析表明,alpha2结构域是多态性最高的区域。基于Mete-UAA与其他物种Ⅰ同源基因的序列比对,也发现alpha3结构域较alpha1和alpha2结构域保守。在Mete-UAA的alpha2结构和alpha3结构域发现的4个高度保守的半胱氨酸残基,可能参与二硫键形成而对维持Ⅰ编码蛋白质的稳定性具有重要作用。
基于MHCⅠα氨基酸序列构建的ML系统发育树分析表明,三角鲂与团头鲂、草鱼、鲫()、鲤和斑马鱼等鲤形目鱼类可形成一个单系群,这与基于团头鲂MHCⅠα氨基酸序列构建的邻接(Neighbor-joining, NJ)树的分析结果一致。虽然鲤形目的鱼类在目水平上为单系群,但在属水平上则表现为复系群。其中,三角鲂样本与草鱼样本(BAD01521)聚类在了一起,这也验证了BLAST比较的结果,但大多数其他草鱼样本则与三角鲂样本表现为并系群关系。大多数草鱼样本可形成2个主要的分支,一个分支由4个草鱼样本构成1个单系群,另一个分支则与团头鲂、鲫等构成一个复系群。此外,目水平上表现为复系群特征的分支,还有鲑形目和鲈形目。鲑形目中由于2个大麻哈鱼MHC基因为非典型结构,独自构成一个分支,而其他鲑形目的鱼类则能够聚类在一起,构成另一个分支。情况最为复杂的是鲈形目,该目的鱼类形成了4个分支。需要注意的是,大多数草鱼样本与三角鲂样本表现为并系群关系,而草鱼样本(BAD01521)与三角鲂样本处在了同一分支中,虽然与BLAST结果一致,但也显示出草鱼MHCⅠα的高度多态性。多个草鱼样本和鲫样本与团头鲂样本聚类在一起,与NJ树的分析结果稍有差异,可能由于马晓茜等构建NJ树所用样本有限。基因树毕竟不同于物种树,图7中草鱼样本(BAD01521)与三角鲂样本处在了同一分支中,以及多个草鱼样本、鲫样本与团头鲂样本聚类在一起,这种情况的产生,有可能是由于用于构建基因树的Ⅰ基因所含有的系统发育信息含量差异造成或是存在长枝吸引假象,也有可能是Ⅰ基因存在趋同进化或物种之间有基因流、不完全谱系发生等情况。
此外,三角鲂感染嗜水气单胞菌24 h后Ⅰ基因表达量呈现出显著下降趋势。与三角鲂不同的是,团头鲂感染嗜水气单胞菌24 h后Ⅰ基因表达量达到峰值,72 h后显著降低。基于三角鲂对Unigenes的BLAST比对结果,推测Ⅰ基因应具有多个或存在可变剪切。如马晓茜等从同一个体中克隆出2个Ⅰ基因。不同的Ⅰ基因其表达趋势不同,如g.Unigene002682在24 h的表达量(FPKM值为18.25)显著高于3 h的表达量(FPKM值为1.55),但Ⅰ基因总体呈现表达下降趋势。因此,Ⅰ基因表达量在团头鲂感染24 h时达到峰值,除去物种差异外,可能与Luo等未分析到其他Ⅰ基因有关。此外,本研究与Luo等研究结果的差异也可能与所研究的组织有关。团头鲂感染后肝、鳃和肠中Ⅰ基因表达量在24 h达到峰值,而肾中则在4 h达到峰值,脾中表达量在24 h后仍然在持续升高。而本研究中,所研究组织为肌肉、肝和血液的混合样品,Luo等未检测Ⅰ基因在团头鲂肌肉和血液中表达情况,因此,三角鲂与团头鲂Ⅰ基因表达时序差异也可能与研究的组织有关。基于转录组学对罗非鱼()感染后的基因表达分析表明,感染24 h后罗非鱼Ⅰ基因表达量与对照组相比显著下降,与本研究结果类似,表明Ⅰ基因在三角鲂抗嗜水气单胞菌感染中发挥了重要作用。
