优化近乎PAM-less腺嘌呤碱基编辑器提升编辑效率
2022-06-27周小钰王晓月
周小钰,王晓月
(中国医学科学院基础医学研究所 北京协和医学院基础学院 生物化学与分子生物学系,北京 100005)
在已知的人类致病遗传变异中,约50%是由单个核苷酸突变引起的[1],因此,开发高效的点突变纠正工具是人类遗传疾病研究和治疗的重要目标。目前,单碱基编辑技术将Cas9蛋白与脱氨酶结合,可以在sgRNA的引导下对基因组靶位点进行单核苷酸突变[2],其中腺嘌呤碱基编辑器(ABE)能将靶位点的腺嘌呤(A)转变为鸟嘌呤(G)[3]。该技术不会引起DNA双链断裂[2],具有更高的安全性。
然而单碱基编辑技术也存在一定的局限性:首先,最常用的SpCas9蛋白识别靶位点时必须首先识别NGG PAM序列[4],极大的限制了其应用范围。其次,编辑效率较低也是阻碍该技术在基因治疗领域发挥作用的重要因素[5]。本研究所采用的PAM-less ABE[6]在SpCas9蛋白上进行了突变使其几乎不受PAM序列的限制,扩大了在基因组的编辑范围。在此基础上通过对ABE其他元件如脱氨酶和连接肽进行替换改造,在基因组上非NGG PAM的位点进行编辑效率验证实验,从中初步评估ABE的编辑效率。
1 材料与方法
1.1 材料
HEK 293T人胚肾细胞系(国家生物医学实验细胞资源库);DMEM高糖培养基(Hyclone公司);gRNA序列(擎科生物科技公司);NeofectTM转染试剂(零客创智公司);lentiGuide-Puro 载体、pCMV-T7-ABEmax(7.10)-SpRY-P2A-EGFP载体(简称为SpRY-ABEmax)(Addgene公司);胎牛血清(Gibco公司);Opti-MEM(Life公司);Tryptone、Yeast Extract(OXOIO公司);Agar(鼎国昌盛公司);Trans5α 化学感受态细胞(全式金生物技术公司); FastAP、MluⅠ、BglⅡ、BsmBⅠ(Thermo Scientific公司);T4 DNA Ligase、T4 PNK、Gibson Assembly® Master Mix、NEBNext® UltraTMⅡ Q5® Master Mix(NEB公司); 琼脂糖凝胶DNA回收试剂盒、DNA maker D2000、质粒提取试剂盒、基因组DNA提取试剂盒(上海天根生化科技公司)。
1.2 方法
1.2.1 PAM-less ABE的改造:首先利用MluⅠ和BglⅡ双酶切SpRY-ABEmax载体,胶回收大片段为载体骨架,然后通过克隆突变得到Tad-8e,退火合成PAPAPA连接肽。利用重叠延伸 PCR将各片段连接,通过Gibson 组装将片段和载体骨架连接成环并转化至大肠杆菌感受态细胞,挑单克隆进行Sanger 测序验证组装是否正确,提取成功组装的质粒并转染HEK 293T细胞, 48 h后观察绿色荧光蛋白表达情况以检验ABE在细胞中是否表达。
1.2.2 sgRNA 表达载体克隆构建:将lentiGuide-Puro 载体的scaffold部分改造以提升sgRNA转录效率,具体方法见参考文献[7]。使用BsmBⅠ酶切lentiGuide-Puro,保留大片段作为载体骨架,将sgRNA的两条oligo退火和磷酸化,使用T4连接酶lentiGuide-Puro载体骨架与sgRNA片段连接,转化至感受态细胞并挑单克隆进行Sanger测序,提取成功连接sgRNA的质粒(表1)。

表1 不同基因组位点的sgRNA序列Table 1 sgRNA sequences at different genomic sites
1.2.3 ABE和sgRNA表达质粒共转染HEK 293T细胞:取60万HEK 293T细胞铺于6孔板,第2天观察当细胞汇合至90%时进行下一步实验。在1.5 mL EP管内添加 200 μL Opti-MEM培养基,每管加入2.5 μg ABE表达质粒和1.5 μg sgRNA表达质粒并加入4 μL NeofectTM转染试剂,混匀室温放置30 min后加入6孔板。转染24 h后加入1.5μg/mL的嘌呤霉素并于72 h后流式分选表达绿色荧光蛋白的细胞,将其冻存于负80或立即进行实验。
1.2.4 提取细胞基因组并使用PCR扩增技术在预期产生编辑的位点进行扩增和Sanger测序:使用BEAT工具[8]对Sanger测序的.ab1格式文件进行分析预估各版本ABE在同一编辑位点的编辑效率,每组设置两次或3次重复实验(表2)。

表2 PCR所用引物的序列Table 2 Sequence of primers used in PCR
1.3 统计学分析

2 结果
2.1 PAM-less ABE转染HEK 293T细胞的编辑效果
在基因组上PAM序列为GCTG的位点进行A到G的编辑,Sanger测序结果显示在第3位和第5位(前间隔序列protospacer的第1位至第20位为sgRNA与基因组DNA互补序列)的编辑效率分别为32.5%、70.5%(图1)。
2.2 PAM-less ABE的改造及验证
以SpRY-ABEmax为初始版本,在此基础上替换Tad-8e脱氨酶和PAPAPA连接肽分别构建SpRY-ABE8e和SpRY-ABE8e-PAPAPA(图2A),将各版本PAM-less ABE分别转染HEK 293T细胞,48 h后荧光显微镜下观察,带有绿色荧光的细胞即为表达PAM-less ABE的细胞(图2B)。
2.3 改造各版本PAM-less ABE编辑效率的比较
与对照组SpRY-ABEmax相比:1)SpRY-ABE8e在PAM序列为GACT和CAAC位点的编辑效率提升,编辑窗口也扩大;2)SpRY-ABE8e-PAPAPA提升了这两个基因组位点的中心位置(GACT-6A或CAAC-5A)的编辑效率,同时编辑窗口内的旁编辑情况相较于SpRY-ABE8e降低(图3);3)SpRY-ABE8e-PAPAPA在基因组PAM序列为GACG的两个位点的整体编辑效率更高(图4)。
2.4 PAM-less ABE对Clinvar致病突变位点的编辑效果
SpRY-ABEmax在Clinvar数据库中BRCA1:p.L1705P和BRCA2:p.D189G两个位点的编辑效率分别为53%和26%,SpRY-ABE8e-PAPAPA在对应两个位点的编辑效率分别为60%和35%(图5)。
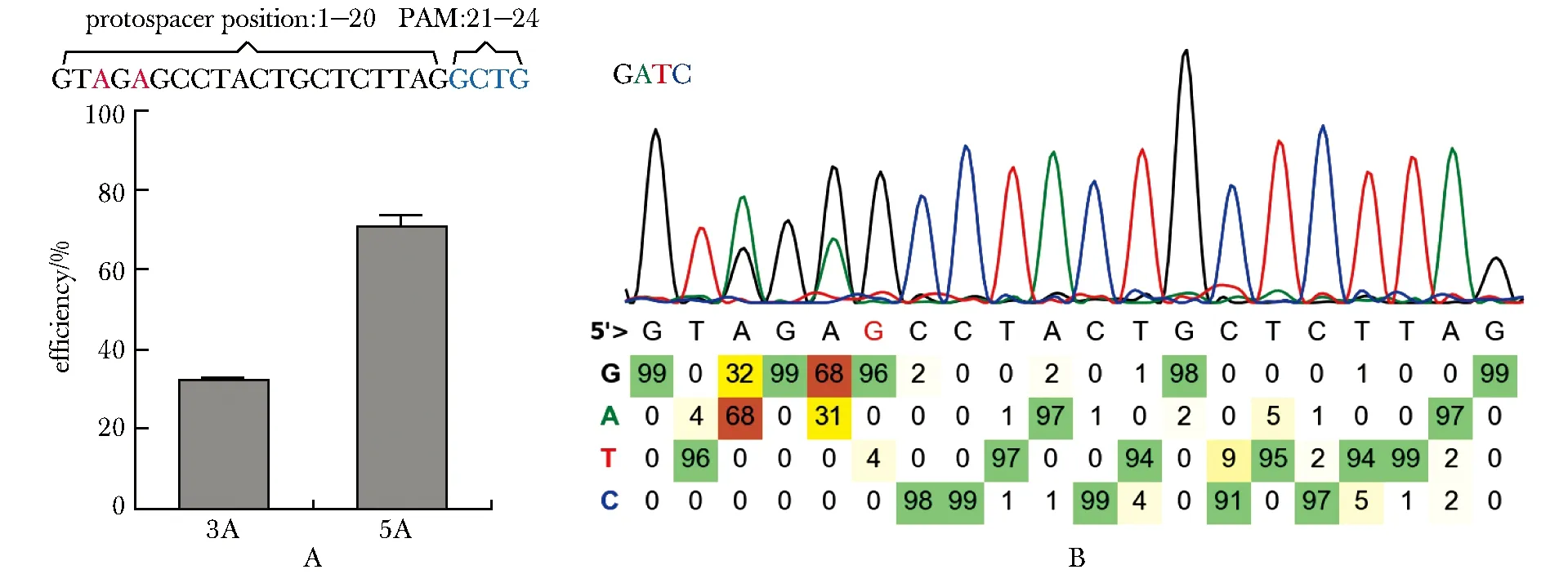
protospacer (black and red) and PAM (blue) sequences are located above the histogram; target As are indicated in redwithin the protospacer
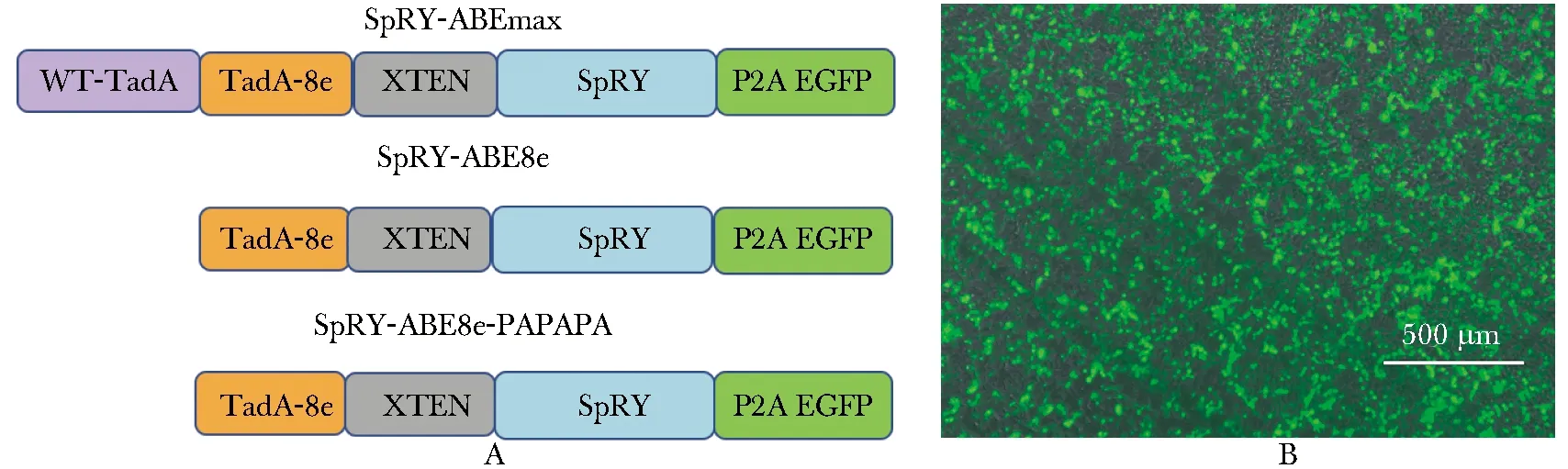
图2 各版本PAM-less ABE的构建 (A)和转染HEK 293T细胞的绿色荧光蛋白表达验证(B)
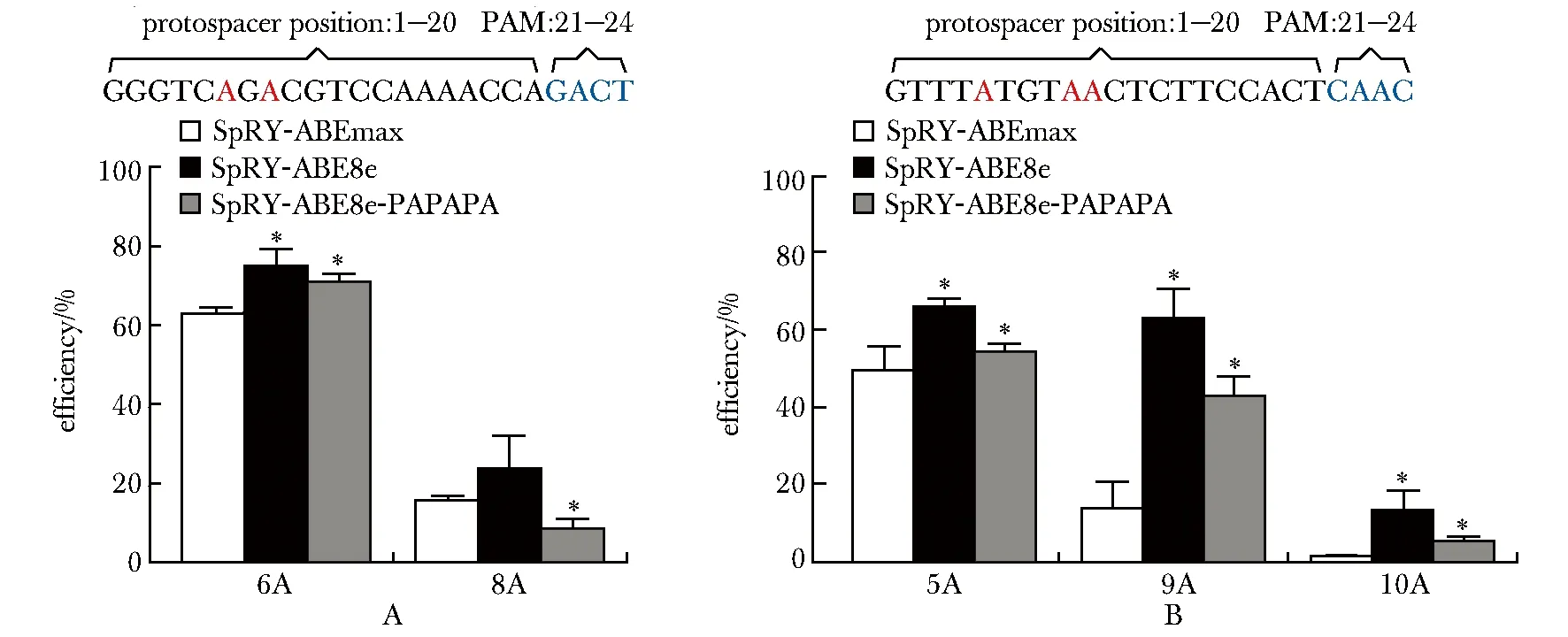
protospacer (black and red) and PAM (blue) sequences are located above the histogram; target As are indicated in red within the protospacer;*P<0.05 compared with SpRY-ABEmax
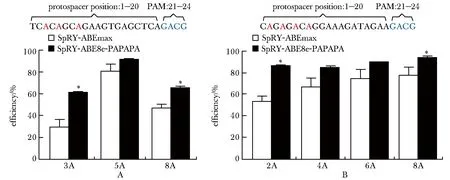
protospacer (black and red) and PAM (blue) sequences are located above the histogram; target As are indicated in red within the protospacer;*P<0.05 compared with SpRY-ABEmax
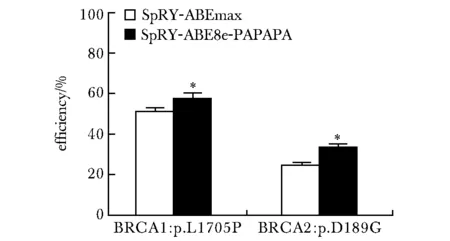
*P<0.05 compared with SpRY-ABEmax图5 PAM-less ABEs 在BRCA1和BRCA2基因上构造致病突变的编辑效率Fig 5 Base editing efficiency of PAM-less ABEs to construct pathogenic mutations in BRCA1
3 讨论
基于CRISPR-Cas系统的单碱基编辑技术可以直接在细胞基因组内实现单个碱基的转换类突变,这种方式避免了DNA双键断裂可能对细胞基因组造成的损伤[8]。但由于PAM序列识别位点的限制,目前主流的碱基编辑器受到NGG PAM序列的限制,只有当靶位点下游13~17 bp范围内存在NGG PAM序列时才能进行编辑,限制了应用范围[4]。本研究中的SpCas9变体SpRY几乎不受PAM序列限制,扩大了基因组的编辑范围[6]。然而ABEmax编辑效率较低[9],因此需要对其进行改造来提升编辑效率以实现更多的应用。
本研究首先验证了SpRY-ABEmax能对非NGG PAM的基因组位点进行编辑,然后尝试替换活性较高的腺嘌呤脱氨酶来改造新版本的PAM-less ABE。根据已有研究显示,最新的ABE8e 进行脱氨的速率快了大约1 100倍[10],在本研究中将ABE8e与SpRY进行组合构建SpRY-ABE8e并与sgRNA表达质粒共转染HEK 293T细胞,通过对几个非NGG PAM的基因组位点进行编辑效率验证实验发现:相较于SpRY-ABEmax,SpRY-ABE8e提升了对应基因组位点的编辑效率,但ABE-8e的高活性导致了编辑窗口扩大,从而产生更多的旁编辑,考虑到这个问题,根据之前的一项研究,缩短连接肽会在一定程度上限制编辑窗口[11],因此本研究采用了PAPAPA连接肽构建SpRY-ABE8e-PAPAPA,在一定程度上保持了中心位点编辑效率的同时限制了ABE8e的编辑范围,进一步将该版本ABE应用在ClinVar致病位点的编辑上[12],结果显示相较于对照组,SpRY-ABE8e-PAPAPA提升了对应位点的编辑效率。本研究所改造的PAM-less ABE有助于进一步对人类单核苷酸变异位点进行研究,由于突破了PAM序列的限制,可以在更多基因组位点进行疾病模型的构建或疾病治疗模型的构建,在基因治疗领域有广阔的前景。
综上所述,本研究证实通过替换活性更高的脱氨酶Tad8e可提升编辑效率;进一步可缩短脱氨酶与SpRY蛋白之间的连接肽来限制编辑窗口。该研究为今后基础研究和基因治疗应用提供了更多选择的碱基编辑工具。
