湖泊沉积物古DNA揭示气候环境变化和生态演化
2022-06-16王晶,徐海
王 晶,徐 海
天津大学 地球系统科学学院,天津 300072
自人类诞生以来,人为活动的持续性影响就以或大或小的力量塑造着地球系统的面貌(孙凯,2020)。尤其是在工业革命以后,科学技术的发展使得人类活动的影响远远超过了地球自然生态系统的演化历程,并因此产生一系列重大的区域性、全球性环境问题和社会问题(Biermann et al,2012;Grundmann,2016; 孙 凯,2020)。近几十年来,气候变化以及人类活动对全球的自然和人类系统造成了重大影响,许多陆地和水生生物已经改变了其地理活动范围和分布格局以及多样性组成和物种相互作用,以此来应对持续的气候和环境变化(IPCC,2014)。湖泊生态系统是地球表层系统中的典型代表和重要组成部分。作为一种重要的自然资源,湖泊对全球气候变化非常敏感 (Adrian et al,2009;Woolway et al,2020),在驱动流域关键生命元素(碳、氮、磷、硫等元素)的生物地球化学循环和水量平衡过程中扮演着重要角色。因此作为水生生态系统、大气圈和陆地生态系统的结合点,湖泊生态过程及生物演变成为人类活动以及区域和全球气候环境变化影响的综合体现。
目前,湖泊已经高度暴露于气候变化和人类活动引起的双重压力之下(Keck et al,2020)。而湖泊生态系统对人类活动扰动以及全球环境改变的响应在不同区域和不同的生态系统演化阶段表现出很大的差异,主要包括湖泊水环境变化、水生动物(包括浮游动物)变化、水生植物(包括浮游植物)生长变化、微生物变化、地层特征和沉积过程变化、生态系统功能特性变化以及生物多样性和生态系统稳定性变化等(Ahmed et al,2018;秦伯强,2020;Ellegaard et al,2020)。尽管气候和环境条件一直在影响着湖泊生态系统,而人类对湖泊的干扰也已经长达数千年之久(Jenny et al,2019),但是对湖泊生物多样性的长期变化仍然知之甚少。只有深刻了解地球历史时期湖泊生态系统对气候变化和人类活动的响应特征,明确其在不同气候环境背景下的生存状态,准确地认知湖泊生物进化以及生态演化过程,才能更好地理解在未来的气候变化情景下,人类活动和全球变化对不同区域、不同类型湖泊生态系统在不同演替阶段的潜在影响。
一般情况下,通常以湖泊水体的沉积物记录来反演和重建地球历史时期的气候和环境变迁以及周边流域生态系统演化和人类活动影响的变化(Giguet-Covex et al,2014;Hou et al,2014;Pedersen et al,2016;Yang et al,2016a;Yang et al,2016b),并结合其他古气候和/或记录人为作用痕迹的代用指标阐述其内在的响应机制。由于湖泊是一个流域的主要汇水区,并聚集了来自周边流域的多种有机物、无机物以及动植物和微生物残体等,对汇入其中的丰富物质和各类信息能够进行充分有效地保存、浓缩和沉积,成为记录气候及人为活动变化的精确历史档案和优良环境载体(Anderson-Carpenter et al,2011;Parducci et al,2017;Ellegaard et al,2020)。因此,它们为古代动物、植物和微生物以及其他一些原生生物和后生动物古DNA(ancient DNA,aDNA)的保存提供了良好的环境条件。随着沉积物的连续累积,通过借助地质年代学方法建立高分辨率的古DNA分子记录,可以全新的视角审视过去的气候环境波动和人为作用的干扰,并为古生物进化和古生态演化的分子历史提供全新的证据,同时可为传统古湖沼学气候和生态重建代用指标提供可靠补充。
近十几年来,随着基因测序技术(下一代测序技术、第三代测序技术)的不断更新和完善,组学(病毒组学、宏基因组、宏转录组、宏蛋白组)技术的进步和发展,以及对功能基因识别和绝对定量(quantitative PCR,qPCR)的实现,从核酸水平研究气候、生态演变历史的精细程度逐渐达到了前所未有的高度。综合湖泊沉积物古DNA和多代用气候指标方法,研究不能形成化石的生物种群遗传变异的表型特征,将能够用于理解重要生物种群进化和生态演化事件的时间尺度,并解决地质历史时期湖泊不同生物种群、群落和整个生态系统对关键气候事件、气候危机以及其他环境变化和人类活动的响应过程,从而揭示物种和生态系统如何适应生存的环境,最终将有利于预测全球变化背景下湖泊生物的响应和适应性特征及其内在变化机理,为理解湖泊生态系统的长期生存状态提供理论基础和数据支撑。
本文首先回顾了最近几十年古DNA研究领域不断发展和进步的历史背景,从古DNA分子丰富来源和基本特征的角度阐述了开展古DNA相关研究工作的重要理论基础,并总结了影响湖泊沉积物古DNA保存和稳定性特征的主要因素。概述了应用于古DNA领域的古基因组生物信息学分析流程,并提出未来宏基因组学将成为探索湖泊沉积物古DNA和生态功能演变的有力工具。在此基础上就湖泊沉积物古DNA在古气候重建和古生态演化历史中的诸多应用和进展做了较系统的回顾和评述,同时还简单介绍了当前开展湖泊沉积物古DNA研究面临的局限和挑战以及组学技术在古DNA研究中的基本概况。最后总结了当前研究存在的不足和今后可能的努力方向,以期为未来湖泊沉积物古DNA及其他相关古基因组学研究领域提供理论、方法上的参考和借鉴。
1 古DNA发展的历史背景和理论基础
1.1 古DNA研究历史概况
过去30多年的古DNA革命,在利用古代生物核酸分子解析古气候波动、生物多样性和种间相互作用、物种灭绝与进化、陆地和水生生态系统演化等方面,推动了不同学科领域相关话题的爆发式增长。1984年首次报道了对长期死亡组织的DNA分析,发表了博物馆保存的伯切尔氏斑马(Equus quagga,一种马科动物,已于19世纪80年代灭绝,是现代斑马的亲缘物种)风干肌肉组织样本线粒体DNA(mitochondria DNA,mtDNA)的229个碱基对序列,并成功完成DNA的分子克隆(Higuchi et al,1984),由此标志着古DNA研究领域的诞生(Cappellini et al,2018)。这项基础研究表明古代灭绝生物的DNA分子能够持续存在,并可借助现代分子生物学和古基因组学工具从其残留遗迹当中恢复出来。而几乎与此同时,在一个距今2400 a的小孩木乃伊上也发现了能够在质粒载体中进行分子克隆的古代DNA(Pääbo,1985),表明这一独特的古代人类遗骸来源可被成功应用于分子遗传学分析。更为重要的是,20世纪80年代末期诞生的聚合酶链反应(polymerase chain action,PCR)技术实现了从有限拷贝数模板DNA分子当中获取大量的扩增产物,通过对其直接测序不仅降低了序列错误率而且避免了分子克隆技术中出现人为产物的可能性,从而为分子考古学和种群生物学的快速进步开辟了广阔前景(Pääbo and Wilson,1988),同时也为古DNA研究领域的蓬勃发展和迅速兴起注入强劲动力。因此,在PCR时代的古DNA研究领域才开始走向成熟,逐渐变得更加科学严谨。此外,研究人员开始同时关注具有人类学和进化意义的重大历史事件,为开展分子演化遗传学的历时性研究创造了有利条件(Pääbo,1989),并且对于古代DNA分子的生存极限以及古基因组学研究工作中涉及的细微差别和遭遇的潜在困境也不断受到广泛关注和深入了解。
然而关于木乃伊古DNA的研究被广泛认为是由污染所导致的结果(Shapiro and Hofreiter,2014),因此更加突出了一个需要克服的重大问题 —— 即现代DNA几乎污染了所有的古代遗迹、提取样本和许多实验室器材及操作环境(Pääbo et al,2004;Peyrégne and Prüfer,2020),并且这也是当今古DNA研究仍然需要面临和解决的主要风险与挑战。尽管古代DNA分子的降解特性和对现代DNA外源污染的极度敏感,大量的早期研究和积极探索仍然极大地激发了不同领域研究人员从古代遗迹和沉积环境当中提取DNA分子的热情,并取得了一系列重大突破和革命性进展(Xu et al,2011;Bellemain et al,2013;Giguet-Covex et al,2014;Green and Speller,2017;Olajos et al,2018;Seersholm et al,2020;van der Valk et al,2021)。过去十几年对古代DNA分子的研究已经彻底改变了人们对地球生命进化史以及生态系统演化过程的理解。
自20世纪80年代的开创性工作以来,古DNA研究经历了快速增长过程(图1),相关研究跨越的空间范围和时间尺度也有了显著增加。目前已能对过去0.8 Ma的环境变化进行重建,并通过保存在冰芯当中的遗传数据档案为来自遥远过去的动植物进化和生态过程提供深刻见解(Willerslev et al,2007)。一直以来,超过百万年尺度的古DNA保存就像一道难以逾越的关卡。2013年古遗传学家利用埋藏于永久冻土当中约0.56 — 0.78 Ma的马腿骨标本进行了基因组测序及氨基酸和蛋白质组学分析,发现虽然在获取的DNA碎片当中最短的片段只有25个碱基对(base pair,bp),但是仍然可以进行古遗传学分析,并获得了当时最古老的动物古基因组草图(Orlando et al,2013)。此后对于样本中古DNA分子可以保存超过1 Ma一直处于猜测阶段。然而最新的一项发掘于西伯利亚永久冻土灭绝猛犸象臼齿样本的研究成果将古DNA保存的上限扩展到了过去的1.65 Ma,并获得了首个“杂交物种形成”的古DNA证据(van der Valk et al,2021)。该研究不仅揭示了一个全新的猛犸象演化谱系,并描绘了比从臼齿形态中获得的更为复杂的物种进化关系,更为重要的是对百万年级别基因组保存壁垒的突破为今后其他相关领域的关键分子演化历史和深时测序研究提供了重要的参考意义。

图1 2020年11月,使用“ancient DNA”作为检索词,并按照不同主题进行分类,从Web of Science数据库中检索到的有关古DNA研究的出版物数量Fig.1 Number of publications retrieved from the Web of Science data bank in Nov.2020, using “ancient DNA” as search term, categorized according to different subjects
古DNA研究领域的快速发展主要得益于近年来高通量测序(high-throughput sequencing,HTS)技术的广泛应用(Mardis,2008;Orlando et al,2015)。高通量测序特指下一代DNA测序(next-generation DNA sequencing,NGS),主要相对于第一代桑格(Sanger)测序而言,致力于快速、价格低廉和精确的大规模生物基因组信息数据产出(Metzker,2010)。在此之前,自动化桑格法已主导该行业领域超过20 a,其测序的生物化学过程发生于“循环测序”反应之中,不断执行着模板变性、引物退火和延伸过程(Shendure and Ji,2008)。这种测序手段本身较长的读段长度(约1000 bp)和超高的测序准确性(>99.999%)优势带动了包括人类基因组常染色体测序(International Human Genome Sequencing Consortium,2004)在内的一系列具有里程碑意义卓越成就的实现。而第二代DNA测序技术主要应用的是循环阵列合成的测序策略,可以概括为通过酶操作和基于图像的数据收集迭代循环,对密集的DNA特征阵列进行测序,涉及模板制备、测序、成像以及下游数据分析和可视化等多个步骤(Metzker,2010;Kircher,2012)。对古DNA提取物或在特定位点进行靶向富集的古DNA序列文库开展高通量测序(Krause et al,2006;Burbano et al,2010),不仅影响了人们对以往演化问题的思考角度和方式,而且还通过为古代遗传物质提供全新窗口而改变了古DNA研究领域的应用范围。从其他层面来看,人们陆续发现古DNA广泛存在于多样化环境和载体当中,例如冰芯、永久冻土、湖泊沉积物、海洋沉积物、粪体化石和泥炭地岩芯(Pedersen et al,2015;Green and Speller,2017;Zimmermann et al,2017)等,进一步为不同古DNA领域创新性成果的获得提供了前所未有的契机。
另外,一系列重要方法学的建立不仅为古DNA的研究开辟了道路,其在古基因组学的不断发展和成熟过程中更起到了至关重要的支撑作用(图2)。例如:1990年发表了第一个细菌DNA宏条形码(Giovannoni et al,1990),该研究对海洋超微型浮游生物的16S核糖体RNA(ribosome RNA,rRNA)克隆文库进行了系统发育分析,在聚类中观察到的16S rRNA多样性表明这些浮游细菌可能是独立谱系的联合体,有着惊人的遥远共同祖先,从而为环境微生物古DNA研究奠定重要的技术和理论基础。尽管此时湖泊沉积物尚未因其是保存植物、细菌、真菌和动物古DNA的绝佳环境载体而引起广泛关注,但是对湖泊沉积环境下植物化石样本叶绿体基因的成功测序和对比验证已为了解现存植物群落与灭绝类群之间的系统发生关系开辟了新的途径(Golenberg et al,1990)。在将近10 a后研究人员从湖泊沉积物当中恢复了过去11000 a的紫硫细菌16S rRNA基因序列(Coolen and Overmann,1998),针对古DNA的核苷酸序列分析开始在重建湖泊过去微生物群落组成和环境条件变化方面表现出巨大潜能。而从2010年以后,古DNA研究领域开始逐渐踏上快速发展的轨道,相关新技术和新方法不断涌现(图2)。

图2 1985年至今古DNA研究关键方法学发展时间轴Fig.2 Timeline of key methodologies of ancient DNA research from 1985 to present
最近10 a,古气候学家和分子生态学家对利用从湖泊沉积物中分离提取的DNA探索晚第四纪环境波动、植被发展历史以及流域生态系统演化过程表现出极大兴趣,一系列有影响力研究成果的报道进一步证实了湖泊沉积物古DNA在重建过去环境变化方面所拥有的巨大潜力(Giguet-Covex et al,2014;Pedersen et al,2016;Keck et al,2020;Liu et al,2021b)。基于以下几点原因,湖泊沉积物毫无疑问成为环境古DNA领域使用最为广泛的自然档案和信息载体。首先,湖沼学以解决生态问题为核心和主线,其系统性的原理和方法基础可为了解湖泊及周边生物集群的动态过程和驱动机制提供建设性观点和见解(Gregory-Eaves and Beisner,2011)。其次,湖泊沉积物古DNA包含了内部水生生物(Capo et al,2015)和陆地流域环境组分(Pansu et al,2015),在分别评估静水生态系统结构和功能以及重建湖泊附近陆地环境演变方面具有独特优势。微生物对有机质的不断分解和矿化加之与大气环境的有效隔绝为湖泊底部和表层沉积物创造了相对稳定的厌氧条件(Sobek et al,2009),大大降低了以往古生态学研究中对生物扰动的担忧。另外,尽管早已在非冻土环境和一些洞穴沉积物当中观察到古DNA分子存在垂直迁移(浸出)的证据(Haile et al,2007;Andersen et al,2012),但是目前已知的研究均未发现湖泊沉积物古DNA具有浸出的迹象(Anderson-Carpenter et al,2011;Sjögren et al,2017),可能与湖泊底部沉积物永久饱和,缺乏形成湖水重力浸透的天然条件有关。由此湖泊沉积物古DNA研究在近10 a左右获得广泛关注并实现快速发展,对全新世植物大化石和湖泊沉积物古DNA分子的可靠测序不仅弥补了传统古生态学和现代遗传学之间的鸿沟(Anderson-Carpenter et al,2011),更将生态信息和环境变化的探索从对传统微体化石和大化石的形态特征识别中解放出来,直接与种群的遗传变化和分子演化关系相联结,为变化环境条件下的生态和演化结果提供了深刻见解。
大量的研究(表1)揭示了湖泊沉积物古DNA如何进行物种识别,并且相比基于花粉和动植物大化石的传统物种鉴定方法,古DNA的分子手段具有更高的生物学分辨率,能够鉴定到种甚至是亚种水平,由此为研究地点提供了重要的生态和气候信息(Parducci et al,2015),有助于一窥局地生物多样性的演变历史并实现对古环境过程的重建。最近马睿等(2021)也对湖泊沉积物古DNA分析在重建古气候、古环境和古生态研究中的优势进行了详细总结,指出古DNA具有不依赖于形态学鉴定、可提供多种分类群的群落视角以及实现对特定生态功能基因进行靶向的独特优势。本文聚焦于湖泊沉积物的古DNA记录(图3),因为湖泊在全球广泛分布,它们的地理背景提供并记录了丰富的跨越时间和空间尺度的可靠古DNA分子信息和自然历史档案。
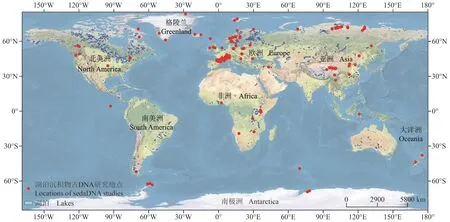
图3 全球已开展湖泊沉积物古DNA研究地点分布Fig.3 Locations of lakes worldwide and research sites where studies have investigated lake sedimentary ancient DNA (sedaDNA)

表1 与湖泊沉积物古DNA研究相关的论文Tab.1 Ancient DNA-related publications from lake sediments
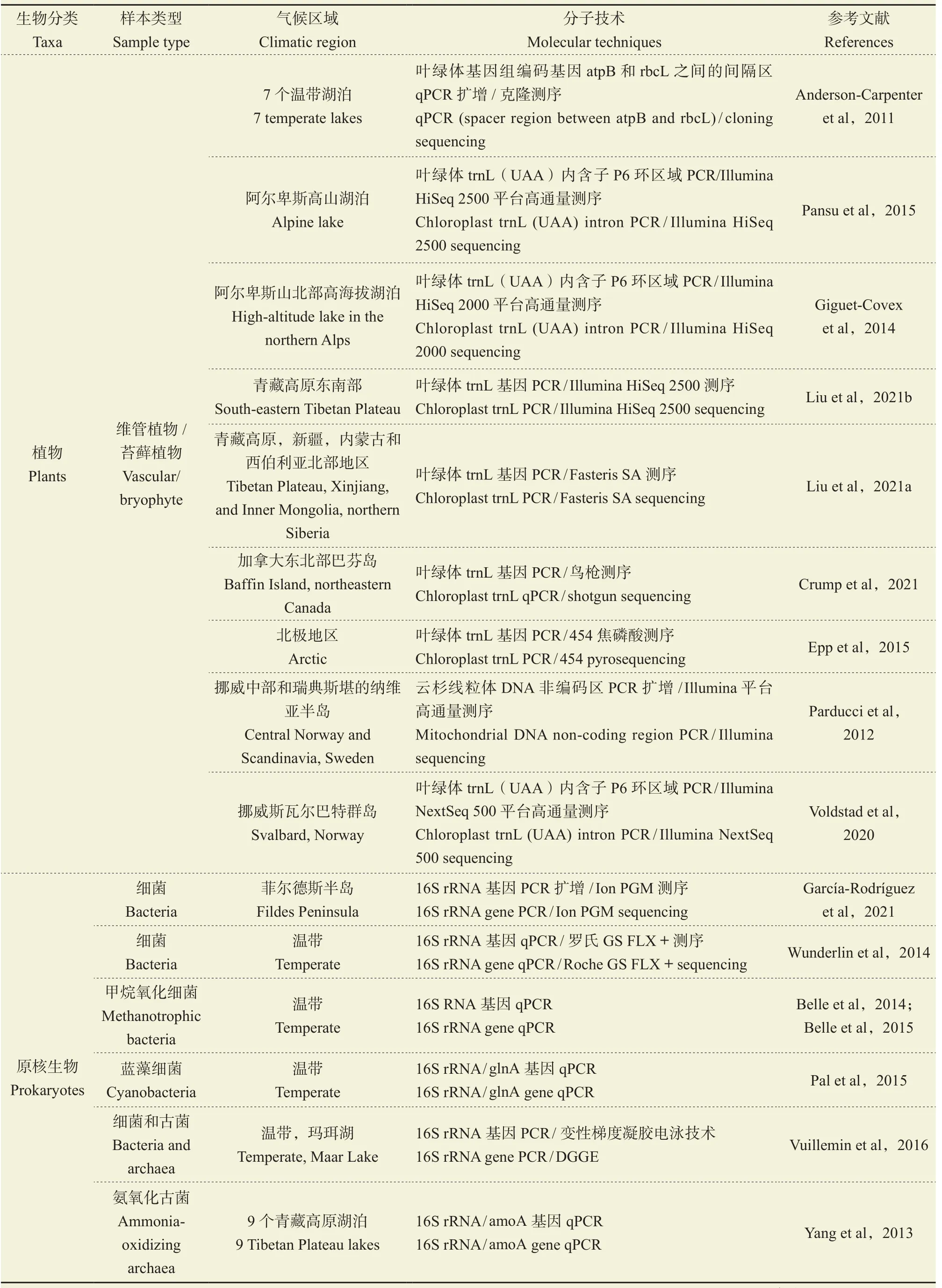
(续表1 Continued Tab.1)

(续表1 Continued Tab.1)
1.2 古DNA研究的理论基础
1.2.1 古DNA分子的来源
早期的古DNA研究主要集中于古代木乃伊、人类大脑、动物毛皮、干燥肌肉和内脏等软体组织样本(Pääbo,1985;Doran et al,1986;Pääbo,1989),从20世纪80年代末期开始以考古学的坚硬组织样本(骨骼和牙齿)古DNA分子(Hagelberg et al,1989;Horai et al,1989) 作 为 研究对象。随后其他一些携带古DNA的样本材料也相继被发现。例如动物古DNA来源:毛发、爪子和羽毛等角质材料(Tito et al,2012)以及灭绝动物的粪体化石(Hofreiter et al,2000)等;植物古DNA来源:炭化、干燥、冰冻和保存于缺氧环境条件下的种子等考古植物学遗迹(Elias and Mock,2013);以及考古发掘的人工样本,如陶瓷制品,代表了食物古DNA的重要来源(Green and Speller,2017),可用于研究古代人类饮食的变化。
除此之外,还在更广泛的沉积环境背景下提取出了植物、动物和微生物的古DNA,包括洞穴沉积物(Haouchar et al,2014)、永久冻土(Willerslev et al,2003)、冰芯(Willerslev et al,2007)以及海洋和湖泊沉积物(Pedersen et al,2016;Wurzbacher et al,2017;Olajos et al,2018)等。然而由于当前研究方法和相关技术条件的限制,仍有许多极有前途的古DNA来源未得到充分探索。微体化石就是其中的重要代表,例如花粉、硅藻和真菌孢子,在全球的一些沉积环境代表了非常丰富的化石类型。但是相比于沉积物古DNA,其在揭示生物多样性历史演变中的生物分类学分辨率要低得多(Boessenkool et al,2014),并且现有的用于微体化石分离的方法大多数还是靠手工操作,因此时间成本较高。
1.2.2 古DNA分子的特征
活细胞死亡后,由于分解代谢酶对胞外DNA的攻击,加之一系列复杂的成岩作用和生物化学反应,导致古DNA的分子结构将会受到明显的改变。尽管DNA分子可与某些矿物基质表面结合而免受微生物的降解,但是其他一些化学过程仍不可避免地影响DNA的保存状况(表2),随着DNA受损过程的不断积累,最终导致其完整性的丧失和被逐渐分解,同时伴随核苷酸序列信息不可逆的损失。

表2 古DNA受损类型(修改自Pääbo et al(2004))Tab.2 Different types of damage in ancient DNA (revised from Pääbo et al (2004))
因此,古DNA通常是高度片段化和经过化学修饰作用的。生物死亡后,其DNA分子首先会经历细胞内源核酸酶溶解作用的破坏,之后很快经历外生降解过程,例如水解作用、氧化作用、辐射作用等,导致DNA分子含氮碱基的改变和糖-磷酸骨架的裂解(Cappellini et al,2018),使得它们的稳定性显著降低且呈现出高度碎片化的状态。DNA分子保存的时间长短和受损状况主要取决于降解速率、样本材料来源和周围的环境条件等因素。其中最有利于古DNA保存的环境分布于永久冻土和冰芯当中(Parducci et al,2017;Cappellini et al,2018),这些区域的古DNA主要保存于生物遗骸和沉积环境样本(如土壤、沉积物),最长可持续存在几十万年(Orlando and Cooper,2014)甚至超过1 Ma(van der Valk et al,2021)。对于湖泊沉积物,寒冷环境条件下古DNA保存的年限多集中于10 — 20000 a的时间尺度(Epp et al,2015;Yang et al,2016a;Zhang et al,2021),而相对温暖气候下的湖泊沉积物古DNA,大多呈现的是十年至百年尺度,最多只有几千年的研究结果(Boessenkool et al,2014)。例如:Martínez de la Escalera et al(2014)从一个亚热带湖泊沉积物提取古DNA进行系统发育分析,以检验人为富营养化对湖泊原核微生物组成的影响,在数十年和百年尺度上发现湖泊发生水华之后不久,演化出了具备合成编码毒素的柱孢藻类群。
综上所述,来自历史和古代组织以及多样化沉积环境中的DNA表现为高度碎片化、化学损伤、内源性DNA大量枯竭,并与周围环境高度混合的明显特征。与长期建立的古DNA真实性标准相一致(Cooper and Poinar,2000;Gilbert et al,2005),这些特征对于验证古DNA研究结果和区分真伪及污染DNA来源具有重要意义。例如:古代DNA分子碱基修饰的一个典型特征是胞嘧啶核苷酸往往会自发地失去一个氨基并转化为脱氧尿嘧啶(Hofreiter et al,2001;Briggs et al,2007)。这样的化学损伤使得在文库制备和测序过程中会发生胞嘧啶-胸腺嘧啶(C-T)的错误匹配,并且脱氨基作用向片段末端迅速增加,磷酸二酯键的水解分裂作用进一步促进单链悬垂的形成。因此,在基因组时代,5’末端大量的C-T错配和3’末端的互补G-A错配被视为古DNA真实性的检验标准(Rohland et al,2015;Kistler et al,2017),同时对于从现代污染性外源DNA分子当中识别受损的古代DNA也具有重要的指导作用。另外,从结果的可信性方面,早在上个世纪末,借助于湖泊沉积物古DNA的16S扩增子分析,已有研究表明DNA文库测序的错误率仅为≤0.5%,这与从现存菌群中提取的DNA相比,测序误差并无明显增加(Coolen and Overmann,1998),进而证实了之前的发现,即扩增16S rRNA基因片段对于分析跨越万年尺度的样本是可靠的(Poinar et al,1996)。另外,Jónsson et al(2013)开发了生物信息工具包mapDamage 2.0,其构建的贝叶斯统计框架为识别和分辨古DNA的关键特征及受损速率提供了定量估计。由此可见,湖泊沉积物古DNA本身所具备的优势和潜能为洞悉过去的生物区系和演化过程提供了真实可信的窗口,而在此之前,涉及相关科学话题的探讨仅仅依赖活体生物DNA测序或古生物学研究是很难实现的。
1.2.3 湖泊沉积物古DNA分子的影响因素
根据文献资料调研的结果(表1),大量的研究成功地从湖泊沉积物当中提取出古DNA分子,并从这些自然档案中推断了过去的生物多样性、气候条件以及流域生态演化等动态变化过程。通过这些文献,可以对湖泊沉积物DNA的保存程度以及控制湖泊沉积物中DNA保存的条件有一些初步了解。根据相关研究论文和综述文章,基本上可以将影响湖泊沉积物DNA持久性的因素分为三大类:(1)DNA自身特性(例如:DNA分子的构象、长度及其与细胞膜和细胞器膜的关联);(2)非生物环境(例如:光照、湖泊盐度、溶解氧、pH、离子化合价、底物的种类和浓度);(3)生物因素(例如微生物群落和胞外酶的组成及活性)(Barnes et al,2014)。对于不同类型的湖泊及其周边多样化的气候环境条件,只要从这3个方面进行综合分析,即可提供湖泊沉积物古DNA的保存状况、存在特点及其形成机制等关键信息。其中稳定的湖泊分层、缺氧条件以及低而稳定的温度应该是促进沉积物DNA保存的最关键因素(Coolen et al,2006;Corinaldesi et al,2008;Corinaldesi et al,2011)。同时其他一些生物(如通过核酸酶的降解)和非生物(如黏土矿物和有机质组成)因素也代表了影响沉积物中DNA保存的重要制约条件。
此外,不同类型的沉积物具有不同的物理和化学特性,这将对DNA的保存产生不同的影响。因此,湖泊沉积物古DNA分子的保存状况可能会因不同湖泊的地理位置、湖泊深度、含盐量、湖泊的分层特征及内部循环特性等因素而存在较大差异(Parducci et al,2017)。而湖泊不同生物类群DNA能够以多样化形式存档于沉积物中,主要取决于其生物来源(Domaizon et al,2017)。湖泊沉积物当中的古DNA分子既可以保存在结构上完整的组织或者细胞内部,也能够以游离态的胞外DNA形式保存。其中较长的古DNA序列(900 — 1400 bp)可能源于保存完整的细胞结构,而较短的片段(150 — 290 bp甚至<50 bp)则反映了古代沉积物中胞外DNA的累积(Vuillemin et al,2016)。其中游离DNA分子可能通过组织脱落、代谢废物的释放以及生物死亡后的分解直接将DNA释放到环境中(Domaizon et al,2017)。根据Corinaldesi et al(2005)得出的结论,海洋沉积物当中胞外DNA的浓度比胞内DNA高出10 — 70倍,并且水生系统沉积物当中的胞外DNA浓度也比水体高出3 — 4个数量级(Turner et al,2015),因此湖泊沉积物中最主要的古代遗传物质应当也是胞外游离的DNA分子。此外,这些高浓度的胞外DNA分子也可能在生物地球化学循环过程中扮演着重要角色,并可通过自然转化实现水平基因转移(Corinaldesi et al,2005),而这种通过水平基因转移实现外源DNA的整合及全新遗传信息的捕获对于细菌的演化也是至关重要的(Nielsen et al,2007)。
1.2.4 古DNA分子的生物信息学分析
高通量测序技术驱动了古基因组学的快速发展,使得可获取的组学数据快速增加,一些测序平台运行一次即可在短时间内产生几百GB甚至超过TB级的组学数据(Metzker,2010;Quince et al,2017)。相比于现代环境DNA,古DNA序列通常长度较短、受损严重且拷贝数较低,因此相对于传统的测序方法(例如一代Sanger测序),高通量测序技术具有无可比拟的优势,使得对湖泊沉积物古DNA提取物的完整表征成为可能。由于不同湖泊的流域形态和地质构造多种多样,其他一些因素(例如河流侵蚀和人为污染物排放)同样会影响DNA来源向沉积物内部的输送过程,导致湖泊沉积物古DNA信号具有显著的变异性和空间异质性特征(Capo et al,2021),因此在提供清晰简洁的沉积物古DNA收集、数据分析和结果解释等方面仍存在较大的困难和挑战。本文仅提供了湖泊沉积物古DNA分析的整体框架(图4),主要包括沉积物岩芯采集、样本古DNA提取、测序文库制备、高通量测序平台测序以及在获取数据之后进行高效可靠的下游生物信息学分析。这些数据后处理过程并不是对单个测序序列进行比对,而是需要对长度在36 — 300核苷酸长度(nt)的短DNA片段进行综合分析(图4)。这里将湖泊沉积物古DNA高通量测序实验的典型数据分析工作流程概括为以下8个方面:(1)将待测提取物当中的随机DNA小片段与特异性接头连接,制备测序文库;(2)采用PCR聚合酶链式反应对构建的DNA文库进行扩增;(3)在Illumina测序平台上进行大规模的平行测序;(4)从多重实验当中分离单个样本;(5)切除接头序列并合并双末端测序数据;(6)从环境背景当中识别内源的古DNA分子;(7)对其他DNA来源的污染进行定量;(8)建立古DNA受损模型。更加详细的组学数据分析、相关常用软件和数据库以及可视化工具参考刘永鑫等(2019)、Bolyen et al(2019)、Chong et al(2020)提供的主流分析方法。

图4 湖泊沉积物古DNA分析流程Fig.4 Workflow of lake sedimentary ancient DNA
目前,湖泊沉积物古DNA研究领域使用最广泛的测序策略主要包括基于标记基因序列(marker gene sequences,例如 16S/18S/ITS)(Yang et al,2016a;Wurzbacher et al,2017)和宏基因组学-鸟枪测序(metagenomics-shotgun sequencing)(Pedersen et al,2016)。例如:Liu et al (2021b)利用植物通用引物g和h靶向横断山脉Naleng湖沉积物DNA叶绿体trnL基因内含子P6环,在完成PCR扩增之后进行Illumina HiSeq平台双末端测序,重建了青藏高原东南部过去18000 a植被丰富度演化时间序列。结果发现与气候变暖相关的高山生境丧失导致植物类群丰富度大幅下降,并据此提出未来青藏高原生物多样性保护工作应充分考虑来自湖泊沉积物DNA所提供的古生态学证据。另外,末次盛冰期之后(约15000 —14000 cal ka BP),位于加拿大中西部的劳伦太德冰盖和科迪勒拉冰盖退却,在二者之间形成了一条长约1500 km的无冰廊道。Pedersen et al(2016)在这条廊道瓶颈口区域的Charlie湖和Spring湖采集了9个湖泊沉积物岩芯,通过对各层沉积物进行放射性碳定年并结合花粉、化石和沉积物样本古DNA宏基因组学分析,研究者分别确定了不同动物、植物在这条无冰廊道开始定居的具体年代,并揭示出在12.6 cal ka BP之前,无论是克洛维斯人还是更早的人群都不可能通过这条廊道进入到美洲境内。随着测序成本的不断下降,基于上述两种测序策略获取样本高覆盖度基因组数据变得越来越容易,湖泊沉积物古DNA研究领域的发展前景也更加广阔。其中,通过高通量测序湖泊沉积物古DNA的16S rRNA/18S rRNA、ITS基因或其他特定功能基因位点,即可分析对应部位的微生物群落和不同动植物的物种组成和相对丰度。根据具体的研究问题和选择的测序区域,利用带有样本标签(barcode)的特异性引物扩增相应的标记基因片段并进行测序分析,即可获悉未知环境样本中多样化的生物组成、丰度及其群落结构。
而宏基因组鸟枪测序则是将提取的DNA分子按照一定的长度随机打断来构建测序文库(Quince et al,2017),进而测定样本中全部微生物的基因组。宏基因组学(metagenomics)最早由Handelsman et al(1998)提出,随着时间的推移,其概念的发展经历了从最初的集合基因组(collective genome)、全基因组储存库(whole genome treasure)等基本含义逐渐过渡到完全被“宏基因组学”所替代的过程(Handelsman,2005)。宏基因组分析以环境样本微生物群体基因组为研究对象,可以直接对群落微生物多样性做出全面和深入的阐释。其最主要的优势是不依赖于PCR扩增,能够对提取的古DNA分子进行完整和无偏的测序,分析微生物群落的物种多样性组成、丰度和群落结构,并能够挖掘和研究新的、具有特定功能的基因以及构建相关的代谢通路。
作为一门既年轻又令人兴奋的前沿学科,未来利用宏基因组学重建湖泊、海洋和其他生态系统演化及其对气候变化和人类活动的响应已经是大势所趋,宏基因组也将成为研究古DNA和古生态演化基因组学的重要工具。而且该方法还改进了对湖泊沉积物古DNA降解(如胞嘧啶脱氨基)的验证和定量,允许对古DNA的末端序列进行测序,通过识别DNA的受损模式来帮助区分现代DNA(来自于沉积物中固有的微生物群以及外源DNA污染)和沉积物古DNA(Capo et al,2021),以此作为古DNA真实性的一种度量(Pedersen et al,2016)。然而,目前宏基因组学在古DNA领域仍未得到充分和广泛的应用,尤其是在研究气候环境变化和人类活动对湖泊生态系统微生物群落结构和物种组成以及功能特性演变过程的影响。并且宏基因组学在当前仍然处于起步阶段,在沉积物古DNA应用中的全部潜力和局限性也有待深入探索。
2 湖泊沉积物古DNA在古气候重建和生态演化中的应用
由于气候变化和人类活动的影响,微生物群落组成、结构和多样性的变化已经在湖沼学研究中获得了广泛的报道(Pesce et al,2008;Qu et al,2019;Keck et al,2020)。沉积物样品的DNA分析作为一种新的方法,已被应用于物种组成响应环境变化的分析当中,并且相关研究涉及到的生态系统类型多种多样,在陆地、海洋和淡水生态系统中均得到了广泛的应用(Coolen and Overmann,2007;Madeja et al,2009;Xu et al,2011;Ahmed et al,2018;Hoshino et al,2020)。最早的关于湖泊沉积物古DNA的研究是Coolen and Overmann(1998)利用针对含有类胡萝卜素奥氏酮嗜盐紫色硫细菌的特定引物结合变性梯度凝胶电泳技术(denaturing gradient gel electrophoresis,DGGE)对加拿大Mahoney湖泊沉积物中不同深度的细菌进行16S rRNA基因测序,重建了过去11000 a细菌群落的动态变化过程和湖泊流域的古环境条件以及湖泊盐度、营养状态和水位等参数的演变历史。接下来主要从以下7个方面来阐述湖泊沉积物古DNA的应用。
2.1 湖泊沉积物古DNA与气候和环境变化
由于湖泊浮游生物和微生物对环境条件变化非常敏感,并且具有较高的周转速率,因此常被用作湖泊沉积物古DNA研究中的环境指标(Hou et al,2014;Domaizon et al,2017;Ellegaard et al,2020)。通过研究保存在湖泊沉积物中同一时期的生物与环境数据之间的相互关系,并将其应用于沉积物记录,即可推断过去的环境条件(Smol,2008)。此外,基于DNA的方法可以为传统古生态学指标研究提供新的、更加全面的信息。例如:在形态学计数数据资料中添加或补充分子遗传多样性数据,可以提供关于过去硅藻生物集群更加完整的生态图谱,因为遗传分析能够揭示出部分隐藏在单一形态背后的种间和种内变异,并且特定遗传世系的出现也很可能与环境因子有关(Stoof-Leichsenring et al,2012)。Clarke et al(2019)在挪威芬马克郡东北部瓦兰吉尔半岛的一个小型湖泊沉积物中提取古DNA,通过特异性引物对植物叶绿体基因组trnL基因P6环进行PCR扩增并结合花粉分析进行补充,重建了该地区植物多样性和环境变化的全新世记录(10700 — 3300 cal a BP)。研究发现植物区系在全新世具有连续性和稳定性,在末次冰消期以后、进入全新世之前,丰富多样的植物就已经开始在此生长。另外,花粉记录为8000 — 7300 cal a BP松属木本植物的出现提供了有力补充,而其出现地点位于现代松树森林界限之外以及特定喜温水生植物(水马齿属)仅生活于10200 — 9600 cal a BP的现象也可能反映了早全新世期间更为温暖的气候特征。
湖泊沉积物不仅记录了过去的气候条件,同时为微生物提供了广泛的生态位,并由此导致沉积DNA极端复杂的组成特征。湖泊底栖微生物群落组成不仅取决于沉积物的环境特征和地球化学条件,而且沉积物降解过程中的个体发生学特性也具有显著影响。Vuillemin et al(2016)基于沉积化石色素和细菌与古生菌的16S rRNA基因测序,通过对巴塔哥尼亚玛珥湖地质微生物进行调查和系统发育分析,表明不同的沉积微生物集群源自于特定气候时期的特定湖泊环境条件体系。同样地,Hou et al(2014)通过对蓝藻细菌和真核藻类23S rDNA进行荧光定量PCR扩增和变性梯度凝胶电泳分析,重建了青藏高原库塞湖过去3100 a的光合浮游生物演化历史;对岩芯和古气候指标的地球化学平行分析表明:季风强度驱动的养分有效性、温度和盐度变化以及轨道驱动的太阳辐射强度变化均可能是湖泊主要浮游植物类群丰度随时间变化的原因。Yang et al(2016a)利用qPCR技术解读了晚更新世和全新世以来青海湖奇古菌门及其内在的编码氨单加氧酶α亚基amoA基因丰度动态变化过程,表明整个青海湖沉积物岩芯中具有编码氨氧化基因的古菌丰度变异与青海湖历史时期营养状态和盐度波动相对应。Epp et al(2015)以格陵兰最北端海岸的Bliss湖泊沉积物古DNA为研究对象,通过针对于藻类DNA的宏条形码分析发现该湖泊在全新世期间经历了海相和湖相条件的转变,其所反映的环境变化与之前Olsen et al(2012)通过多代用指标研究包括微型硅藻分析得出的结论相一致。因此,湖相沉积物古DNA不仅为补充古环境指标和追踪数万年的沉积后成岩过程提供了必要的遗传信息,而且也是反映类似于北极和高海拔地区一些低生产力湖泊和周边流域生态系统古气候、古环境及古生态变化的有力工具。
2.2 湖泊沉积物古DNA与生物及生态演化
湖泊沉积物DNA除了可以重建过去的气候和环境变化,还可以研究湖泊历史时期的生物多样性演变并指示湖泊微生物群落组成的构建过程以及不同部位的能量信息。之前由于缺乏长期的监测调查数据,导致对影响和调节水生生态系统化学元素和养分循环重要参与者的年际动态以及多样性变化控制因子的了解不够充分,而湖泊沉积物古DNA为解决类似的古生态学问题提供了机会。例如:Domaizon et al(2013)以法国阿尔卑斯山西北边缘布尔歇湖(Lake Bourget)沉积物岩芯作为研究对象,将基于DNA的古湖沼学方法应用于沉积物的历史记录档案,利用16S rRNA基因和转录间隔区(internally transcribed spacer,ITS)的系统进化分析,调查了过去100 a聚球藻(Synechococcus)的长期多样性变化和动态特征。Wurzbacher et al(2017)在位于德国北部的Stechlin湖中,将通过沉积物DNA反演的微生物群落组成与一系列生物地球化学参数联系起来,证明了微生物类群转换是驱动不同地层沉积物中生物群落组成构建的主要机制,并且这一过程很可能与形成高度生物类群周转的微生物活性指示参数(CO2、CH4的浓度,细菌蛋白产量)紧密相关。从沉积物上层以真菌和细菌为主到下层以古菌为主的优势微生物种类的转变,也说明了湖泊底层沉积物的相对低能量环境和生物丰富度的损失。这与之前在海洋沉积物系统中的发现相类似(Hoehler and Jørgensen,2013;Parkes et al,2014),并且湖泊沉积物中的古生菌和部分细菌世系与深海环境存在很大部分的重叠,表明将海洋系统的一般概念和研究结论转移和推广到湖泊沉积环境的可能性。尽管这两个生态系统的盐度和硫酸盐含量存在较大的差异(Capone and Kiene,1988;Comte et al,2014),但是由于深海研究的巨大成本,湖泊沉积物可能为针对这些“偏远”和“嗜极”微生物的研究问题提供了一种可选的替代方案。另外,对于湖泊生态系统重要初级生产者以及其他生物类群(例如哺乳动物、鱼类和两栖类动物等)的调查不仅为其生物组成和群落结构的长期历史演变特征提供了全新的视角,而且为耦合分子工具和传统湖沼学手段重建湖泊生物多样性历史指明了光辉的前景。未来大尺度(包括不同地理背景和多个湖泊调查)的分子生物学 — 古湖沼学研究将能提供史无前例的数据,实现对不同生物类群历史地理分布的重建,并且对于探索生物多样性对环境参数变化的响应机制也具有重要意义。
更新世末期以及更新世—全新世转变期间一系列显著的气候转型事件深刻改变了北半球的生态系统。这些显著的气候变化不仅直接影响过去的湖泊生态系统,而且通过季节性变化和流域过程调节陆地生态系统溶质和颗粒物向湖泊的迁移,进而间接影响过去不同环境背景下的湖泊生态系统发展轨迹(Leavitt et al,2009;Fritz and Anderson,2013)。然而,由于缺乏覆盖这一关键地质历史时期的精确定年的生物化石和孢粉记录,目前针对更新世晚期的气候振荡和随后的全新世快速变暖所引起的湖泊生物更替和生态演化的速度及其规模一直难以评估。而来自沉积记录的古代DNA作为一种新兴的工具,近年来在重建这一时期的生物多样性动态研究中得到了非常广泛的应用(Parducci et al,2013;Willerslev et al,2014;Parducci et al,2015;Parducci et al,2017;de Schepper et al,2019)。例如:Ahmed et al(2018)利用古代环境DNA宏基因组学和古菌细胞膜脂质化石重建了13.9 — 10.8 cal ka BP湖泊沉积物主要古菌类群的时间演替序列,表明古菌的群落组成和多样性演变可归因于影响底水条件和沉积物—水界面过程的湖水温度变化、快速沉积物填充以及湖冰覆盖的持续时间等因素。尽管湖泊沉积物古DNA为流域古生态过程的环境重建提供了有说服力的间接证据,然而由于考古样本和骨质化石材料的缺乏,其在解释人类迁徙与气候变化之间的耦合关系以及二者对局部尺度上大型生物多样性动态和物种灭绝影响相对贡献的效力等方面比较薄弱。近年来,针对考古发掘材料以及洞穴沉积物的古DNA提取与纯化技术取得重大改进和突破性进展(Slon et al,2017;Zhang et al,2020),使得痕量的化石残骸、动物毛发和耳骨等古代生物材料都可用于古DNA测序和分析。经典定义的化石材料将不仅限于提供地质年代和物理参数等非生物学推论,新旧观念和技术的紧密相连与发展以及与沉积环境古DNA信息解读的深层次融合,为揭示自然选择、环境压力下的人类演化路径和生物多样性改变贡献了史无前例的全新认识与见解。例如:Seersholm et al(2020)在位于干旱半干旱气候区的爱德华兹高原一处洞穴中收集了骨质材料和沉积物样本,通过古DNA提取以及针对脊椎动物线粒体和植物叶绿体DNA进行PCR扩增和Illumina MiSeq平台测序,检测到100种脊椎动物和45种植物类群。研究结果表明:整个更新世 — 全新世过渡期间的气候波动使该地区产生剧烈的生态系统转变,气候的波动导致陆地景观格局以及生境的变化,进而影响到生物生存的环境条件并形成优势动植物种的消长态势。但是气候变化本身并不能完全解释巨型动物在更新世末期的灭绝,新仙女木期间陆生植物与脊椎动物群落对气候的显著差异性响应结果表明人类在该区域的出现也可能导致当地某些动物多样性的永久性丧失。
2.3 湖泊沉积物古DNA与物种定殖及外来物种入侵
了解自然种群的年龄对于研究基本生态和进化过程的速率至关重要,例如物种的定殖、灭绝、入侵、适应和趋异进化等(Hugueny et al,2011;Öhlund,2012;McGee et al,2016),其中如何有效区分本地物种和外来物种对于物种入侵保护目标的制定通常是关键的考虑因素。然而,由于人类活动对物种分布的多样化影响(Crees and Turvey,2015),在超过百年—千年以及万年时间尺度上对于本地种和外来种的定义和区分仍然不够明确,而湖泊沉积物中的DNA检测有望成为研究这些过程的有力工具。近年来一系列的研究发现,在过去万年尺度甚至更长时间内形成的湖泊沉积物中可以检测到古代DNA分子(Jørgensen et al,2012;Giguet-Covex et al,2014;Capo et al,2016)。并且这些研究中发现的生物范围非常广泛,不仅包括水生生物,也包括了陆生动物、植物和微生物等(Giguet-Covex et al,2014;Pedersen et al,2016;Yang et al,2016a)。这是对研究特定环境条件下生物定殖、入侵和适应性特征的演变而向前迈出的关键一步,由此代表了湖泊沉积物古DNA在该领域应用的实质性进展。因此,湖泊沉积物古DNA分析将极大地延长观察生物群落变化的时间窗口,从几十年到几万年甚至可以扩展到更加久远的时间尺度。
研究表明利用湖泊沉积物中的古DNA记录可以确定某一特定或者某些物种确切的定殖时间。例如:Olajos et al(2018)利用在瑞典中部的两个湖泊(Stora Lögdasjön湖、Hotagen湖)沉积物中保存的DNA分子档案,再现了末次冰期之后不同鱼类在湖泊中的定居情况。该研究使用三对特异性引物,通过对沉积物中普遍存在的DNA序列进行扩增分析,追踪了湖泊环境历史时期白鲑鱼的生存特征。研究结果表明白鲑鱼在10000 a前就来到了西博滕省的Stora Lögdasjön湖,原因是由于当时内陆冰川逐渐融化,使得Stora Lögdasjön湖与波罗的海相连;而位于耶姆特兰省的Hotagen湖在冰川消退之后的很长一段时间(距今约2200 a)才发现了白鲑鱼存在的痕迹,并推测此时白鲑鱼的出现很可能与大约2000 a前定居于此的狩猎者和渔民的人为引入有关。该研究首次利用湖泊沉积物古DNA 结合扩增测序技术进行物种识别,为利用环境古DNA信息重建历史时期自然综合体的物种定殖或者外来物种入侵提供了新思路和新方法。通过解析湖泊沉积物中DNA的存在状况和内部特征,为了解湖泊的历史演化提供了全新的角度,更加清晰地认识到自然状态和人为影响下的湖泊生态系统是如何在一段较长的时间内发展起来的。因此通过研究历史气候变化对湖泊生态系统的影响,可以为更好地理解当前的全球变暖将如何影响生态系统提供重要线索。
除了研究动物的定殖与入侵过程及其对湖泊和周边流域生态系统的影响,湖泊沉积物DNA还可作为一种重建过去植被动态变化的手段,帮助研究不同类型植被的发展历史(Parducci et al,2013;Epp et al,2015)、外来植物种的构建与引入(Sjögren et al,2017)、过去植物耕作、驯化及农业和人类饮食的变化(Schlumbaum et al,2008),成为了解过去环境条件(Epp et al,2015)及生态、经济、移民和贸易活动(Schlumbaum et al,2008)的代用指标和有力工具。本世纪初,Willerslev et al(2003)首次研究了保存在永久冻土和洞穴沉积物中的古代植物群落的遗传特征,随后Willerslev et al(2007)利用对植物叶绿体DNA标记基因(cpDNA的rbcL基因和trnL基因内含子)进行PCR扩增和测序,进一步证明了在相当长的时间跨度内(>400000 a),可以从少量的沉积物样本中检索到多样化的植物类群。这种方法不依赖于可识别的植物化石的保存,对于那些无法被传统古生态学方法发现的植物种是非常有利的。不过值得一提的是,湖泊沉积物古DNA宏条形码技术在解析过去植被群落演化研究中只是提供了花粉分析的补充工具(Pedersen et al,2013),而非代替工具。另外,几项观察表明:从沉积物当中获得的植物DNA序列大多是当地起源的,而不是远距离传播的结果,但是植物花粉则可能来源于更加广阔的地区(Willerslev et al,2007;Boessenkool et al,2014;Sjögren et al,2017)。综上所述,继花粉调查之后,湖泊沉积物古DNA分析提供了来自周围环境的最全面的信号特征。
2.4 湖泊沉积物古DNA与人类活动
重建由人类活动所驱动的、塑造地球形态的动力学过程对于理解过去人类和环境的相互作用并帮助应对目前的全球变化至关重要。例如:影响侵蚀的景观塑造很可能代表了最古老的人为地质过程(Ruddiman,2003),而与此同时,气候的变化也会影响景观的转变。因此,区分气候和人类活动对环境变化的影响往往具有一定的困难和挑战性。而应用于湖泊沉积物古DNA的宏条形码技术则能够以较高的分辨率再现区域生物演化的详细历史以及人类活动对景观的塑造过程。Giguet-Covex et al(2014)以法国阿尔卑斯山北部Anterne湖泊沉积物DNA作为研究对象,利用特异性引物和通用引物对哺乳动物线粒体16S基因和植物叶绿体trnL(UAA)基因内含子进行PCR扩增及高通量测序分析,首次在物种层面上以高分辨率重建了6000多年来的植被覆盖和畜牧业发展历史。通过与之前的流域侵蚀事件频率重建资料相比较,研究者发现由于人为森林砍伐和对牛羊的过度放牧,在铁器时代晚期和罗马时期的流域侵蚀程度最为强烈,并引发了Anterne流域全新世以来最严重的侵蚀危机。湖泊沉积物古DNA不仅以更加细腻的程度重构区域生物的演化历史和生态过程的发展动态,而且相比于经典的古环境研究手段,其在区分塑造流域景观格局的气候变化和人类驱动因子的应用中更是具有无可比拟的优势。因此,湖泊沉积物古DNA分析在未来很可能成为重建全新世古环境演变的一种全新方法,从而帮助人们更加全面和深入地了解人类世期间气候、环境和人类活动之间的复杂相互作用。
虽然长期的时间序列研究已经表明人为因素能够对湖泊造成巨大的压力和威胁,但是对于在人类世期间湖泊生物多样性如何响应环境条件的波动以及在何种程度上发生变化,目前的研究尚不充分,尤其是对于微生物的变化。而古基因组的重建,包括以微生物为主的真核生物古基因组数据的挖掘,使人们对古生态系统和环境的发展有了更全面的认识,从而为了解过去的生态系统过程提供更加深刻的见解。即使来自相对较小湖泊的沉积物古DNA也是记录过去湖泊生态系统及其周围流域景观的良好档案,由此成为描述人为压力作用下湖泊及其流域古生态演化的一种可靠工具和手段。Kisand et al(2018)通过采集拉脱维亚东部雷泽克内区Lielais Svētiņu湖泊沉积物样品,对真核生物18S rRNA的V4区进行PCR扩增及高通量测序,并结合烧矢量和花粉分析,重建了过去14500 a以来湖泊生物多样性及周边陆地古生态系统的演变过程。研究发现“人类世”(<2000 cal a BP)期间湖泊浮游生物以绿藻类为主,而在全新世之前和早全新世期间则主要是真菌和高等植物占优势;另外,尽管当前的全球变暖与全新世大暖期均使湖泊中的一些浮游生物丰富度增加,但是二者的响应机制存在明显的区别:中全新世水生生物的繁荣与温暖气候条件下湖泊更高的营养状态紧密相关,而过去2000 a以来湖泊真核生物丰富度的增加则受到人类活动的潜在影响。该相对原始的湖泊沉积物中栽培品种植物花粉和沉积炭屑的增加,指示了当地和区域植被的相对开放程度。Keck et al(2020)利用在48个温带湖泊沉积物岩芯中保存的古DNA分子档案,重建了现代真核微生物群落与19世纪的微生物集群。该研究发现在两个时间点当中,湖泊微生物群落组成和所有的功能类群均发生了显著的变化。在全球范围内,低地湖泊明显受到当地人类活动影响,人为因素的驱动和干扰尤其有利于光合自养和混合营养微生物的繁荣,而处于偏远地区的高海拔湖泊受到的影响则相对较小。这项研究提供了一幅前所未有的自19世纪以来湖泊真核微生物多样性的演变图景,有力地说明了保存在湖泊沉积物中的DNA是补充传统气候变化代用指标和研究人类活动干扰及环境变化背景下生物多样性动态和生态系统运行轨迹的重要工具。
2.5 湖泊沉积物古DNA与生态功能基因演化
潜在的功能基因通常允许推断细菌和古菌的功能特征。以蓝藻为代表的具有固碳(Domaizon et al,2013)和固氮(杨文焕等,2020)功能的微生物类群,在介导全球湖泊生态系统关键地球化学元素的物质循环过程中发挥着至关重要的作用,并且对于调节水生生态系统平衡、改善水质以及维持湖泊的生态系统服务功能和生态健康具有重要意义。然而,由于人类活动导致的全球变暖和湖泊养分有效性的不断增加,全球湖泊遭受了前所未有和广泛的蓝藻水华危害(Schindler et al,2012;Michalak et al,2013;张娇等,2016;鲁韦坤等,2017;唐晓先等,2017)。相关的研究报道甚至已经发现有些蓝藻能够分泌有毒物质(Martínez de la Escalera et al,2014;Monchamp et al,2016),进而导致湖泊水质的破坏和降低,尤其是在富营养化的条件下。例如:Monchamp et al(2016)利用保存在瑞士格里芬湖(Greifensee,已经处于富营养化阶段)和苏黎世湖(Lake Zurich,处于中营养化阶段)分层沉积物中的DNA重建了过去200 a的蓝藻细菌群落结构和系统发育特征,发现格里芬湖中的蓝藻在最近十几年开始出现编码微囊藻毒素(microcystin)的功能基因mcyA,而在苏黎世湖中,mcyA基因的存在已有超过百年的历史。类似地,Martínez de la Escalera et al(2014)根据一个亚热带淡水生态系统沉积物岩芯中的古代DNA重建了自1846年以来的微生物集群演变过程,结果表明21世纪的微生物样本组成明显区别于19世纪中期,并且仅在最近的岩芯中发现了合成蛤蚌毒素(saxitoxin)功能基因(sxtU)的存在。
此外,这种方法也可以扩展到其他一些编码重要生态功能的基因,例如涉及氮、磷等元素的吸收、碳的固定、烷烃的分解、甲烷的还原与氧化、对抗生素和污染物的抗性以及氨氧化基因(Krause et al,2014;Yang et al,2016a;刘洋荧等,2017;王朱珺等,2018;Liu et al,2019)等。诸多研究表明:通过对湖泊沉积物中保存的古DNA历史档案进行深入分析,探究湖泊历史时期微生物群落组成和结构以及功能特性的演变,进而可以帮助探讨湖泊生态系统的长期变化历程,解决一系列环境变化和人类活动影响下湖泊生态系统的响应以及各因子之间的互作等相关的重大生态议题。
2.6 湖泊沉积物古DNA在细胞器基因组研究中的应用
细胞器基因组包括两种类型,即叶绿体(chloroplast)和线粒体(mitochondrion)中的基因组。细胞器DNA研究生命特征和进化关系的主要优势在于其结构比较简单,并且具有单倍性和高拷贝数的特点。细胞器基因组已成为古DNA研究领域的重要工具,并对相关成果的获得起到了至关重要的促进作用(Krause et al,2010;Sjögren et al,2017;Seersholm et al,2020)。尤其是近年来高通量测序技术的推动,完成测序的细胞器基因组数量不断增加(图5)。而古DNA研究领域正是从灭绝动物(Higuchi et al,1984)和埃及木乃伊线粒体DNA(Pääbo,1985)分子克隆正式开启的。线粒体基因组DNA遗传信息的表达受到线粒体本身以及细胞核基因组的共同调控作用,因此线粒体只是一种半自主性细胞器(Anderson et al,1981)。叶绿体普遍存在于所有的植物和一部分原生生物当中,与光合作用紧密相关,并且叶绿体基因组DNA序列存在较高变异的微卫星(microsatellite)区域,在植物的分子进化以及群体遗传研究当中具有非常广泛的应用。

图5 叶绿体和线粒体细胞器基因组全长序列数量逐年统计(来自GenBank数据库)Fig.5 Full length sequence of organelle genome in GenBank database
在湖泊沉积物古DNA研究领域,重建植物演化历史通常以叶绿体细胞器基因组作为主要研究对象。例如:Golenberg et al(1990)在中新世湖泊浅水沉积物当中发现了木兰属的植物叶片化石,从中获取了其叶绿体rbcL基因并完成对DNA序列的PCR扩增和测序。基于建树方法和系统发育推断分析以及该基因不同于任何已知的rbcL序列的事实,研究者将其归结为首个测定的中新世植物古DNA序列,从而将植物古DNA保存年代的上限追溯到数百万年以前,并为进化生物学的发展做出了划时代的贡献。近年来测序技术的进步更是允许对来自化石样本的植物古DNA进行分析,并因此得以实现对古植物群系的分子重建(Parducci et al,2017)。除了对以化石形式保存的物种开展演化和生态变化古基因组研究,对没有形成化石记录的生物类群细胞器基因组进行全基因组分析同样具有揭示种群遗传信息的潜力。例如:Lammers et al(2021)对两个末次盛冰期的湖泊沉积物样本古DNA进行宏基因组鸟枪测序,重构了微拟球藻(Nannochloropsis)的全叶绿体和线粒体基因组,以探索湖泊内部的种群基因组变异特征。结果表明每一个细胞器基因组可以分配到对应的已知湖沼微拟球藻类群,并且存在至少3个单倍型(haplotypes)。由此可见,湖泊沉积物古DNA不仅可应用于种群基因组分析,同时也为环境古基因组研究打开了一扇全新的窗口,最终将能够发挥湖泊沉积物古DNA的全部潜能。
由于古代冻土拥有保存DNA分子的天然优良条件,因此成为迄今为止最常用的植物古DNA分子来源。但是在没有地层扰动和再沉积过程发生的前提和条件下,冻土中提供的植物古DNA分子仅仅代表了位于采样点附近范围内的古代植物类群。而湖泊由于具有较大的流域面积,最近的研究表明来自湖泊沉积物的植物古DNA在植物群系发展历史的重建过程中,成为更加可靠的工具(Epp et al,2015;Pansu et al,2015;Domaizon et al,2017;Parducci et al,2017)。并且湖泊几乎在全球所有环境当中都有分布(图3),因此为研究不同气候和环境条件下的植被生态演变提供了便利。湖泊沉积物古DNA分析为古代植物种群和群落演化研究提供了全新的视角,最终将提供更高的分类学分辨率和更精确的丰度估计。
2.7 湖泊沉积物古DNA在古生态学时间序列分析中的应用
近年来随着环境DNA研究的深入发展以及新技术和新观点的不断涌现(Pedersen et al,2015;Epp,2019;Ruppert et al,2019),湖泊沉积物古DNA开始为在百年 — 千年尺度甚至万年尺度上研究种群、群落和生态系统的演变动态提供强有力的时间数据资料支撑。因为环境古DNA并不依赖于在形态学上可辨认的生物遗骸,几乎所有在环境当中留有DNA的生物均可被检测到,从而填补了古生态演化过程的重要数据空白。例如:Zhang et al(2021)利用沉积物古DNA宏条形码和qPCR技术首次重建了中国东北部沙尘区一个火山湖浮游植物群落过去2000 a的演化历史。研究发现AD 1400之前湖泊蓝藻细菌丰度上升主要归因于温暖干旱的气候条件,而AD 1400之后蓝藻生物量和丰度升高主要是由于西伯利亚高压增强导致北半球中纬度地区沙尘通量显著增加,包括磷、铁和钙在内的重要生态养分经由沙尘沉降物携带输送进入湖泊,从而造成浮游植物群落结构的转变。因此过去几百年的风成降尘对中国北方湖泊生态系统的改变很可能已经超过自然气候变异的影响。然而相比于水生生态系统,极地沉积环境能够在更长时间尺度上解析动植物的演化过程,尽管这些样本只包含了周围生物多样性的很小部分。例如:Willerslev et al(2003)通过对西伯利亚东北部永久冻土进行遗传分析,完成了对400000 a前白令陆桥植被和大型动物古DNA的提取和扩增,第一次在晚更新世或全新世动植物群遗骸以外的地层沉积物当中检测到古群落的遗传记录及其组成和类群多样性的剧烈变化。由于当时的技术手段限制,PCR产物分析只能依赖于耗时昂贵的克隆和Sanger测序。随着下一代测序技术的发展,逐步实现了将Willerslev et al(2003)的研究成果拓展到解析更大规模和尺度上的古代动植物群落演变。例如:Willerslev et al(2014)利用从21个北极高海拔地点收集的242个永久冻土沉积物样本开展了首个极地附近植物多样性的大尺度古DNA宏条形码研究,重建了过去50000 a的北极植被演化历史,同时以线虫多样性作为模拟植被覆盖和土壤质量的代用指标,并探索了大型食草哺乳动物的饮食行为。而Zobel et al(2018)则根据Willerslev et al(2014)中的植物古DNA数据集结合现有的已发表菌根信息为每一种植物类别分配对应的菌根类型,展现了过去50000 a植物与异养微生物(菌根和固氮细菌)互利共生关系的详细演化过程,表明植被—异养生物互惠共生特性结构的改变可能是对过去历史时期环境条件变化的响应。然而这一方法具有可行性的一个基本假设与前提是当今的植被与共生生物之间的关系与历史时期并无显著差异,即植被—异养生物共生关系随时间保持稳定,且在现代环境条件下没有发生空间上的解耦。而利用针对于植物及其共生生物的环境古DNA来调查二者之间的长期共存模式则可以很好地验证这一假设,由此也代表了以湖泊沉积物古DNA为基础,根据现代生态学理论研究地质历史时期的种间关系、群落动态以及生态演化的全新方向。
环境古DNA不仅提供了高分类学宽度的长期历史记录(图6),而且能够对一些重要的生态理论、生物多样性假设和模型进行有效检验(Bálint et al,2018),这对于理解全球变化以及人类活动对湖泊及其周边生态系统的影响至关重要。此外,通过对湖泊沉积物古DNA记录的挖掘,不仅可以揭示出流域生态与演化的变化趋势,而且也能用于理解生态过程与演化事件之间的动态特征(即出现于相似时间尺度上发生的演化和生态过程之间的相互反馈作用)。然而作为一种时间生态数据资料的重要来源,湖泊沉积物古DNA仍然未被充分利用和挖掘。

图6 时间生态学中环境古DNA时间序列的生态位(根据Bálint et al(2018)重绘)Fig.6 A niche for environmental ancient DNA time series in temporal ecology (redrawn according to Bálint et al (2018))
近年来在高通量测序技术方面的进步促进了记录不同环境中微生物群落时间变化纵向测序数据集的增加(Gilbert et al,2012;Shade et al,2013;David et al,2014),从而为研究湖泊沉积物中微生物群落的稳定性及其对扰动(气候变化和人类活动)的响应历史提供了独特的时间生态学视角。例如:Fuhrman et al(2006)利用高分辨率分子指纹图谱阐述了过去4.5 a南加利福尼亚海岸海洋浮游细菌群落组成具有高度可重复的时间模式,不同微生物类群的丰度和分布格局可进行预测并受到一系列生物和非生物环境因子的显著影响,表明细菌群落可能具有调节生态系统功能和响应的潜力。类似地,Gilbert et al(2012)通过连续6 a、以每隔一个月为时间间隔采集位于英国普利茅斯温带海岸近表层水体样本,利用高分辨率16S rRNA标签焦磷酸测序描述了迄今为止最长的现代微生物时间序列分析研究。结果显示细菌群落具有强烈的重复性季节模式,在所有年份中细菌多样性均在冬季达到峰值,日长变化解释了>65%的群落多样性变异,并且环境变量的季节性变化比营养相互作用更加重要。除此之外,从全球各大洋到人类微生物组等多样化的环境条件,一系列纵向时间测序数据分析技术在基于标记基因扩增子和宏基因组学分析中的成功应用,为揭示历史时期湖泊微生物动态特征和生态演化的周期性模式,建立预测模型以及量化影响群落行为的不规则因素,结合其他相关气候环境变化和人类活动代用指标阐述其内在响应机制等方面提供了独特的范例和可供解决的研究方案。
3 开展湖泊沉积物古DNA研究面临的局限和挑战
活体生物内部的完整修复系统和运行机制保证了面对外界破坏作用和环境压力时能够及时高效地完成双链DNA分子结构的损伤修复,从而保证DNA复制的忠实性和准确性,帮助维持生命活动的正常运转。然而生物死亡后,耗能过程的酶修复途径不再发挥作用,持续的微生物降解导致多种DNA损伤类型不断积累(表2),呈现出碎片化和多种化学修饰的典型特征。因此,处于高浓度现代DNA环境中的湖泊沉积物样本,古DNA分子的低含量和降解特性使其分析变得更加复杂(Peyrégne and Prüfer,2020),成为利用湖泊沉积物古DNA序列数据开展生态演化过程研究的第一个主要障碍。此外,相比于无处不在的现代环境DNA,古DNA研究领域复杂性的首要挑战源于提取的湖泊沉积物样品中内生DNA含量过低,造成PCR扩增目标序列的起始拷贝数较小,进而出现大量外源污染DNA优先扩增的不利现象(Gansauge and Meyer,2013;Rohland et al,2018)。这些敏感性污染源包括沉积物样品内部和表面附着生长的微生物以及渗透进入样品中的现代环境DNA(如空气悬浮颗粒),例如不当的人为触摸和操作带来的交叉污染、实验室设备和试剂菌群污染(Llamas et al,2017;de Goffau et al,2018)等。只有样品中污染DNA分子含量足够低,才能保证PCR循环进入指数阶段后顺利实现大量真实古DNA的快速扩增。因此古DNA分析过程的每一个步骤都必须采取特定的预防措施(Fulton,2012),将污染发生的可能性和程度降到最低。
以下3种方法可使上述问题得到有效缓解:(1)为尽最大可能避免实验室污染,样本前处理、DNA提取、文库制备和PCR扩增应分别在特定的洁净室进行操作,尽量远离现代分子生物实验室(Pääbo et al,2004),同时实验人员应全程穿戴防护服、防护帽和专用手套;(2)对所有试剂和设备进行紫外光照射或氧化剂处理,保证将潜在污染源DNA充分降解(Pääbo et al,2004;Pilli et al,2013);(3)上述措施仍不可避免一些短DNA分子片段的残留,因此可对所有试剂、DNA提取和文库制备过程设置重复处理和阴性对照,以有效检测潜在低浓度DNA污染(Parducci et al,2017;Rohland et al,2018)。由于污染的普遍存在以及有效区分潜在污染和真实古DNA序列之间的技术挑战,湖泊古代微生物群落演化研究的可靠性风险被进一步放大。加之环境微生物极端复杂,从湖泊沉积物中获取到的新核酸序列,很有可能代表的是未被确认的世系而不是新的古代物种或基因型(Fulton and Shapiro,2019)。因此湖泊沉积物古DNA研究起始阶段的风险评估变得极为重要,传统的真实性检验标准可能不再具有充分性保证,相关领域研究人员在设计和执行古DNA研究时需要详细阐明组学数据的获取过程并给出序列真实性的具体评判标准(Gilbert et al,2005;Briggs et al,2007;Pedersen et al,2016)。
4 组学技术在古DNA研究中的应用及进展
近年来,随着古DNA恢复技术的进步和不断完善,特别是下一代测序技术在古DNA分析中的广泛应用,涉及古DNA的研究经历了一场真正的技术革命(Orlando et al,2015;Parducci et al,2017;Kistler et al,2020)。而DNA高效分离方法的开发和应用以及一系列防治外源污染的措施对古DNA革命的成功至关重要。然而由于技术瓶颈的限制,以前的研究仅仅限于分析有限数量的个体遗传信息,而从古DNA研究领域开创至今,古DNA研究已经发展到对越来越多的古代个体、灭绝物种、粪体化石、湖泊沉积物、洞穴沉积物、永久冻土、极地冰芯等环境古DNA进行标记基因数据分析和全基因组测序,在近年来甚至已经深入到古表观基因组特征的挖掘(Orlando et al,2015;Cappellini et al,2018),从而赋予了其在描述整个进化时间尺度上进行调控变化的潜力。并且古DNA的研究重点也已经从专注于线粒体DNA小片段检索单个物种(Shapiro et al,2004)发展到多个物种(Lorenzen et al,2011),再到包括上百个样本的全基因组鸟枪测序(Allentoft et al,2015)以及基于单核苷酸多态性(single nucleotide polymorphisms,SNPs)捕获的群体基因组学(Haak et al,2015),至此,古DNA革命对气候变迁、人为活动和生态演化以及人类的迁徙与融合等历史事件的解析和还原程度已经令人惊叹不已。例如:关于人类演化历史研究中的基因组革命就源自于全基因组数据的深入探索(Green et al,2010),这意味着人们将能对所有的遗传信息进行一次性的大规模解读,而不再局限于对线粒体或叶绿体基因组DNA这样的小片段进行分析。上述的一系列进展使现在能对百万年尺度上的环境样本古DNA进行提取并测序,而新兴测序技术的进步更是为古DNA研究领域的发展注入了新的活力,由此古DNA技术让诸多细致入微的历史重建工作成为可能。
5 湖泊沉积物古DNA研究展望
过去30多年的古DNA研究逐步确立了地质时期生命遗传物质保存的基本预期和上限(Parducci et al,2017;van der Valk et al,2021),不同古DNA相关的研究领域采用特定的针对性策略进行了富有成效的、跨越时间和空间尺度的探索,并通过基因组学方法将覆盖不同时空尺度的多样性和演化动态纳入分子遗传的整体框架,使得古DNA研究取得长足进步 —— 从对晚全新世样本线粒体短片段的恢复发展到追溯百万年尺度上全基因组序列的组装,从而深刻改变了人们对地球早期生命形式的起源与演化、适应性与灭绝过程以及塑造当今生物多样性格局的重大气候变异、历史迁徙与混合事件的理解。以湖泊沉积物为中心的古DNA研究为重建古气候环境,揭示湖泊生物多样性动态及流域生态系统演化提供了独特的分子生物学视角,不仅为传统孢粉学研究提供了高分辨率的有力补充,更为从核酸水平窥探更新世 — 全新世气候振荡和强烈人为干扰之下湖泊生态系统的响应特征和内在机制及其多样化的发展轨迹提供了全新的分子古生态学窗口,在湖泊分子生态重建研究的不断成熟过程中扮演着关键角色。基因组革命的发展进一步为湖泊重要的演化过程带来新的方向和见解,例如农业作物的驯化、外来动植物的入侵及其对流域生态系统的影响、生态功能基因的演化等,有助于从根本上评估和验证长期以来存在的不同模型和假说。
尽管最近科学界对湖泊沉积物古DNA分析表现出极大的兴趣,然而利用古DNA宏条形码技术或者宏基因组学洞察过去的群落变化仍然存在一些技术层面的挑战,主要包括污染的有效控制、特定标记基因的选择和针对性的生物信息学分析策略等。目前对于开展沉积物古基因组学探索跨越不同领域的新兴问题和存在已久的困惑仍然处于起步阶段。基于前文回顾,本文认为在未来数年亦或数十年之内,湖泊沉积物古DNA的研究或许可从以下几个方面努力,以期取得突破:(1)深入了解不同沉积档案中物种特异性古DNA的埋藏过程;对于处于非冰冻状态的水生沉积系统,影响古DNA沉积和保存的确切物理 — 化学过程仍然知之甚少,亟需实验层面的多角度、跨分类群沉积物古DNA的解释。(2)将湖泊沉积物古DNA分析与其他技术手段(如有机质分子组成分析)进行交叉对比。例如:可通过与湖泊沉积物有机分子组分傅立叶离子回旋共振质谱(Fourier-transform ion cyclotron resonance-mass spectrometry,FT-ICRMS)分析技术对接,利用FT-ICR-MS解析湖泊生态系统不同演化阶段的有机分子组合,通过化合物中脂肪族和木质素的相对占比探究有机质的内外来源,追踪历史环境条件和土地利用方式对有机质分子水平变化的影响,从分子组成角度剖析湖沼生态系统对自然和人为活动的响应。因此,从古DNA层面和有机分子组成层面反演古气候环境和湖泊生态演化过程的结合不仅可以对各自的真实性、可靠性进行相互验证,对于深入了解其内在的变化机理和提供多角度的证据也具有重要意义。(3)除了古基因组学的发展,以古蛋白组学和古脂类组学为代表的新兴组学技术在近年来不断焕发出新的活力。二者分别以古代生物蛋白和脂质分子为研究对象,致力于古蛋白质组学测序和脂质代谢组学研究,通过直接观测关键进化时刻的古蛋白序列来增强蛋白质结构和功能演化的理解,并利用高分辨率脂质分析揭示不同化合物之间生物化学及生物合成过程的联系或者发现新的生物标志物。因此未来湖泊沉积物古DNA研究结合古蛋白组学和古脂类组学的多代用指标方法,将为处理更加错综复杂的湖泊生物分类群和功能特性及其与气候环境变化和人类活动影响之间的深层次关联提供更加综合的方法,并促成对地球生命演化历史的全新见解。
