基于线粒体Cytb基因序列的山瑞鳖 保种群体遗传多样性分析
2022-06-14黄松茂赵玉姣张铭枭刘霄姚创孔令富武祥伟
黄松茂 赵玉姣 张铭枭 刘霄 姚创 孔令富 武祥伟

摘要:为研究云南地区山瑞鳖(Palea steindachneri)保种群体遗传多样性和遗传结构。该研究基于线粒体细胞色素b(Cytb)基因,利用PCR扩增和测序技术,分析了2个云南地区山瑞鳖保种群体的单倍型分布情况、遗传距离、遗传结构和群体历史动态等,扩增得到F3代群体和F4代群体共45个个体的线粒体Cytb基因序列。用DNAStar和MEGA软件剪切校对后序列长度为1000bp,碱基A、T、C和G的平均含量分别为31.28%、27.19%、30.18%和11.35%。在两个群体中共检测到9种单倍型,其中F3代群体有7种单倍型,F4代群体有3种单倍型,单倍型间没有明显的谱系结构,亲缘关系较近。两个群体内遗传背景单一,个体间平均遗传距离较小,随着保种代数的增加群体的遗传多样性水平也在逐渐降低,近交现象明显。中性检验和核苷酸歧点分布图显示,F3代群体和F4代群体历史上可能经历过扩张。研究结果表明,该保种群体的群体内遗传多样性较低,在今后的保种过程中应采取不同的保种策略,避免近亲繁殖。
关键词:山瑞鳖;Cytb基因;保种群体;遗传多样性;遗传结构
中图分类号:Q343.2;S968.3文献标志码:A
山瑞鳖(Palea steindachneri),隶属于龟鳖目(Testudines)、鳖科(Trionychidae)、山瑞鳖属(Palea)[1],保护等级为国家二级保护动物,在中国分布范围不广,云南省是山瑞鳖的主要栖息分布地之一。山瑞鳖形态与中华鳖(Pelodiscus sinensis)相似,个体较中华鳖大且生长快,性格比较温顺,属于亚热带鳖类,有较高的药用价值[2],早些年人为捕捞对山瑞鳖种群结构破坏较大。再加上近年来修建水利工程等活动严重破坏了山瑞鳖原有的栖息地,使其在自然界的竞争中处于劣势状态,现在野外环境下已很难觅其踪迹。云南省野生山瑞鳖的资源量严重下降,保护山瑞鳖现有种群,恢复山瑞鳖自然种群结构已刻不容缓。
有关山瑞鳖的研究主要集中在生物学、驯化养殖和人工繁育等方面[3],分子水平的研究主要是对其亲缘关系和系统学地位进行鉴定[4]。作为珍稀濒危保护动物,关于云南地区山瑞鳖群体的遗傳多样性研究还未有人报道。该研究团队自2008年开始搜集云南山瑞鳖野生群体,以自然繁殖的方式进行人工保种,随着人工保种年限的增长,本研究团队发现保种后代的繁殖性状出现明显的衰退,主要表现在受精率降低、受精后孵化率减小且孵化出膜后幼体存活率低,初步推测是由于亲本的遗传多样性低,亲缘关系近,可能存在近交衰退现象。因此,了解山瑞鳖保种群体的遗传多样性和遗传结构,有助于为山瑞鳖制定科学的保种方式,制定不同的繁殖策略,防止种质资源退化。本研究采用线粒体细胞色素b(Cytb)基因作为分子标记,利用PCR和测序技术获得2个山瑞鳖群体样本的线粒体Cytb基因序列,通过序列比对、系统进化树构建和群体遗传结构分析等,对云南地区山瑞鳖保种群体的遗传多样性和遗传结构进行了初步研究。克服了形态特征鉴别困难、受环境影响变化较大的缺陷,可以更准确地梳理群体的系统进化关系,为云南地区山瑞鳖持续保种提供科学依据。
1 材料和方法
1.1 试验材料与总DNA提取
实验所用山瑞鳖样本为云南地区人工驯化养殖群体。其中F3代群体12只,F4代群体33只,剪取少量裙边组织保存于95%酒精中备用。总DNA提取参照常规的酚-氯仿[5]方法进行提取。使用1%琼脂糖凝胶电泳检测DNA质量,并采用超微量分光光度计(NP80Touch)测定DNA的浓度,于-20℃保存待用。
1.2 线粒体Cytb基因序列扩增
使用已发表文献中的山瑞鳖线粒体Cytb基因扩增引物,引物序列为turtle-cytb-F1:GGACTYTAACCAAGACCAATG和turtle-cytb-R1:TCAATCTTTGGTTTACAAGAC[6],引物由上海生物工程有限公司(Sangon)合成。PCR反应体系总体积为20μL,其中12.6μL超纯水,2.5μL10×PCR buffer,dNTP Mixture(2.5mM each)1.5μL,上、下游引物(10 μmoL /L)各0.5μL,Taq DNA聚合酶(5U/μL)0.4μL、DNA模板2μL。PCR反应程序为95℃预变性5min;95℃变性1min,52℃退火40s,72℃延伸1min,35个循环;最后72℃充分延伸10mins,4℃保存。取5μL PCR扩增产物用1%琼脂糖凝胶电泳检测,选择电泳条带清晰的PCR产物直接委托北京擎科生物科技有限公司昆明分公司进行双向测序。
1.3 数据统计与分析
Cytb基因序列测序结果使用DNAStar[7]软件包中的SeqMan程序先对测序结果的ab1文件手动剪切首尾两端不准确的序列,之后对正反引物序列进行比对拼接,并对拼接结果检查校正。使用MEGA10.1.8[8]中的clustalw(参数为默认参数)对齐序列,计算序列的碱基组成、变异位点和遗传距离等,并构建单倍型最大似然树(参数为:使用bootstrap method算法,检验次数为500次,遗传距离计算使用Kimura 2-parameter model,插入/缺失碱基处理方式为partial deletion50%)等。对齐后的序列用DnaSP6.12.03[9]软件分析群体的单倍型数目、单倍型分布、单倍型多样性指数和核苷酸多样度等,并绘制核苷酸歧点分布图,使用gengrate程序生成单倍型信息文件用于Network10.2.0.0[10]软件绘制单倍型网络图,分析单倍型之间的进化关系。利用Arlequin3.5[11]软件进行群体分子方差分析(AMOVA),计算群体内和群体间的自由度、方差总和、变异组分和变异贡献率以及Tajima's D[12]和Fu's Fs[13]中性检验,结合核苷酸歧点分布图检测群体内和群体之间的遗传分化和群体历史扩张等。4C1F193C-5322-4D34-A264-0695750EB7AF
2 结果与分析
2.1 山瑞鳖Cytb基因碱基组成和变异分析
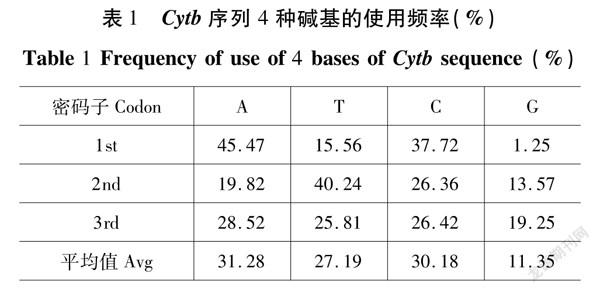
经对齐校对获得2个群体45条山瑞鳖Cytb基因序列,序列长度为1000bp。序列中四种碱基A、T、C和G的平均含量分别为31.28%、27.19%、30.18%和11.35%,碱基A+T的含量(58.47%)高于G+C的含量(41.53%)。在密码子第1位表现出了对碱基A和C的偏好性,使用频率分别为45.0%和37.5%。在密码子第2位,碱基T的使用频率(40.2%)最高,分别是A和C的2.0倍和1.5倍,更是G的3.0倍。而在密码子第3位上,四种碱基使用频率的差别不大(表1)。在这1000bp 序列中,共检测到9个多态位点,占分析位点总数的0.90%。其中5个为单一多态位点,4个为简约信息位点。
2.2单倍型分布情况和遗传多样性分析
45个个体共检测到9种单倍型(表2),编号为Hap_1~Hap_9。单倍型Hap_1出现频率最高(82.22%),分布于F3代群体和F4代群体,其余单倍型均为某个群体的个体特有,F3代群体共有(Hap_1~ Hap_7)7种单倍型,F4代群体(Hap_1、Hap_8和Hap_9)有3种。使用Network做出单倍型的网状关系图发现,单倍型Hap_4位于所有单倍型的中间,其他单倍型都是从单倍型Hap_4分化出去的,从单倍型Hap_4分化出单倍型Hap_8再分化出最大的单倍型Hap_1。单倍型Hap_2、Hap_3、Hap_4和Hap_8联系密切,从单倍型Hap_4分化出单倍型Hap_5和单倍型Hap_7,再由Hap_5分化出单倍型Hap_9(图1)。
基于2个山瑞鳖群体Cytb基因单倍型构建的最大似然树特点可以看出,2个群体体明显形成了2大分支,单倍型Hap_7单独聚为一支,单倍型Hap_1、Hap_6和Hap_8聚为一支,单倍型Hap_2、Hap_3和Hap_4聚为一支,单倍型Hap_5和Hap_9聚为一支,F4代群体单倍型与F3代之间没有明显的分化(图2)。结合单倍型的网状关系图可以看出,虽然单倍型Hap_7单独聚为一支,但是这种单倍型还是由单倍型Hap_4分化出去的,两个群体不存在明显的谱系结构。
2.3 群体遗传多样性
根据山瑞鳖2个群体的遗传多样性参数可以看出,在单倍型参数中,F3代群体的单倍型数量(12)和单倍型多样性(0.773)最高,F4代群体的单倍型多样性(0.119)较低;核苷酸多样性参数表明,F3代群体的核苷酸多样性为0.00184,遗传多样性比F4代群体(0.00042)高(表3),F4代群体可能出现了退化;两个群体都表现出了高单倍型多样性和低核苷酸多样性的特点。
F3代群体个体间的平均遗传距离为0.00183,F4代群体个体间的平均遗传距离为0.00042,F3代群体的遗传多样性比F4代群体高,F3代群体与F4代群体的群体间遗传距离为0.00138(表4),表明群体内、群体间遗传距离较小,亲缘关系较近。
2.4遗传结构和群体历史动态
山瑞鳖群体AMOVA分析结果表明,群体间自由度为1,群体内自由度为43,群体间方差总和(2.923)较群体内(16.811)小,群体内的变异组分(0.39094 Vb)和变异贡献率(73.10%)较群体间的变异组分(0.14385 Va)和变异贡献率(26.90%)高(表5)。表明变异来源主要在群体内,群体间差异不大,两个群体亲缘关系较近。
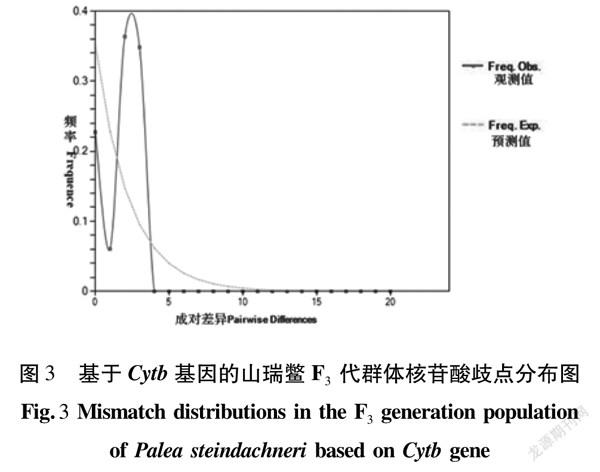
基于山瑞鳖Cytb基因单倍型对两个群体分别进行中性检验,检验显示Tajima′s D test和Fu′s Fs test均为负值(表6),F4代群體Tajima′s D test和F3代群体Fu′s Fs test差异显著(P<0.05),F3代群体Tajima′s D test和F4代群体Fu′s Fs test差异不显著(P>0.05)。核苷酸歧点分布图表明,F3代和F4代群体核苷酸歧点分布图为单峰(图3,图4)。结果表明,这个山瑞鳖群体历史上可能发生过扩张。
3 讨论
3.1 山瑞鳖群体遗传多样性分析
在自然群体中保持了大量的遗传变异,在DNA水平上关于这种变异的信息可以从核苷酸之间的差异获得[12]。线粒体碱基对替换率不受母系遗传和营养分离,在涉及短时间跨度的进化研究中更有用[14]。本研究获得了45条山瑞鳖线粒体Cytb基因同源序列(1000bp),序列中四种碱基T(27.19%)、A(31.28%)、G(11.35%)和C(30.18%)的平均含量碱基C含量最低,与李小三[4]得到的山瑞鳖线粒体Cytb基因序列四种碱基平均含量相似,验证了山瑞鳖Cytb基因碱基分布情况。山瑞鳖Cytb基因序列碱基组成C+G含量较A+T低、具有明显的反G偏移等特点在中华鳖[15]和太湖大银鱼[16]中也有类似体现,与大部分水产动物Cytb基因序列特征一致。群体内突变位点占比小(0.90%),个体间变异较小,亲缘关系较近。
遗传多样性是生物多样性的形式之一,群体遗传多样性是反映群体适应性的重要指标[17]。线粒体基因组(mitochondria DNA,mt DNA)是真核细胞的第二遗传系统,动物线粒体基因由15-20kb的双链环状DNA分子构成,具有进化速率快、母系遗传、无组织特异性和序列差异在群体中的个体间存在等遗传特点,现已成为研究种内、种间遗传多样性的重要依据[18,19]。Cytb基因是线粒体基因组的13个开放阅读框之一,在进化上较为保守,在水产动物上通常被用于分子进化和遗传多样性的研究。从研究结果来看2个群体都表现出了高单倍型多样性和低核苷酸多样性的特点,说明该群体可能经历过快速的群体增长和突变[20]。F3代群体(0.00184)和F4代代群体(0.00042)的核苷酸多样性均在0.0015~0.0047之间,表明这2个群体的遗传多样性处于较低水平,群体内遗传变异较小[21]。但F3代群体的单倍型多样性和核苷酸多样性较F4代群体高,F3代群体的遗传多样性较F4代丰富,群体内平均遗传距离也较F4代高得多,F4代群体可能在保种过程中出现了种质资源退化。两个群体整体核苷酸多样性为0.00090,群体间平均遗传距离(0.00138)较低。综上所述,两个群体的亲缘关系较近,保种过程中群体内的变异逐渐减小,遗传距离随着保种年限的增加越来越小,在今后的保种过程中要根据亲本的亲缘关系制定繁殖策略,尽可能使雌雄搭配合理,避免近亲繁殖。4C1F193C-5322-4D34-A264-0695750EB7AF
3.2 山瑞鳖群体遗传结构分析
种群遗传结构的研究有助于评估不同的种群的进化轨迹,或在多大程度上保持了基因流的联系,是了解物种进化的先决条件[22]。山瑞鳖2个群体中共存在9种单倍型,其中F3代群体有7种单倍型,F4代群体有3种单倍型,两个群体间共享单倍型仅有Hap_1,除了单倍型Hap_1有较多个体外,其余单倍型均只有1个个体,说明个体间同源性较高。从单倍型网状关系图可以看出,Hap_4单倍型可能为两个群体的祖先单倍型,群体间不存在明显的遗传分化。根据进化树推断,F4代群体中的单倍型Hap_5和Hap_9亲缘关系较近,单倍型Hap_1和Hap_8与Hap_6亲缘关系较近,在繁殖过程中应尽量避免各单倍型内的个体间交配,进化树结果也与单倍型网状关系图中这些单倍型的联系相对应。后续应理清所有用于人工保种群体个体间的遗传关系,根据每个群体内的遗传距离和遗传结构制定繁殖策略,提高后代群体的遗传多样性。
3.3 群体动态分析
群体中多态性的模式不仅受到突变和随机漂移的影响,还受到自然选择的影响,遗传分化系数的引入为研究种群结构提供了一种方便的方法[23]。该研究山瑞鳖群体AMOVA分析结果显示,该保种群体的变异来源主要来自群体内,群体间变异贡献率为26.90%。中性检验结果显示,两个群体的Tajima′s D值和Fu′s Fs均为负值,说明这个山瑞鳖保种群体存在大量的低频等位基因位点,古代群体可能经历过扩张。据李大命[24]等对个体间碱基不配对分布曲线的解释,在山瑞鳖两个保种群体中,核苷酸歧点分布图为单峰,F3代群体和F4代群体历史上可能发生过扩张,与中性检验结果相符。
群体遗传多样性水平反映了其在自然界的生存能力和进化潜力。根据研究结果可以看出,山瑞鳖两个保种群体内的遗传多样性较低,亲缘关系较近,群体遗传多样性水平退化明显。在接下来的保种过程中应采取不同的保种策略,尽量避免近亲交配,利用分子生物技术的方法辅助保种,进而保护云南土著龟鳖种质资源,为山瑞鳖人工保种和保护云南生物多样性提供科学依据。
参考文献:
[1]王剀,任金龙,陈宏满,等. 中国两栖、爬行动物更新名录[J]. 生物多样性,2020,28(02): 189-218.
[2]郑文彪,粱淡茹,肖智,等. 山瑞鳖與中华鳖的养殖生长和形态比较[J]. 淡水渔业,1998,28(01):40-41.
[3]唐大由,李贵生,李海. 山瑞鳖的生物学特性及人工养殖技术[J]. 中药材,1997,20(04):168-171.
[4]李小三,汪玲,周科,等. 基于线粒体Cytb基因全序列探讨两爪鳖和山瑞鳖的系统进化关系[J]. 生命科学研究,2010,14(05):413-418.
[5]SAMBROOK J, FRITSCH E P, MANIATIS T. Molecular Cloning: A Laboratory Manual [M]. Beijing: Science Press, 1996.
[6]陈合格,刘文彬,李建中,等. 三种鳖线粒体DNA细胞色素b基因序列的比较分析[J]. 水生生物学报,2006,30(04):380-385.
[7]BURLAND T G. DNASTAR′s Lasergene sequence analysis software[M]//Bioinformatics methods and protocols. Humana Press, Totowa, NJ, 2000: 71-91.
[8]KUMAR S, STECHER G, LI M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular biology and evolution, 2018, 35(6): 547-1549.
[9]ROZAS J, FERRER-MATA A, SANCHEZ-DELBARRIO J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular biology and evolution, 2017, 34(12): 3299-3302.
[10]FORSTER P, TOTH A, BANDELT H J. Evolutionary network analysis of word lists: Visualising the relationships between Alpine Romance languages[J]. Journal of Quantitative Linguistics, 1998, 5(3): 174-187.
[11]EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular ecology resources, 2010, 10(3): 564-567.
[12]TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595.4C1F193C-5322-4D34-A264-0695750EB7AF
[13]FU Y X, LI W H. Statistical tests of neutrality of mutations[J]. Genetics, 1993, 133(3): 693-709.
[14]BIRKY JR C W, MARUYAMA T, FUERST P. An approach to population and evolutionary genetic theory for genes in mitochondria and chloroplasts, and some results[J]. Genetics, 1983, 103(3): 513-527.
[15]张卉琴,陈志男,李戈锐,等.汉寿中华鳖线粒体细胞色素b基因克隆及其遗传多样性分析[J].激光生物学报,2018,27(04):359-366.
[16]李大命,张彤晴,唐晟凯,等. 太湖大银鱼(Protosalanx chinensis)细胞色素b基因序列多态性分析[J].江苏农业学报,2015,31(4):840-845.
[17]REED D H, FRANKHAM R. Correlation between Fitness and Genetic Diversity[J]. Conservation Biology, 2003, 17(1):230-237.
[18]廖顺尧,鲁成. 动物线粒体基因组研究进展[J]. 生物化学与生物物理进展,2000,27(05):508-512.
[19]闫华超,高岚. 线粒体DNA遗传特性的研究新进展[J]. 生物技术,2003,13(06):63-65.
[20]GRANT W A S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of heredity, 1998, 89(5): 415-426.
[21]LAN H, SHI L. The origin and genetic differentiation of native breeds of pigs in southwest China: an approach from mitochondrial DNA polymorphism[J]. Biochemical genetics, 1993, 31(1): 51-60.
[22]DUMINIL J, FINESCHI S, HAMPE A, et al. Can population genetic structure be predicted from life-history traits?[J]. The American Naturalist, 2007, 169(5): 662-672.
[23]WEIR B S. Estimating F-statistics: A historical view[J]. Philosophy of science, 2012, 79(5): 637-643.
[24]李大命,张彤晴,唐晟凯,等. 洪泽湖野生河蚬(Corbicula fluminea)线粒体CO Ⅰ基因序列的遗传多样性分析[J]. 渔业科学进展,2015,36(05):81-86.
Genetic diversity analysis of Palea steindachneri conservation populations based on mitochondrial Cytb gene sequence
Abstract: To study the genetic diversity and genetic structure of Palea steindachneri conserved populations in Yunnan. In this study, we analyzed the haplotype distribution, genetic distance, genetic structure and population history dynamics of two populations of Palea steindachneri conserved in Yunnan, based on the mitochondrial cytochrome b(Cytb) gene, using PCR amplification and sequencing techniques, and amplified the mitochondrial Cytb gene sequences of 45 individuals from the F3 and F4 generation populations. The sequence length was 1000 bp after cut and aligned with DNAStar and MEGA software, and the average contents of bases A, T, C and G were 31.28%, 27.19%, 30.18% and 11.35%, respectively. A total of nine haplotypes were detected in the two populations, including seven haplotypes in the F3 generation population and three haplotypes in the F4 generation population, with no obvious genealogical structure between haplotypes and close kinship. The genetic background within the two populations was single, and the average genetic distance between individuals was small. The genetic diversity level of the populations was gradually decreasing with the increase of the number of conserved generations, and the inbreeding phenomenon was obvious. The neutrality test and nucleotide manifold distribution maps indicated that the F3 and F4 populations may have undergone expansion historically. The results indicated that the conserved population had low intra-population genetic diversity, and different conservation strategies should be adopted to avoid inbreeding in the future conservation process.
Key words: Palea steindachneri; Cytb gene; conservation population; genetic diversity; genetic structure
文章編号:16742419(2022)03000307
基金项目:国家自然科学基金(32060827)资助 [Supported by National Natural Science Fund(32060827)]。
作者简介:黄松茂(1997-),男,云南农业大学动物科学技术学院在读硕士研究生,主要从事水产动物养殖与繁育研究。E-mail:2359282510@qq.com。
通信作者:孔令富(1975-),男,云南昭通人,硕士,云南农业大学副教授。研究方向:水产种质资源保护与利用。E-mail:wzlklf@sina.com;武祥伟(1984-),男,山东菏泽人,博士,云南农业大学讲师。研究方向:云南土著鱼类种质资源保护与利用。E-mail:wxw9559@126.com。4C1F193C-5322-4D34-A264-0695750EB7AF
