基于叶绿体基因组重建中国野生葡萄的系统发育关系
2022-06-06张丽娜何华勤王亚男郑英杰
张丽娜, 何华勤, 田 甜, 王亚男, 吴 题, 郑英杰
(福建农林大学生命科学学院,福建 福州 350002)
葡萄(Vitisvinifera)是一种具有重要经济价值的水果,可鲜食,可做成果脯,也可用来酿酒,约在8 000年前开始被驯化[1].葡萄属约有60个种,主要分布在温带树林中[2].很多学者从形态特征和基因组方面对其开展了研究,以澄清葡萄属内部类群间的系统发育关系[3-6].葡萄属按染色体数目、种子形态等被划分为2个亚属,即葡萄亚属(subgenusVitis)和圆叶葡萄亚属(subgenusMuscadinia).大部分的葡萄属种类归属于葡萄亚属,仅2个种属于圆叶葡萄亚属,且在中国没有分布[7].目前,系统发育研究将葡萄亚属分为北美葡萄、欧洲葡萄和东亚葡萄3个分支[5,8].东亚葡萄和北美葡萄囊括了绝大部分的野生葡萄类群[6,9],且东亚葡萄中的大部分种类只分布在中国,约37个种[2,7].
学者们基于传统分类方法对于中国野生葡萄的分类结果存在较大差异:刘崇怀[3]将中国葡萄分为8个组,鸡足葡萄(V.lanceolatifoliosa)单独位于基部分支,第3组又分为5个亚组;段来军[4]将中国葡萄分为3个组,其中,菱叶葡萄(V.hancockii)单独位于基部分支,第1和2组各分为3个亚组,鸡足葡萄位于第1组第3亚组.
随着高通量测序技术的发展,植物基因组得到广泛研究.Ma et al[6]基于核基因组构建了具有较高分辨率的中国野生葡萄系统发育树,将中国野生葡萄分成了4个分支,基部分支由山葡萄(V.amurensis)和变叶葡萄(V.piasezkii)组成.该结果与上述两种传统分类结果不同:山葡萄在传统分类中属于第3组的第1亚组[3],变叶葡萄属于第5组[3]或第1组的第3亚组[4].Wen et al[9]基于23种42个个体的叶绿体基因组重建了北美葡萄的系统发育关系,该结果与Ma et al[6]基于核基因组的研究结果也不一致.质核冲突普遍存在于绿色植物分类中[10-14],杂交或基因渐渗、不完全谱系分选、基因选择或孤雄生殖等,都可能会引起质核冲突[15-17].
目前,NCBI上公布的中国野生葡萄叶绿体基因组只有10个,为明确中国野生葡萄系统发育关系是否存在质核冲突问题,本研究从NCBI上下载了已经公布但未组装出叶绿体基因组的中国野生葡萄重测序数据,从中组装出叶绿体基因组,并构建中国野生葡萄系统发育树.
1 材料与方法
1.1 数据下载及基因组组装和注释
中国野生葡萄约37种,从NCBI上下载了26个重测序数据,这些数据都来源于PRJNA490319,覆盖度在20倍左右.所有数据在NCBI上的接受号见表1.
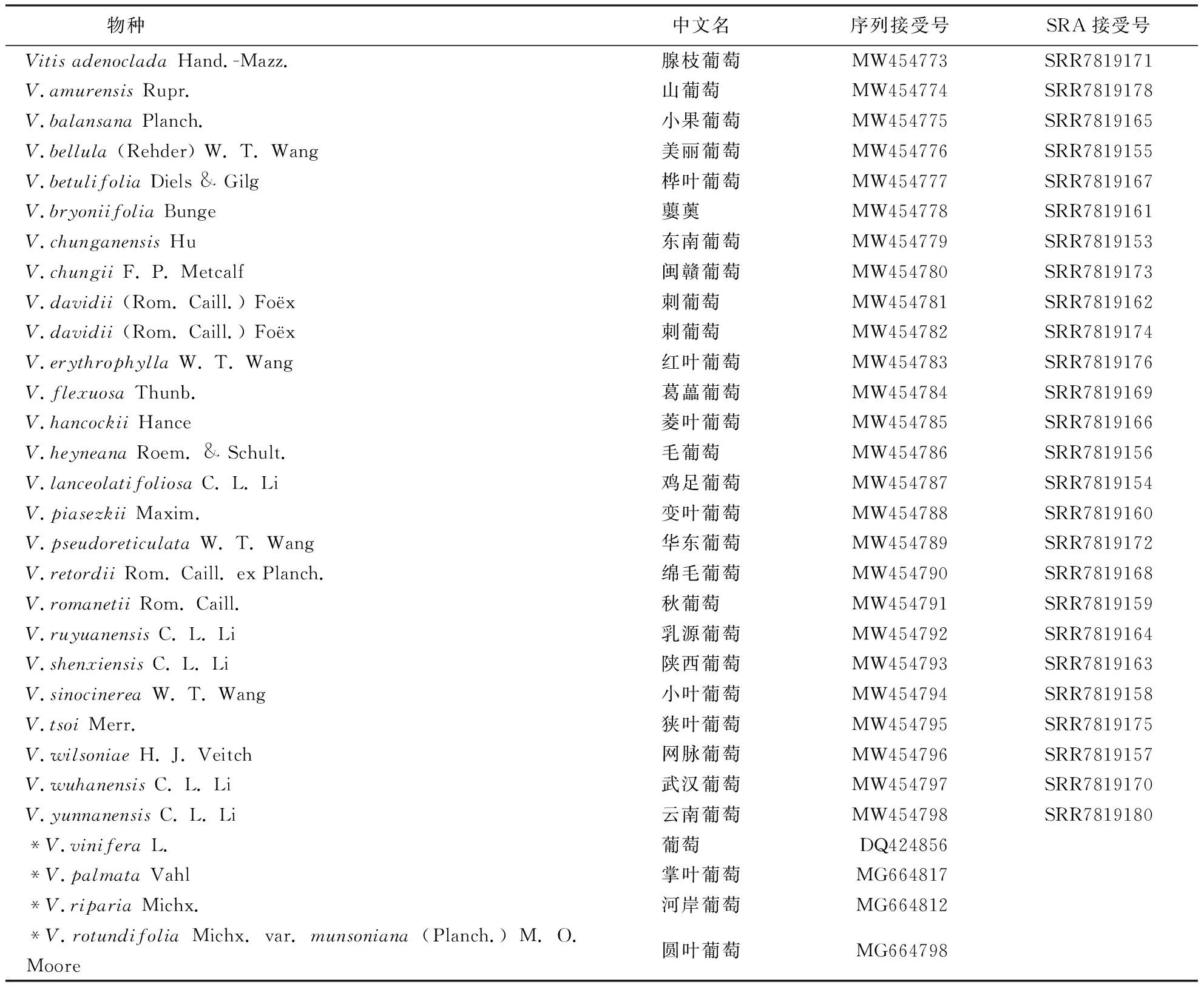
表1 30种葡萄叶绿体基因组序列的接受号1)Table 1 Accession number of chloroplast genomes from 30 grape varieties
原始数据经过fastp-0.19.4[18]过滤后,以发布的葡萄叶绿体基因组(DQ424856)为参考序列组进行组装.具体的组装过程:将过滤后的reads利用bwa-0.7.15[19](K=32)进行mapping产生sam文件,该sam文件通过samtools-0.1.18[20]转换为bam文件,借助ConsensusFixer0.4[21]生成相应的叶绿体基因组序列;通过IGV-Win-2.4.14[22-23]对组装的序列,特别是一些SNP(single nucleotide polymorphism)和插入缺失的位点进行人工校对.将校对好的26个叶绿体基因组序列以DQ424856为参考,使用PGA软件[24]进行注释.将注释完成的26个叶绿体基因组上传到GenBank,相应的序列接受号见表1.
1.2 序列比对和系统发育分析
本研究下载了葡萄、2个北美葡萄V.riparia和V.palmata及圆叶葡萄(V.rotundifoliavar.munsoniana)的叶绿体基因组序列(对应的接受号见表1),作为系统发育分析的外类群,加上新组装的26个叶绿体基因组,总共为30个叶绿体基因组.将这些基因组序列输入mafft-7.407[25]中进行比对,然后用GENEIOUS 4.8.5[26]进行人工校对,比对好的序列用于后续的分析.采用最大似然法(maximum likelihood, ML)及贝叶斯推理法(Bayesian inference, BI)进行系统发育分析.通过IQ-TREE 1.6.12[27]进行ML分析,使用的模型为TESTMERGE,1 000次重复;通过MrBayes-3.2.7a[28]进行BI分析,参数设置参考jmodeltest-2.1.10[29]的最优模型参数,马尔科夫链(Markov chain Monte Carlo, MCMC)设置的代数为1 000 000代,确保离频率(split frequencies)平均标准差值小于0.01,每隔1 000代抽样一次,最后计算一致树(consensus tree)及后验概率.
为探讨叶绿体基因组的编码(coding sequences, CDs)区和非编码(non-coding sequences, NCs)区揭示的中国葡萄的系统发育关系是否与叶绿体全基因组的结果一致,本研究分别提取CDs区和NCs区的序列进行分析.鉴于ML和BI分析显示的系统发育框架非常相似,本研究仅使用ML分析CDs区和NCs区的系统发育关系,方法同上.
对以上3套数据(全基因组、CDs区和NCs区)分别进行QS(quartet sampling)[30]分析,以推测相应系统发育树上一些分支支持率较低的原因,每次分析都进行1 000次重复.
选取至少存在一个SNP位点的基因用于后续分析,共筛选出56个基因,以V.riparia和V.vinifera为外类群,对28个类群进行系统发育分析.将56个基因分别输入IQ-TREE 1.6.12,进行200次重复分析.将分析产生的基因树和bootstrap文件都输入ASTRAL-4.7.12[31]中,进行基于溯祖理论的物种树推断.另外,将56个基因联合起来进行ML分析,利用软件IQ-TREE 1.6.12分别运算分区(partitioned)和不分区(unpartitioned)模型.
2 结果与分析
2.1 叶绿体基因组的组装和注释
26个中国野生葡萄叶绿体基因组的长度为160 879~160 961 bp.26个基因组的结构与葡萄[32]几乎一致,即由2个IR(inverted repeat)区、1个SSC(small single copy)区和1个LSC(large single copy)区组成的一个环状结构.这4个区的序列长度变化不大[除了桦叶葡萄(V.betulifolia)的LSC区与其他葡萄相差约50 bp外].26个叶绿体基因组都包含114个基因,GC含量均为37.40%(表2).
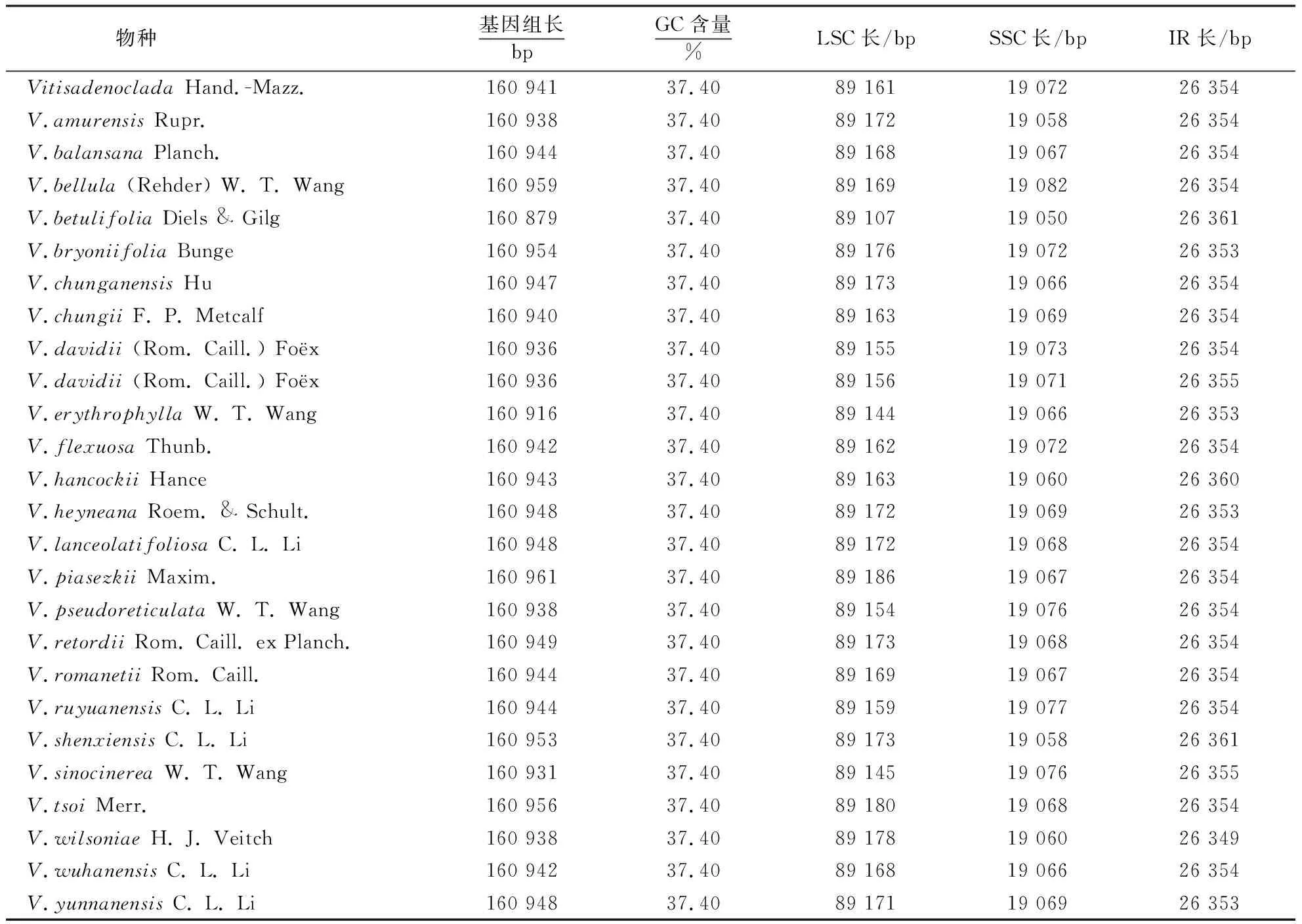
表2 26种中国野生葡萄叶绿体基因组信息1)Table 2 Information on chloroplast genomes from 26 Chinese wild grape varieties
2.2 基于叶绿体基因组3套数据的系统发育分析
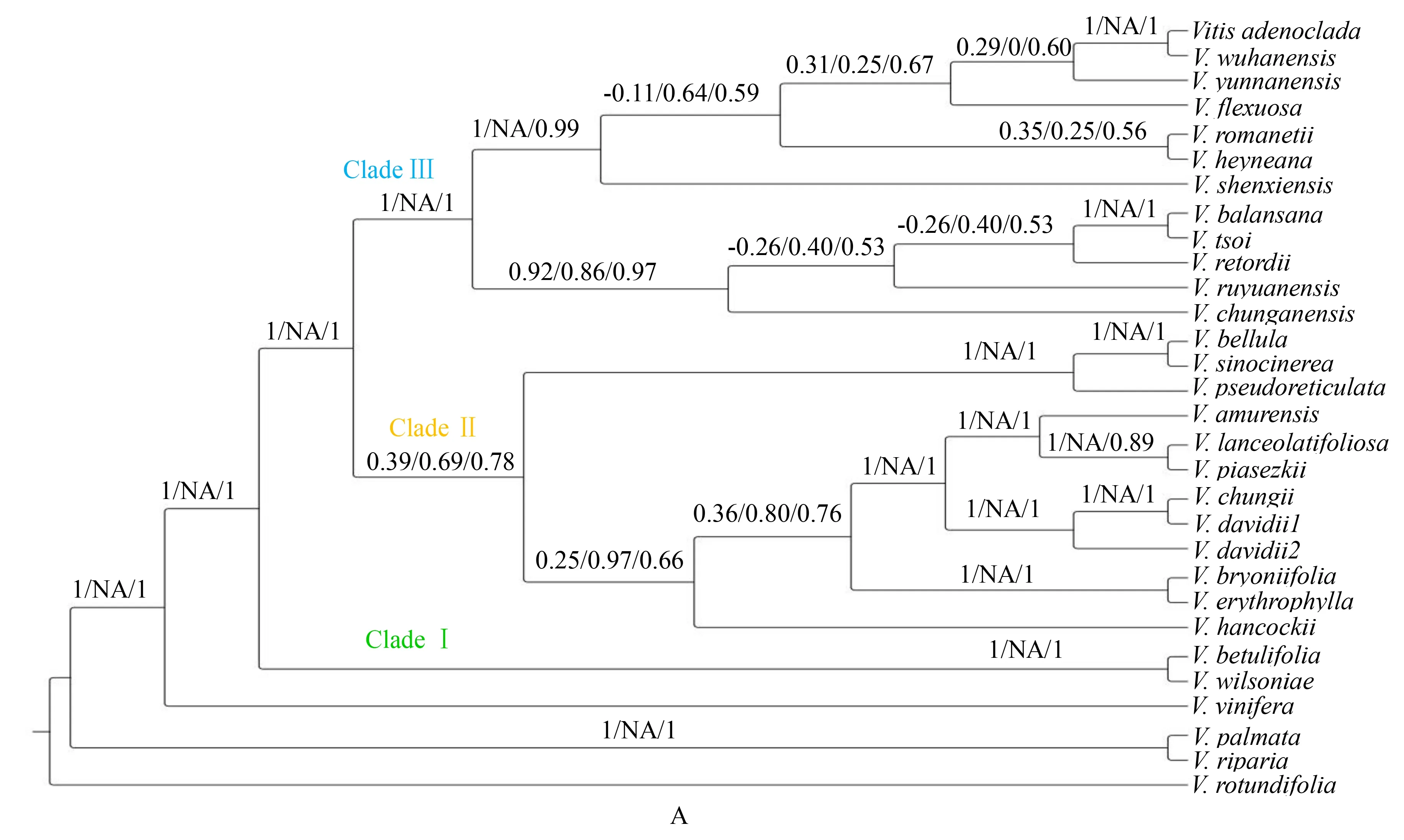
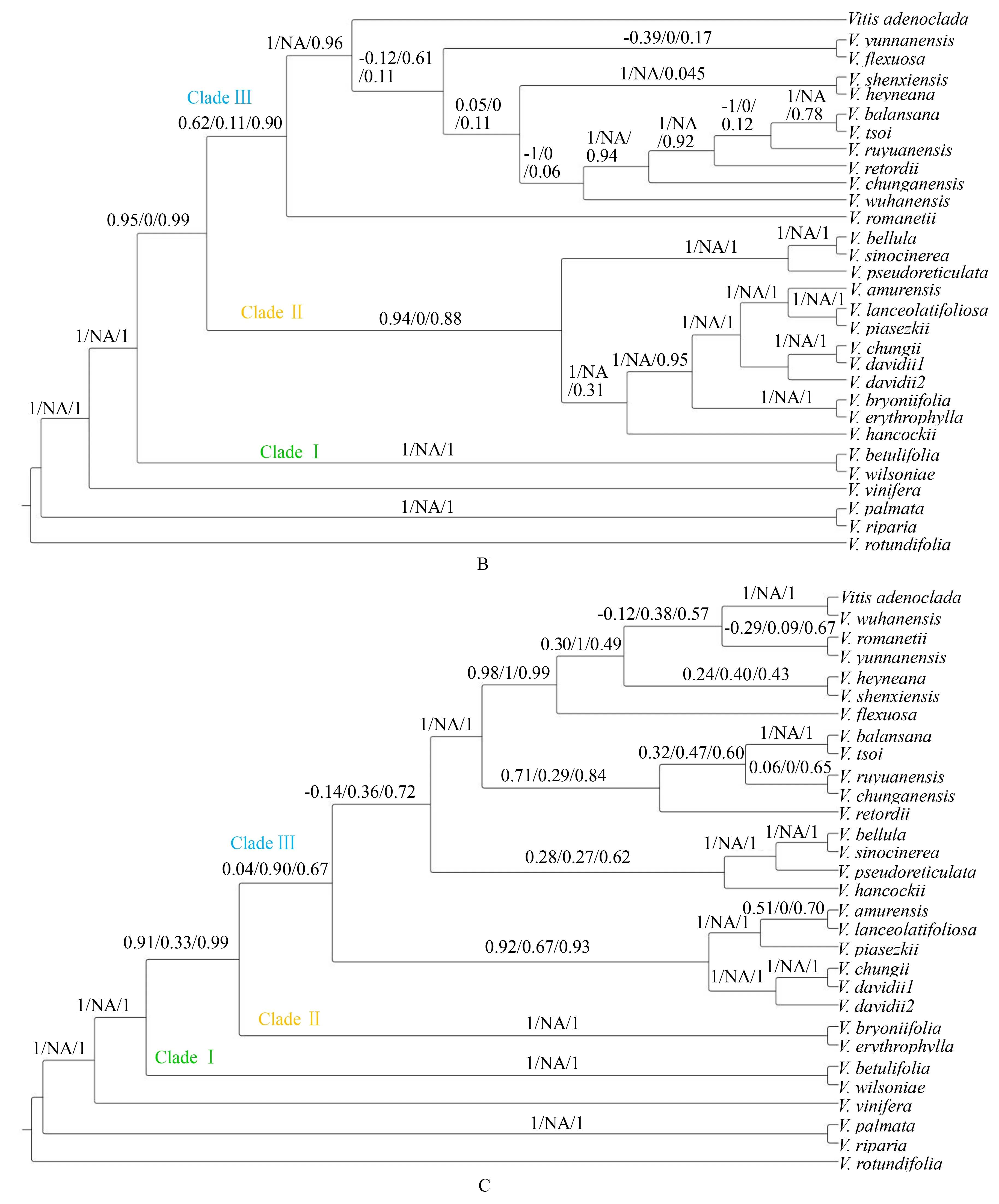
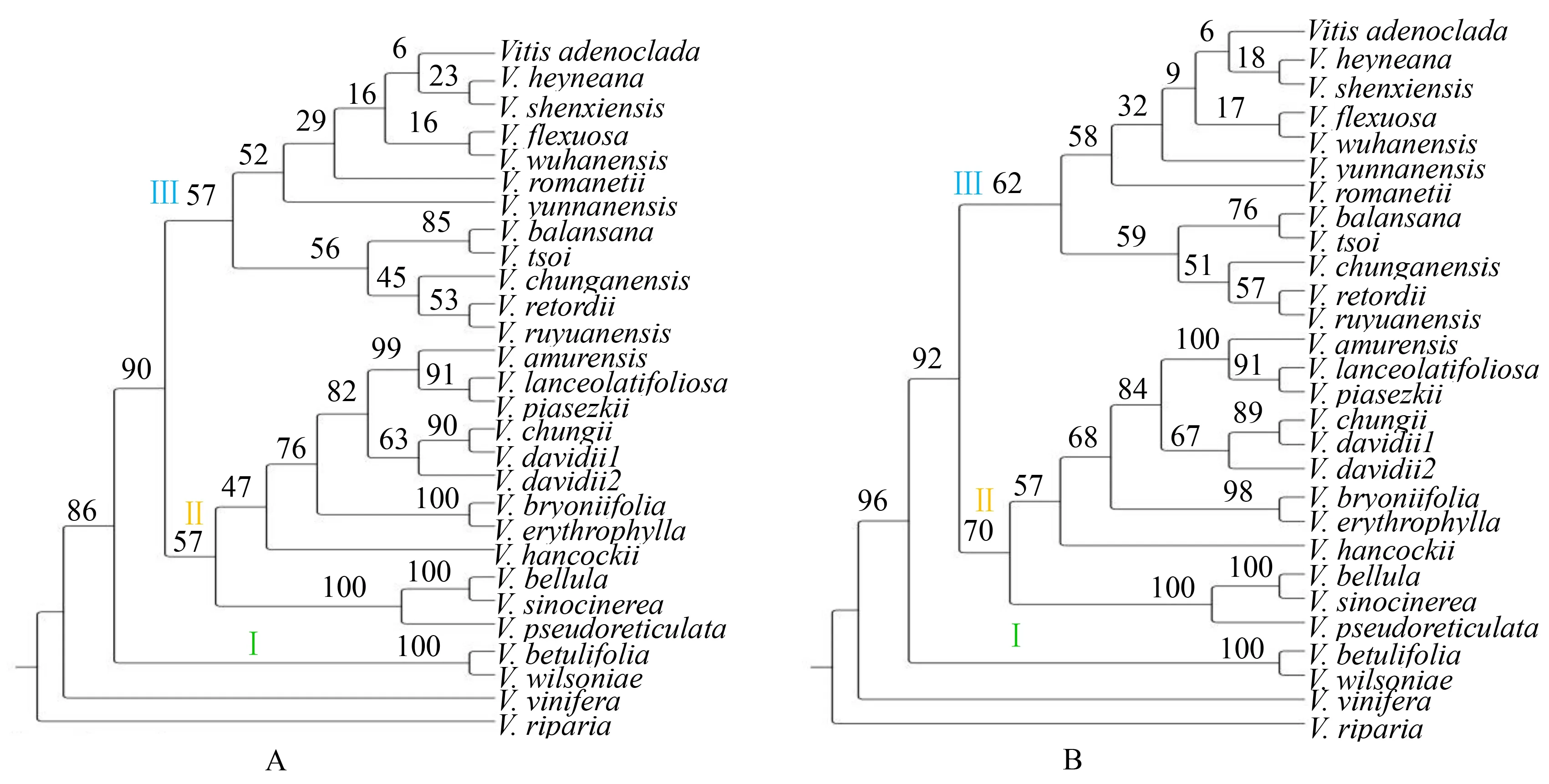
ML和BI分析所得的中国野生葡萄系统发育树的拓扑结构非常相似(图1),因此,下面仅对ML的结果进行分析.从图2可以看出,基于3套数据的中国野生葡萄系统发育树均分为3个分支(clade).其中,基于叶绿体全基因组构建的系统发育树上每个分支的支持率都比较高.V.betulifolia和V.wilsoniae组成基部分支,即cladeⅠ;cladeⅡ由5个亚分支构成,但在基于NCs区构建的系统发育树上,cladeⅡ仅由一个亚分支构成.3棵系统发育树上,cladeⅢ的差异比较大:在全基因组系统发育树上,分成2个亚分支;在CDs区系统发育树上分成5个亚分支,其中4个亚分支只有1或2个种组成,且分支的支持率非常低;在NCs区系统发育树上,由4个亚分支组成,这种拓扑关系的支持率非常低,但其中有2个亚分支组成的拓扑结构与全基因组非常相似,且该分支的支持率比较高.
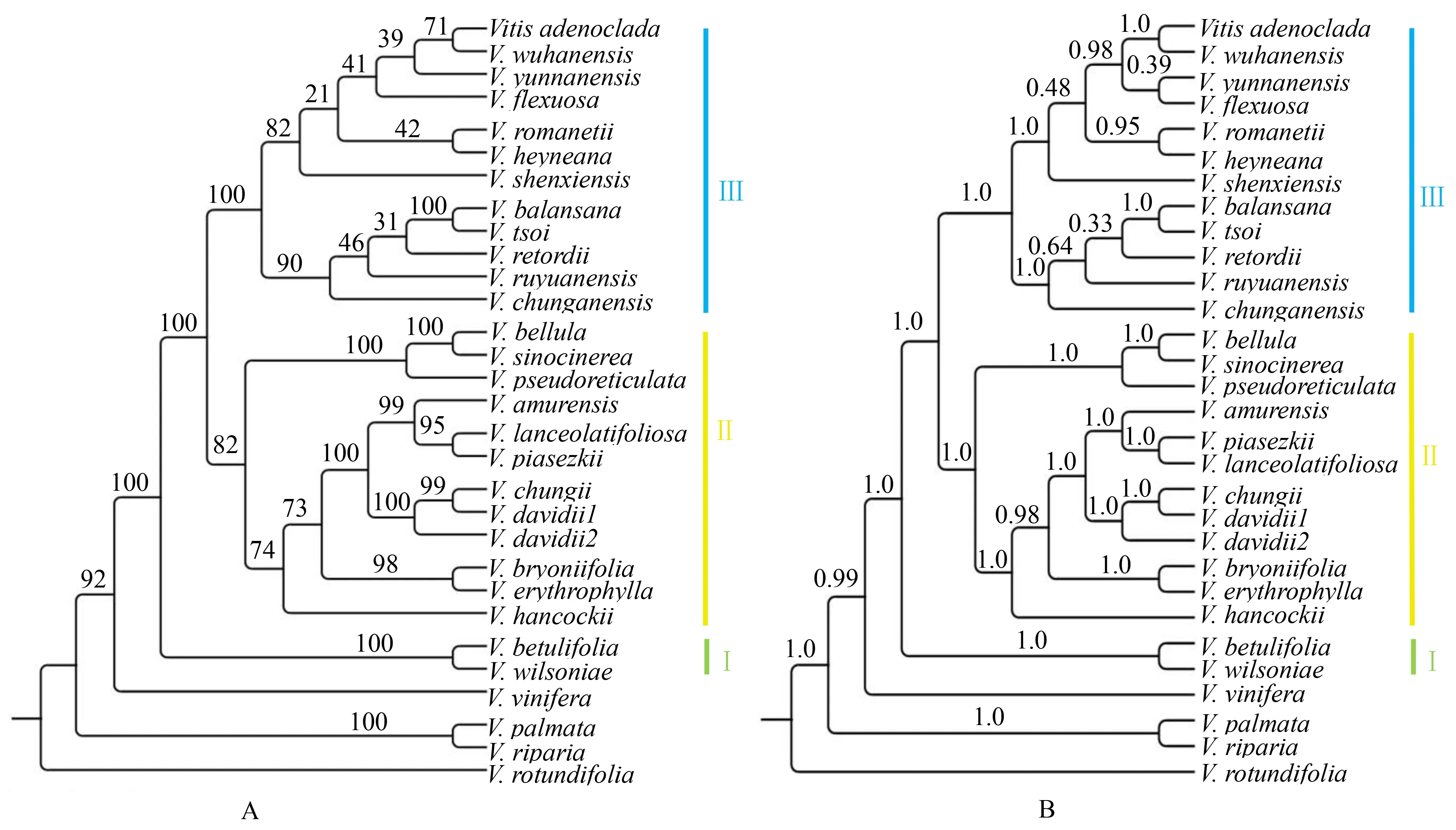
图1 利用最大似然法(ML)(A)和贝叶斯推理法(BI)(B)构建的中国野生葡萄系统发育树Fig.1 Phylogenetic tree of Chinese wild grapes using maximum likelihood (ML)(A) and Bayesian inference (BI)(B) analysis
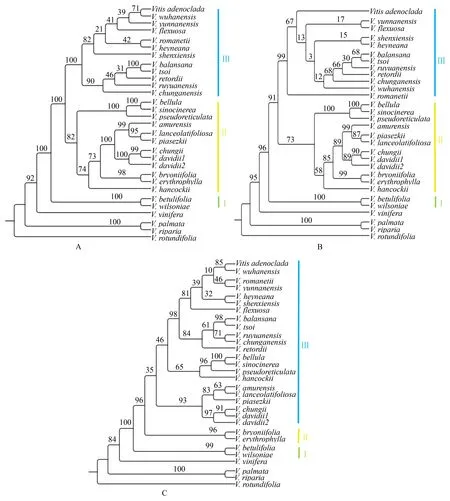
A.基于全基因组构建;B.基于编码区构建;C.基于非编码区构建.图2 基于3套叶绿体基因组数据构建的中国野生葡萄ML系统发育树Fig.2 Phylogeny tree of Chinese wild grapes based on 3 chloroplast genome datasets using ML analysis
另外,本研究构建的中国野生葡萄系统发育树与基于核基因组构建的系统发育树[6]存在比较大的差异(图3).在核基因树上,中国野生葡萄分成4个分支,但cladeⅠ落在叶绿体基因树的cladeⅡ上,cladeⅡ分布在叶绿体基因树的cladeⅢ上,cladeⅢ和cladeⅣ上的类群则被分散在叶绿体基因树的3个分支中.
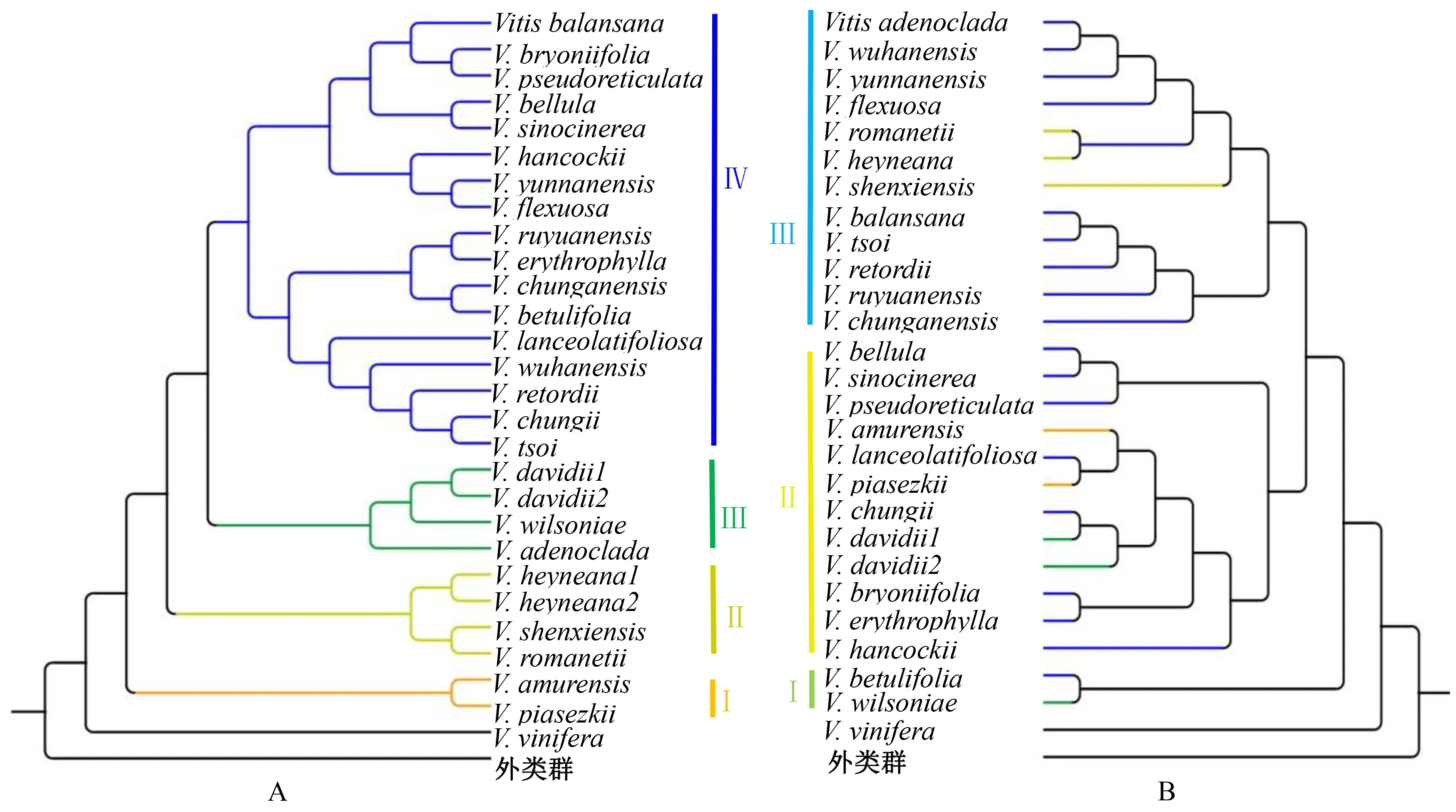
基于核基因组的系统发育树参考Ma et al[6]的结果.叶绿体基因组系统发育树上分支的不同颜色与核基因组系统发育树上的颜色相对应.图3 基于核基因组(A)和叶绿体基因组(B)构建的中国野生葡萄系统发育树Fig.3 Phylogeny tree of Chinese wild grapes based on nuclear genomes (A) and chloroplast genomes (B)
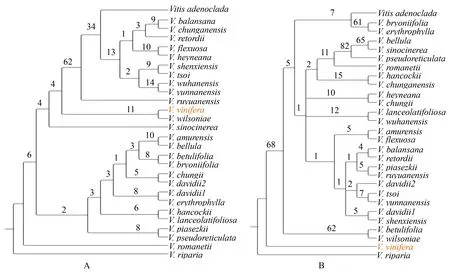
在QS分析中,QI(quartet informativeness)、QC(quartet concordance)和QD(quartet differential)分别表示分支的系统发育信息量及这些信息的一致性和分歧度.本研究中,除了cladeⅢ,大部分分支都得到了中高分的QI和QC值(图4).3棵系统发育树上的cladeⅠ都得到了较高支持率,QC和QI值都为1,QD值为NA(表示分歧度为空值,即没有分歧).由cladeⅡ和cladeⅢ组成的分支,得到了接近于或等于1的QC和QI值;在全基因组和CDs区的系统发育树上QD值为NA和0,但在NCs区系统发育树上QD值为0.33.cladeⅡ在全基因组系统发育树上的QC、QD和QI值分别为0.39、0.69和0.78;在CDs区和NCs区系统发育树上的QC和QI值比较高,QD值为0或NA.cladeⅢ在全基因组系统发育树上的QC和QI值都为1,QD值为NA;在CDs区系统发育树上,QC、QD和QI值分别为0.62、0.11和0.90;在NCs区系统发育树上,QC、QD和QI值分别为0.04、0.90和0.67.通过QS分析可知,这3套数据对中国野生葡萄系统发育关系的解析能力有所不同.从整体上看,全基因组的解析能力最佳,NCs区数据在重建cladeⅡ和cladeⅢ分支的类群关系时存在相对较多的信号冲突,以致与另外2套数据的结果差异较大.
A.基于全基因组构建;B.基于编码区构建;C.基于非编码区构建.分支上的数值分别代表QC(quartet concordance)/QD(quartet differential)/QI(quartet informativeness)值.图4 中国野生葡萄系统发育的QS分析Fig.4 Quartet sampling analyses for the phylogeny of Chinese wild grapes
续图4Continued Fig.4
2.3 基于联合分析和溯祖分析的系统发育树
在进行溯祖分析前,分别对56个基因单独构建了系统发育树(如图5A),结果显示,单个基因构建的系统发育树分辨率非常低,甚至不能将中国野生葡萄类群与外类群区分.分析显示,单个基因的系统发育信号非常弱(表3).56个基因中,只有10个基因可以将中国野生葡萄与外类群区分,但支持率很低,这10个基因分别为atpF、atpI、ndhF、ndhH、petD、rbcL、rpl22、rpl32、rpl33 和ycf1(如图5B).56个基因联合分析得到的系统发育树与全基因组系统发育树的拓扑结构几乎一致.另外,基于分区和不分区分析得到的系统发育关系基本一致,仅个别分支的支持率不同(图6).
A.基于accD构建;B.基于ndhF构建.图5 基于单个叶绿体基因构建的中国野生葡萄ML系统发育树Fig.5 Phylogeny tree of Chinese wild grapes based on individual chloroplast gene using ML analysis
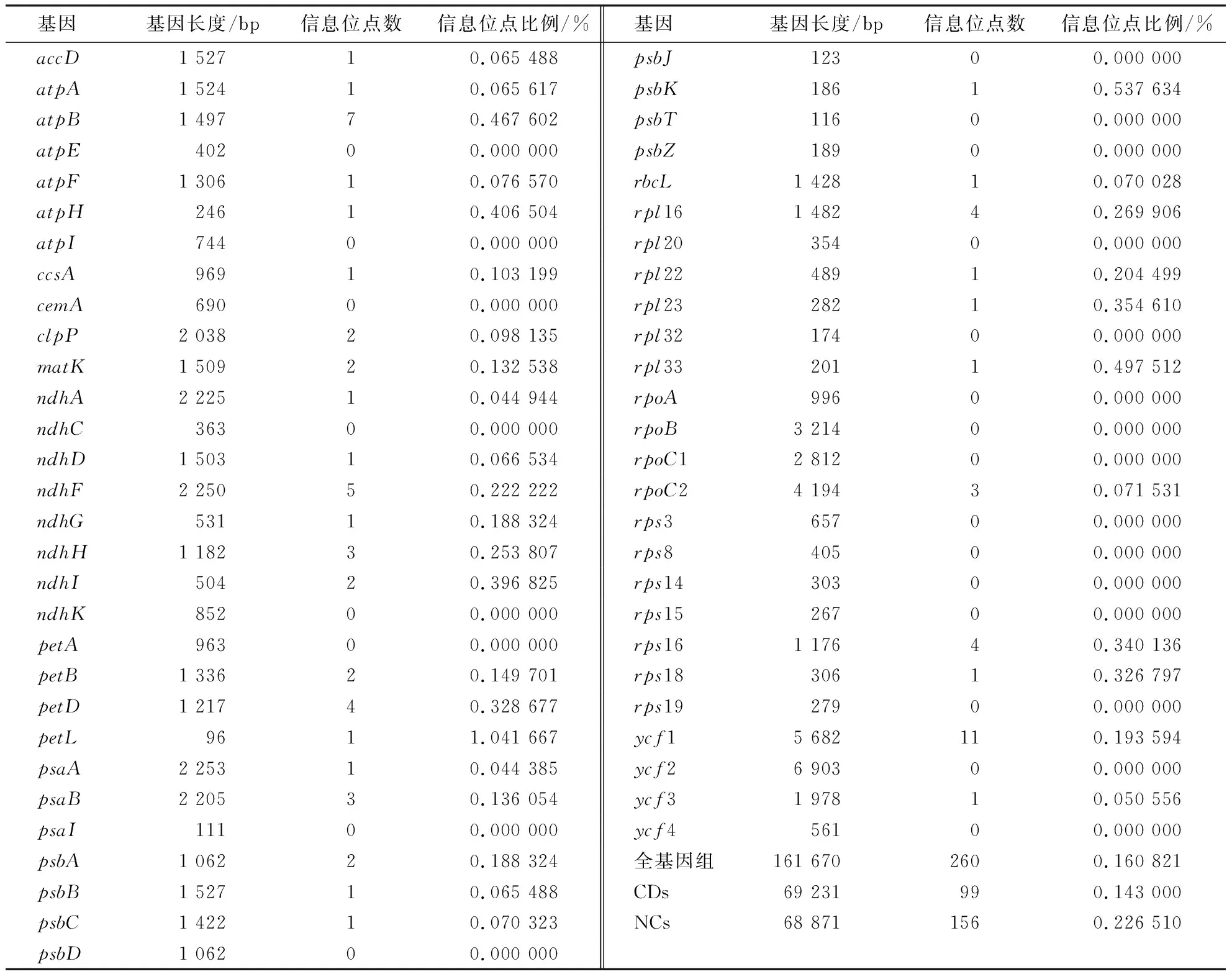
表3 28种葡萄的56个叶绿体基因的信息Table 3 Information of 56 chloroplast genes from 28 grape species
A.基于不分区数据构建;B.基于分区数据构建.图6 基于56个叶绿体基因联合构建的中国野生葡萄ML系统发育树Fig.6 Phylogeny tree of Chinese wild grapes based on 56 chloroplast genes using ML analysis
溯祖分析得到的系统发育树(图7),将中国野生葡萄分成2个分支,支持率分别为81和70,且其中大部分内部分支的支持率都非常低;外类群葡萄(V.vinifera)也在其中,中国野生葡萄类群不为单系,该分支的支持率仅52.可见,溯祖分析得到的系统发育树与联合分析的系统发育树存在非常大的差异.
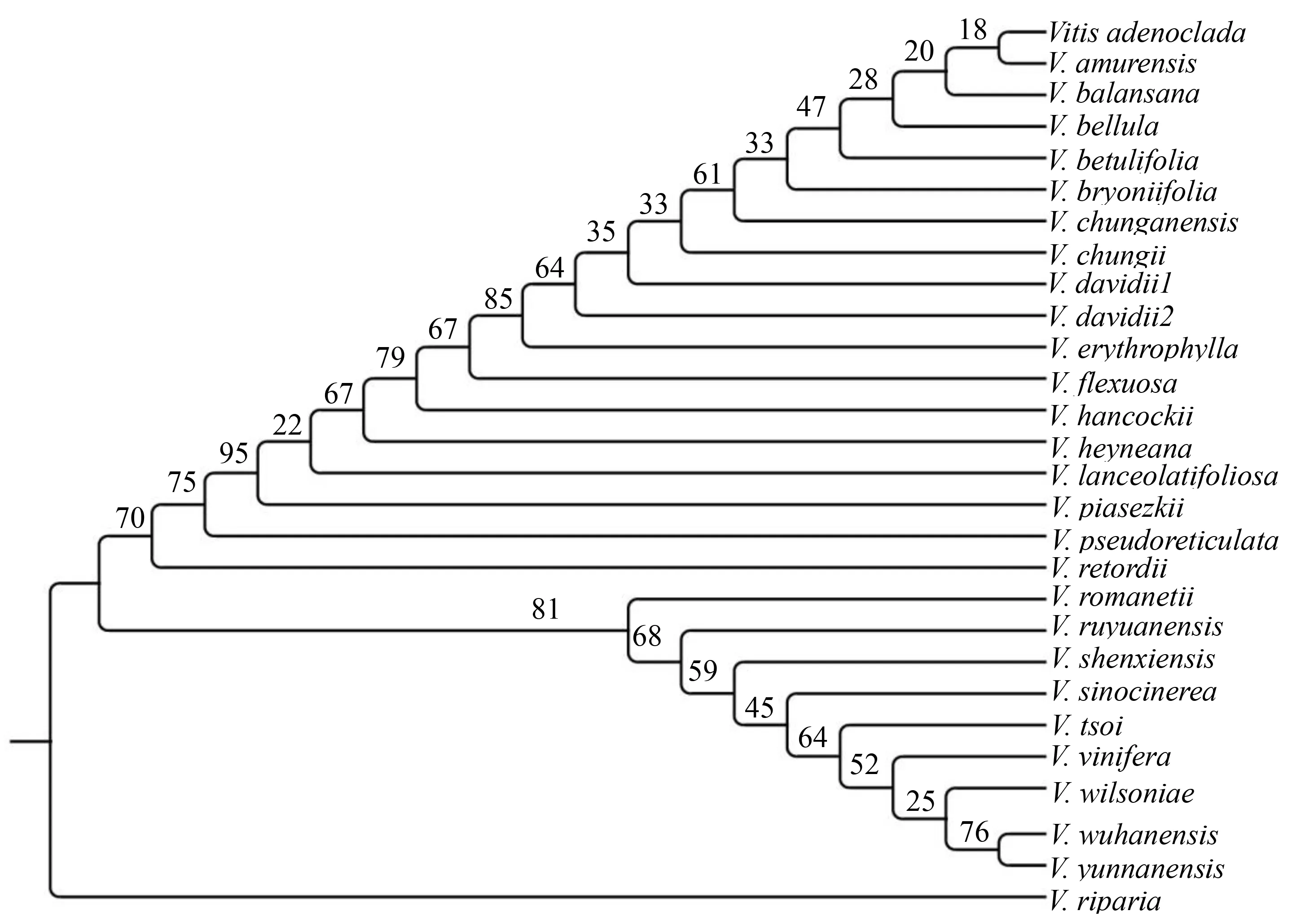
图7 基于56个叶绿体基因用ASTRAL构建的中国野生葡萄系统发育树Fig.7 Phylogeny tree of Chinese wild grapes based on 56 chloroplast genes using ASTRAL
3 讨论
3.1 基于3套叶绿体基因组数据得到的中国野生葡萄系统发育关系的比较
cladeⅠ在全基因组、CDs区和NCs区的系统发育树上都得到了支持,cladeⅡ得到了全基因组和CDs区系统发育树的支持,而cladeⅢ中有2个亚分支得到了全基因组和NCs区系统发育树的支持.通过QS分析发现,系统发育树上支持率比较低的分支,通常都有比较低的QC值,部分QI值也比较低.因此,本研究中低支持率的分支可能主要是由于系统发育信号不一致或比较弱所造成.
基于高阶层如科或目水平的的系统发育研究表明,CDs区能提供更多的系统发育信息,而NCs区由于容易经历饱和信号(signatures of saturation)而可能提供错的系统发育信息[33].在一些属水平上的系统发育研究中,NCs区相对于CDs区表现的更为可靠,但全基因组的表现最好[34].本研究也表明,基于全基因组的中国野生葡萄系统发育关系整合了CDs和NCs区分辨率最高的部分,能更好地反映中国野生葡萄的系统发育关系.
3.2 中国野生葡萄叶绿体基因组联合分析和溯祖分析比较
联合分析和溯祖分析是目前系统发育分析中最为常用的两种方法.联合分析是将所有的基因联合成一个超级大矩阵,这可能会提供更多的系统发育信息.然而,有研究发现,这也可能会构建错误但支持率高的系统发育关系,因为联合分析忽略了每个基因不同的演化过程[35-36].对于具有不同演化背景基因的分析,溯祖分析更具有优势.现有的大部分溯祖分析方法(如BEAST[37],BUCKy[38]和ASTRAL等)主要是针对不完全谱系分选,对于杂交或基因渐渗也具有一定的容忍度.由此推断对于没有经历复杂演化历程的类群的系统发育关系分析,联合分析和溯祖分析可能都比较适用[39].叶绿体基因组表现为单亲遗传且整个基因组可被当成一个基因处理,因此被认为是用于推测系统发育关系非常理想的分子标记[40-42].但是,叶绿体基因组可能经历复杂的演化历史,如不完全谱系分选、重组,甚至不是非常严格的单亲遗传[43],叶绿体基因组上的基因也可能经历不同的演化历程[44].鉴于此,本研究采用联合分析和基于溯祖理论的ASTRAL对中国野生葡萄进行了系统发育分析.
但是,利用ASTRAL构建的系统发育树中,外类群聚到了内类群中.这可能是由于叶绿体基因组在种间的变异程度不是很高,大部分基因提供的信息有限,不利于溯祖分析.而联合分析重建了分辨率较高的系统发育树,其中3大分支的支持率都比较高,相应的QC和QI值也很高.
3.3 中国野生葡萄间的质核冲突
本研究比较了基于叶绿体基因组和核基因组[6]数据分别构建的系统发育树,两种结果存在非常大的差异;同时,不管是核基因组的系统发育关系还是叶绿体基因组的系统发育关系都不完全支持传统分类的处理[3-4].叶绿体基因树上的V.betulifolia和V.wilsoniae的姐妹群关系与传统分类[3]一致,但与核基因树[6]存在差异;核基因树[6]上V.tsoi和V.chungii的姐妹群关系也与传统分类[3]一致,但与叶绿体基因树不一致.
很多植物的分类都存在质核冲突现象,造成这种冲突的原因可能主要是杂交和不完全谱系分选[11,13,45].叶绿体捕获是比较特殊的杂交方式[46].Ma et al[6]构建的系统发育树是基于联合的直系同源基因的SNP数据,未考虑每个基因可能有不同的演化历程.有研究显示,中国野生葡萄可能存在快速辐射分化和基因渐渗现象[47].这些复杂的演化背景使得中国野生葡萄的系统发育关系比较难被澄清.因此,要解析中国野生葡萄质核冲突的原因,需要开展更多的研究,如增加野生葡萄每个种的样本数、采用合适的系统发育分析方法等.
本研究通过从NCBI上下载重测序数据,组装并注释了26种中国野生葡萄的叶绿体基因组,并利用多种方法重建了中国野生葡萄的系统发育关系.通过比较发现,全基因组的解析效果最好.溯祖分析不适用于信息比较缺乏的系统发育分析.
