基于迁移学习和支持向量机的乳腺癌分子分型预测
2022-05-31赵清一林勇
赵清一,林勇
上海理工大学健康科学与工程学院,上海 200093
前言
乳腺癌是女性常见的癌症,早期筛查及准确的诊断和治疗是应对乳腺癌的重要方法[1]。乳腺癌分子分型标准是一种基于基因表达和免疫组织化学分析方法的乳腺癌分类标准。1999年,美国国家癌症研究中心首次提出乳腺癌分子分型概念[2]。2000年,Perou 等[3]首次将乳腺癌分为雌激素受体(Estrogen Receptor,ER)阳性和ER 阴性。2009年,Cheang 等[4]用孕激素受体(Progesterone Receptor,PR)、ER、人类表皮生长因子受体2(Human Epidermal Growth Factor Receptor 2,HER-2)和Ki-67 4 种免疫组化指标将乳腺癌分为luminal A、luminal B、HER-2+和Basallike 4 种类型。以上4 种不同的乳腺癌分子分型在治疗方案和预后有较大的差异,因此准确的诊断乳腺癌分子分型对乳腺癌的临床治疗有重要的参考作用[5-7]。
目前传统的乳腺癌分子分型检测方法为免疫组织化学分析方法,该方法需要进行穿刺检查不仅会对患者造成创伤,并且有一定误诊几率[8]。美国临床肿瘤协会指出,全世界大约20%的免疫组织化学分析法得到的结果是不正确的[9]。因此,研究者尝试通过医学影像对乳腺癌分子分型进行诊断,并且已经得到一些研究成果。王世健等[10]提出半自动特征提取方法,提取DCE-MRI图像中形态特征、纹理特征以及动态增强特征等65维影像特征。利用逻辑回归方法评估影像特征和分子分型之间的关联性。但王世健等[10]提出的半自动特征提取方法较为复杂且需要人工选择特征,存在一定的主观性,难以科学准确地预测乳腺癌的分子分型。任湘等[11]通过卷积神经网络预测乳腺癌分子分型,但其仅使用了结构较为简单的卷积神经网络,并且使用的MRI 图像只有乳腺癌结构信息不包含乳腺癌分子信息,得到的曲线下面积(Area Under Curve,AUC)值最高为0.697,预测效果一般。以上研究存在方法繁缛,模型简单且准确率较低等不足。
针对以上方法的不足,本文创新性地基于迁移学习,选取Xception 深度卷积神经网络,使用基于ImageNet 数据集的预训练权重进行参数微调,再从网络中提取特征输入到支持向量机(Support Vector Machine,SVM)中,从而实现对乳腺癌分子分型分类预测。通过对75 例样本进行训练和测试,测试实验结果表明,本文提出的基于Xception 网络结合SVM的方法有效提高了乳腺癌分子分型预测准确率。
1 基于Xception+SVM的乳腺癌分子分型预测
1.1 数据集及预处理
本文回顾了2012年2月~2018年7月75 例乳腺癌患者的PET/CT 图像和乳腺癌分子分型信息,PET/CT 图像来源于复旦大学附属肿瘤医院,使用18F-FDG 显像剂,德国西门子公司PET/CT 机器进行采集,其中CT 图像的大小为512×512,PET 图像的大小为168×168。使用总样本的80%作为训练集,20%作为测试集。luminal B 型是最常见的乳腺癌分子分型且诊疗方案和预后与其它3 种分子分型有较大的差别,并且luminal B 型乳腺癌淋巴结转移率明显高于其它分子分型[12]。因此本文将75例病例分为luminal B 和非luminal B 两类,对luminal B 型和非luminal B型进行二分类预测研究。在75例乳腺癌患者中,luminal B 型有40例;非luminal B 型有35例,其中HER-2+有16 例,Basal-like 有17 例,luminal A有2例。
数据预处理步骤有肿瘤标记、RGB 多通道图像融合、数据扩增和归一化操作。首先由资深医生使用专业医学软件ITK-SNAP 在PET 图像上对肿瘤区域进行分割标记,肿瘤区域标记有助于去除图像中的无关信息,使得训练集和测试集的图像包含更多的肿瘤信息,有助于提升模型的准确率。CT 图像、PET图像、肿瘤标记图像如图1所示。
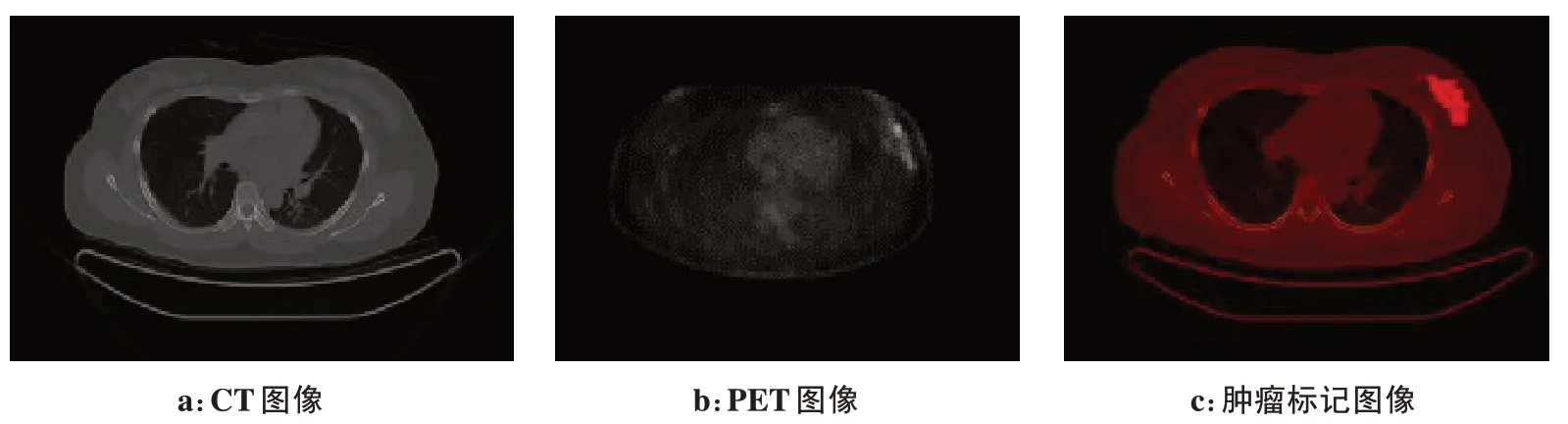
图1 CT图像、PET图像、肿瘤标记图像Figure 1 CT image,PET image and tumor marker image
文明等[13]提出医学图像融合可以将人体组织和器官的功能、代谢以及相应的解剖结构相结合,利用多种成像方法的各自优势,从而显著提高影像诊断的准确性和临床治疗水平。本文中的乳腺癌PET 图像包含了生物分子代谢、受体及神经介质活动等信息,包含了肿瘤分子层面的信息[14-16]。CT 图像则包含了生物组织结构信息和纹理信息[17-20]。医生标记的病灶图像则包含了肿瘤病灶信息。本文使用专业图像处理软件Image J对以上3种图像进行RGB多通道融合,生成多种关联生物信息的图像,便于之后图像的特征提取和分类[21]。
在进行肿瘤标记和图像多通道融合之后,截取包含肿瘤区域在内的大小为66×66 的图像作为感兴趣区域。同时,选取同一序列肿瘤横截面最大的图像,并且选取其前后各两张图像,一共5 张图像切片作为构建网络模型的图像数据。75 例样本一共得到375张原始图像。再对375张原始图像采用旋转和镜像翻转的方法进行数据扩增,首先将原始图像顺时针旋转30°、60°、90°、120°、180°、210°、240°、270°、300°,得到变换后的图像,其次将图像进行镜像翻转。原本样本数量为375 张,数据扩增10 倍之后扩充至4 125 张,其中训练集3 300 张,占总数据量的80%,测试集825 张,占总数据量的20%。最后为了有效地收敛到最优,找到最优解,本文使用最值归一化将所有数据归一化到0 到1 之间。归一化之后的数据使得网络收敛时更加平顺[22-23]。
1.2 模型构建
研究表明,应用卷积神经网络提取影像特征,再通过机器学习进行分类可提高医学图像分类的准确性。例如Teramoto等[24]提出在肺结节诊断中,首先使用卷积网络提取特征,再使用机器学习方法进行分类可以有效提高肺结节诊断准确率。因此,本文采用ImageNet 数据集预训练的Xception 网络提取乳腺癌的特征,并通过机器学习中经典的SVM 算法实现luminal B和非luminal B的二分类。
1.2.1 分析流程设计Xception+SVM 方法的分析流程如图2 所示。首先是数据预处理,该步骤在1.1 中已经说明,不再赘述。预处理后的标注图像作为训练集对在ImageNet 数据集训练过的Xception 网络参数进行微调,得到最优网络模型。然后使用SVM 替代Xception 网络的全连接层,使用不含全连接层的Xception 网络提取特征,再将特征作为训练数据,对SVM进行训练,最后使用测试集测试网络的性能。

图2 本文方法的分析流程图Figure 2 Flowchart of analysis by the proposed method
1.2.2 网络训练Xception+SVM 方法对乳腺癌非luminal B 和luminal B 进行二分类预测,Xception 是Chollet 等[25]在Inception V3 基础上改进的卷积神经网络。Xception 在基本不增加网络复杂度的前提下提高了模型的性能。Xception 网络在ImageNet 数据集Top5 的精准率达到了0.945,性能非常优秀。该网络利用深度可分离卷积的设计思想,使用可分离卷积(SeparableConv)来替代Inception V3 网络中的卷积层。SVM 是一种二元线性分类器,按照监督学习的方法对数据进行分类,通过训练集在特征空间中寻找正负样本超平面之间的最佳差异。因为SVM在小样本分类上的优异性能,本文选择SVM 对图像进行分类。
本文对Xception 网络和SVM进行训练,首先将预处理后的图像和分子分型信息作为训练集训练在 ImageNet 预训练过的Xception 网络。使用随机梯度下降(SGD)算法训练至收敛,并自动保存最佳网络模型。SGD 算法随机从训练集中选择样本,在样本选择上不需要使用全部的样本进行学习,只需要选择部分样本进行学习,所以其学习速度较快,可以快速更新。
在得到Xception 网络最优模型之后,使用从去除全连接层的Xception 网络提取的图像深度特征对SVM 进行训练。采用高斯核函数(RBF),设置惩罚系数C为1,核函数系数gamma为10。本文同时使用Vgg16+SVM 和GoogleNet+SVM 作为对比组,对Vgg16+SVM 和GoogleNet+SVM 进行训练和测试。Xception 网络分为3 层,依次为输入层、中间层以及输出层,其中输入层有6 个SeparableConv,中间层有3 个SeparableConv,并且中间层需要循环8次,输出层有4 个SeparableConv。Xception 网络输入层的尺寸为(229,229,3)。Xception+SVM 网络结构和参数如图3所示。
2 实验结果与分析
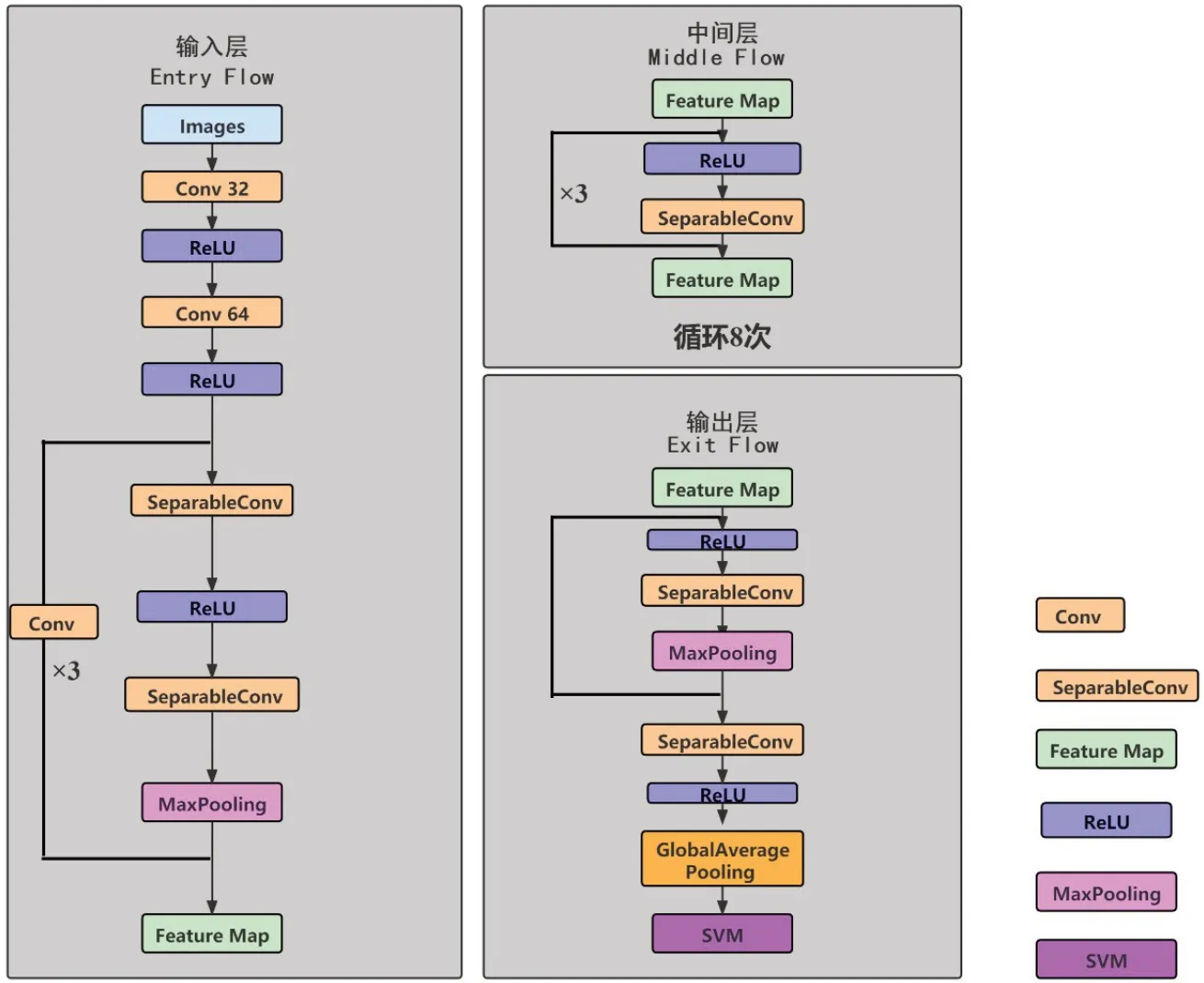
图3 Xception+SVM 网络结构图Figure 3 Xception+SVM network structure diagram
实验的硬件平台中央处理器为英特尔至强Gold 6142 处理器,主频为2.6 GHz。运行内存为32 G。实验软件环境为Windows 10,集成开发软件为Jetbrains公司的Pycharm,版本为2020.2,python 版本号为3.6,深度学习框架为Keras,版本号为2.1.6。
2.1 评价标准
采用以单张图像为单元的评价方法,其评价标准包括精确率(Precision)、准确率(Accuracy)、召回率(Recall)、受试者工作特征曲线及其AUC。设luminal B 为阳性,非luminal B 为阴性。乳腺癌图像对应的分子分型为阳性,预测为阳性,记作真阳性(True Positive,TP);乳腺癌图像对应的分子分型为阳性,预测为阴性,记作假阴性(False Negative,FN);乳腺癌图像对应的分子分型为阴性,预测为阳性,记作假阳性(False Positive, FP);乳腺癌图像对应的分子分型为阴性,预测为阴性,记作真阴性(True Negative,TN)。召回率为真阳性占整个阳性样本的比例,公式如下:

精确度为真阳性样本占所有预测阳性样本的比例,公式如下:

准确率是所有预测正确的样本占总样本的比例,公式如下:
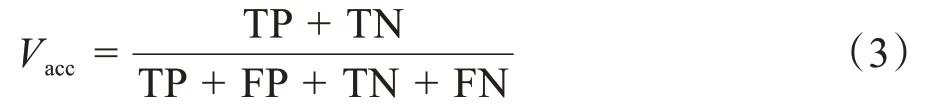
2.2 结果分析
实验采用10 折交叉验证,本文数据集在不同模型中的乳腺癌分子分型分类预测结果如表1所示。
表1 3种模型预测结果比较(± s)Table 1 Comparison of prediction results obtained by 3 models(Mean±SD)
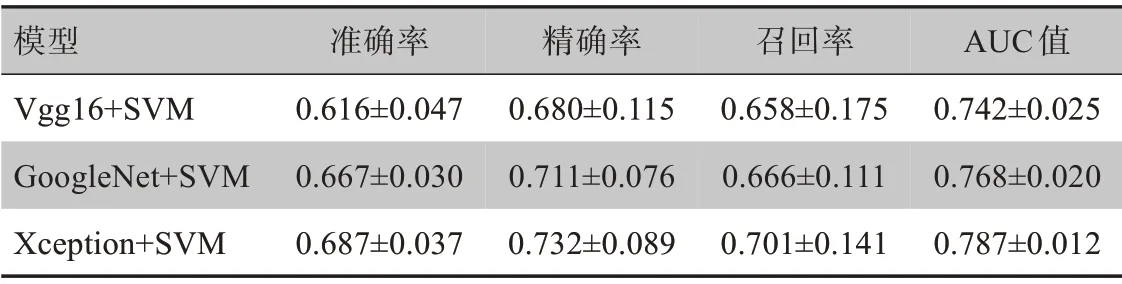
表1 3种模型预测结果比较(± s)Table 1 Comparison of prediction results obtained by 3 models(Mean±SD)
模型Vgg16+SVM GoogleNet+SVM Xception+SVM准确率0.616±0.047 0.667±0.030 0.687±0.037精确率0.680±0.115 0.711±0.076 0.732±0.089召回率0.658±0.175 0.666±0.111 0.701±0.141 AUC值0.742±0.025 0.768±0.020 0.787±0.012
由表1可以看出,本文使用的Xception+SVM模型的分类准确率为0.687,相比于使用Vgg16+SVM的分类准确率0.616和使用GoogleNet+SVM的分类准确率0.667,分别提高了7.1%和2.0%。使用Xception+SVM模型的精确率为0.732,相比于使用Vgg16+SVM的精确率0.680和使用GoogleNet+SVM的精确率0.711,分别提高了5.2%和2.1%。使用Xception+SVM模型的召回率为0.701,相比于使用Vgg16+SVM的召回率0.658和使用GoogleNet+SVM 的召回率0.666,分别提高了4.3%和3.5%。基于以上数据,可以得出本文构建的Xception+SVM模型的分类准确率、精确率、召回率均高于另外两种模型,分类效果最优异。本文提出的Xception+SVM模型的分类准确率为0.687,接近传统方法的0.8,高于任湘等[11]提出方法的0.653。召回率为0.701,高于任湘等[11]提出方法的0.647。
为了比较上述模型的分类性能,绘制各个分类器的ROC曲线,具体的ROC曲线图如图4所示。同时基于ROC曲线得出AUC。AUC的值越高表明分类效果越好。使用Xception+SVM、Vgg16+SVM、GoogleNet+SVM模型的AUC分别为0.787、0.742、0.768。基于各个网络的AUC数据,可以发现本文提出的Xception+SVM模型的乳腺癌分子分型预测效果优于Vgg16+SVM模型和GoogleNet+SVM模型。本文提出的Xception+SVM模型AUC 达到了0.787,高于任湘等[11]提出方法的0.671。测试结果表明,本文提出的Xception+SVM模型的准确率、召回率和AUC值均高于任湘等[11]提出的模型,有效地提高了乳腺癌分子分型预测效果。
3 总结与展望
准确诊断乳腺癌的分子分型对乳腺癌的治疗尤为关键。本文从临床的需求出发,以乳腺癌PET/CT图像为研究对象,创新性地将迁移学习技术和SVM应用于乳腺癌的分子分型预测研究,构建可信任的乳腺癌分子分型模型,预测其分子分型。实验结果表明,本文采用的基于Xception网络和SVM建立的模型对乳腺癌分子分型预测有一定的效果。为无创预测分子分型开拓了新的方向。本文在乳腺癌分子分型预测模型构建中,利用迁移学习思想,解决了小数量级的数据集训练卷积神经网络效果不佳的问题,将已经在ImageNet数据集训练完成的卷积神经网络进行迁移,并且用SVM替换原网络的全连接层构建乳腺癌分子分型预测模型。实验结果证明,迁移学习的使用有效地提升了网络的性能。同时在无创诊断乳腺癌分子分型方面进行了探索性研究。通过迁移学习和SVM建立的预测模型有一定的预测效果,该方法能有效减少患者的痛苦,为无创诊断乳腺癌分子分型提供了重要的价值。

图4 3种模型ROC曲线对比图Figure 4 Comparison of ROC curves of 3 models
但本研究仍然存在一些不足。例如由于医学图像的获取和标注较为困难,导致本文采集到的样本数量较少。另外本文提出的网络虽然有一定的预测效果,但参数的优化还需要进一步的提升,预测的准确率与传统方法相比还有一定的差距。乳腺癌分子分型预测研究依然存在巨大的挑战。在未来的研究中,可以通过改进算法、优化参数以及建立大数据量的已标注乳腺癌图像数据集来提高乳腺癌分子分型预测模型的性能。