基于Camoco 算法挖掘谷子叶鞘和叶枕颜色相关基因
2022-05-20杨宇琭罗韶凡郄倩茹张雅坤段明李亚军孙蓉韩渊怀李旭凯马芳芳
杨宇琭,罗韶凡,郄倩茹,张雅坤,段明,李亚军,孙蓉,韩渊怀,李旭凯*,马芳芳*
(1.山西农业大学 农学院,山西 太谷 030801;2.山西农业大学 生命科学学院,山西太谷 030801;3.山西农业大学 实验教学中心,山西 太谷 030801)
谷子(Setaria italic),禾本科狗尾草属,古称稷、粟、粱禾,为中国古代的五谷之首。其具有耐干旱、耐贫瘠、高光效等特点,是典型的环境友好作物,完全有可能重新成为主栽作物和主要粮食[1]。但是谷子产量没有较大的突破,经济效益低,种植面积增长幅度小[2]。影响作物产量的因素之一是光合作用,而叶绿体、碳代谢途径、以及多种酶类等都会影响光合速率[3-4]。其中叶绿素是光合作用的主要色素之一。在高等植物的叶绿体中,形成叶绿素a 和叶绿素b 的过程中涉及到多种酶、多个基因[5-6]。叶绿素含量减少或者过多都会影响作物的生长发育,有些甚至会导致植株死亡。研究表明叶鞘中合成的光合产物主要运输到作物穗等部位,对产量约有15%的贡献[7]。研究人员通过研究不同施氮条件下水稻叶鞘中与衰老相关的非结构性碳水化合物(non-structural carbohydratem,NSC),发现叶鞘中NSC 的积累与转运对水稻增产稳产具有重要意义[8]。因此,研究控制谷子叶鞘颜色和叶枕颜色的遗传机制作为挖掘植株颜色的切入点,对进一步提高谷子光合效率,提高谷子产量具有现实意义。
颜色性状是一种重要的标记性状,早在1980年就有人利用叶片颜色筛选谷子的有性杂交种[9]。一些研究人员按照叶鞘颜色对谷子品种类型进行了初步区分[10-11]。叶鞘颜色作为形态标记在水稻和小麦育种中的应用更广[12-13]。在高粱中结合叶鞘颜色开发了SRR(Simple Sequence Repeats)标记用于划分类群[14]。另外,谷子叶鞘颜色和叶枕颜色相对丰富,因此研究控制谷子叶鞘颜色和叶枕颜色的基因,对开发谷子形态标记和筛选谷子品种具有重要意义。前期,刁现民等对916 份谷子种质资源进行了重测序,对谷子的多种性状进行了统计,包括叶鞘颜色和叶枕颜色,为本研究奠定了基础[15]。
近年来,高通量测序技术快速发展,转录组测序在植物研究上也逐渐被普及[16],与此同时,生物信息分析工具和方法也在不断的发展与更新[17]。全基因组关联分析(Genome-wide association studies,GWAS)已在作物上广泛应用,但传统的GWAS 由于连锁不平衡的特点,经常会出现数十个甚至上百个基因座与性状相关联,而这些基因座中往往包含数百个SNP 位点,其中许多SNP 位点距离真正导致表型变异的位点还比较远,这些多余的信息会干扰确定候选基因[18]。另外很多物种的基因注释信息并不完善,这也成为鉴定和确认功能基因的障碍。因此本研究使用了美国明尼苏达大学的研究团队开发的Camoco(Co-analysis of molecular components)的算法,其原理是将全基因组关联分析(GWAS)和共表达网络整合起来,通过识别GWAS 筛选出的显著SNP 位点附近的基因并对判断优先级,确定共表达较强的基因,将结果与随机网络进行比较,把P值分配给候选基因,筛选出高置信度的候选基因。已有研究使用此方法对玉米17个性状进行分析,用3个基因表达数据集和两种共表达网络验证此方法,并对筛选出的高置信候选基因使用突变体进行了功能验证[19-20]。
1 材料与方法
1.1 试验材料
GWAS 数据:刁现民研究员团队916 份谷子种质资源重测序数据[15]。
转录组数据:名优品种晋谷21(JG21)种植于山西农业大学试验田,在其生长周期中,对18个组织时期取样,每个样设置3个重复,进行转录组学分析。具体组织时期有:JG21-发芽种子-3 天、JG21-幼苗-2 周、JG21-植株-两叶一心、JG21-未成熟小穗-S2、JG21-未成熟小穗-S4、JG21-旗叶-灌浆期S3、JG21-旗叶叶鞘-灌浆期S3、JG21-倒4叶-灌浆期S3、JG21-倒4 叶叶鞘-灌浆期S3、JG21-根-灌浆期、JG21-茎杆-灌浆期S3、JG21-倒2、3 叶-30 天、JG21-倒2、3 叶-抽穗、JG21-未成熟种子-S1、JG21-未成熟种子-S2、JG21-未成熟种子-S3、JG21-未成熟种子-S4、JG21-未成熟种子-S5。(S1:籽粒颜色为鲜绿色,质地为乳状;S2:籽粒颜色为暗绿色,质地为乳状;S3:籽粒颜色为黄绿色,质地中出现粉状物;S4:籽粒颜色为暗黄色,质地为固态)。
1.2 试验方法
1.2.1 GWAS 数据分析
以豫谷1号基因组为参考,依据上述916 份谷子种质资源GWAS 结果选取候选SNP 位点上下游500 bp 的序列,通过Blast 比对到谷子高质量基因组xiaomi上[21],定位到新基因组位置。
1.2.2 转录组数据分析
基于Illumina 技术得到的双端测序数据,利用FastQ[22]软件对数据进行质控,并使用Trimmonatic[23]软件依据碱基质量对fastq 文件进行修剪,过滤低质量的reads,得到clean data。利用Hisat2[24]软件将上一步数据比对到xiaomi基因组[21]。用featureCounts[25]获得数据的reads 计数,之后计算TPM(transcripts per million)值。
1.2.3 Camoco 分析及流程
利用Camoco 将基因表达量数据和GWAS 分析获得的SNP 位点信息结合[19],定位到基因。具体参数设定可参谷子的设定,指定50 kb(SNP 位点向上下50 kb)范围和1个侧翼基因,以及范围外的1个侧翼基因。如果指定的范围不覆盖任何基因,侧翼基因允许使用最近的基因。candidatewindow-size 自行根据所研究物质的连锁不平衡(LD)范围改变。
1.2.4 加权共表达网络分析
使用R 软件(R version 4.0.2)中的WGCNA(R version 1.6.6)包[20],构建加权基因共表达网络并划分相关模块。采用一步法构建共表达矩阵,使用WGCNA 包中的pickSoftThreashold 计算权重值,根据表达量的相关性构建基因聚类树。设置基因模块中最少基因数为30,按照混合动态剪切的标准划分模块,并计算每个模块的特征向量值,对模块进行聚类分析,将距离较近的模块合并为新的模块。
1.2.5 模块的GO 富集分析
提取模块中的基因,利用TBtools 进行GO 富集分析[26],确定候选基因的主要生物学功能。使用Cytoscape(version 3.6.1)对模块中的基因进行可视化[27]。
1.2.6 谷子叶鞘颜色和叶枕颜色候选基因的表达模式分析
在山西农业大学谷子多组学数据库(http://sky. sxau. edu. cn/MDSi. htm)中输入候选基因的信息得到候选基因在谷子各组织时期的表达模式[21]。
2 结果与分析
2.1 基于GWAS 定位谷子叶鞘颜色和叶枕颜色相关基因
通过筛选GWAS 结果,发现叶鞘颜色和叶枕颜色与多个种植地区GWAS 结果关联位点显著且一致。2种性状在不同环境下的GWAS 曼哈顿图[15]结果显示:在5种环境下谷子叶鞘颜色和叶枕颜色的GWAS 结果中,位于7号染色体和4号染色体上存在显著大于阈值的SNP 位点(图1)。筛选鉴定出与谷子叶鞘颜色和叶枕颜色显著的SNP 位点,初步获得颜色性状相关的候选基因(增强出版附件1)。

图1 谷子叶鞘颜色和叶枕颜色在不同环境下的GWAS 曼哈顿结果图Fig.1 GWAS Manhattan Result Map of leaf sheath color and pulvinus color in Different Environment
使用Camoco 算法中SNP 到基因位点定位(SNP to gene mapping)的功能,对上述基因进行筛选,得到一个高置信度的候选基因列表(表1)。在叶鞘和叶枕中分别筛选到了8个和5个与颜色相关的候选基因。利用CandiHap[28],绘制候选基因的结构图,并绘制出基因所在的染色体位置,非编 码 区UTR(Untranslated Region)、编 码 序 列CDS(Coding sequence)的位置和大小,包括相应发生在外显子区域的突变位置(增强出版附件2)。
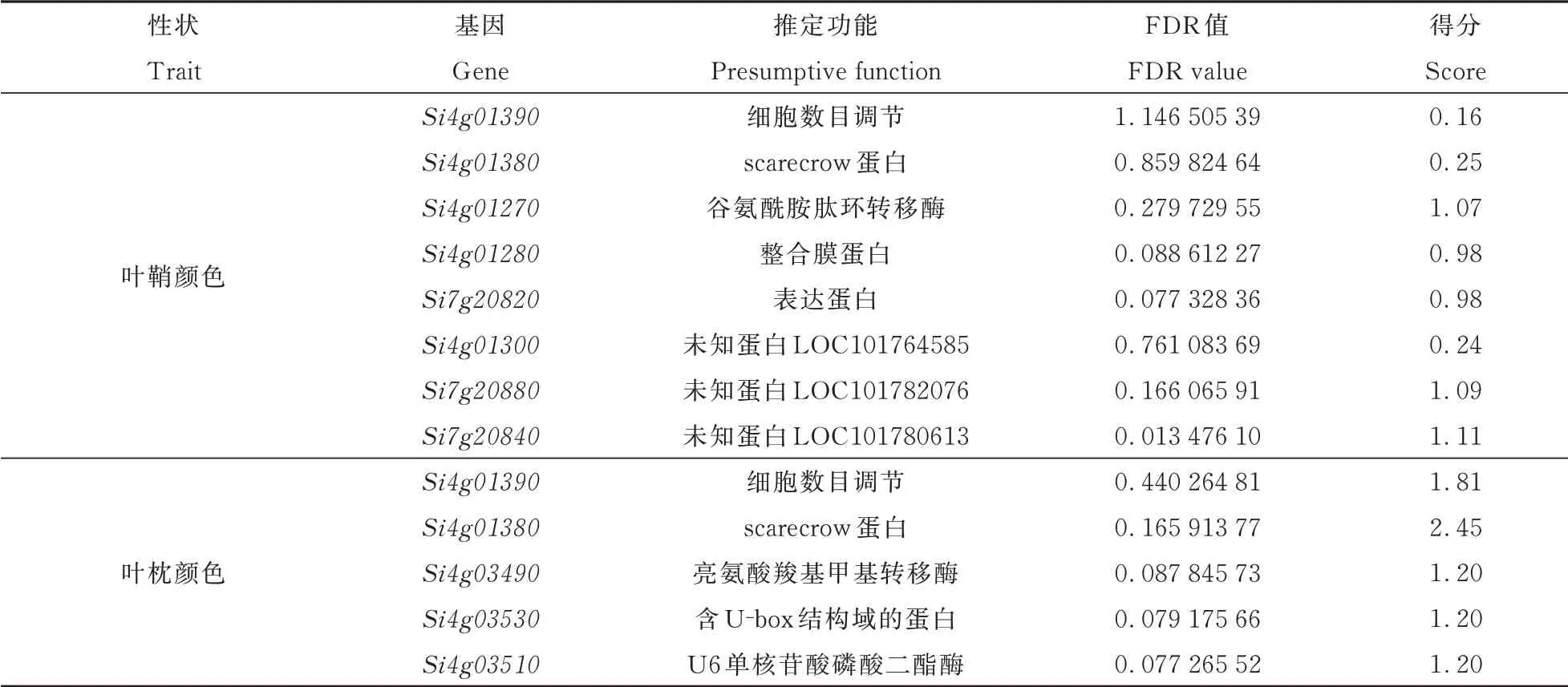
表1 Camoco 筛选结果Table 1 Camoco screening results
2.2 基因共表达网络模块的构建及筛选结果
对表达矩阵中的基因进行过滤,获得31 980个高表达基因。通过WGCNA 算法,选择权重值β=10 构建无尺度网络,按照混合动态剪切的标准划分模块,最终获得37个模块(图2)。将Camoco分析中得到的候选基因与模块中的基因进行比较,确定候选基因在模块中的分布情况(图3)。

图2 基因聚类树和样品分割Fig.2 Gene cluster dendrograms and module detecting
2.3 相关模块及候选基因分析结果
对谷子叶鞘颜色和叶枕颜色候选基因所在的模块分别进行GO 功能富集分析,并使用R 语言中ggplot2 包,将其进行可视化,发现2 者共同富集到了与质体(GO:0009536),叶绿体(GO:0044434),四吡咯生物合成过程(GO:0033014),色素的生物合成过程(GO:0046148),光反应(GO:0019684)等生物学过程(图4,增强出版附件2 图3,附件3 表1)。以上的富集结果有力的证明了Camoco 挖掘的基因较为全面。

图3 候选基因在模块中的分布情况Fig.3 The distribution of candidate genes in the module
整合Camoco 分析得到的基因和GO 富集分析的结果(增强出版附件3),本研究共获得了10个与颜色相关的候选基因,推测谷子中Si4g01380、Si4g01390、Si4g01300、Si7g20820、Si7g20840、Si7g20880对叶鞘颜色起调控作用,Si4g01380、Si4g01390、Si4g03530、Si4g03510对谷子叶枕颜色起调控作用。
2.4 谷子叶鞘颜色和叶枕颜色候选基因的表达模式分析
对谷子叶鞘颜色和叶枕颜色这2种性状候选基因进行表达模式分析,发现这些基因在整个生育期都有表达,但普遍在苗期中表达量较高,说明在这些基因的调控下谷子表型在苗期开始出现差异(增强出版附件2,图4~图11)。如谷子叶鞘颜色的候选基因Si401300谷子全生育期都有表达但在苗期中表达量最高,在旗叶叶鞘和倒4 叶叶鞘中的表达量分别为90.225 和114.745(图5)。

图4 与谷子叶鞘颜色相关的3个模块中共表达基因的GO 功能富集Fig.4 GO function enrichment of co-expressed genes in 3 modules related to the color of foxtail millet leaf sheath.

图5 谷子Si4g01300 基因表达模式Fig.5 Expression pattern of Si4g01300 gene in foxtail millet
2.5 互作网络的构建及枢纽基因功能注释
选取叶鞘颜色候选基因所在的模块并将其作为核心基因使用Cytoscape 网络图工具,选择软阈值作为连通性计算方法,构建局部共表达网络图(图6)。同理构建谷子叶枕颜色的共表达网络图(增强出版附件2 图12)。同时,用WGCNA 深入挖掘相关模块内的枢纽基因,并对未知功能的基因进行功能注释。本试验对共表达网络图中的枢纽基因进行了功能注释(增强出版附件4 表1),为以后验证未知功能的基因提供参考。如在叶鞘颜色 的 green 模 块 中Si1g38040、Si1g18850、Si2g07320等27个枢纽基因都注释到与叶绿体相关;Si3g17790、Si3g17800直接注释到细胞色素P450;Si3g31460、Si2g36540等基因注释与水解酶相关,水解酶与植物的衰老密切相关。

图6 谷子叶鞘颜色基因在相应的3种模块中的局部共表达网络图Fig.6 Local co-expression network of sheath color genes in the corresponding 3 modules
3 讨论
GWAS 分析与传统的连锁分析相比较具有耗时少、广度大、精确度高等优势,但是GWAS 研究中很容易出遗传性缺失现象,以往为解决此问题通常通过增加样本量,提高检测效率,或以整个代谢通路或其功能类型作为单位进行关联以及富集分析,通常不关注个体的功能基因。另外从SNP位点到基因的定位通常是人工检查筛选,因此增加了很多人为因素,很可能错过真正对性状起调控作用的基因。因此有人利用一些功能基因组数据的互补性来解释GWAS,在人类和拟南芥的研究中建立了这样的共表达网络[29-30],此方法被称为GWAB(genome-wide association boosting),但大部分作物功能基因的基因注释数据都不完整,此方法普遍应用较为困难。因此本文使用Camoco算法利用基因表达量筛选候选基因,既大大减少了候选基因的数量,又可以将表达模式相似的基因筛选出来,减少GWAS 分析定位到假阳性基因的概率。本研究通过GWAS 分析,在叶鞘颜色中筛选到10个相关的SNP 位点,叶枕颜色中筛选到11个相关的SNP 位点,此区段共预测到相关基因278个。Camoco 算法计算后共得到12个基因,进一步结合WGCNA 分析共得到10个候选基因,且这些基因都直接或间接与植物体内叶绿体的功能有关[31-36]。如谷子叶鞘颜色的候选基因Si4g01300编码了一种DUF3754 域蛋白质,其在谷子中的具体功能还不是很明确,但有研究人员发现一些DUF 家族基因与植物发育和细胞的生长、叶绿体运动等功能有关[37],已有研究表明叶鞘颜色会受多对隐性基因或者单显性基因控制,并证明这些基因调控的代谢途径会受光照等因子的影响[38]。另外谷子叶枕颜色的候选基因Si4g03530的推定功能是泛素连接酶家族,E3 泛素连接酶,在玉米的穗位叶的叶绿体中检测到该基因的表达[39],并且该连接酶家族可能与植物细胞的自然凋亡相关。双形状共定位到的基因Si4g01380则与半胱氨酸合成酶在叶肉细胞叶绿体中对无机硫的催化有关,在基因GO 注释中主要表达于质体组织等细胞组分内,其编码GRAS 家族转录因子,该家族中有多个转录因子参与光敏色素(phytochrome)信号传导[40~42],光敏色素为色素蛋白结构,是叶绿素等四吡咯物质的基本类型。除此之外,通过共表达网络挖掘到的枢纽基因也很重要,其中一些基因如Si3g31460、Si2g36540没有直接富集到与色素合成相关的GO term,但富集到与蛋白质水解,水解酶活性,转移酶活性和激素相关的GO term,这些与植物衰老有密切关系,植物衰老对谷子的颜色性状有显著影响。
4 结论
本研究基于谷子叶鞘颜色和叶枕颜色的全基因组关联分析,初步确定相关的SNP 位点,利用Camoco 软件初步确定的SNP 位点进行筛选,挖掘到高置信度的候选基因,对其进行功能注释,并构建共表达网络,对相关模块进行GO 富集分析,得到了具有生物学意义的功能富集结果。总之,本研究利用Camoco 算法结合共表达网络确定了10个候选基因,其中叶鞘颜色6个,叶枕颜色4个,Si4g01380和Si4g01390是两个性状共同定位到的候选基因。这些发现为深入探索调控谷子相关颜色的分子机制提供一定的理论依据,对培育高产谷子品种、发掘谷子形态标记以及开发各种功能性谷子具有重要意义。
