玉蜀黍尾孢菌VelB 基因敲除及生物信息学分析
2022-05-20路媛媛孙承龙谭玉琴魏文杰史咸春宋艳波李丽刘振宇
路媛媛,孙承龙,谭玉琴,魏文杰,史咸春,宋艳波,李丽,刘振宇
(1.山西农业大学 生命科学学院,山西 太谷 030801;2.山西农业大学 园艺学院,山西 太谷 030801;3.山西农业大学 信息科学与工程学院,山西 太谷 030801)
玉米灰斑病是世界玉米种植区重要真菌性病害之一,其病原菌为玉蜀黍尾孢菌(Cecrospora ze⁃ae⁃maydisTehon & Daniels),属子囊菌门球腔菌属真菌[1]。自1991年首次在我国辽宁丹东地区发现该病害以来,玉米灰斑病菌在我国多次爆发流行,近些年在东北地区大面积发生,危害程度达3~5 级,严重地块达7 级,给玉米生产造成了惨重损失[2]。随后在山东、吉林、云南等地区相继发生,成为我国东北和西南玉米产区一种重要病害[3]。
病原菌致病性分化与变异是引起病害流行的主导因素之一。对病原菌进行致病性研究发现,病原菌侵染后显症速度不同,且出现的病斑类型也存在明显差异。通过血清学和蛋白质组学研究证明了病原菌存在分化现象[4]。对玉蜀黍尾孢菌致病性的研究主要集中在尾孢毒素方面。长期以来,致病学家和化学家研究内容主要集中在尾孢毒素的分离、纯化[5]和化学结构鉴定[6]等方面。目前对尾孢毒素的提取仍采用浓缩浸提法[7],研究发现该毒素对寄主保护酶及玉米胚根的生长有抑制作用,对玉米胚根细胞膜具有伤害,且能够引起叶片的电渗值发生变化[8-10]。但目前对该毒素的主要成分仍有争议[11-12]。由上可知,对玉蜀黍尾孢菌致病分子机理的研究较少。因此,尽快寻找发掘相关致病基因对于摸清玉米灰斑病菌致病性分子机理,以及玉米灰斑病的防治具有重要意义。
Velvet 蛋白家族最早发现在模式真菌Asper⁃gillus nidulans中[13],是一类真菌特异的具有Velvet 结构域的蛋白。该家族含有VeA(Velvet),VelB(Velvet like B),VosA(Viability of spores A)和VelC(Velvet like C),具有调控真菌次生代谢及生长和发育的作用。其中Vel B 是Velvet 家族中唯一包含2个Velvet 结构域的成员[14]。近些年研究结果表明,VelB可通过调控Cochliobolus sati⁃vus、Fusarium oxysporum、Curvularia lunata等 子囊菌的次生代谢产物、孢子产生及子实体的形成,最终调控致病性[15-17]。本课题组前期研究利用农杆菌介导遗传转化技术,获得玉米灰斑病菌PH6WC(强致病类型)突变体库,通过形态学和致病性分析,从突变体库中获得一个产孢量下降,致病力明显减弱的菌株,通过Tail-PCR 分析,获得T-DNA 插入位点为VelB基因的启动子区域[18]。
因此,本文克隆玉蜀黍尾孢菌的CzVelB基因序列,并进行生物信息学分析,研究CzVelB基因与玉蜀黍尾孢菌致病性之间的关系。构建ATMT敲除载体,获得敲除转化子,为研究CzVelB对玉蜀黍尾孢菌致病性的分子机制奠定基础,同时也为加快该重大植物病害的防控理论研究提供新的思路和策略。
1 材料和方法
1.1 试验菌株、载体及试剂
本试验所用的玉米灰斑病菌菌株Cz-1,大肠杆菌菌株DH5α、农杆菌为EHA105 由山西农业大学生命科学学院保存,载体构建所需的pPZP100和pCT74 购买于武汉淼灵生物科技有限公司。
本文中所使用2×Es Taq Master Mix,所用试剂盒及抗生素等试剂购于沈阳森宇生物科技有限公司。
限制性内切酶、T4 连接酶、PMD19-T Vector购于Takara 生物技术有限公司。
1.2 试验方法
1.2.1 生物信息学分析
根据NCBI 基因组数据库中Colletotrichum graminicola中 的VelB 保 守 区 序 列,从JGI 数 据 库中Cecrospora zeae-maydis 基因组中查询到相似度较高的基因,采用DNAMAN 软件对C. zeae-maydis 基因组中的VelB 蛋白序列进行同源性比对分析,最终确定其基因序列和拷贝数。根据所得到的基因序列,利用多种生物信息学软件对CzVelB基因的性质、结构及功能进行预测。
生物信息学分析软件如表1所示,分别对Cz-VelB 蛋白的氨基酸组成、理化性质、疏水性、跨膜结构、磷酸化位点、亚细胞定位、保守结构域以及信号肽进行分析。在NCBI 数据库中Blast 搜索其他病原真菌VelB 基因序列,使用MEGA 5.0 软件,利用NJ 法构建系统发育树,Bootstrap 分析重复数为1000。

表1 生物信息学分析工具及网址Table 1 Bioinformatics analysis tools and websites
1.2.2 CzVelB 侧翼序列的获得
首先采用CTAB 法[19]对玉米灰斑病菌进行基因组DNA 的提取。其次根据CzVelB 序列,利用DNAMAN 和BioXM 软件设计特异性引物,并结合PCT74 载体上标记基因序列和pPZP100 骨架载体序列,在特异性引物的5'端加入酶切位点,最后利用高保真PCR 技术获得侧翼序列。
引物如下,其中gcc为保护碱基,小写字母并带有下划线为酶切位点碱基。
CzVelB-5F-F1: gccgtcgacTGTGTACCCGACCTCAGTG
CzVelB-5F-F2:gcctctagaTTCCATAAATCGACCGTGAG
HphGFP-F: gcctctagaCAATTAACCCTCACTAAAGG
HphGFP-R: gccggtaccTAATACGACTCACTATAGGG
CzVelB-3R-R1: gccggtaccTCAACCTCGTCCGTCATTCG
CzVelB-3R-R2: gccgagctcAGGTGACAAATTAGGCGCTT
高保真PCR 反应体系:DNA 1.0 μL,5×Phusion HF 10.0 μL,dNTP mixture(10 mM)1.0 μL,primer(10 μM)2.5 μL,最 终ddH2O 定 容 至50.0 μL。
PCR 反应程序:98 ℃,30 s;98 ℃,10 s;Tm,30 s;72 ℃,30 s 共30个 循 环;72 ℃,10 min;4 ℃,保存。
1.2.3 CzVelB 敲除载体构建及验证
以野生菌Cz-1为模板,分别采用CzVelB-5FF1/CzVelB-5F-F2、CzVelB-3R-R1/CzVelB-3RR2 和HphGFP-F/HphGFP-R 扩 增 上 游、下 游 和标记基因片段,扩增产物连入pMD19-T 载体,大肠杆菌转化后,用质粒试剂盒提取质粒,酶切验证。上游片段和pPZP100 经SalⅠ和XbaⅠ酶切后连入,构成重组质粒pPZP100CzVelB-5F。随后用KpnⅠ和SacⅠ酶切下游片段和重组质粒pPZP100CzVelB-5F,构成重组质粒pPZP100Cz-VelB-5F3R,最后分别采用HindIIⅠ和KpnⅠ酶切重组质粒pPZP100CzVelB-5F3R 和标记基因片段,形成敲除载体质粒pPZP100HphGFP-Cz-VelB(图1)。

图1 敲除载体pPZP100HphGFP-CzVelBFig.1 Delection vector pPZP100HphGFP-CzVelB
1.2.4 CzVelB 敲除突变体获得及验证
将上述构建好的敲除载体质粒pPZP100Hph-GFP-CzVelB,利用冻融法转化到农杆菌感受态EHA105 细胞中[18]。将疑似含有敲除载体质粒pPZP100HphGFP-CzVelB 的农杆菌,置于28 ℃培养至菌液浑浊,随后进行PCR 验证。
通过农杆菌介导的方法,将含有敲除载体的农杆菌转化到玉蜀黍尾孢菌基因组中,获得敲除突变体[19]。将转化所得拟敲除突变株进行单胞分离,继代培养后,取长势较好的疑似转化子接种在250 μg·mL-1头孢霉素、200 μg·mL-1潮霉素的PDA培养基中进行验证。随后提取疑似转化子DNA,最终利用PCR 的方法进行验证。
2 结果与分析
2.1 CzVelB 基因序列的获得
根据NCBI 中Colletotrichum graminicola中的VelB 保守区序列(XM_008095148.1),利用JGI 数据库中Cecrospora zeae-maydis 基因组序列,分析获得1个相似度高达95%的VelB 基因,将该基因定为CzVelB。该基因全长为1184 bp。
2.2 CzVelB 生物信息学分析
2.2.1 CzVelB 的蛋白质组成及理化性质
利用DNAMAN 对CzVelB 编码的氨基酸组成进行分析,结果表明该基因编码氨基酸为385个,含有一个开放阅读框。蛋白序列如图2所示。

图2 CzVelB 基因碱基序列及氨基酸序列Fig.2 Gene and amino acid sequence of CzVelB gene
利用ProtParam 对CzVelB 编码的氨基酸进行理化性质分析,结果表明该蛋白42.136 45 kDa,等电点(PI)为9.05,正(Asp+Glu)/负(Arg+Lys)的电荷残基数分别为31 和26,分子式为C1884H2887N519O561S11,总原子数为5862,不稳定性指数值为58.27(<40 稳定;>40 不稳定[20]),因此该蛋白属于不稳定蛋白,脂肪指数为67.90,总平均亲水性是-0.434,为亲水性蛋白。
2.2.2 CzVelB 蛋白的亲水性/疏水性分析
利用ProtScale 对CzVelB 蛋白亲/疏水性进行预测分析,结果如图3所示。CzVelB 蛋白在第268位氨基酸上得分为1.15,有最强疏水性;而在第12位上亲水性最强,最低值为-2.10。玉米灰斑病CzVelB 蛋白的亲水性总平均值为-0.434,表明该蛋白为亲水性蛋白。

图3 CzVelB 蛋白的疏水性和亲水性分析Fig.3 Hydrophobic and hydrophilic analysis of CzVelB
2.2.3 CzVelB 蛋白跨膜结构域分析
利用TMPRED 对CzVelB 蛋白的跨膜结构域进行预测分析(图4)。得分越高,则说明该蛋白质位于某一部位的概率就越大。根据预测结果可知该蛋白处于膜外,没有跨膜结构区。

图4 CzVelB 蛋白质跨膜结构域分析Fig.4 Transmembrane analysis of CzVelB
2.2.4 CzVelB 蛋白磷酸化位点分析
磷酸化多数情况下是发生在丝氨酸(Serine,Ser)、苏氨酸(Threoine,Thr)、酪氨酸(Tyrosine.Tyr)等氨基酸的残基上。利用NetPhos 3.1 在线软件在线分析CzVelB 蛋白的磷酸化位点,结果如图5所示。发现在该蛋白上存在有17个丝氨酸激酶磷酸化位点(第21、44、146、149、157、158、169、200、274、285、298、307、323、346、356、365、372 位氨基酸)、5个苏氨酸激酶磷酸化位点(第97、111、122、137、280 位氨基酸)以及7个酪氨酸激酶磷酸化位点(第82、172、202、206、221、283 和360 位氨基酸)。该结果说明,CzVelB 能够被激酶磷酸化,从而实现其功能的调控。
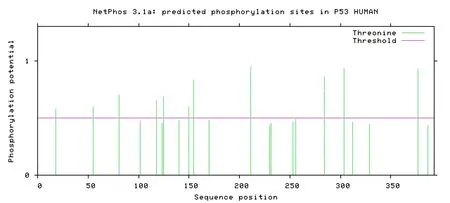
图5 CzVelB 蛋白磷酸化位点分析Fig.5 Phosphorylation sites analysis of CzVelB
2.2.5 CzVelB 蛋白的亚细胞定位
采用PSORT 对玉米灰斑病CzVelB 的亚细胞定位进行预测,结果显示:CzVelB 在细胞核(nuclear)、细胞质(cytoplasmic)、线粒体(mitochondrial)、过氧化物酶(peroxisome)、高尔基体(Golgi apparatus)的分布比例分别为15.5%、7.5%、2%、1%、1%,说明CzVelB 蛋白定位在细胞核中。
2.2.6 CzVelB 蛋白功能结构域
利用NCBI 对CzVelB 蛋白进行蛋白质结构域分析(图6)。其氨基酸序列含有2个Velvet 结构域,具有Velvet 蛋白的典型特征。

图6 CzVelB 蛋白结构域分析Fig.6 Conserved domain analysis of CzVelB
2.2.7 CzVelB 蛋白信号肽分析
利用软件SignalP-4.1 预测玉米灰斑病Cz-VelB 编码蛋白信号肽。C-score、S-score、Y-score分别代表着剪切位点的得分、信号肽的得分和综合酶切位点的得分。信号肽预测结果计算方式为以上3个值均为最大值的位点才是可信度最高的信号肽剪切位点,由图7可知得出该蛋白不存在信号肽位点,目前可以推测该蛋白为非分泌蛋白。

图7 CzVelB 蛋白的信号肽分析Fig.7 The signal Peptide graph analysis of CzVelB
2.2.8 CzVelB 系统进化分析
结合GenBank 和JGI 数据库中Curvularia lu⁃nata等多种植物病原菌的VelB保守区序列,利用MEGA5 对CzVelB进行系统进化分析(图8)。A.flavus、A. niger、Cecrospora zeae-maydis、Emeri⁃cella nidulans聚为一类,Curvularia lunata、Collet⁃otrichum graminicola和Cochliobolus heterostro⁃phus聚为一类,其中Cecrospora zeae-maydis 与A.flavus、A. niger亲 缘 关 系 较 近。CzVelB的 同 源 性分析进一步验证了该基因具有保守性。即推测出的CzVelB功能与A. flavus、A. niger的VelB基因功能相似,参与调控玉蜀黍尾孢菌致病性。

图8 CzVelB 与其他病原菌的系统进化分析Fig.8 Evolutionary analysis of CzVelB amino acid system
2.3 CzVelB 敲除载体构建及验证
根据CzVelB 基因序列设计特异性引物,分别扩增上游片段315 bp 和下游片段281 bp(图9-A,B)。利用HphGFP-F/HphGFP-R 扩增出带有HPH 和GFP 的标记基因片段2600 bp(图9-C)。
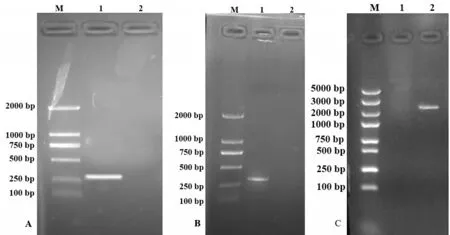
图9 CzVelB 侧翼序列和标记基因获得Fig.9 Flanking sequence of CzVelB and HphGFP sequence
利用HindIIⅠ和KpnⅠ验证标,2 条片段大小分别为2915 bp 和6917 bp,说明载体构建成功(图10)。

图10 pPZP100HphGFP-CzVelB 载体验证Fig.10 Confirmation pPZP100HphGFP-CzVelB by digested
2.4 农杆菌转化验证
将疑似含有敲除载体质粒pPZP100HphGFPCzVelB 的农杆菌进行PCR 验证,分别利用Cz-VelB-5F-F1/CzVelB-5F-F2、 CzVelB-3R-R1/CzVelB-3R-R2 和H phGFP-F/HphGFP-R 对 农杆菌进行菌落PCR,最终分别获得315、281 和2600 bp 大小的目标片段(图11)。以上结果表明pPZP100HphGFP-CzVelB 敲除载体已成功转入农杆菌菌株中。

图11 PCR 验证敲除载体pPZP100HphGFP-CzVelB 转入农杆菌Fig.11 PCR verification of transformation of pPZP100HphGFPCzVelB into Agrobacterium
2.5 CzVelB 敲除突变体获得及验证
经含有250 μg·mL-1头孢霉素、200 μg·mL-1潮霉素的PDA 抗性培养基筛选获得敲除突变体(图12)。并根据玉蜀黍尾孢菌VelB基因载体构建过程中设计的特异性引物,对ΔCzVelB进行PCR 验证。利用CzVelB-5F-F1/CzVelB-3R-R2 引物,以野生型菌株Cz-1 基因组为模板,扩增出的全长片段大小为616 bp,突变体扩增出片段大小为3196 bp;利 用CzVelB-5F-F1/HphGFP-R 引 物,以野生型菌株Cz-1 基因组为模板为扩增出条带,但突变体扩增出2915 bp 大小的片段,与预期大小一致(图13),证明获得了CzVelB的敲除突变体。

图13 PCR 验证敲除突变体Fig.13 PCR identification of ΔCzVelB
3 讨论与结论
本文采用生物信息学手段分析CzVelB 蛋白保守结构域表明,其氨基酸序列含有2个VelB 蛋白的保守结构域-Velvet 结构域,具有Velvet 蛋白的典型特征。已有研究表明,Aspergillus nidu⁃lans、Fusarium oxysporum、Aspergillus flavus等 真菌的VelB 蛋白均存在Velvet 结构域,且结构域保守[16,21]。通过结合系统发育树进一步分析,C. ze⁃ae-maydis 与A. flavus、A. niger亲 缘 关 系 较 近。因此,玉蜀黍尾孢菌具有典型的VelB 蛋白功能。
另外,针对CzVelB 蛋白理化性质、亲水性、疏水性等分析,结果表明该蛋白为亲水性蛋白,无跨膜结构区,这与Ustilago maydis和Cochliobolus heterostrophus的分析结果相一致[22-23]。
亚细胞定位结果表明,在分析玉米大斑病菌VelB 蛋白时发现,该蛋白位于细胞质中,因此可能需要从细胞质中转运至细胞核中,进而发挥作用[24]。但本文分析CzVelB 蛋白位于细胞核中,且不存在信号肽,这与杨峥的研究结果不一致。针对蛋白磷酸化结果分析,均发现CzVelB 蛋白存在大量的磷酸化位点,能被激酶磷酸化,从而实现其功能的调控。因此推测CzVelB 蛋白参与调控玉蜀黍尾孢菌刺激代谢途径。
国内外研究表明,ATMT 技术是目前丝状真菌遗传转化最常用的手段,因此获得目标基因的敲除突变体为后续研究该基因的功能奠定基础。本文根据玉蜀黍尾孢菌的VelB基因序列,设计特异性引物,扩增出侧翼片段和标记基因片段,并成功连入骨架载体中,构建CzVelB基因的敲除载体pPZP100HphGFP-CzVelB,最终利用ATMT 技术获得ΔCzVelB,为进一步研究该基因的功能分析提供基础材料。
