林麝FASN基因CDS区克隆及序列分析
2022-05-16谌颖莲赵贵军封孝兰张承露吴佳勇曾德军
谌颖莲 竭 航 赵贵军 封孝兰 张承露 吴佳勇 曾德军
(重庆市药物种植研究所,特色生物资源研究与利用川渝共建重点实验室,重庆,408435)
麝香在心脑血管疾病及神经系统疾病中具有较好的疗效,在香料行业中具有较高的经济价值[1-3],其主要来源途径为林麝(Moschusberezovskii)的人工养殖及其活体取香,但麝香的生物合成机制尚不明确,限制了麝香产业的发展。目前对麝香主要基于微生物宏基因组[4]、代谢组[1]和转录组[5]测序研究,麝香化学组分或前体组分的生物合成是麝香形成的关键步骤,分析与其合成有关的基因可为解析麝香的生物合成机制提供基础数据。根据麝香的化学组分构成分析,其含有大量的棕榈酸、软脂酸、长碳链环骨架、固醇物质和甾类等物质[1,4]。研究表明,脂肪酸合酶(Fatty acid synthase,FASN)对脂肪酸、长碳链骨架和甲羟戊酸的合成具有重要作用[6],因此林麝FASN基因mRNA的克隆及序列分析可为解析麝香的生物合成机制提供参考[7]。本研究通过转录组测序及Trinity拼接[8]获取林麝FASN基因的mRNA序列,并通过反转录PCR扩增及PCR产物Singer测序验证序列的准确性,对序列进行一系列的生物信息学分析,以麝鼠(Ondatrazibethicus)、牛(Bostaurus)、山羊(Caprahircus)和绵羊(Ovisaries)的FASN序列为参考找出与麝香合成有关的位点信息,为掌握林麝的FASN基因mRNA序列信息及解析麝香的生物机制提供基础数据。
1 材料与方法
1.1 材料
在5—6月泌香盛期,采取饲养于重庆市药物种植研究所林麝养殖基地的健康成年雄性林麝(5岁)香腺组织样本,置于RNA保存液中-80 ℃保存;送样至生工生物工程(上海)股份有限公司采用HiSeq 2500对mRNA转录组进行测序。
1.2 方法
1.2.1 RNA提取
取250 mg左右组织块利用液氮研磨后转至2 mL离心管中,加入样品1 mL,TRIzol冰上裂解5 min并摇匀,加入200 μL的氯仿,充分混匀后12 000 r/min 离心15 min,将上清转移至1.5 mL离心管中,加入等体积的异丙醇,至冰盒中摇床放置20 min,取出后10 000 r/min 离心10 min,去上清并晾至白色沉淀湿润为止,加入400 μL 75%乙醇洗涤,7 500 r/min 离心5 min后取出晾至白色沉淀湿润,加入50 μL的DEPC水溶解,置于水浴锅内85 ℃水浴5 min后放置冰箱中-80 ℃保存。
1.2.2 cDNA合成
取1 μg的RNA样品利用DNA酶处理2 min后,75 ℃处理5 min去酶活,加入多聚dT引物和随机引物各1 μL(10 μmol/L),65 ℃孵育5 min,加入dNTP、反转录酶、反应Buffer及灭菌水,42 ℃孵育1 h,75 ℃处理5 min去酶活,cDNA样品于冰箱中-20 ℃保存。
1.2.3 引物设计及PCR反应
利用Primer 5.0、GenBank数据库(www.ncbi. nlm.nih.gov)及转录组拼接序列,对林麝FASN基因mRNA序列进行PCR引物设计,以RNA逆转录cDNA为模板分段克隆基因CDS序列,详细引物信息见表1。反应体系20 μL,包括Mix 10 μL、上游和下游引物各1 μL(10 μmol/L)、cDNA模板1 μL、灭菌水7 μL,置于PCR仪反应。反应程序为95 ℃预变性5 min;95 ℃变性30 s,55~65 ℃退火30 s,35个循环;72 ℃终延伸10 min。用1%琼脂糖凝胶电泳检测反应产物片段大小,将PCR反应产物送重庆擎科生物科技有限公司进行Singer测序。
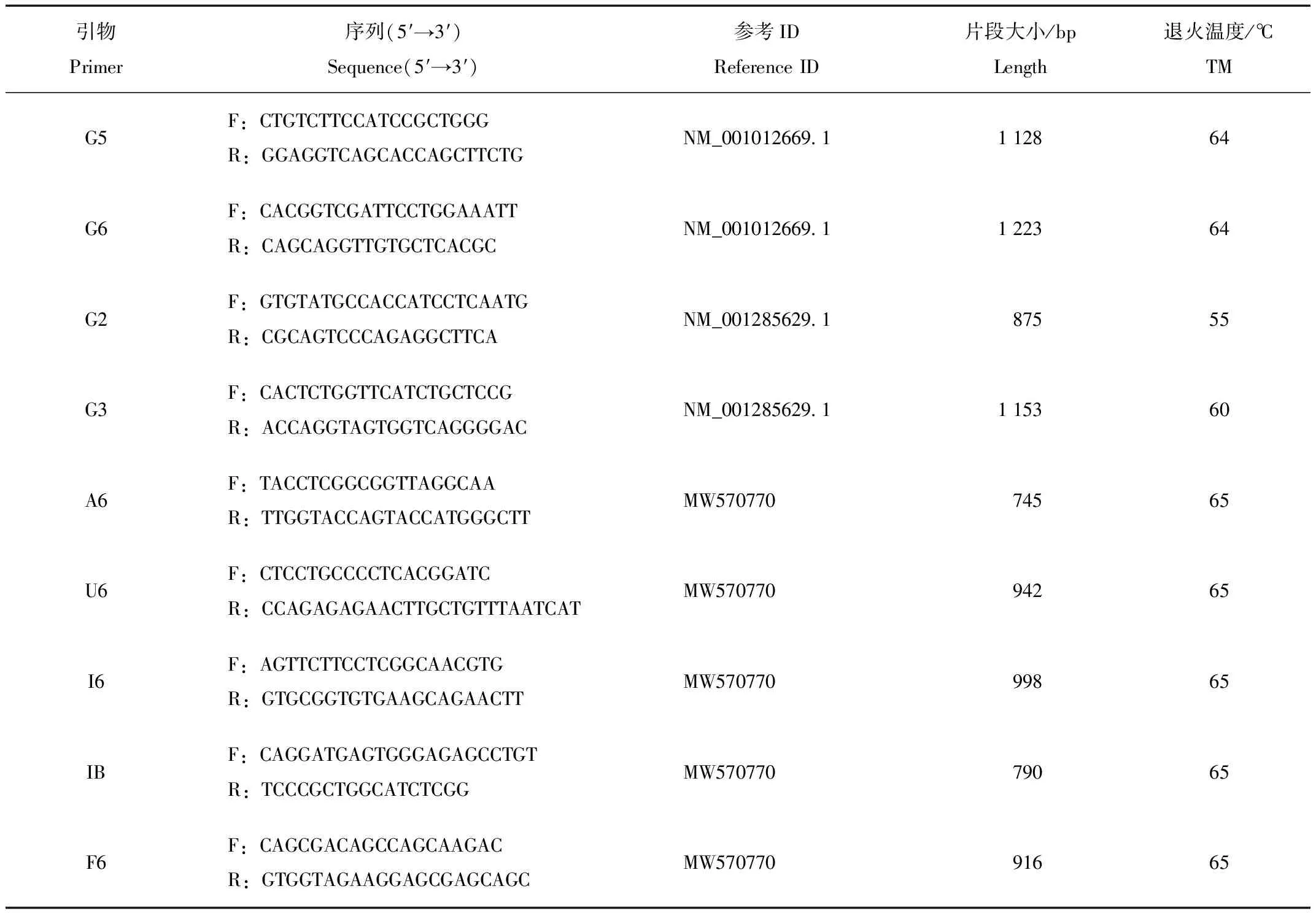
表1 FASN扩增引物信息
1.2.4 Singer测序和Trinity拼接
以转录组测序Trinity拼接结果FASNmRNA序列与牛(NM_001012669.1)、山羊(NM_001285629.1)和绵羊(XM_027974304.1)序列比对为参考,筛选林麝的FASN基因mRNA序列,用扩增片段与拼接结果进行匹配,验证Trinity拼接的FASNmRNA序列准确性,差异的碱基位点以Singer测序结果为准,进行下一步序列分析。
1.2.5 FASN进化树构建
对FASNmRNA序列用BioXM 2.6进行ORF分析及序列比对,以最长的ORF判定为CDS,通过脊椎动物标准氨基酸密码子翻译林麝FASN蛋白序列。从GenBank数据库下载并选择人(HomosapiensNP_004095.4)、阿肯色镖鲈(EtheostomacraginiXP_034717176.1)、小家鼠(MusmusculusNP_032014.3)、红色原鸡(GallusgallusNP_990486.2)、野猪(Susscro-faNP_001093400.1)、牛(NP_001012687.1)、山羊(NP_001272558.1)、绵羊(XP_027830105.1)、扬子鳄(AlligatorsinensisXP_025050442.1)、豹(PantherapardusXP_019321392.1)和旋毛虫(Trichinellasp.T9 KRX63946.1)序列,结合转录组测序拼接的麝鼠(Ondatrazibethicus)FASN蛋白序列,利用MEGA 6.0软件和最大似然法构建进化树,Bootstrap检验计算支持百分率。
1.2.6 蛋白序列分析
利用DNASTAR 7.1分析林麝FASN蛋白序列的二级结构、SwissModel(Swissmodel.expasy.org)分析三维结构、String(string-db.org)预测与FASN相互作用的蛋白、Scansite 4.0(scansite4.mit.edu/4.0/#home)预测林麝FASN可能的磷酸化位点。以能够生产麝香的麝鼠FASN蛋白序列为参考,同时用与林麝遗传距离较接近的牛、山羊和绵羊FASN蛋白序列比对,在林麝FASN蛋白序列中,选择与麝鼠相同与牛、山羊和绵羊不同的氨基酸位点作为麝香合成相关的关键,在mRNA序列中标出,最后通过SWChen(https://github.com/YLCHEN1992/SWChen)使用双阈值预测(全局110分+种子80分)调控林麝FASN表达的miRNA,使用SSNChen(https://github.com/YLCHEN1992/SSNChen)绘制林麝FASNmRNA 基因序列图。
2 结果与分析
2.1 林麝FASN基因mRNA序列获取及进化树构建
利用转录组测序拼接结果及牛、羊序列比对,成功获取了林麝FASN基因的mRNA序列,利用反转录PCR扩增和Singer测序验证。由于使用了2个不同的个体样本分别进行转录组测序拼接和Singer测序,序列中存在4个不一致的碱基位点,分别为G2片段上的T/C(AGT/AGC,CDS第1 425位,编码丝氨酸)变化;G3片段上的A/G(ACA/ACG,CDS第2 031位,编码苏氨酸)变化;A6片段上的T/C(ATT/ATC,CDS第738位,编码异亮氨酸)变化;I6片段上的C/T(GAC/GAT,CDS第738位,编码天冬氨酸)变化。变化的碱基位点并不改变编码氨基酸。利用GenBank数据库下载的各物种FASN蛋白序列构建遗传进化树(图1),结果显示林麝FASN序列与牛、山羊和绵羊遗传距离最为接近,其次为野猪、豹,与扬子鳄、红色原鸡、阿肯色镖鲈遗传距离较远,与麝鼠差异较大,麝鼠与小家鼠归为一支且接近人种,FASN蛋白序列在同源物种中具有较高的保守性。
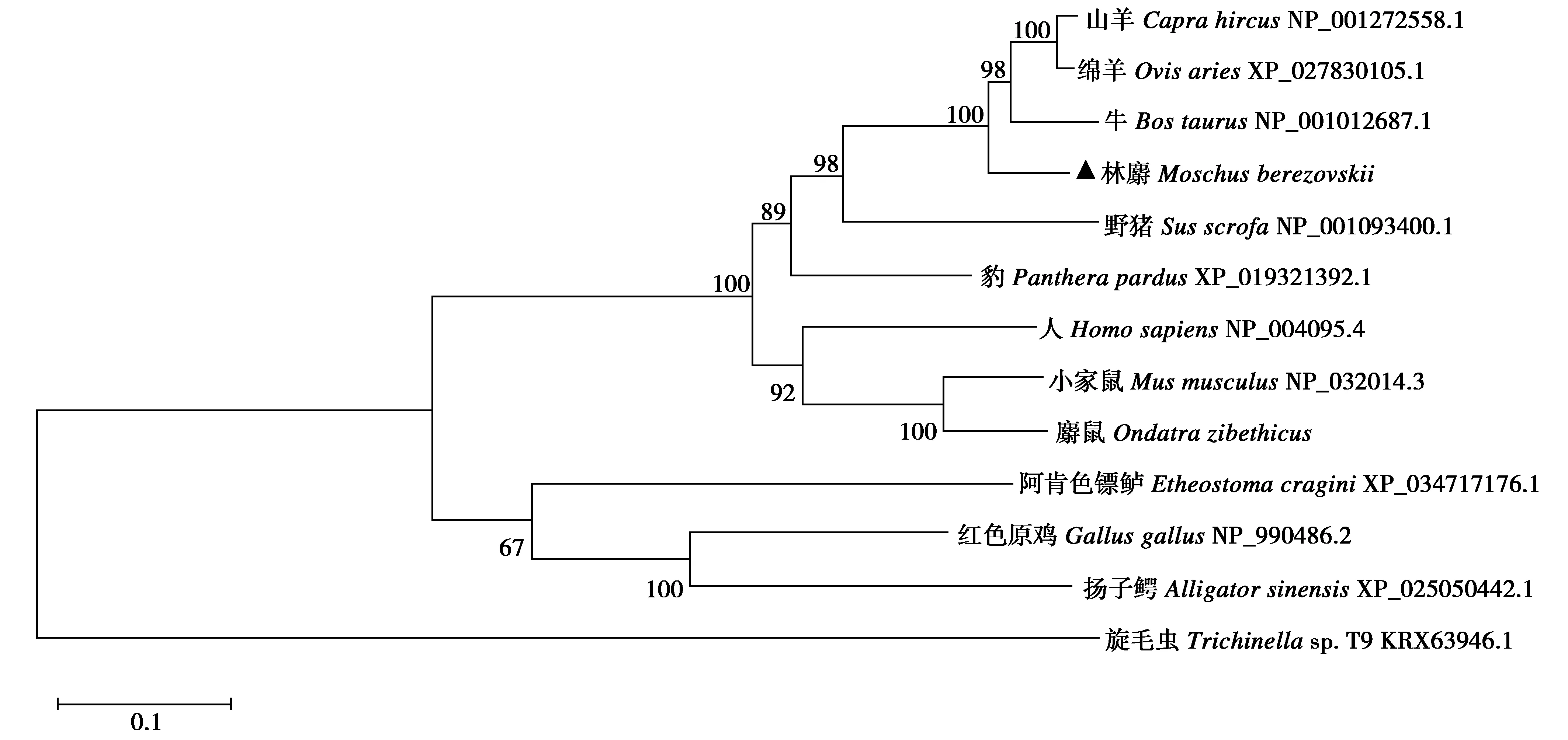
图1 基于FASN蛋白序列构建的系统发育树Fig.1 Phylogenetic tree construction based on FASN protein sequence 注:分支上的数据表示Bootstrap检验的支持百分率,以旋毛虫为外群;▲表示林麝FASN蛋白序列,其中麝鼠FASN序列通过转录组测序Trinity拼接序列翻译获取;标尺表示氨基酸替代率 Note:The data on the branch represented the percentage of support for the Bootstrap test.Trichinella sp.was the outgroup.The “▲” represented the FASN protein sequence of forest musk deer.The FASN protein sequence of muskrat was obtained by transcriptome sequencing,Trinity splicing and following protein translation.The ruler represented amino acid substitution rate
2.2 林麝FASN蛋白序列分析
通过序列比对发现,林麝FASN基因mRNA的CDS区包含9个结构域,其中6个区域为酶活性区,3个区域为结构区,CDS区域全长7 545 nt,5′-UTR和3′-UTR全长分别为99 nt和576 nt,详细结果如图2所示。对林麝FASN蛋白的基本性质、磷酸化位点、互作蛋白、二级和三级结构的预测结果显示,相对分子质量为274 284.44,等电点为5.70~5.80,预测发现11个候选磷酸化位点如图2所示。通过String网站预测与林麝FASN相互作用蛋白(图2),选取试验评分和高置信度的判定规则方法,发现细胞分化循环5样蛋白(Cell division cycle 5-like protein,CDC5L)、胆固醇羟化酶(Sterol 26-hydroxylase,CYP27A1)、热休克蛋白ATP酶激活剂同源1蛋白(Activator of 90 kDa heat shock protein ATPase homolog 1,AHSA1)和L-氨基乙二酸-半醛脱氢酶-磷酸泛酰巯基乙胺基转移酶(L-aminoadipate-semialdehyde dehydrogenase-phosphopantetheinyl transferase,AASDHPPT)能够与FASN相互作用;SWChen双阈值分析显示miR-1307-5p和miR-669为调控其表达的miRNAs。二级结构预测结果显示,林麝FASN蛋白含有多个α螺旋、β折叠、转角、无规卷曲结构及较多的可变域(图3)。三维预测结果显示林麝FASN呈现二聚体结构,存在多个位点的丝氨酸和苏氨酸位点(图4A),以及2个NADPH结合位点,三维结构呈现X型(图4B)。
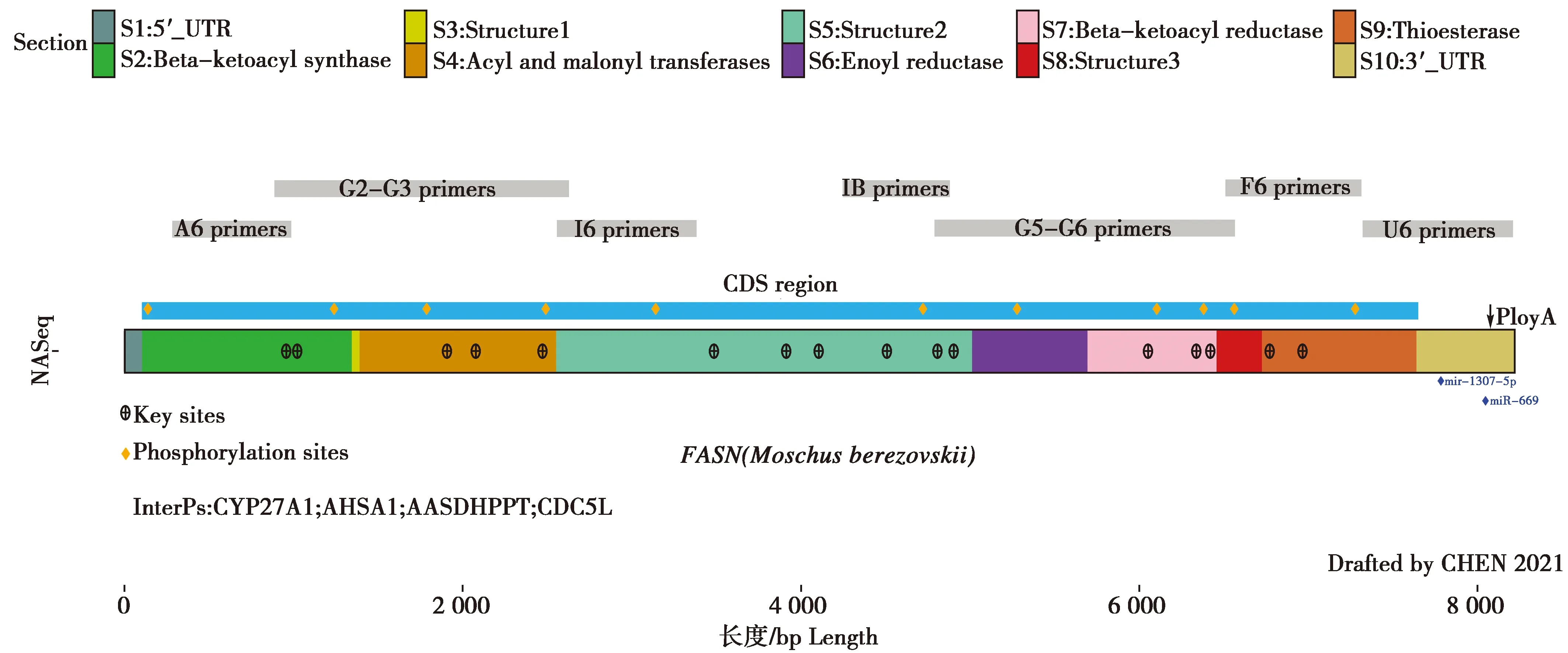
图2 林麝FASN基因mRNA序列信息Fig.2 The mRNA sequence information of FASN gene in Moschus berezovskii 注:下标尺度为序列长度;中间柱形图为FASN基因的mRNA序列,不同条带颜色显示不同的区域结构,关键的碱基位点在条带内部被标记出来,CDS区使用蓝色条带标出,其黄色点显示Scansite 4.0预测的蛋白磷酸化位点,灰色条带为对应引物的扩增区域;使用String预测的互作蛋白被标记在图左下方,互作的miRNAs位置及名称被标记在3′-UTR下方 Note:The subscript scale is sequence length.The middle column has shown the mRNA sequence of FASN gene that different stripe colors show different regional structures and key base sites are marked.The CDS region is marked with a blue band,with yellow dots showing the protein phosphorylation sites predicted by Scansite 4.0.Grey bands are the amplification regions of corresponding primers.The interacting proteins predicted by String software are labeled at the lower left,and the positions and names of the interacting miRNAs are labeled below 3′-UTR region
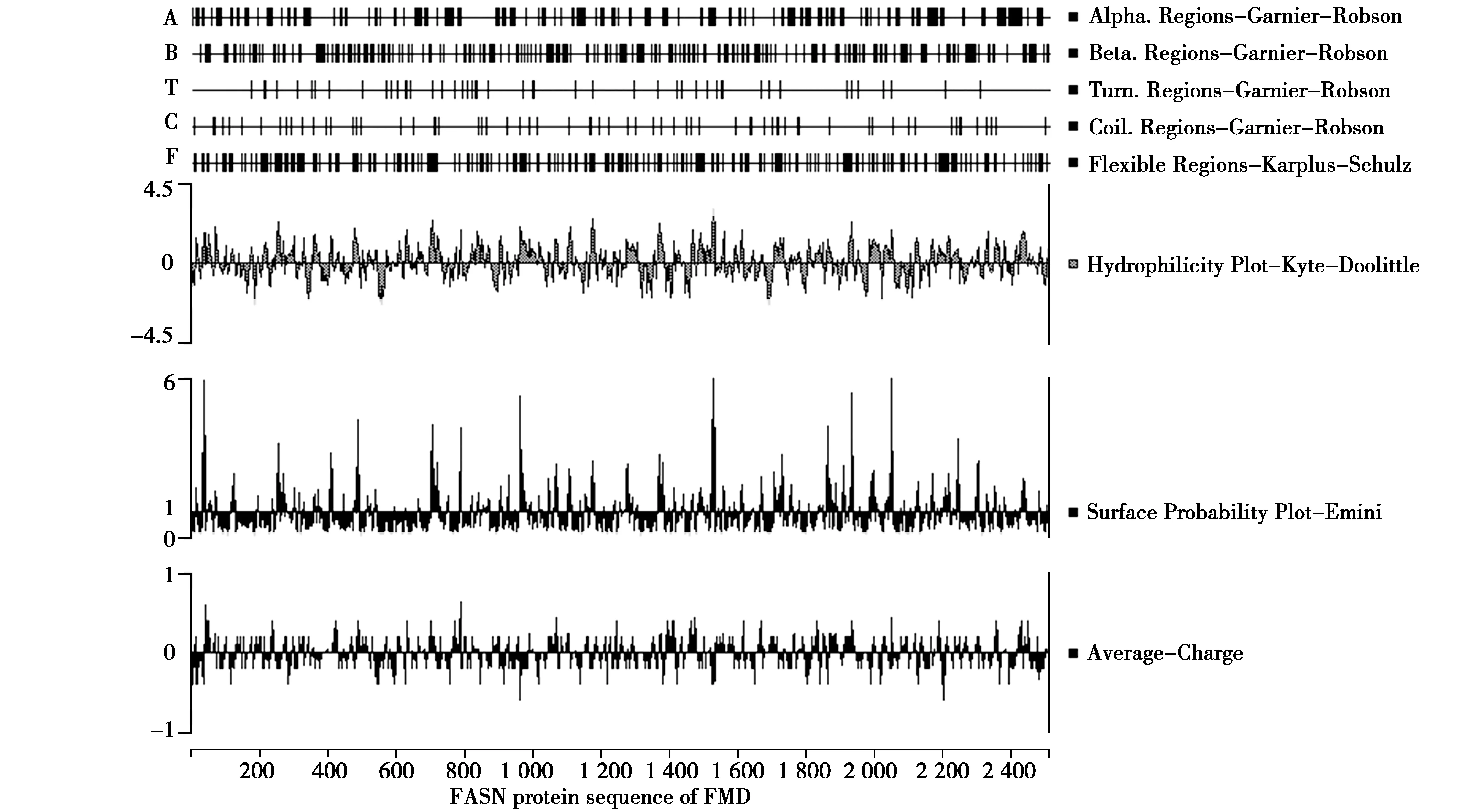
图3 林麝FASN蛋白二级结构Fig.3 Second structure analysis of FASN protein in Moschus berezovskii
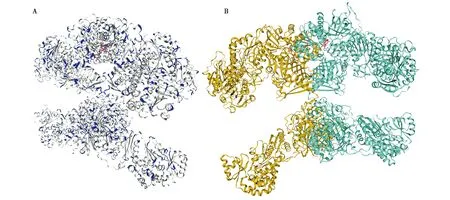
图4 林麝FASN蛋白三维结构模型Fig.4 3D model of FASN protein in Moschus berezovskii 注:A.丝氨酸/苏氨酸标识FASN二聚体,蓝色部分为丝氨酸或苏氨酸位点;B.FASN二聚体,绿色和黄色为各自的单体结构 Note:The A represents the dimer of FASN protein with Ser/Thr marking,and the blue part is the site of serine or threonine.The B represents the dimer of FASN protein where green and yellow are respective monomer structures respectively
2.3 林麝FASN关键位点筛选
通过物种中的氨基酸变化筛选关键氨基酸位点,其中有4个位点涉活性羟基变化,如FASN蛋白α螺旋上的第307位氨基酸在林麝和麝鼠中为丝氨酸,牛羊中为丝氨酸,与第265位(距离:8.90 Å)和第274位(距离:9.89 Å)的丝氨酸临近;α螺旋上的第602位氨基酸为丙氨酸,牛羊中为丝氨酸,临近第693位(距离:13.19 Å)苏氨酸;第657位氨基酸在牛羊中为丝氨酸,在麝鼠和林麝中为丙氨酸,临近第652位(距离:10.62 Å)丝氨酸;而α螺旋上的第1 334位氨基酸在牛羊中为甘氨酸,林麝和麝鼠为丝氨酸,且与第1 331位(距离:5.23 Å)苏氨酸位于同一测螺旋上;关键蛋白氨基酸位点信息见表2和图2。
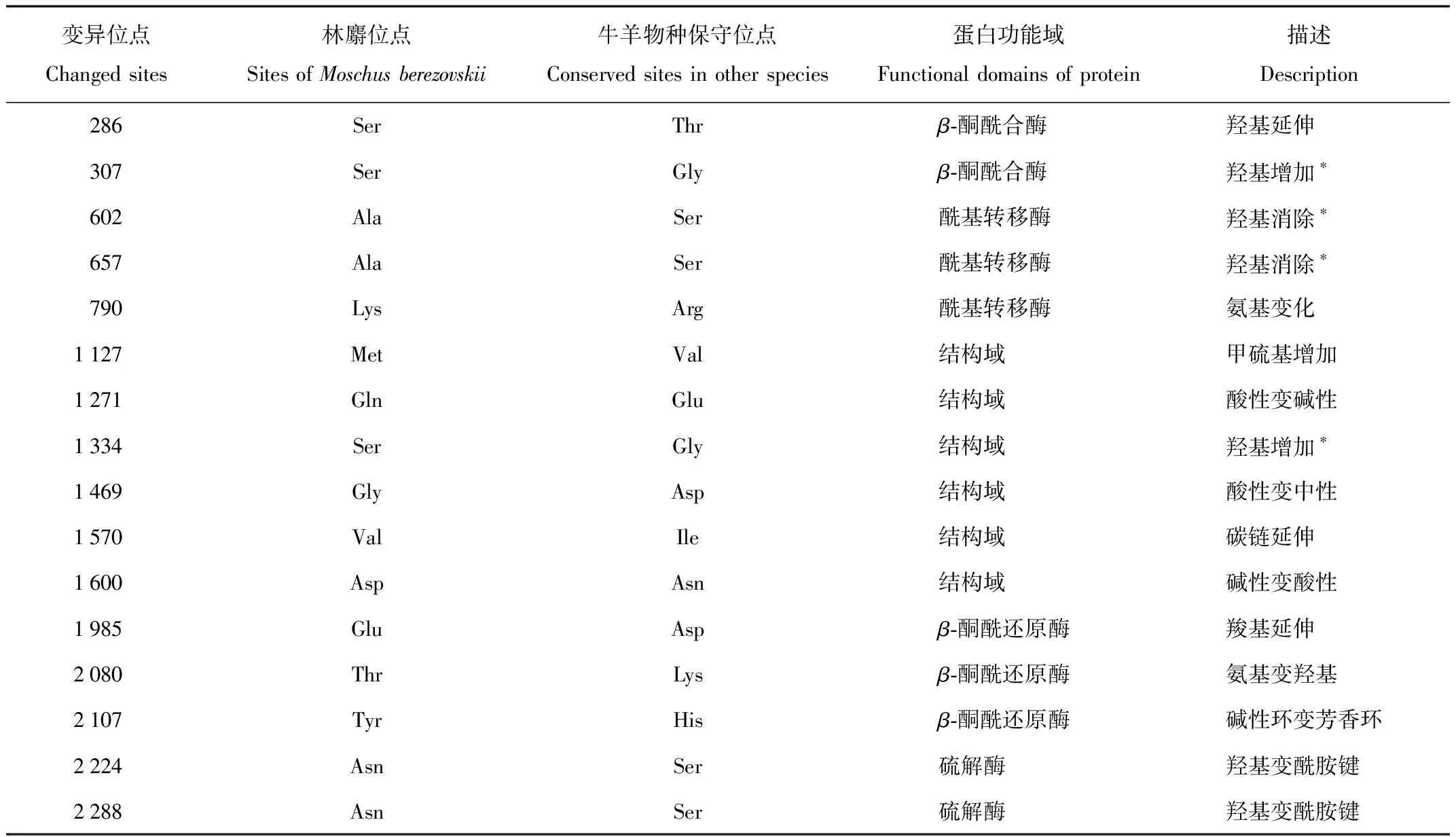
表2 FASN差异氨基酸位点描述
3 讨论
麝香具有较高的药用和香用价值,但目前麝香的生物合成机制尚不明确。根据现有的报道显示,麝香中的化学成分与碳链骨架、固醇和甾类物质具有较大的关联性,而FASN基因是其合成的关键酶之一。本研究通过转录组测序拼接获取林麝FASN基因的全长mRNA序列,利用反转录PCR及Sanger测序进一步验证序列的准确性,进行生物信息学分析,为解析麝香的生物合成机制提供基础数据。
由于基因转录后存在可变剪切现象,Trinity拼接的Chrysalis过程中会认定未加工完成的mRNA为unigene[8]。为保证获取序列的准确性,本研究利用牛、羊的mRNA序列作参考,选择长度接近、无内含子且CDS序列连续的片段为林麝的FASN基因mRNA序列,避开Trinity拼接的Chrysalis过程,得到的林麝FASN基因为加工的成熟mRNA序列;为保证序列的准确性后续使用反转录PCR扩增及Sanger测序进行验证,结果显示存在4个位点的差异,但并不改变编码的氨基酸序列,不一致的4个位点可能为潜在的SNP多态位点,需要进一步的试验验证。对于大片段的扩增具有难度且耗时,难以扩增出全长序列,林麝的FASN基因mRNA序列全长为8 221 nt。本研究通过片段拆分扩增,缺少5′-UTR区域、第100~280位和第3 378~4 244位碱基(CDS第1~60位和第1 093~1 382位氨基酸)的PCR扩增测序,后期可能需要获取高质量的RNA进行反转录扩增测序进一步验证,但根据其他片段的测序结果显示出的FASN基因mRNA序列可靠并可用于下一步的序列分析。
林麝FASN基因mRNA序列的获取及克隆验证对于以FASN角度研究麝香的生物合成机制具有重要参考价值。FASN参与脂类物质的合成[9],在进化过程中具有较高的保守性,因此与牛羊归为一支;林麝FASN的二级结构和三级结构显示,林麝脂肪酸合酶呈现二聚体结构,与牛羊等哺乳动物FASN结构相似,但变化的氨基酸位点可能影响蛋白之间的相互作用和蛋白的磷酸化,例如,与CDC5L、CYP27A1、AHSA1和AASDHPPT蛋白的相互作用,通过改变底物传递速度、酶构象或蛋白磷酸化能力,影响终产物的生成[10-13]。进一步对靶向调控林麝FASN基因表达的miRNA通过SWChen系统利用双阈值进行预测[14-17],结果显示miR-1307-5p和miR-669与FASN基因mRNA的3′-UTR具有较高的亲和能力,暗示3′-UTR可能通过海绵机制影响miRNA功能的执行[18],参与麝香合成过程。预测分析林麝FASN的二级结构、三级结构、磷酸化、蛋白相互作用和靶向miRNAs对了解FASN在麝香合成过程中的功能具有参考作用。
麝鼠也能合成和分泌能够替代麝香的麝鼠香[19],通过牛、山羊、绵羊、麝鼠和林麝的FASN蛋白序列比对,找出林麝与牛羊具有差异且与麝鼠相同的氨基酸位点,可能与麝香合成有关,同时丝氨酸和苏氨酸的R基团羟基对于酶的活性、蛋白磷酸化等具有关键作用[9,20]。本研究通过找到16个差异位点进行分析,变化的氨基酸位点不含磷酸化位点,暗示物种FASN磷酸化位点具有较高的保守性;而第307、602、657和1 334位氨基酸位点能够增添或消除羟基基团,其中第307位氨基酸位于FASN蛋白β-酮酰合酶的α螺旋上,在牛羊中为甘氨酸,在林麝和麝鼠中为丝氨酸,与第265位和第274位丝氨酸临近,临近的丝氨酸群有一定的概率组合成新的指环区域影响酶学活性,如BLM解旋酶的精氨酸指环结构[21]和RecQ的半胱氨酸指环[22-24];而第602和657位氨基酸由丝氨酸变为中性丙氨酸,羟基基团的消除可能影响该区域酰基转移酶的活性,β-酮酰合酶和酰基转移酶能够对脂肪酸的碳链加成和延伸[6,9],第307、602和657位点的变化有可能对脂肪酸碳链的活化和麝香酮的成环具有重要作用;FASN约1/3的区域为结构区域,对蛋白的正确折叠功能发挥等具有重要作用[9],第1 334位丝氨酸与同一螺旋结构上第1 331位的丝氨酸存在相互影响,而该位点属于结构区域,氨基酸变化可能影响其他蛋白域的活性,因此4个具有羟基变化的氨基酸位点提示与麝香的生物合成具有较强关联性,但还需要进行进一步的试验验证,如模型细胞的定点突变验证等。通过以牛、山羊、绵羊、麝鼠和林麝的FASN蛋白序列比对,筛选出与麝鼠相同且与牛羊不同的位点可为麝香的生物合成机制研究提供参考数据。
4 结论
林麝FASN对于麝香的化学组分的合成密切相关,通过转录组测序拼接及Singer测序验证获取了林麝FASN基因的mRNA序列,进行进化树构建、二级结构、三级结构、蛋白相互作用、蛋白磷酸位点化及靶向miRNAs分析,结果显示,林麝FASN具有较高的物种保守性,16个关键位点与麝鼠相同且与牛羊不同,其中第307、602、657和1 334位氨基酸位点能够影响活性羟基的变化与麝香的合成密切相关;FASN的3′-UTR与miR-1307-5p和miR-669具有较高的亲和能力,林麝FASN基因序列与其他物种之间的差异信息可为解析麝香生物合成机制提供参考。
