基于复合物结构和网络拓扑特性的关键蛋白质识别算法
2022-05-08卢鹏丽蔚京娟
卢鹏丽, 蔚京娟
(兰州理工大学 计算机与通信学院, 甘肃 兰州 730050)
蛋白质是构成一切细胞和组织结构必不可少的成分,而关键蛋白质是生物体生存和繁殖所必须的,在生命活动中扮演着重要的角色[1],通过基因剔除式突变将关键蛋白质移除会导致细胞死亡或无法繁殖[2].关键蛋白质的识别有助于从分子水平上理解细胞的基本要求[3],因此识别关键蛋白质对生物体的生命活动很重要.在生物学上,关键蛋白质的识别主要依靠生物实验方法,如条件敲除[4]、RNA干扰[5]和单基因敲除[6].通过生物学实验预测关键蛋白质虽然成果有效且准确性高,但需要消耗大量时间、成本和资源.
随着蛋白质相互作用PPI(protein-protein interaction)网络的日益完善,基于PPI网络,采用计算机技术从大量的实验中预测关键蛋白质成为生物信息学领域的重要研究方向[7-8].Jeong等[9]提出的“集中性-致命性”规则证明了蛋白质的关键性与PPI网络拓扑结构有关.因此,出现了大量基于网络拓扑结构的中心性指标[10].利用图的拓扑指数构造的节点中心性反映复杂网络的整体特性,可以很好地识别复杂网络的节点重要性[11].从节点的角度出发,度中心性(degree centrality)[12]将节点的邻居个数作为该节点的中心性度量,仅仅考虑了节点的局部信息;在酵母蛋白质网络中,存在很多具有较高介数但较低度的节点在网络中起着关键的作用,基于这一发现,提出了介数中心性BC(betweenness centrality)[13];节点的紧密中心性CC(closeness centrality)[14]反映了节点在网络中居于中心的测度,节点越居于网络的中心,其重要程度越高;特征向量中心性EC(eigenvector centrality)[15]和子图中心性SC(subgraph centrality)[16]分别从网络全局和局部考虑节点的重要程度;信息中心性IC(information centrality)[17]用于通过分析每一对节点间的信息来考虑节点的重要程度;局部平均连通性LAC(local average connectivity)[18]考虑了节点在诱导子图中的特性.以上这些中心性方法从节点拓扑特性考虑忽略了网络中边的重要程度.
从边拓扑特性的角度出发,边聚类系数ECC(edge clustering coefficient)[19]考虑了两个节点与公共邻居节点间构成的三角形个数来寻找关键蛋白质;集成边缘权重IEC(integrated edge weights)[20]和网络中心度NC(network centrality)[21]通过结合边聚类系数以及节点自身的性质来衡量蛋白质节点的重要程度.基于拓扑结构的方法虽然能够提高关键蛋白质的识别效率,但却忽略了蛋白质信息数据对节点关键程度的影响.
高通量生物信息数据的增加极大地丰富了传统的PPI网络,这些生物信息主要包括基因表达、语义相似性、亚细胞定位以及复合物信息等[22],利用生物信息能够提高鉴定关键蛋白质的准确性.PeC方法利用基因表达数据构建了加权网络,并结合边缘聚类系数来提高识别关键蛋白质的准确率[23];esPOS方法主要采用了基因表达数据和亚细胞定位来提高识别精度[24].
蛋白质复合物是指在相同时间和空间通过相互作用组成一个多分子机制的一组蛋白质.目前已有大量研究表明,蛋白质复合物和关键蛋白质之间存在密切的联系.李敏等[24]证明了复合物中关键蛋白质的出现频率高于整个网络中的频率;Hart等[25]发现关键性是蛋白质复合物的属性,蛋白质复合物能够更好地确定关键蛋白质;UC(united complex centrality)方法[26]考虑了蛋白质出现在复合物的频率和边缘特性来提高预测能力;CED方法[27]结合了皮尔逊相关系数、边聚类系数以及复合物信息来提高关键蛋白质的识别能力;PSCD方法[28]利用皮尔逊相关系数和边聚类系数构建了加权网络,并运用复合物信息提高了识别关键蛋白质的能力.因此,结合复合物信息是提高关键蛋白质识别率的一种有效手段.
为了提高关键蛋白质的识别能力,本文一方面考虑了复合物信息在关键蛋白质中发挥的重要作用,另一方面结合拓扑结构的全局特性用于考虑不存在于复合物信息中的蛋白质节点,提出了扩展信息中心性EIC(extend information centrality)来预测关键蛋白质.为评估该算法预测关键蛋白质的准确性,选用酵母蛋白质的相关数据集MIPS和DIP中进行实验,并与已有算法BC、DC、EC、SC、LAC、NC以及UC进行对比,结果表明EIC方法在F-度量、准确率、特异性、敏感度等方面具有良好的性能.
1 相关算法定义及EIC算法
蛋白质相互作用网络可以表示为无向简单图G(V,E),V={v1,v2,…,vn}表示蛋白质节点的集合,E{(vi,vj)|vi,vj∈V}表示蛋白质节点之间相互作用的集合.A是蛋白质相互作用网络对应的邻接矩阵,如果节点vi和vj之间存在连边,则其元素Aij=1,否则Aij=0.
1.1 相关研究
以下是已有的基于网络拓扑结构的DC、BC、EC、SC、LAC、NC算法和基于复合物信息的UC算法介绍,在后续实验中要与这些算法进行对比.
1) 度中心性DC
节点的度中心性定义为与该节点相邻的节点数目,公式如下:
(1)
其中:N是网络中节点的总数.度中心性是最简单、最直观的测度,节点的度越大,则节点对于整个网络产生的影响也越大.
2) 介数中心性BC
节点的介数中心性表示蛋白质网络中经过某个节点v的最短路径数目在所有最短路径数目中所占比例,公式如下:
(2)
其中:σst是节点s和节点t之间的最短路径数目;σst(v)是通过节点v的最短路径数目.节点的介数中心性主要功能是识别该节点在网络拓扑结构中是否处于枢纽位置.
3) 特征向量中心性EC
节点v的特征向量中心性指网络邻接矩阵的主特征向量的第v个分量,公式如下:
EC(v)=αmax(v)
(3)
其中:αmax为对应于网络邻接矩阵的最大特征值λmax的特征向量;αmax(v)为αmax的第v个分量.
4) 子图中心性SC
节点的子图中心性表示节点参与网络闭回路的总数,公式如下:
(4)
其中:μk(v)代表以节点v为起点和终点且长度为k的闭回路数目.
5) 局部平均连通性 LAC
局部平均连通性用来评估邻居节点形成的诱导子图中节点的紧密程度,公式如下:
(5)
其中:Nv表示节点v的邻居节点集合;Cv表示节点v的邻居节点构成的诱导子图;对于子图Cv中的节点u,用dCv(u)表示节点u在子图中的度.
6) 网络中心度NC
节点的网络中心度表示了节点周围边的聚类特性,公式如下:
(6)
其中:Nv为节点v的邻居节点的集合;Zu,v为由节点u和v构成的三角形的个数;dv和du分别为节点v和u的度.
7) 联合复合物中心性UC
节点的联合复合物中心性表示节点的邻居在复合物中的出现频度以及边的聚类特性,公式如下:
(7)
其中:fu为节点u在复合物中出现的次数;fM为蛋白质节点在复合物中出现的最大次数;Zu,v为由节点u和v构成的三角形的个数;dv和du分别为节点v和u的度.
1.2 EIC算法
本文提出了一种识别关键蛋白质的方法——扩展信息中心性EIC.其基本思想如下:首先,在复合物内定义一种新的中心性指标用于寻找复合物中的关键节点;其次,整合复合物中节点的关键性;最后,结合识别网络整体拓扑结构的信息中心性,提出识别关键蛋白质的算法.
信息中心性 IC定义为
(8)
其中:Iv,u=(cv,v+cu,u-2cv,u);而C=(cv,u)=(D-A+J)-1;D为度对角矩阵;J为全一矩阵.
首先,基于节点与邻居节点的聚类程度以及节点本身的特性,提出了LC(local centrality)方法,用于复合物结构中判定复合物中节点的重要程度:
(9)
其中:Ev为节点v的邻居节点之间实际存在的边数;dv为节点v的度.
考虑到节点在复合物中出现的频度不同,结合复合物信息定义了ILC(in-local centrality):

(10)
其中:ComplexSet(v)表示包含蛋白质v的复合物子集;LC(v)i表示蛋白质节点v在第i个复合物中对应LC的值.因此在复合物信息中,节点的重要性不仅与其在每个复合物中的中心性有关,还与节点出现在各个复合物中的频率有关.
然而,在蛋白质网络中,有的节点不存在于复合物中,单单考虑节点在复合物中的特性不足以区分其他节点的重要程度.因此不仅要考虑复合物中蛋白质的重要程度,也要考虑网络的全局特性.结合节点在复合物中的特性以及网络全局特性,提出了EIC来提高识别关键蛋白质的准确率:
(11)
其中:a为[0,1]的随机数;ILCmax代表在所有节点中ILC的最大值;ICmax代表所有节点中IC的最大值.运用EIC算法来识别关键蛋白质的实验流程如图1所示.
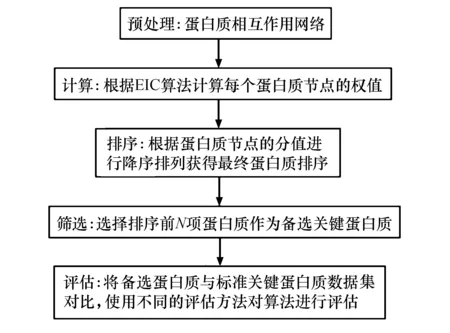
图1 实验流程图Fig.1 Experimental flowchart
2 实验数据与评估方法
2.1 数据集
为评估EIC算法的性能,选用相对可靠完整的酵母蛋白质相互作用网络数据作为实验数据,具体使用两种子集MIPS[29]和DIP[30].通过去除重边和自相互作用,得到网络的属性如表1所列.标准的关键蛋白质数据由四个数据集得到,分别是MIPS(munich information center for protein sequences)[31]、
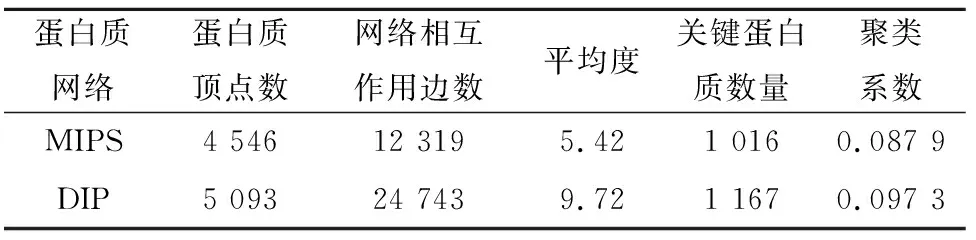
表1 蛋白质网络MIPS和DIP的属性
SGD(sacharomyces genome database)[32]、DEG (diethylene glycol)[29]和SGDP(simons genome diversity project)[4].蛋白质复合物信息集来源于CM270[31]、CM425[33]、CYC408[34]以及CYC428[35]数据集,包含745个蛋白质复合物子集(即2 167个蛋白质).
2.2 评估方法
为了评估算法的有效性和准确性,首先对算法赋予的节点权值进行降序排列,得出对应蛋白质节点的排序结果.选用不同规模的蛋白质节点作为备选的关键蛋白质,通过对比标准关键蛋白质数据,可以得到备选关键蛋白质被真正识别为关键蛋白质的数量.由于识别关键蛋白质本质上是一个二分类问题,可以通过六种评估方法和精准回归方法对二分类问题进行评估.六种评估方法包括敏感度SN(sensitivity)、特异性SP(specificity)、阳性预测值PPV(positive predictive value)、阴性预测值NPV(negative predictive value)、F-度量(F-measure)和准确率ACC(accuracy).敏感度(SN)衡量了分类器识别正确关键蛋白质的能力,该值越大,分类器越好.特异性(SP)衡量了分类器识别正确非关键蛋白质的能力.阳性预测(PPV)和阴性预测(NPV)同样可以反映识别正确关键蛋白质和非关键蛋白质的能力,值越大越好.F-measure代表精度和灵敏度的谐波均值.精度(ACC)越高,也表示分类器越好.这六个评估方法的定义如下[36]:

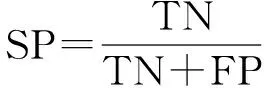
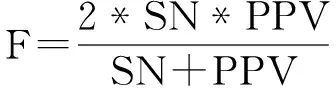

其中:TP表示备选关键蛋白质被正确地预测为关键蛋白质的数量;FN表示为备选关键蛋白质被错误地预测为非关键蛋白质的数量;FP表示为备选的非关键蛋白质被错误地预测为关键蛋白质的数量;TN表示为备选的非关键蛋白质被正确预测为非关键蛋白质的数量.
查准率-查全率曲线(P-R曲线)也是一种评估算法性能的统计方法,根据精准度(precision)和回归值(recall),可以拟合P-R(精准回归)曲线,通过曲线反映识别关键蛋白质的算法效果,曲线越高表示效果越好.其x轴代表Recall,y轴代表Precision,具体定义如下:
在给定网络中对关键蛋白质的预测可以看作一种二分类问题,且是非平衡分类,性能评估指标ROC曲线(receiver operating characteristic)是一种衡量分类不平衡的有力工具,常用于评估二分类器的优劣.其x轴代表FPR,y轴代表TPR,具体定义如下[37]:
3 实验结果分析
3.1 算法参数设定与选择
为设定参数a的值,选取DIP和MIPS两种网络进行实验.根据“排序-筛选”方法,首先对各个方法得出的节点权值进行降序排列,在序列中分别选取top 100、top 200、top 300、top 400、top 500和top 600节点作为备选的关键蛋白质;对比标准关键蛋白质数据,得出的这些备选关键蛋白质中真正的关键蛋白质数量.通过比较能够识别的真正关键蛋白质数量,来寻找a的最优解.首先在区间[0,1]选择取值的跳步.实验中,初步设定跳步为0.01,而实验数据表示了a在间距为0.01内识别的关键蛋白质数量几乎不变;加大跳步将其设定为0.05,由于数据量较大,抽取了其中部分结果作为比较,以便于找到a的最优解.实验结果见表2.
由表2数据可得,当a为0.8时可以得到较好的实验结果.从系数设定可以看出,当节点存在于复合物中时,起主导作用的是节点在复合物中的特性.
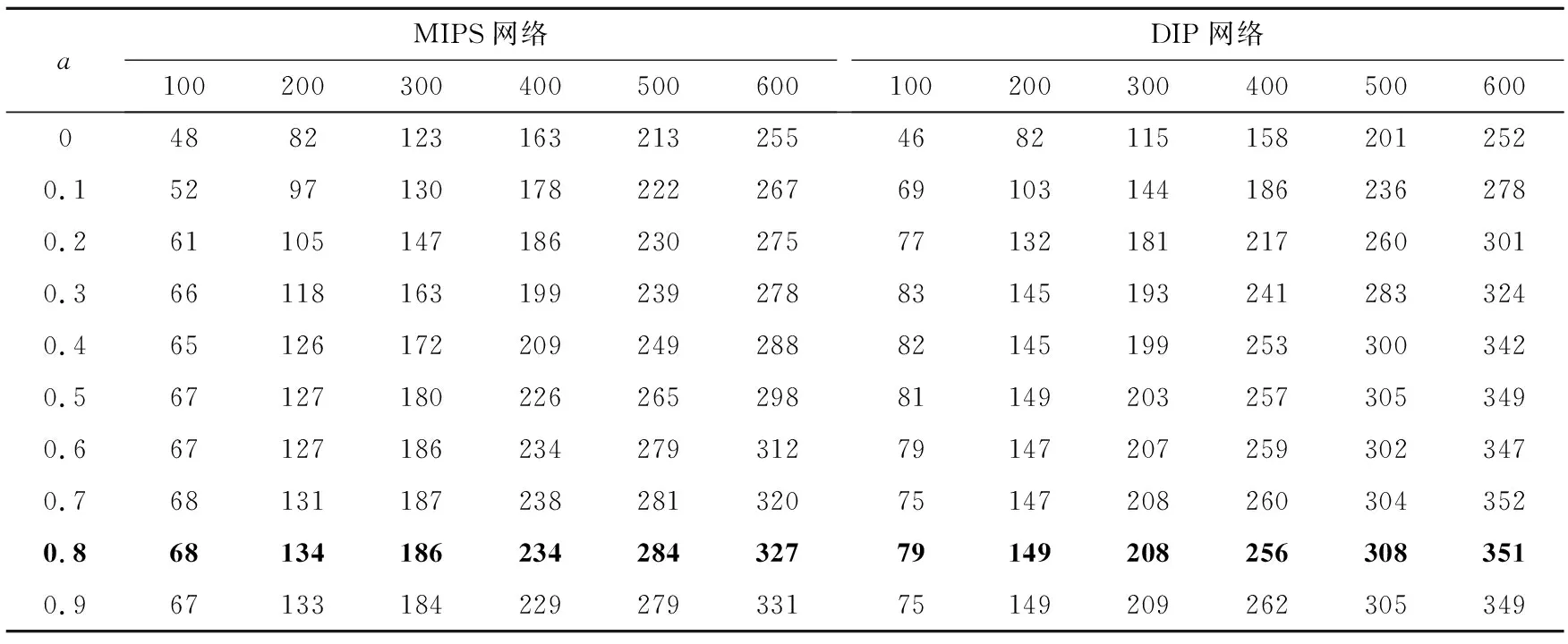
表2 在MIPS和DIP网络中参数a的选取
3.2 与其他算法的比较
为评估算法的有效性,使用在实验数据部分描述的MIPS和DIP作为数据集,将EIC方法与现有方法DC、BC、EC、SC、LAC、NC和UC进行对比.根据“排序-筛选”方法,实验方法与3.1节的实验方法相同.实验结果如图2和图3所示.

图2 MIPS网络中使用EIC方法以及其他七种方法识别关键蛋白质的数量
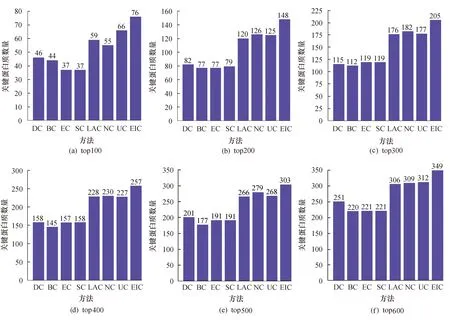
图3 DIP网络中使用EIC方法以及其他七种方法识别关键蛋白质的数量Fig.3 The quantity of essential proteins determined by EIC and other seven methods in DIP networks
通过对比现有方法DC、BC、EC、SC、LAC、NC和UC,可以看出EIC方法能够识别更多的关键蛋白质.在MIPS网络中EIC方法在排序top100~top600中分别能识别68、132、186、233、281以及322个关键蛋白质,均高于其他方法识别的关键蛋白质数量.在这八种方法中,识别能力较差的是SC和EC方法.与识别能力较好的UC比较,本文的方法EIC在排序top100~top600中识别关键蛋白质的能力分别提高了1.19倍、1.59倍、1.29倍、1.26倍、1.07倍和0.87倍.同样,在DIP网络中,EIC方法识别的关键蛋白质数量最多.因此EIC方法可以更好地识别关键蛋白质.
3.3 使用六种统计方法以及精准召回曲线评估
为了综合评估EIC方法的整体性能,利用了在2.2节介绍的六个统计分析参数,即:敏感度SN、特异性SP、阳性预测值PPV、阴性预测值NPV、F-度量和准确率ACC.首先,将不同算法得到的蛋白质分值降序排列,选择序列中前20%的蛋白质作为备选的关键蛋白质,其余的80%作为备选的非关键蛋白质.在两个网络MIPS和DIP中,将EIC方法与已有算法DC、BC、EC、SC、LAC、NC和UC进行比较,实验结果见表3.在两个网络中,算法EIC的六个评估指标值均高于其余算法,表明EIC方法可以更好地识别关键蛋白质.
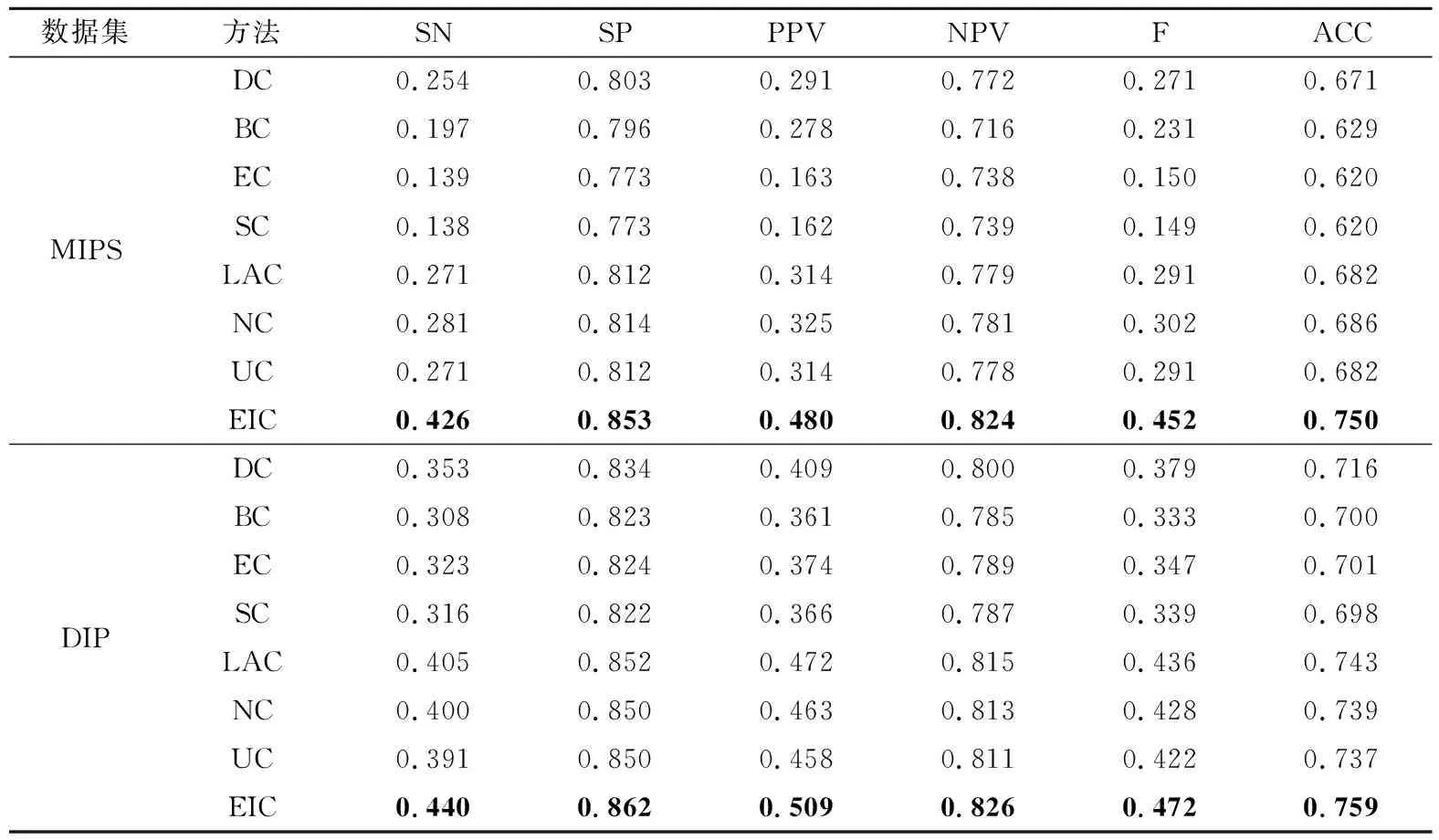
表3 EIC和其他方法在六种统计指标下的性能比较
此外,利用查准率-查全率曲线(P-R曲线)可以评估算法性能的统计方法,实验结果如图4所示.在MIPS和DIP网络中,EIC方法均高于其他算法,因此能够提高识别关键蛋白质的准确率.

图4 两种网络中使用EIC算法和其他七种算法的精准召回P-R曲线
3.4 基于ROC曲线评估
由于蛋白质相互作用网络中非关键蛋白质的数目比关键蛋白质数目高出三到四倍,因此识别关键蛋白质是一种不平衡的二分类问题,通过性能评估指标ROC曲线来评估算法的优劣是一种较好的方法.
选定每种方法预测降序排序结果的前20%节点作为备选的关键蛋白质,网络中余下的80%节点作为备选的非关键蛋白质.在MIPS和DIP数据集中比较,由图5所示,EIC算法比其他七种方法的曲线更高,能够更加有效地识别关键蛋白质.

图5 两种网络中使用EIC算法和其他七种算法的ROC曲线
3.5 基于折刀方法评估
折刀算法是由Holman等[33]提出的一种有效通用的评估方法.其x轴表示备选的蛋白质数量,y轴表示备选蛋白质中标准关键蛋白质数量.根据不同的算法得出的权值对蛋白质节点排序,选取排序后的top0~top600节点作为备选关键蛋白质节点.通过对比标准关键蛋白质数据,得出在top0~top600能够识别的准确关键蛋白质数量,绘制的折刀曲线如图6所示.图6(a1~a7)和图6(b1~b7)分别是在MIPS和DIP网络中使用EIC算法以及其他七种算法的折刀曲线.

图6 MIPS和DIP网络中使用EIC算法以及其他七种算法的折刀曲线
在MIPS和DIP网络中,EIC方法识别的关键蛋白质普遍高于其他七种方法,随着备选关键蛋白质数量的增多,本文提出的算法能够正确识别的关键蛋白质数目与其他方法的正确识别数目间的差值越大.因此EIC算法能够提高识别关键蛋白质的准确率.
4 结论
识别关键蛋白质在生物医学中具有重要的意义,当前提出并应用了许多预测关键蛋白质的方法,然而提高关键蛋白质的识别精度是一项艰巨的任务.本文在复合物信息和网络全局拓扑特性的基础上,提出了一种新的方法扩展信息中心性EIC.选用相对可靠完整的酵母蛋白质中MIPS和DIP数据集,运用“排序-筛选”法、敏感度SN、特异性SP、阳性预测值PPV、阴性预测值NPV、F-度量和准确率ACC的统计方法、精准召回曲线、ROC曲线和折刀方法,通过对比DC、BC、EC、SC、LAC、NC和UC等七种已有方法,表明EIC的方法优于这七种预测关键蛋白质的方法.
在后续研究中,可以结合其他生物信息例如基因表达、语义相似性,还可结合机器学习和深度学习中的算法来提高关键蛋白质的识别精度.
