传统自然发酵酱油细菌群落结构特征分析
2022-05-06邓岳梁丽静迟原龙孙群
邓岳,梁丽静,迟原龙,孙群
(1.泸州职业技术学院,四川 泸州 646000;2.四川大学 轻工科学与工程学院,成都 610064;3.四川大学 生命科学学院 生物资源与生态环境教育部重点实验室,成都 610064)
酱油作为中国使用大豆为主要原料的传统的发酵制品之一,因其独特营养价值与风味品质而备受全球人民尤其是亚洲人民的喜爱,中国作为最早酿造酱油的国家之一,其酿造历史可以追溯到周朝时期[1]。传统酱油酿造过程中主要包括以曲霉发酵代谢活动为中心的制曲阶段和以耐盐性乳酸菌和耐盐性酵母菌为中心的固液发酵阶段,并且酱油中的重要特征风味物质,如乙醇、甘油和醇类物质以及芳香族化合物都在此阶段生成,从而赋予酱油特殊的风味特征。在酱油固液发酵过程中,以耐盐性乳酸菌和耐盐性酵母菌为主的微生物对酱油色、香、味和体态的形成和品质起重要作用[2]。
目前,国内酱油酿造行业中工业生产酱油主要采用沪酿3.042作为发酵的工业菌种[3],该菌种具有产酶能力强、生长快、抵抗污染能力较强等优点。但添加单一菌种制曲生产的酱油风味和口感远不如传统自然发酵酱油。传统自然发酵酱油采用的是长周期开放式的酿造工艺,酱醅发酵过程涉及到多种有益微生物的联合协同作用,通过微生物分解原料中的碳水化合物、蛋白质、脂肪等大分子化合物产生醇类、醛类、酸类、酯类、酚类等小分子风味物质,是酱油独特产品风味形成的主要途径[4]。因此,研究传统发酵酱油中微生物多样性对揭示酱油品质形成显得尤为必要。
四川省泸州市先市镇的先市酱油,采用手工作坊式的天然多菌种混合发酵工艺,在自然环境中日晒夜露3~5年,所产酱油具有独特风味和显著的区域特色[5]。该酿造技艺于2014年被列入中国“非物质文化遗产”名录进行重点保护,其生产场所也被列为“四川重点文物保护单位”名录。本文拟对先市酱油酿造技艺过程中的细菌群落结构进行分析,为揭示传统自然发酵过程中的细菌菌群结构和动态变化规律,传统自然发酵酿造技艺调整,解释传统自然发酵酱油风味等品质形成和潜力菌株筛选提供了理论基础。
1 材料与设备
1.1 样品采集
样品:由四川省泸州市合江县先市酿造食品有限公司提供,分别采自整个发酵过程中的第0.5,1,2,3,4年,具体取样点和样品命名见图1。每个年份由3个随机分布的酱缸中间位置取样,样品选择遵循随机原则,不同酱缸样品经充分混合后,于4 ℃保存备用。
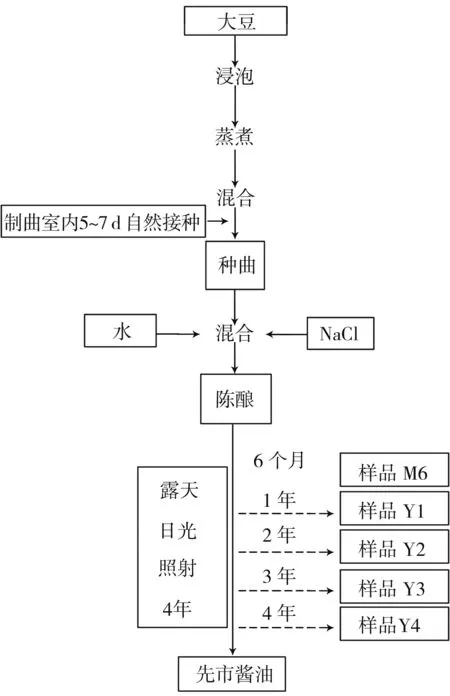
图1 先市酱油生产工艺流程和试验样品取样点Fig.1 The production process of Xianshi soy sauce and sampling points of test samples
1.2 主要试剂
FastPfu聚合酶:TransGen Biotech Co., Ltd.;2%琼脂糖凝胶:Biowest公司;DNA抽提试剂盒(Omega-soil DNA kit):Omega公司;Illumina MiSeq平台:美国Illumina公司;其余试剂:均为分析纯。
1.3 主要仪器设备
NanoDrop 2000超微量分光光度计 Thermo Fisher Scientific公司;ABI GeneAmp®9700型PCR仪;ABSON MiFly-6小型离心机;Eppendorf 5424R高速台式冷冻离心机;DYY-6C电泳仪;Illumina MiSeq测序仪;BioTek ELx800酶标仪 美国BioTek公司;UV-2450紫外分光光度计 日本岛津公司。
1.4 DNA 抽提和 PCR 扩增
采用 E.Z.N.A.®soil DNA kit (Omega BioTek, Norcross, GA, U.S.)提供的标准DNA提取方法,对传统工艺发酵酱油中微生物群落总DNA进行抽提,并使用浓度为1%的琼脂糖凝胶电泳对提取的DNA进行质量检测,并使用NanoDrop 2000对提取的测定DNA浓度和纯度进行分析检测。
1.5 PCR扩增
以传统工艺发酵酱油中提取的基因组DNA作为模板,对该基因组的16S rDNA序列V3-V4区域进行扩增,使用的引物如下:
338F:5′-ACTCCTACGGGAGGCAGCAG-3′;
806R:5′-GGACTACHVGGGTWTCTAAT-3′。
DNA扩增程序:95 ℃预变性 3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),72 ℃稳定延伸10 min,并置于4 ℃保存,每个样本3个重复。
1.6 Illumina MiSeq测序
将传统发酵酱油中同一样本的PCR产物混合后,进行PCR产物回收与纯化、检测定量后,使用Illumina公司的MiSeq PE300/NovaSeq PE250平台进行测序。
1.7 数据处理
使用fastp[6]、FLASH[7]、UPARSE[8]等分析软件对测序数据进行统计分析,并基于分类学信息,讨论传统自然发酵酱油细菌群落结构。
2 结果分析
2.1 PCR扩增结果
以传统自然发酵酱油中提取的基因组DNA作为模板,用通用引物对其16S rDNA V3-V4区进行扩增,扩增后的目的片段见图2。

图2 PCR扩增产物电泳图Fig.2 The electrophoretogram of PCR amplified products注:M为DNA分子质量标准;1为发酵6个月;2为发酵1年;3为发酵2年;4为发酵3年;5为发酵4年。
由图2扩增产物的电泳图可知,条带清晰,可以满足后续测序实验要求。
2.2 测序数据分析
传统自然发酵酱油中扩增DNA样本,经Illumina MiSeq高通量测序、质控并优化后共得到212762 条有效序列,且每组的有效序列数均在37000以上,占比80%以上,表明本次测序达到传统自然发酵酱油中后续微生物多样性分析的要求。
2.3 α多样性分析
根据传统自然发酵酱油中97%相似性水平下的OTU信息,对传统自然发酵酱油中的微生物物种丰富度和多样性进行分析,结果见表1,在本次传统自然发酵酱油中样本测序覆盖率均在99%以上。

表1 不同发酵时长酱油醅料中α多样性指数Table 1 α diversity indexes in the soy sauce mash fermented for different time
Shannon-Wiener曲线见图3,在本次传统自然发酵酱油的所有样本中,曲线逐渐稳定。

图3 Shannon-Wiener曲线分析Fig.3 The analysis of Shannon-Wiener curve
2.4 群落多样性分析
对传统自然发酵酱油在不同发酵时长下不同样品获得的OTU属水平上进行物种注释,统计分析结果见表2,其中属水平上相对丰度大于1%的菌群柱状分布见图4。

表2 不同发酵时间样品菌群在属水平上的分布Table 2 The distribution of bacterial community in samples at the genus level with different fermentation time

图4 不同发酵时长样品细菌的分布Fig.4 The distribution of bacteria in samples with different fermentation time
由表2可知,在发酵6个月的酱醅中发现27个菌属,其中丰度较大的菌群为芽孢杆菌属(Bacillus)、肠球菌属(Enterococcus)、葡萄球菌属(Staphylococcus)、肠杆菌属(Enterobacter)、盐厌氧菌属(Halanaerobium)、魏斯氏菌属(Weissella)、大肠杆菌志贺氏菌(Escherichia-Shigella)、色盐杆菌属(Chromohalobacter)、片球菌属(Pediococcus)、四联球菌属(Tetragenococcus),在发酵1年的酱醅中发现22个菌属,主要为芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter),在发酵2年的酱醅中发现26个菌属,主要为芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter)、魏斯氏菌属(Weissella)、色盐杆菌属(Chromohalobacter),在发酵3年酱醅中发现8个菌属,在发酵4年的酱醅中发现3个菌属的,其主要菌属都为芽孢杆菌属(Bacillus),这与之前传统自然发酵豆制品的研究结果相似[9-11]。表明在传统自然发酵酱油过程中,酱醅中细菌菌群处于动态变化中,且早期出现大肠杆菌志贺氏菌(Escherichia-Shigella)表明传统自然发酵卫生条件不佳[12],随着发酵时间延长,酱醅中细菌群落多样性呈现下降趋势,群落结构趋于稳定,形成以芽孢杆菌为绝对优势菌属并均趋于稳定。其中芽孢杆菌属(Bacillus)在酱油发酵过程中可以协助真菌分解原料中蛋白质和淀粉,是酱油中酱香风味成分形成的基础和关键[13];肠球菌属(Enterococcus)、四联球菌属(Tetragenococcus)、片球菌属(Pediococcus)、魏斯氏菌属(Weissella)等乳酸菌属是一类存在于酱油、鱼酱、豆制品等多种发酵食品及糖浆中的耐盐或耐高渗压的乳酸菌,它们在食品发酵过程中可提高食品中有机酸、醛类和酯类等风味物质含量,在提升产品风味、维持产品色泽、拮抗有害杂菌、促进有益酵母生长和提高产品功能性等方面具有重要作用[14]。
对传统自然发酵酱油不同发酵时长的酱醅中各微生物丰度的相似性进行聚类,传统自然发酵酱油中微生物总丰度热图见图5。

图5 各样本细菌聚类分析热图Fig.5 The heat map of the cluster analysis of bacteria in each sample
结合聚类分析结果可知,5个样本聚成3类,其中Y1和Y2这2个样本聚为一簇,Y3和Y4样本聚为一簇,表明两两之间样本相似,传统自然发酵酱油在发酵过程中细菌结构可以分为3个阶段,并于第3年传统自然发酵酱油进入稳定期。综上,发酵初期酱油细菌群落结构变化都较大,发酵中期(1~2年)趋于较稳定,发酵后期(3~4年)形成以芽孢杆菌为绝对优势菌属并均趋于稳定的细菌菌落结构。
实验采用Canoco for Windows 5.0对传统自然发酵酱油过程中细菌菌群,传统自然发酵酱油在发酵过程中各优势菌随着发酵时间的动态变化与分布见图6。

图6 传统自然发酵酱油发酵过程中细菌菌群PCA分析Fig.6 PCA analysis of bacterial community during the fermentation process of traditional natural fermented soy sauce
由图6可知,横、纵坐标的PC1和 PC2的贡献率分别为76.32%和14.98%,并且在传统自然发酵酱油中,第1年和第2年结构相似,第3年和第4年菌群结构相似,呈现出分为3个阶段的细菌群落结构变化规律。
3 结论
本研究对传统自然发酵酱油中不同发酵时长的细菌群落结构及多样性进行分析,共发现包括芽孢杆菌属(Bacillus)、肠球菌属(Enterococcus)、葡萄球菌属(Staphylococcus)、肠杆菌属(Enterobacter)、盐厌氧菌属(Halanaerobium)、魏斯氏菌属(Weissella)、片球菌属(Pediococcus)等在内的41个属。聚类分析结果显示,在传统自然发酵酱油过程中,酱醅中细菌菌群处于动态变化中,且早期出现大肠杆菌志贺氏菌(Escherichia-Shigella)表明传统自然发酵卫生条件不佳,随着发酵时间延长,酱醅中细菌群落多样性呈现下降趋势,形成以芽孢杆菌为绝对优势菌属并趋于稳定;传统自然发酵酱油发酵过程中,细菌群落结构的动态变化呈现出3个发酵阶段,并于第3年进入以芽孢杆菌为绝对优势菌的发酵后期,同时形成趋于稳定的细菌菌落结构。本研究对传统自然发酵酱油中的细菌群落结构进行了高通量测序分析,后续将对传统自然发酵酱油中真菌群落结构进行解构,进一步揭示传统自然发酵过程中的微生物菌群随着发酵时间的动态变化规律,为揭示传统自然发酵酱油独特的产品品质形成、潜力菌株筛选和传统自然发酵酿造技艺优化提供了理论依据。
