基于2b-RAD基因测序技术遗传图谱构建和QTL定位
2022-04-08高杰
高 杰
(河北农业大学海洋学院,中国水产科学研究院南海水产研究所)
简化基因组测序(Reduced-representation sequencing)是一种从第二代测序发展而来的测序技术,利用实验手段,如酶解,降低物种基因组的复杂性,对基因组的特定区域进行测序以反映结构信息的测序技术。
通常简化基因组测序,是指利用限制性内切酶对基因组DNA进行裂解,并分离出其中的某些特征片段,利用二代测序技术,可以用于全基因组遗传标记的开发和分型。利用简化的基因组测序技术进行大规模高通量SNP分型是目前国际上动植物基因组学研究的趋势。
使用时间最长的简单基因组分析技术是由美国Oregon大学发明的限制性位点关联DNA(RAD)技术。 然而,RAD技术有两大技术缺陷:繁琐的文库构建过程,涉及多轮的DNA分离和纯化,严重影响了技术的可重复性和可靠性;RAD技术产生的DNA具有不同长度的限制性片段,但PCR技术本身具有对图案的长度有偏好(即优先扩增较小的DNA片段)。而文库构建阶段的PCR和测序阶段的emulsionPCR,会严重影响扩增DNA片段的基因组代表性。
2b-RAD技术有效地克服了上述RAD技术的固有缺点。它使用IIB型限制性内切酶,通过全基因组消化产生33-36bp的等长标记,这些标记被富集并用于下游的高通量测序反应,通过生物信息分析进行高通量全基因组SNP筛选和分型分析。
1 简化基因组技术介绍
随着信息技术的不断提升,从2008年至今已经发展出至少以下7种简化基因组测序技术。

表1 简化基因测序技术发展顺序

技术名称 出现年份2b-RAD(IIB restriction sit e Associated DNA) [4] 2012 年SLAF(specific-locus amplified fragment sequencing) [5] 2013 年Multi-isoRAD(2b-RAD 升级版)[6] 2016 年Super-GBS(GBS 改进版)[7] 2018 年
2 2b-RAD技术
针对RAD技术的一系列缺点,2012年Wang等推出基于IIB型限制性内切酶的2b-RAD技术。其核心是IIB型限制性内切酶的应用。将适合于高通量测序平台的接头连接于标签两侧后,再经过PCR扩增,即可上机进行测序。
2.1 技术特点
与其他测序技术相比2b-RAD技术优势更加明显,如表2。其所有酶切片段都用于测序,避免了像其他RAD技术那样,必须经过片段选择而导致部分信息的损失;目前已经成功实现严重降解的中华绒螯蟹、中国对虾、等样品的建库测序[8];如果DNA量非常少,常规情况下无法开展个体水平的研究,2b-RAD对1-10ng级的DNA依然可建库成功。
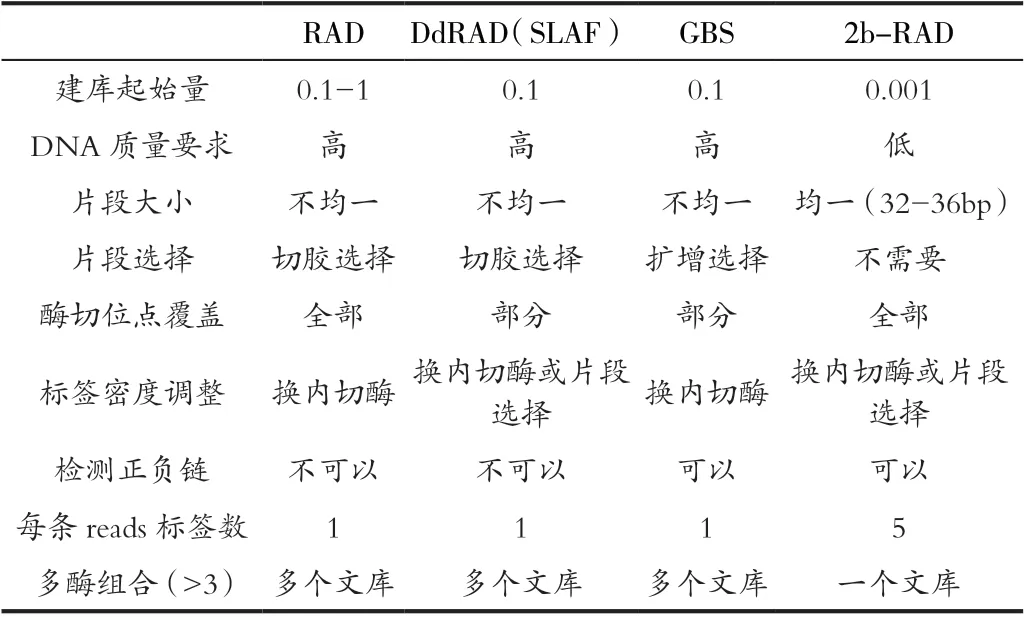
表2 常用简化基因组测序技术比较
2.2 遗传图谱和 QTL定位
遗传图谱(genetic map)又叫连锁图谱(linkage map),是根据重组率估算出来的基因或遗传标记在染色体上相对位置的线性排列图。遗传图谱的构建就是用重组率来表示基因的遗传距离,图距单位用厘摩(Centi-Morgan,cM)表示,1cM的大小大致表示1%的重组率。
QTL(数量性状基因座)定位是指控制数量性状的基因在基因组中的位置,是利用数理统计的方案,分析整个基因组DNA分子标记和数量性状表型值的关系。
利用2b-RAD技术,对某物种合适的作图群体进行测序,获得酶切位点附近的基因序列信息,运用生物信息学方法开发SNP,并计算标记间的遗传连锁距离,绘制连锁图谱,可用于后续QTL定位、辅助基因组组装等方向的分析。能够降低基因组的复杂度,数据量低,操作简便,节约成本,特别适合大样本量的研究。
3 结语
随着科学技术的发展基因测序技术也取得了一定的成就,2b-RAD技术具有信息全面、重复性好不受PCR偏好性影响等优点,其为我们更加深层次和更快捷了解物种某一性状提供了理论依据和技术手段。此技术的完善与发展将为未来基因测序提供了新的技术支持,推动简化基因技术进一步地发展。
