山羊CIDE家族基因克隆分析及互作蛋白预测鉴定
2022-03-22杨昌恒林亚秋朱江江
李 琪,杨昌恒,王 永,林亚秋,,向 华,朱江江,
(1.青藏高原动物遗传资源保护与利用四川省重点实验室,西南民族大学,四川 成都 610041;2.青藏高原动物遗传资源保护与利用教育部重点实验室,西南民族大学,四川 成都 610041)
动物的生长、发育及繁殖过程皆会受到脂代谢的影响。简州大耳羊是我国培育的第2个肉用山羊品种,作为我国重要的山羊品种资源,有关其脂质代谢的相关基因的克隆、功能分析、生物学特性方面的研究可以为改良其遗传特性提供理论依据。最初发现CIDE蛋白家族具有前凋亡蛋白的功能,后逐渐被发现是一类脂滴相关蛋白,在机体脂质平衡中也起到了重要的作用,且该家族基因还是人类肥胖的候选基因之一。探索CIDE家族基因调控网络及其在山羊脂代谢过程中的作用具有重要意义。CIDE(Cell death-inducing DFF45-like effectors)家族刚开始被发现时,被认为与细胞凋亡相关,并且因为与DFF45(DNA fragmentation factor,DFF45)的 N 端结构域高度同源而得名[1-3]。随着人们不断地研究发现,CIDE 家族蛋白主要参与调控细胞的脂代谢,是一类和脂类代谢密切相关的蛋白。CIDE家族共有3个成员,在小鼠中为 CIDEA、CIDEB、FSP27(Fat-specific protein 27)[4],在人体中为 CIDEA、CIDEB、CIDEC[5]。CIDE 家族蛋白的氨基酸序列十分保守,其 N 端(CIDE-N)为一段保守的碱性序列,后面紧跟着一段酸性序列,CIDE蛋白的N 端序列之间可以形成相互作用,也可以通过该结构与其他蛋白形成相互作用。CIDE 蛋白的 C 端可以介导脂滴的融合,且其疏水区具有脂滴定位功能[6-9]。脂肪组织是机体储存能量的主要部位,包括白色和棕色脂肪组织。白色脂肪组织细胞的大部分空间被一个大脂滴所占据,棕色脂肪组织细胞中的脂滴比较小,但通常数量较多[10]。脂滴(Lipid droplet,LD)是细胞内存储脂质的主要亚细胞器,其核心由中性脂质构成,外围为单层磷脂膜,对体内的脂质平衡起重要的调控作用[11-13]。脂滴的膜上分布有多种蛋白质,能够合成或分解脂滴中的磷脂及中性脂,从而调节脂滴的大小及功能[14]。CIDE家族的蛋白均定位于脂滴表面,能够在脂滴与脂滴之间的接触位点上形成脂滴融合复合物(Lipid droplet fusion complex,LDFC),连通2个脂滴,促进脂滴间的融合[10],从而调控脂滴的大小。当过表达CIDE家族基因时,可使细胞内脂滴增大,而在细胞或组织中敲除或是沉默CIDE家族基因时,会导致脂滴变小[15-16]。然而,目前有关脂滴形成的精确机制以及CIDE家族在其中的调控网络还尚不清楚。本研究以简州大耳羊为研究对象,通过基因克隆及生物信息学分析,分析了CIDE家族基因的生物学特性,为其功能研究奠定了基础。并通过实时荧光定量PCR(Real-time quantitative PCR,RT-qPCR)检测了CIDE家族基因在山羊不同组织中的表达水平,发现了与CIDE家族基因表达量具有相关性的基因,可为进一步研究精确的脂滴形成机制以及进一步揭示CIDE基因在脂代谢中的调控网络提供参考和理论基础。
1 材料和方法
1.1 试验材料
1.1.1 试验动物 随机选取成年(1周岁)健康的简州大耳羊10只为试验动物(购自四川省简阳市大哥大牧业有限公司)。屠宰后采集其心、肝、脾、肺、肾、瘤胃、背最长肌、股二头肌等组织样品,将采集的各组织分割成黄豆大小,用DEPC水清洗干净后迅速装入无RNase的冻存管中,置于液氮中保存,用于后续组织RNA的提取。
1.1.2 试验试剂 DNA回收试剂盒、DNA聚合酶、大肠杆菌DH5α感受态细胞购自天根生化科技有限,TRIzol、SYBR®Premix Ex TaqTM(2×)试剂盒、pMD-19T Vector购自TaKaRa公司,反转录试剂盒购自Thermo公司。
1.2 试验方法
1.2.1 组织总RNA提取及反转录 用 TRIzol 法提取简州大耳羊的心、肝、脾、肺、肾、瘤胃等组织的总RNA,测定总RNA的OD260/280值,当该值在 1.8~2.0说明可正常使用,然后通过琼脂糖凝胶电泳检测其完整性。利用反转录试剂盒将提取的RNA反转录合成 cDNA,反应体系及条件:总 RNA 1 μg,Oligo(dT)Primer 1 μL,加 DEPC 水至 12 μL,65 ℃ 5 min;然后加入 dNTP mix 2 μL,5×Reaction Buffer 4 μL,RiboLock RNase Inhibitor 1 μL,Revert Aid RT 1 μL,42 ℃ 60 min,70 ℃ 5 min。-20 ℃保存,用于后期基因克隆及qPCR。
1.2.2 山羊CIDEB、CIDEC基因克隆 根据GenBank上山羊的CIDEB(XM_005685207.3)、CIDEC(XM_018038446.1)基因预测序列,利用Primer Premier 5.0软件设计PCR引物(表1)。RT-PCR反应总体系:2×Taq Master Mix 12.5 μL,上下游引物(10 μmol/L)各1 μL,模板cDNA 1 μL,加水补至25 μL。PCR扩增程序:预变性(98 ℃,3 min);变性(98 ℃,30 s),退火(CIDEB56 ℃/CIDEC59 ℃,30 s),延伸(72 ℃,1 min),共35个循环;延伸(72 ℃,10 min),最后12 ℃保存。1%的琼脂糖凝胶检测后利用胶回收试剂盒对目的片段进行回收和纯化,将纯化后的产物连接到pMD-19T载体上,并通过热激转化到感受态细胞(DH5α)中,然后接种于涂有IPTG和X-gal的LB固体培养基(含Amp)上,37 ℃过夜培养后挑取白色阳性菌落,进行菌液PCR鉴定后送至成都擎科生物技术有限公司进行测序。

表1 克隆引物Tab.1 Primers for cloning
1.2.3 山羊CIDEs基因生物学特性分析 使用ProtParam在线工具对CIDE家族蛋白序列进行分析,使用Prot Scale(http://web.expasy.org/protscale)预测CIDE蛋白的亲疏水性,利用ExPASy(https://embnet.vital-it.ch/software/TMPRED_form.html)预测CIDE家族蛋白的跨膜区域。利用EMBL-EBI(https://www.ebi.ac.uk/Tools/pfa/phobius/)及TargetP-2.0(http://www.cbs.dtu.dk/services/TargetP-2.0/)进行CIDE家族蛋白信号肽的预测。使用SOPMA分析该家族蛋白的二级结构,通过SWISS-MODEL预测蛋白三级结构,利用STRING分析与其相互作用的蛋白。
1.2.4 山羊CIDE基因组织表达差异分析 根据NCBI上的序列设计CIDEA(KP120985)基因的RT-qPCR引物,根据克隆获得的CIDEB、CIDEC基因序列设计其RT-qPCR引物(表2)。
利用RT-qPCR技术检测CIDEs在各个组织中的表达差异,以UXT为内参基因矫正目的基因的相对表达水平,每个组织样本设置3个重复。RT-qPCR结果采用 2-ΔΔCt法进行分析。
1.2.5 CIDE与其互作蛋白相对表达量相关性分析 利用STRING分析得到与CIDE家族基因相互作用的蛋白质,分别设计各互作蛋白的定量引物(表2)。RT-qPCR检测以山羊心脏组织的Ct值为对照,结果用2-ΔΔCt法进行统计分析,检测得到CIDEs基因在山羊中表达量最高的组织,并检测该组织中与其相互作用的基因的表达量,用SPSS软件分析CIDE基因的表达量与其相互作用的基因的表达量的相关性,P<0.05时为差异显著,P<0.01时为差异极显著。

表2 定量引物Tab.2 Primers for quantitative real-time PCR(qPCR)

表2(续)
2 结果与分析
2.1 山羊CIDEB、CIDEC基因克隆
本试验以山羊的肝脏组织cDNA为模板,RT-PCR扩增获得山羊CIDEB基因序列923 bp(图1),其中5′UTR为 146 bp,CDS区660 bp,3′UTR为117 bp,编码219个氨基酸。CIDEC基因1 043 bp(图1),其中5′UTR 为23 bp,CDS区714 bp,3′UTR 为306 bp,编码237个氨基酸。
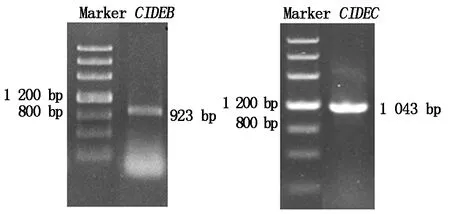
A.克隆所得山羊CIDEB 923 bp;B.克隆所得山羊CIDEC 1 043 bp。A.Cloned goat CIDEB 923 bp;B.Cloned goat CIDEC 1 043 bp.
2.2 山羊CIDE基因生物学特性分析
2.2.1 蛋白理化性质分析 对山羊CIDEB蛋白序列进行分析,预测其蛋白分子式为C1090H1759N311O322S8,分子质量为24.629 33 ku;理论等电点(Theoretical pI)为9.30,不稳定指数为64.96,亲水性总平均值为-0.372,推测该蛋白为碱性亲水性不稳定蛋白。山羊CIDEC蛋白分子式为C1190H1905N321O350S11,分子质量为26.661 82 ku;理论等电点为9.27,不稳定指数为40.44,亲水性总平均值为-0.258,推测该蛋白为碱性亲水性不稳定蛋白(图2)。CIDE家族蛋白信号肽预测结果如图2及表3。

表3 CIDE蛋白信号肽预测Tab.3 Signal peptide of CIDE protein

图2 CIDE蛋白的亲疏水性、跨膜结构以及信号肽的预测Fig.2 Hydrophobic,transmembrane structure and signal peptide of CIDE protein
2.2.2 蛋白质结构及亚细胞定位预测 山羊CIDE家族蛋白均具有 2 个丝氨酸(Serine)磷酸化位点、1 个苏氨酸(Threonine)磷酸化位点和 3 个酪氨酸(Tyrosine)磷酸化位点。
CIDEA二级结构预测显示,79(39.11%)个氨基酸残基可能形成 α-螺旋(h),34(16.83%)个氨基酸残基可能形成β-折叠(e),76(37.62%)个氨基酸残基可能形成无规则卷曲(c),13(16.83%)个氨基酸残基可能形成β-转角(t)(图3-A)。CIDEB二级结构预测显示,102(46.58%)个氨基酸残基可能形成 α-螺旋(h),34(15.53%)个氨基酸残基可能形成β-折叠(e),71(32.42%)个氨基酸残基可能形成无规则卷曲(c),12(5.48%)个氨基酸残基可能形成β-转角(t)(图3-B)。CIDEC二级结构预测显示,102(46.58%)个氨基酸残基可能形成 α-螺旋(h),34(15.53%)个氨基酸残基可能形成β-折叠(e),71(32.42%)个氨基酸残基可能形成无规则卷曲(c),12(5.48%)个氨基酸残基可能形成β-转角(图3-C)。三级结构预测显示,山羊和绵羊的CIDE家族蛋白的三级结构相似,与牛的CIDE家族蛋白三级结构略有不同。山羊CIDEB和CIDEC的三级结构相似,但其CIDEA的三级结构与CIDEB和CIDEC差别较大,而牛的CIDEC的三级结构与CIDEA和CIDEC差别较大,猜测可能与其功能的差别有关,但CIDEB蛋白的三级结构在不同物种中都很保守(图4)。采用STRING 交互式数据库搜索可能与CIDE家族蛋白各成员相互作用的蛋白,结果如图5。

蓝色线条.α-螺旋;红色线条.β-折叠;紫色线条.无规则卷曲;绿色线条.β-转角。Blue line.α-helix;Red line.β-sheet;Purple line.Random curl;Green line.β-turn.
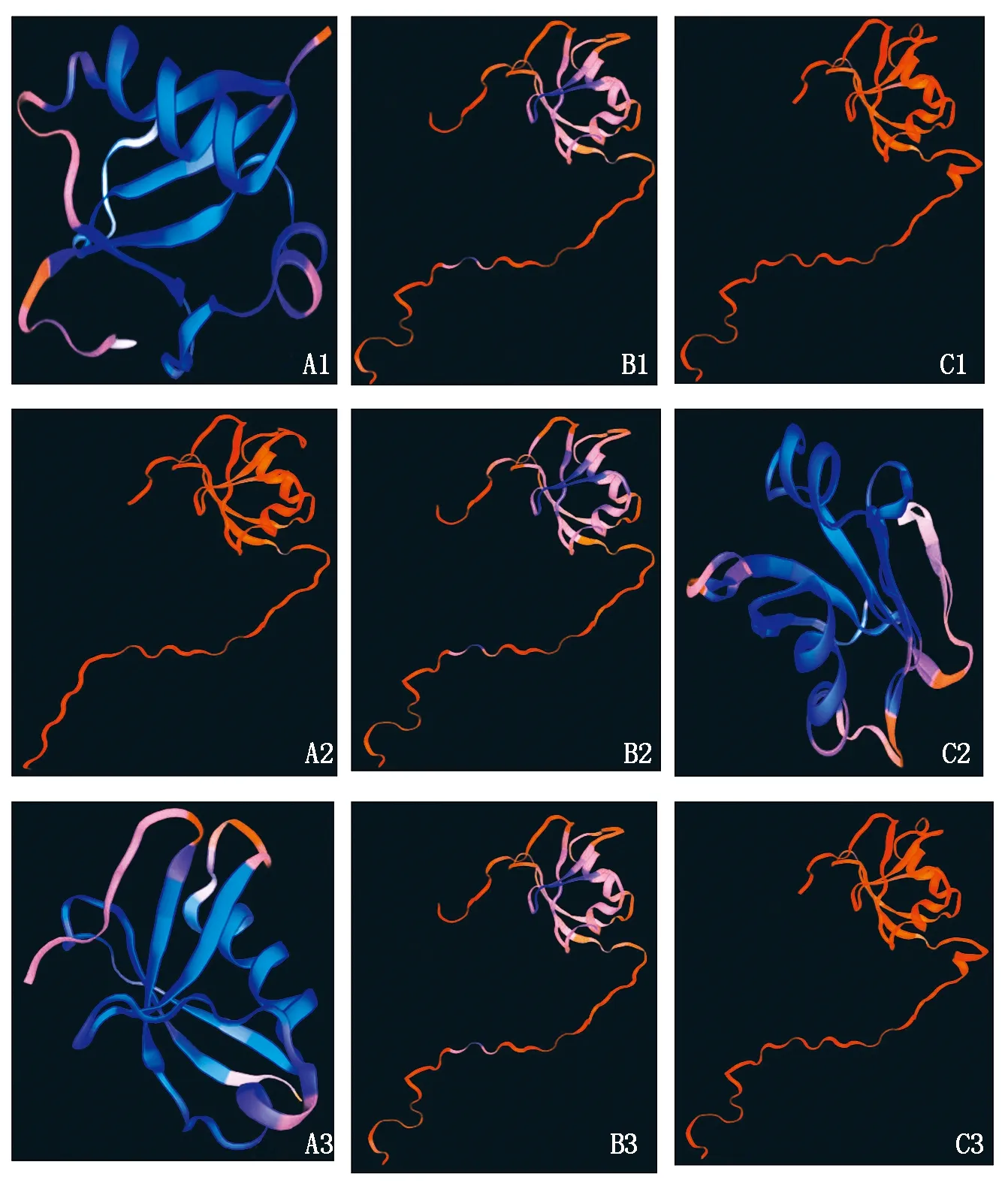
A2、B2、C2分别为牛的CIDEA、CIDEB、CIDEC三级结构预测;A3、B3、C3分别为绵羊的CIDEA、CIDEB、CIDEC三级结构。A2(CIDEA),B2(CIDEB)and C2(CIDEC)are predicted tertiary structure of the cattle;and A3(CIDEA),B3(CIDEB)and C3(CIDEC)are predicted tertiary structure of the sheep.
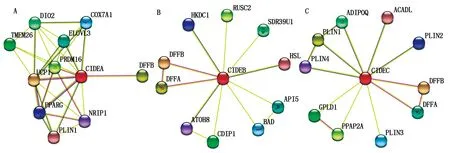
图5 山羊CIDEA(A)、CIDEB(B)和CIDEC(C)蛋白相互作用蛋白预测Fig.5 Prediction of proteins interacting with goat CIDEA(A),CIDEB(B)and CIDEC(C)protein
2.2.3 氨基酸序列同源性比对及系统进化树构建 利用NCBI对不同物种CIDEA的氨基酸同源性进行比较,发现简州大耳羊与绵羊、牛、猪的同源性分别为99.01%,96.04%,83.17%。CIDEB与绵羊、牛、猪、人的同源性分别为100%,96.80%,87.67%,83.56%。CIDEC与绵羊、牛、人的同源性分别为99.16%,93.25%,84.87%。利用MEGA 5.0分别构建山羊CIDEA、CIDEB和CIDEC氨基酸序列系统进化树,结果如图6所示,山羊CIDEA、CIDEB、CIDEC均与绵羊亲缘关系最近。

图6 山羊 CIDEA(A)、CIDEB(B)、CIDEC(C)氨基酸序列系统进化树Fig.6 Phylogenetic trees of goat CIDEA(A),CIDEB(B)and CIDEC(C)amino acid sequence
2.3 山羊CIDE基因组织表达差异分析
以山羊心脏组织为对照,UXT为内参,分析qPCR定量结果可知CIDE家族基因在山羊各组织中均有表达,其中CIDEA在山羊瘤胃中高表达(图7-A);CIDEB在肝脏中高表达(图7-B);CIDEC在小肠中有较高水平的表达(图7-C)。

差异显著(P<0.05)用不同小写字母表示;差异不显著(P>0.05)用相同小写字母表示。The significant difference(P<0.05)was indicated in different lowercase letters;and the insignificant difference(P > 0.05)was indicated in the same lowercase letters.
2.4 山羊CIDE与其互作蛋白表达量相关性分析
本研究利用RT-qPCR检测了CIDEs在山羊不同组织中的表达量,发现CIDEA、CIDEB、CIDEC分别在山羊瘤胃、肝脏、小肠中表达量最高,进而检测了在这些组织中与其相互作用的基因的表达量,通过SPSS软件来分析CIDE基因的表达量与其相互作用的基因的表达量的相关性。由数据分析可以看出,CIDEA与DFFB(R=0.723,P=0.009)和ELOVL3(R=0.724,P=0.009)在瘤胃中的表达量具有极显著正相关性(P<0.01),与DIO2(R=0.560,P=0.046)、TMEM26(R=0.660,P=0.019)、PRDM16(R=0.694,P=0.013)、PLIN1(R=0.640,P=0.023)具有显著正相关性(P<0.05)(表4)。CIDEB与DFFA(R=0.694,P=0.013)和SDR39U1(R=0.655,P=0.020)在肝脏中的表达量具有显著正相关性(P<0.05)(表5)。CIDEC与PLIN2(R=0.596,P=0.034)、GPLD1(R=0.554,P=0.048)、ADIPOQ(R=0.578,P=0.040)在小肠中的表达量具有显著正相关性(P<0.05)(表6)。

表4 CIDEA与其互作蛋白相关性分析Tab.4 Correlation analysis of CIDEA and its interacting proteins

表5 CIDEB与其互作蛋白相关性分析Tab.5 Correlation analysis of CIDEB and its interacting proteins

表6 CIDEC与其互作蛋白相关性分析Tab.6 Correlation analysis of CIDEC and its interacting proteins
3 结论与讨论
影响机体脂代谢平衡的最主要细胞器是脂滴。Richard在1890年发现了脂滴的存在,但直到1991年,脂滴表面蛋白Plin1被发现后,才真正开始了有关脂滴的研究[17]。脂滴的外部是一层磷脂膜,内部为中性脂,但不同类型的细胞,其脂滴内部所含有的中性脂也会有所不同[18],脂滴表面分布有许多功能蛋白。随着越来越多的研究发现,脂滴的功能不仅仅局限于贮存脂质,还能够参与脂质运输、细胞之间、细胞内以及细胞膜与细胞器之间的信号传递过程等[19]。脂滴发生的异常[13]及CIDEs表达的异常[20]都会引发脂肪肝[21]、肥胖[22]等脂代谢疾病。而CIDEs在脂滴的形成及调控中起到了重要作用,该家族蛋白能在脂滴之间形成脂滴融合复合物(LDFC),将中性脂从供体脂滴转运到受体脂滴中,介导脂滴的融合[23-24],促进大脂滴的形成。且其对脂滴的调控能力也不同,有研究发现CIDEA 和 CIDEC 融合脂滴的能力相同,CIDEB的能力较弱[25-26],可能不同成员通过不同的调控通路来调节脂滴大小。
利用该试验获得CIDEB和CIDEC基因序列,可以为后续研究CIDEs调控山羊脂质代谢及在脂滴形成中的作用奠定基础。并通过相关性分析,检测到了不少与CIDEs表达量相关的基因,且大多与脂代谢或脂滴相关。如CIDEA与ELOVL3(R=0.724,P=0.009)的表达量具有极显著正相关性,而ELOVL3 为极长链脂肪酸延长酶3,属于脂肪酸延长酶(ELOVLs)家族,可以催化 C16、C18、C20 等极长链脂肪酸的伸长[27-28]。且在分化的牛肌肉卫星细胞(MDSCs)中 ELOVL3 与脂滴的亚细胞定位具有一致性,ELOVL3 可能通过催化脂肪酸的延伸来调节 TAG 的形成,从而促进脂滴的积累[29]。PLIN1 是分布于脂滴表面含量最多的脂滴包被蛋白,该基因的表达量也与CIDEA呈显著正相关性(P<0.05),该基因的过表达可在mRNA和蛋白水平促进脂肪代谢相关基因的表达,促使脂肪细胞中的脂滴数量、体积和甘油三酯含量增加[30]。CIDEC与PLIN2(R=0.596,P=0.034)的表达量具有显著正相关性,PLIN2是脂滴包被蛋白2,是胞内脂滴表面相关蛋白之一,对脂滴的合成和降解具有重要作用[31],且其可以上调SREBP2的表达,促进巨噬细胞内脂质积累[32]。由此可以推测,CIDE家族不同成员可以通过调控不同的基因或者受到不同基因的调控来调节生物体脂质代谢和脂滴的形成,通过检测与其表达量相关的蛋白可以从侧面验证与其存在相互作用的脂代谢相关蛋白,并可能由此发现其调控通路。
RT-qPCR数据检测未发现CIDE家族蛋白与其他蛋白表达量的相关性,且利用软件也只分析到了部分与其相互作用的蛋白质,但越来越多的蛋白、转录因子被发现能够调节CIDE家族蛋白,从而调控脂滴的发生和大小,影响机体的脂质代谢。CIDEA的表达会受到转录水平的调节,该基因的启动子含有PPARα和 PPARγ 的应答元件,可被PPARs 的激动剂激活[33]。CIDEA可以调控棕色脂肪组织中的UCP1(解偶联蛋白),从而影响机体的脂代谢[34]。CIDEA还可与AMPK 的β亚基发生相互作用。CIDEA敲除小鼠的AMPK 蛋白表达水平及酶活性都有所升高,能够促进脂肪酸的β氧化[35]。CIDEB能够促进TAG 从脂滴向极低密度脂蛋白(VLDL)转运,该蛋白C端的结构可以和位于VLDL 上的 ApoB-100相互作用,提高了VLDL的脂肪组装、成熟及分泌过程[36]。PGC1β 能够促进CIDEB转录水平的调节作用,进而调控 VLDL 的分泌[37],影响脂滴的大小。对脂肪细胞中内源性的 Plin 和 Fsp27进行免疫共沉淀,发现Plin 中心结构域中的氨基酸 291—321部分介导了 Plin 与 Fsp27 的相互作用[38],Plin1 可以与 CIDEC相互作用促进脂滴的融合[39]。由此可见,无论是软件分析还是RT-qPCR检测结果,只能发现部分与CIDE家族蛋白相互作用的蛋白,探索CIDE家族完整的调控网络还需要更进一步的研究。
越来越多的蛋白被发现能够与CIDE家族蛋白发生相互作用,参与脂滴的发生,通过研究与其相互作用的蛋白质、转录因子等,可进一步了解CIDE家族调控网络及其在脂质沉积、脂质代谢中的作用,也有助于更进一步地掌握脂滴形成的精确机制。且通过研究影响脂滴生长的相关基因,可以为肥胖、脂肪肝等脂代谢相关疾病的治疗提供了理论信息。
本研究成功克隆获得山羊CIDEB基因CDS序列660 bp,编码219个氨基酸,CIDEC基因CDS区714 bp,编码237个氨基酸。CIDEA、CIDEB、CIDEC基因分别在山羊瘤胃、肝脏、小肠中表达量最高。CIDEA与DFFB和ELOVL3在瘤胃中的表达量具有极显著正相关性,与DIO2、TMEM26、PRDM16、PLIN1具有显著正相关性。CIDEB与DFFA和SDR39U1在肝脏中的表达量具有显著正相关性。CIDEC与PLIN2、GPLD1、ADIPOQ在小肠中的表达量具有显著正相关性。该结果为进一步阐明CIDEs在脂质代谢及脂滴形成中的作用以及CIDE家族调控网络提供理论基础。
