基于QTL相关SSR标记分析黄淮海和南方大豆品种的遗传多样性及群体遗传结构
2022-03-16刘嘉霖谢慧敏张峥杨柏云罗火林龚贵如熊冬金
刘嘉霖,谢慧敏,张峥,杨柏云,罗火林,龚贵如,熊冬金
(南昌大学生命科学学院,江西 南昌,330031)
大豆(Glycine max(L.) Merr.)含有约40%的蛋白质和20%的脂肪,是重要的粮食作物。我国是大豆的原产地,拥有十分丰富的大豆种质资源[1~3]。黄淮海和南方是我国重要的大豆产区,与国内其他的大豆生态区域种质交流频繁,选育出了大量优异的大豆品种[4]。这些育成品种是大豆选育的重要资源,对大豆遗传研究具有十分重要的意义。Zhou等[5]和Guo 等[6]利用SSR 标记分析大豆种质资源,认为黄淮海夏大豆的遗传多样性最高。盖钧镒等分三个育种时期(1923-1995、1995-2005、2005-现在)对我国大豆育成品种进行了品种收集及系谱分析等工作[7~9],其中2005 年之前的1200 多个优良品种被广泛应用于我国大豆生产[10]。研究大豆遗传多样性和遗传基础,对大豆育种的亲本选配和遗传改良具有重要指导意义。近年来,分子标记和数量性状(QTL)定位发展迅速,在大豆方面,QTL 定位了大豆耐盐、大豆叶绿素、大豆籽粒大小、脂肪含量[11]、蛋白质[12]、产量[13]等性状。Karikari 等[14]报道了亚非欧三个区域大豆资源的遗传多样性,共有21 个SSR 标记与开花天数和百粒重相关。Patil 等[15]对野生、栽培大豆利用连锁定位技术鉴定出18个与蛋白质、油脂和蔗糖含量相关QTL,部分QTL 与大豆驯化相关基因组位点重合。随着基因组重测序和全基因组关联分析的发展[16,17],QTL定位数量激增。利用功能分子标记及与产量性状相关的功能分子标记来研究大豆主要家族的育成品种遗传基础报道较少。本试验选用与QTL相关SSR标记对来源于中国黄淮海江苏的滨海大白花(A034)和铜山天鹅蛋(A231),山东的即墨油豆(A133)三个祖先亲本家族共105份大豆育成品种进行遗传多样性与群体遗传结构研究,研究不同时期群体的遗传多样性水平。探索优异等位变异在大豆品种选育中的利用途径及功能基因相关位点标记的遗传结构,充分了解现有重要祖先亲本家族育成品种的遗传基础,挖掘利用大豆遗传多样性信息,指导育种亲本创制和选择,及品种间的合理布局,提高育种的可预见性,丰富种质遗传多样性。
1 材料与方法
1.1 材料
根据熊冬金等[18]对我国1923-2005 年育成的1300 个大豆的研究信息,选择12 个省份105 份中国黄淮海产区的大豆育成品种作为试验材料(表1)。其中滨海大白花(A034)和铜山天鹅蛋(A231)是江苏的祖先亲本,即墨油豆(A133)山东祖先亲本,105份大豆育成品种名称及相关信息详见首页OSID附表。
表1 105份大豆实验材料Table 1 105 cultivars used in this study
1.2 方法
1.2.1 DNA的提取 选取4~5叶期的大豆叶片,采用改良的CTAB 法[19]提取DNA。用1.2%琼脂糖凝胶电泳检测其完整性,使用核酸定量仪Qubit 2.0测定浓度与纯度,根据检测结果将DNA 稀释至20 ng·μL-1,-20℃保存备用。
1.2.2 QTL 相关SSR 标记引物筛选 根据http://soybase. org[20]资料查阅已经被定位的与产量性状QTL 紧密连锁的SSR 标记作为功能基因标记位点。选择分布于20 条染色体的99 个多态性高、带型清晰、重复性好的SSR 标记。以上引物序列在http://soybase.org查询并由生工生物工程(上海)股份有限公司合成。PCR 反应总体系15 μL,含DNA 60 ng,上下游引物0.1 μmol·L-1,Taq 酶1.5 U,dNTPs 0.2 μmol·L-1,MgCl20.12 μmol·L-1,ddH20 补齐反应体系。PCR 程序为:94℃3 min;94℃30 s,50~59℃1 min,72℃1 min,共30 个循环;72℃10 min。扩增后的PCR 产物用8%非变性聚丙烯酰胺凝胶电泳检测,再用银染显影后观察结果并拍照。
1.3 数据分析
1.3.1 群体遗传多样性分析 通过SSR 标记的主带分子量大小,对105 份供试材料进行基因分型并输入Excel。利用Powermarker V3.25[21]软件计算多态性信息含量(PIC)和基因多样性(gene diversity)系数。用Gen Al Ex6.5[22]软件进行标记位点的遗传多态性分析,计算等位变异位点数(Na)、有效等位变异位点数(Ne)、Shannon’s 信息指数(I),并计算群体的分子方差(AMOVA)估测遗传变异在亚群间和亚群内的分布。
1.3.2 群体遗传结构分析 进一步通过Power-Marker 软件分析各品种之间的遗传相似性,用非加权配对法(UPGMA)进行聚类分型,经i-TOL[23]软件转化为聚类无根树状图;用Gen Al Ex6.5 进行主坐标分析(PCoA)。利用Structure 2.3.4[24]软件估算群体的遗传结构,并计算材料相应Q 值(第I 材料属于第k 亚群的概率)。根据structure Harvester(http://taylor0. biology. ucla. edu/structure Harvester/#)确定合适的K值。
2 结果与分析
2.1 引物扩增多态性结果分析
从20 条染色体上选取了99 对扩增效果较好,数据缺失少的SSR 引物进行分析(图1),最少的11号染色体上仅含1个标记位点,最多的4号、6号和7号染色体上含有8 个标记位点,平均每个染色体上含有4.95 个标记位点。所有位点都检测到等位变异数,每个位点的等位变异数5~24 个,平均每个位点11.54个等位变异。每条染色体的等位变异数9~94个,平均每条染色体上57.10个等位变异(表2)。

表2 引物扩增多态性结果Table 2 Polymorphism result of primers
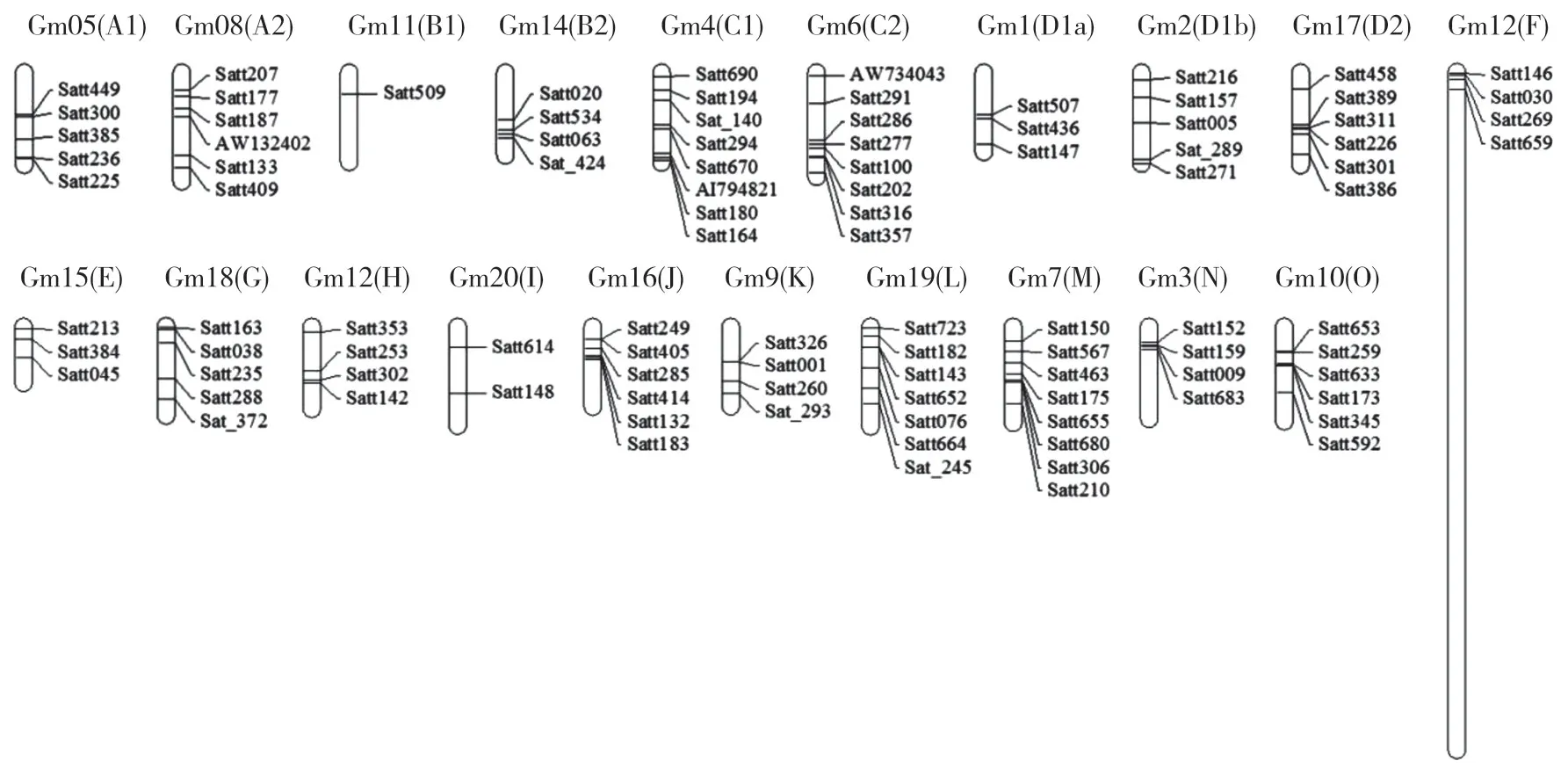
图1 QTL相关的SSR标记位点在染色体上的分布Fig.1 Distribution of SSR marker sites on chromosomes
计算分析每个位点等位基因频率,9 号染色体上Satt326 等位基因频率最高为0.486,3 号染色体上Satt009 最低为0.095,平均等位基因频率0.238。所有的105 个材料上所有等位位点都是纯合基因型,没有杂合基因型,说明所选大豆材料都是高度纯合的育成品种。
2.2 时期亚群间遗传多样性分析
将105 份大豆材料按育成时期划分为4 个亚群,以下简称时期亚群。由表3 可以看出4 个时期亚群和整个群体的遗传多样性,1991-2000 亚群的观测等位基因数(9.121)、Nei's基因多样性(0.839)、多态性信息含量(0.820)最高,而2001-2004 亚群的有效等位基因数(0.6088)最高。1963-1980 亚群的观测等位基因数(6.152)、有效等位基因数(4.668)、Nei's基因多样性(0.628)、多态性信息含量(0.562)都最低。以上结果说明1991-2000亚群的遗传多样性最高,而2001-2004 亚群的材料分布更均匀。1963-1980 亚群的遗传多样性最低可能是由样本数偏少造成的。
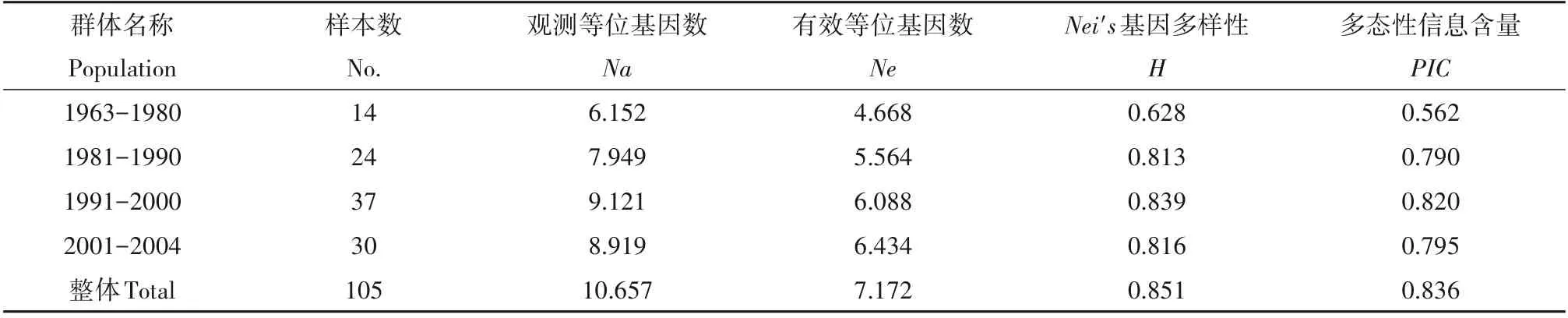
表3 时期亚群间遗传多样性Table 3 Genetic diversity of period populations
2.3 时期亚群相似性系数聚类分析与分子方差分析
表4 相似性系数结果表明亚群间相似性系数(GI)≥0.679,亚群间遗传距离(GD)≤0.387。说明分时期亚群间的遗传差异较大。表5 和图2 可以看出1991-2000 和2001-2004 两个亚群的相似性系数高(GI=0.821),分化小,亲缘关系相近。而1963-1980和1991-2000 两亚群的遗传距离较大,亲缘关系也较远(GD=0.387)。分时期亚群的相似性系数聚类分析和遗传多样性分析结果相似。但综合考虑亚群内样本数目和材料分布等因素的影响,尚不能说明大豆育成品种的基因遗传多样性和育种时间的关系。
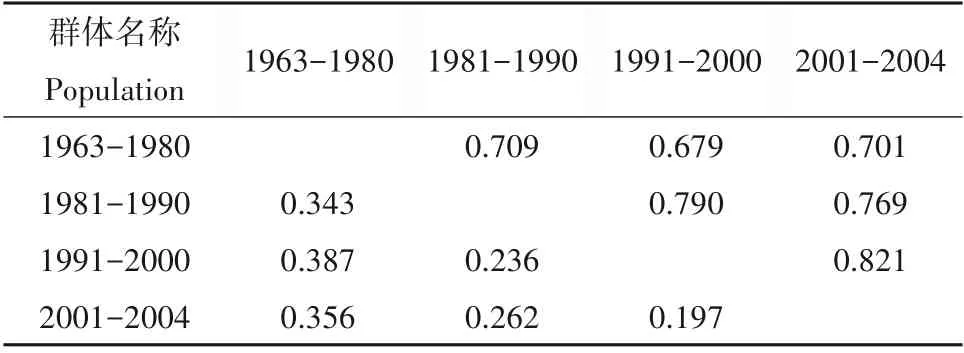
表4 时期亚群间Nei's遗传距离与相似性系数Table 4 Nei's genetic distance and genetic identity between period populations

表5 大豆材料在3个类群中的分布Table 5 Distribution of populations in 3 groups
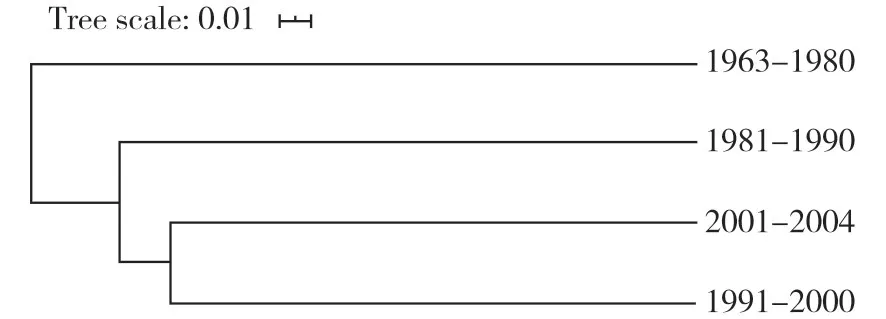
图2 基于Nei's遗传距离大豆时期亚群聚类图Fig.2 Period populations dendrogram of soybeans based on Nei's genetic distance
分子方差分析(AMOVA)分析结果表明,大豆时期亚群内分子变异占99%,群体间变异占1%,而群体分化系数Fst=0.013,P=0.010,组间差异显著。说明亚群间的遗传差异主要是由于亚群内的大豆材料遗传差异较大造成的。分析大豆材料遗传差异较大可能与大豆育成品种主要来自3个不同主要家族,且每个祖先亲本对该品种的遗传贡献值差异造成的。
2.4 大豆材料聚类分析
UPGMA 聚类分析显示(图3),在相似系数D1=0.42 处将105 份大豆材料分为3 个类群,第Ⅰ类群包含49 份材料,主要来自1991-2000 和2001-2004两时期。第Ⅱ类群含有25 份材料,其中17 份大豆材料来自1963-1980、1981-1991 这2 个相对较早的时期。第Ⅲ类群共31份材料,大豆各时期材料分布相对均匀。
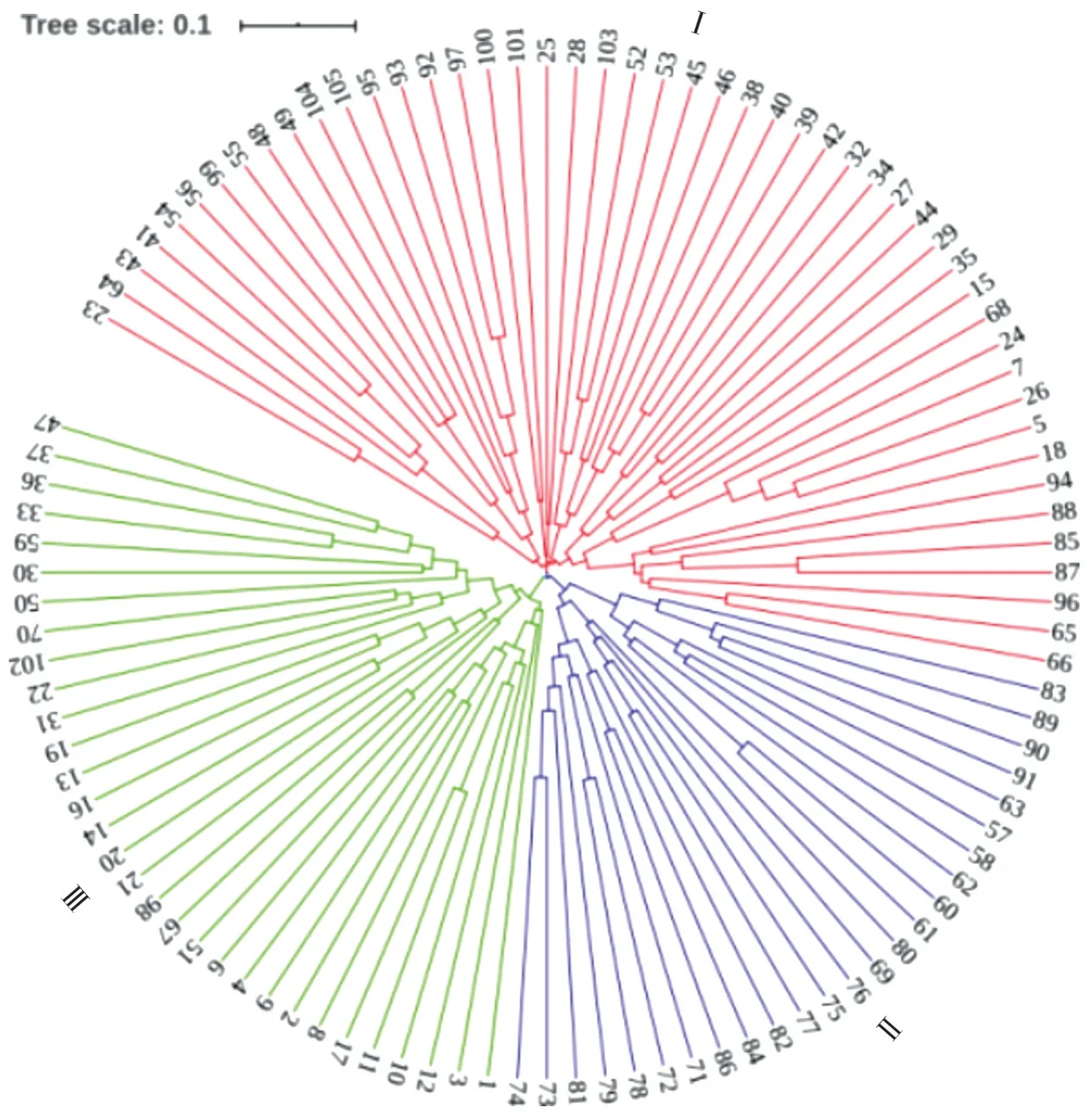
图3 105份大豆材料的UPGMA聚类图Fig.3 UPGMA dendrogram of 105 accessions
从各时期亚群分布来看,4 个时期亚群分散聚集到3 个类群中,且各亚群有其聚类的偏好性。1991-2000、2001-2004 这两个相对较晚的时期主要分布于第Ⅰ类群,这两个时期的大豆资源具有一定的遗传相似性。1963-1980 时期的大豆材料有50%聚集在第Ⅱ类群,且第Ⅱ类群68%的大豆材料来自1963-1980、1981-1990 这2 个时期,说明相对较早时期的大豆材料偏向聚集在第Ⅱ类群。1981-1990时期的大豆均匀分布于第Ⅱ、Ⅲ类群,说明1981-1990时期的大豆材料遗传资源集中度相对较低。
2.5 群体遗传结构分析
大豆材料的遗传结构分析表明K 取值范围为2~9,当K=3 时,△K 最大且较稳定(图4),大豆群体分为3 个类群(图5)。分别对3 个类群内大豆材料统计(表6),第Ⅰ类群包含44份材料,第Ⅱ类群含有25份材料,第Ⅲ类群共36份材料。群体遗传结构分布表明,遗传结构分布与UPGMA 聚类结果具有高度一致性。有100份大豆材料的结构分布与聚类结果一致。UPGMA 聚类第Ⅰ类群中4 份四川省大豆材料(贡豆5 号、贡豆8 号、贡豆10、贡豆11)和江西省大豆材料赣豆4号在群体遗传结构分析中分布在第Ⅲ类群。
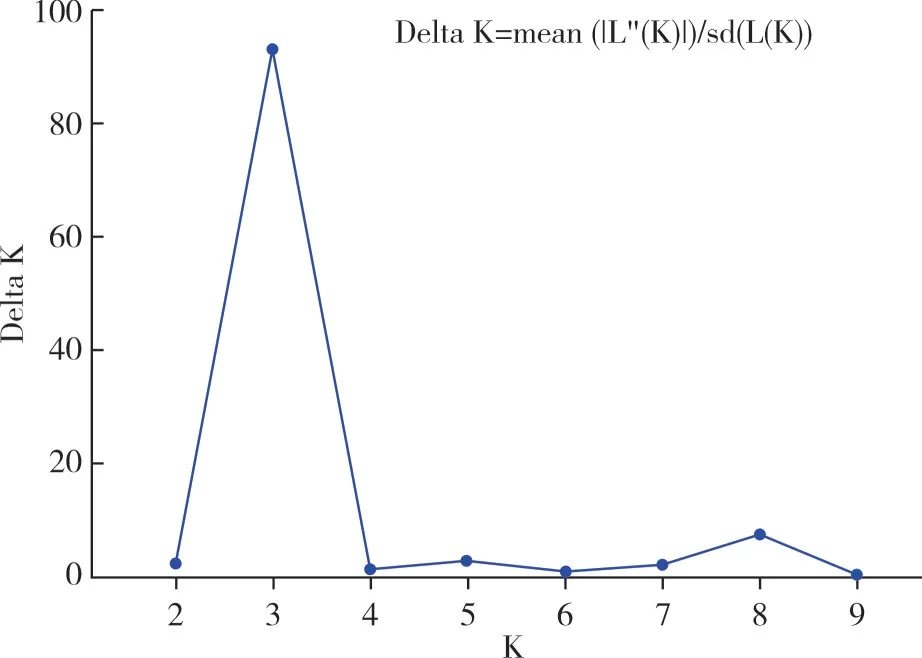
图4 似然对数的变化折线图(ΔK)Fig.4 Line chart of ΔK with change of K value

图5 105份大豆材料群体遗传结构Fig.5 Population structure of 105 accessions

表6 大豆材料遗传结构分布Table 6 Distribution of population structure
2.6 群体主坐标分析
分时期亚群的主坐标分析(图6)表明,第一、二、三主成分分别解释了4.12%、3.61%和2.90%的总变异。相对较后时期(1991-2000 和2001-2004)的材料基本聚集在同一区域,只有少数品种在其他区域,而1963-1980 分布于不同区域。说明大豆材料具有一定的时期特征,早期的大豆材料与相对较后两时期的大豆材料有一定的遗传差异,而这两时期的遗传差异小,亲缘关系较近。
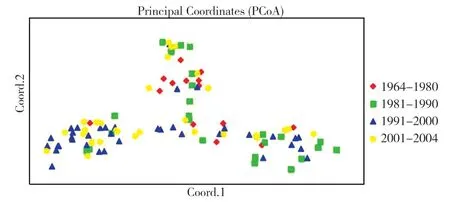
图6 主坐标分析Fig.6 Principal coordinates analysis of subgroups
3 讨论
3.1 QTL相关的SSR位点多态性分析
大豆种质资源研究中不少功能分子标记在遗传变异或多样性分析中都有广泛应用。实验室早期选择ScoT[25]、EST-SSR[26]等标记研究黄淮海和南方地区大豆的遗传多样性,平均位点的等位变异数为8.81 和4.81。SSR 标记与这些功能分子标记相比,等位变异数为11.54,多态性更高,检测更灵敏。此外,熊冬金等[18]也曾用161 个SSR 分子标记对209份大豆育成品种遗传多样性分析,多态性信息含量平均0.819,低于本研究中的0.836。这一结果与通常认为的样本越大、标记数量越多则遗传多样性越高这一结论不符。通过比较发现,本研究中SSR 位点均是与QTL 相关且基因型丰富的位点,平均等位变异数11.54。而熊冬金等研究中SSR 位点数目多,包含与QTL 相关的SSR 位点141 个和QTL 无关的SSR 位点20 个。QTL 无关的SSR 位点大多基因型多态性简单,与QTL 相关位点放一起,平均的等位变异数11.07,影响整体的遗传多样性水平,因此低于本实验的群体遗传多样性。
通过Soybase网站查询各位点信息,每个位点皆与几个不同QTL 相关联。例如基因型丰富的satt009 位点与大豆产量、开花数、种子异黄酮、种子硬度等10余个QTL性状相关。在后期驯化培育中,QTL 连锁区位点的基因型有自身局限性和连锁阻力,理应遗传多样性小。但在育种过程中,育种者极大地保存了位点自身的遗传多样性,在探索基因型时引入新的多样性,增加品种的低频多样性也是长期受驯化和近交的结果[27]。
3.2 育成品种大豆资源的遗传多样性分析
本研究选择99 个QTL 相关的SSR 标记位点,对105 份大豆育成品种进行分时期亚群的遗传多样性分析,Nei's基因多样性范围为0.628~0.839,平均0.774。多态性信息含量0.562~0.820,平均0.742。时期亚群大豆育成品种遗传多样性高低顺序为1991-2000>2001-2004>1981-1990>1963-1980。时期亚群相似性系数聚类分析表示,与遗传多样性最高的1991-2000时期的亚群间遗传距离大小顺序为1963-1980(0.387)>1981-1990(0.236)>2001-2004(0.197)。说明1963-1980 与1991-2000 两时期的亲缘关系最远,而2001-2004 与1991-2000 两时期的亲缘关系最近。大豆分时期亚群的遗传多样性呈现先增后减的趋势与改革开放后我国从国外引入大量的大豆材料有关,我国大豆育成品种遗传基础有所拓宽。而在现有的遗传基础上再增加大豆遗传多样性,需在今后的育种工作中进一步拓宽亲本来源。
分子方差分析表明大豆材料99%为亚群内变异,仅1%为亚群间变异。亚种群内的高变异象征着对经济上重要的性状更频繁地被选择,以致各主要家族的祖先亲本对该品种的遗传贡献值有所差异。育种亲本越复杂的品种,有多个主要家族的遗传贡献,多态性位点的等位变异数增加的几率越大,品种的遗传多样性越高。然而,育成品种所利用的种质资源还是相对较狭窄,一方面种质育种不能囊括优质亲本所有的优势表型,另一方面这与我国引进外来种质资源无法满足育种需求不无关系。其次大豆材料为高度纯合的育成品种也部分影响着遗传变异结果。
3.3 育成品种大豆的群体遗传结构分析
本研究对3 个主要家族的105 份大豆材料的聚类分析和遗传结构分析都认为它们明显分为3 类群,且两方面的分析结果高度一致,也与Dong 等[28]利用53 对SSR 标记对100 份菜豆品种的UPGMA 亚群与结构亚群分析结果具有高度一致性。这些群体的聚类表现出极大的一致性,与大豆育成品种在主要家族系谱中代数及各时期亚群的遗传基础粗略相关。例如类群2 中大豆育成品种在A034、A133和A231 家族系谱中代数在3 代以前,类群1 中大豆品种则在3 个主要家族中代数靠后,主要为4 代以后品种。类群2 在亲缘关系上更贴近3 个主要家族,而类群1 与3 个主要家族亲缘关系不如类群2,其他的遗传背景影响更大。
从各时期亚群的遗传基础来看,且各亚群有其聚类的偏好性,遗传基础相似的亚群有聚在同一类群中的趋势,例如1991-2000 和2001-2004 两个时期内的大豆材料遗传相似性高,所以主要集中在类群1中,1963-1980和上述两时期大豆材料的遗传相似性低,则偏向于集中在类群2 中。关于大豆育成品种遗传组分受到分时期遗传基础影响这一结论有待明确,目前尚无文献报道,但普遍认为有生态区划分遗传基础[29,30]。
聚类后出现的相同来源的种质分别聚类在不同类群中或不同来源的种质相聚在同一类群的现象,常见于群体遗传结构的研究中[31,32]。这与种质的遗传背景和种质间遗传差异相关。不同来源的种质可能遗传背景相似,相同来源的种质也可能遗传差异较大。说明大豆育种不能简单的异地杂交育种,考虑种质间的遗传相似性才能有效利用大豆种质资源。
4 结论
利用99 个与大豆QTL 性状相关的SSR 位点,对黄淮海和南方产区的105份大豆育成品种进行遗传多样性和群体结构分析。按品种育成时期将群体划分为4 个亚群,研究表明不同时期内大豆品种基因交流频繁,UPGMA 聚类和群体遗传结构分析均将105 份材料分为3 个类群,且结果高度一致,大豆育成品种遗传多样性水平具有一定的时期特征,总体呈递增趋势。
