不同剩余采食量水平的安格斯牛下丘脑单核苷酸多态性和可变剪接分析
2022-03-11周小南马应杨朝云丁燕玲张岩峰赵志艳康晓龙
周小南, 马应, 杨朝云, 丁燕玲, 张岩峰, 赵志艳, 康晓龙
(宁夏大学农学院,宁夏 银川 750021)
剩余采食量(residual feed intake, RFI)是指实际采食量和预期的饲料需要量的差值。RFI水平与饲料转化率和干物质采食量显著相关[1]。当RFI的数值为负数时,表明畜禽实际采食量低于预期采食量,该畜禽为高饲料利用率动物,反之则为低饲料利用率动物。RFI水平不仅反映了个体由遗传背景所决定的代谢差异,也校正了试验个体的代谢体质量。杨朝云等[2]对2个RFI水平(高/低)的安格斯牛与生长性状的相关性分析发现,低RFI水平组的安格斯牛比高RFI水平组节约8.8%的饲料。CONNOR等[3]研究发现,当低RFI水平组肉牛的饲料摄入量减少10%~12%,此时温室气体排放量减少25%~30%,粪便中的养分损失减少15%~17%。单核苷酸多态性(single nucleotide polymorphism site, SNPs)是指单核苷酸碱基变异,由转换(C/T或G/A)或颠换(C/G、C/A或T/A、T/G)引起[4]。近年来,许多研究利用表达数据进行表型相关表达基因的SNP分析[5],鉴定差异表达基因单核苷酸变异的潜在作用,为进一步整合表达数据与基因组信息以及分析其与表型相关性提供了参考。SAATCHI等[6]通过对4个独立肉牛群体进行全基因组关联研究和功能分析,鉴定出影响肉牛生长的主效数量性状基因座(quantitative trait loci,QTL)。在植物方面,王赛等[7]基于SNP遗传图谱定位玉米雄穗分枝数和主轴长QTLs。研究表明,基因的不同剪接方式也会影响机体发育和生物功能,可变剪接(alternative splicing, AS)是Pre-mRNA通过不同的剪接方式形成序列不同的mRNA剪接异构体的过程,其中单个基因通过AS产生多个mRNA异构体和蛋白质亚型,从而增加蛋白质多样性和复杂性[8]。周扬[9]对不同发育阶段和不同性别秦川牛的皮下脂肪组织内AS相关基因表达特征进行分析发现,有28%的基因在牛脂肪组织中出现AS事件。在植物中,AS相关基因参与调控植物的抗性特征[10]。下丘脑作为动物采食量调节中心,存在复杂的调节食欲和能量平衡的神经元网络[11]。因此,本研究对安格斯牛下丘脑组织进行高通量测序,分析不同RFI水平安格斯牛下丘脑组织的SNPs和表达基因对应的AS事件,梳理下丘脑中由于RFI水平不同而发生的AS事件及AS相关基因的功能和参与的通路,以期挖掘AS在肉牛剩余采食量表型调控中的潜在生物学作用,为后续解析肉牛饲料利用效率相关的遗传学基础提供理论支撑。
1 材料与方法
1.1 试验动物
试验动物选自宁夏地区某养殖场的安格斯牛,初始体质量为(266.7±35.7)kg,所有安格斯牛(n=30)均按照标准程序饲养,饲喂时期为81 d。饲养结束后,依据标准方法计算RFI水平,选择RFI水平最高(high residual feed intake, HRFI)和水平最低(low residual feed intake, LRFI)个体各5头,分别编号为H1、H2、H3、H4、H5和L1、L2、L3、L4、L5,试验牛只颈部放血屠宰开颅后,立即从下丘脑组织收集1 cm3的组织,并用生理盐水冲洗干净,剪碎后装入无酶的冻存管,于-80 ℃保存备用。试验遵循宁夏大学动物保护委员会的指导原则,所有组织均按照无害化处理方法和原则进行科学处理。
1.2 RNA的提取和测序
取适量下丘脑组织样品,采用TRIzol法分别提取HRFI组和LRFI组样本的总RNA,将提取到的RNA以质量分数1%的琼脂糖凝胶进行检测,利用 Nano Photometer®分光光度计测定OD260/280及OD260/230比值,将OD260/280介于1.9和2.0之间的RNA样品用于RNA测序。构建cDNA文库,将构建合格的不同文库按照有效浓度及目标下机数据量的需求富集后进行Illumina测序,RNA测序由北京诺禾致源科技股份有限公司完成。
1.3 测序数据分析
对测序得到的原始数据(raw reads)进行质控,过滤去除reads中的接头序列、带N碱基以及低质量reads序列,得到clean reads。
1.4 SNP及可变剪接分析
将比对结果进行染色体坐标排序、去掉重复的reads等处理后,通过变异检测软件GATK2分析各个样品的SNP位点,同时对原始结果进行过滤。使用rMATS软件对差异表达的基因进行AS分析,以错误发现率(false discovery rate,FDR)<0.05作为差异表达AS事件的筛选标准。经rMATS软件分析包括5种可变剪接类型(图1),分别为外显子跳跃(skipped exon,SE)、5’端可变剪接(alternative 5’ splice site,A5SS)、3’端可变剪接(alternative 3’ splice site,A3SS)、外显子互斥(mutually exclusive exon,MXE)和内含子保留(retained intron,RI)。
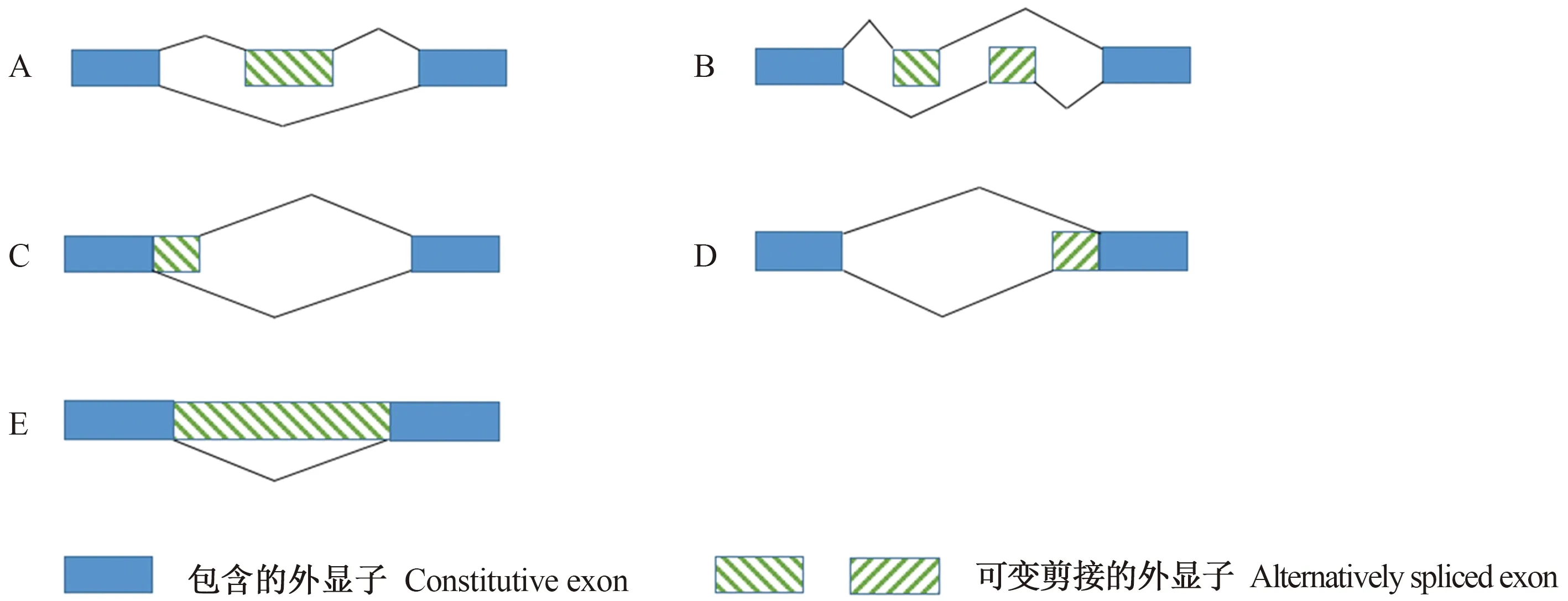
注:A,外显子跳跃;B,外显子互斥;C,5′端可变剪接;D,3′端可变剪接;E,内含子保留。
1.5 差异表达AS相关基因功能富集分析
为确定差异表达AS相关基因的功能,采用在线分析软件g:profiler (https://biit.cs.ut.ee/gprofiler/)进行基因本体(gene ontology, GO)通路富集分析,同时利用DAVID (https://david.ncifcrf.gov/)功能注释进行京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)通路富集分析,显著性水平设置为P<0.05。
1.6 实时荧光定量PCR验证试验结果
为验证转录测序的可靠性,随机挑选7个差异表达AS相关基因(PARP2、ITGB1、BPIFBI、LRP11、USP34、MGAT1和KCNN2)进行实时荧光定量PCR(quantitative real-time PCR,qRT-PCR),以GAPDH基因作为内参基因,引物委托上海生工生物工程有限公司合成(表1)。配置20 μL的反应体系: TB Green Taq II 10 μL,Rox Dye II 0.4 μL,cDNA 2 μL,上下游引物各0.8 μL,dd H2O 6 μL。反应程序:95 ℃ 预变性 4 min;95 ℃变性 10 s,最佳退火温度30 s,72 ℃延伸10 s,共39个循环;65 ℃ 5 s,95 ℃ 5 s。使用 2-△△Ct法对基因的表达量进行标准化计算。差异性分析使用SPSS 2.2软件,以P<0.05 为差异显著判断标准。

表1 荧光定量PCR验证引物序列Table 1 Primer sequences verified by fluorescence quantitative PCR
2 结果与分析
2.1 高/低RFI水平安格斯牛下丘脑组织转录组测序数据质量分析
对HRFI组和LRFI组的安格斯牛下丘脑组织RNA样品进行测序,结果如表2所示。所得的raw reads在145 924 188~159 000 376之间,所有样本平均clean reads比例高于88.48%,Q30碱基比例介于89.48%~93%之间,表明文库构建成功且转录组测序得到的结果质量良好,测序数据可靠,可用于后续分析。
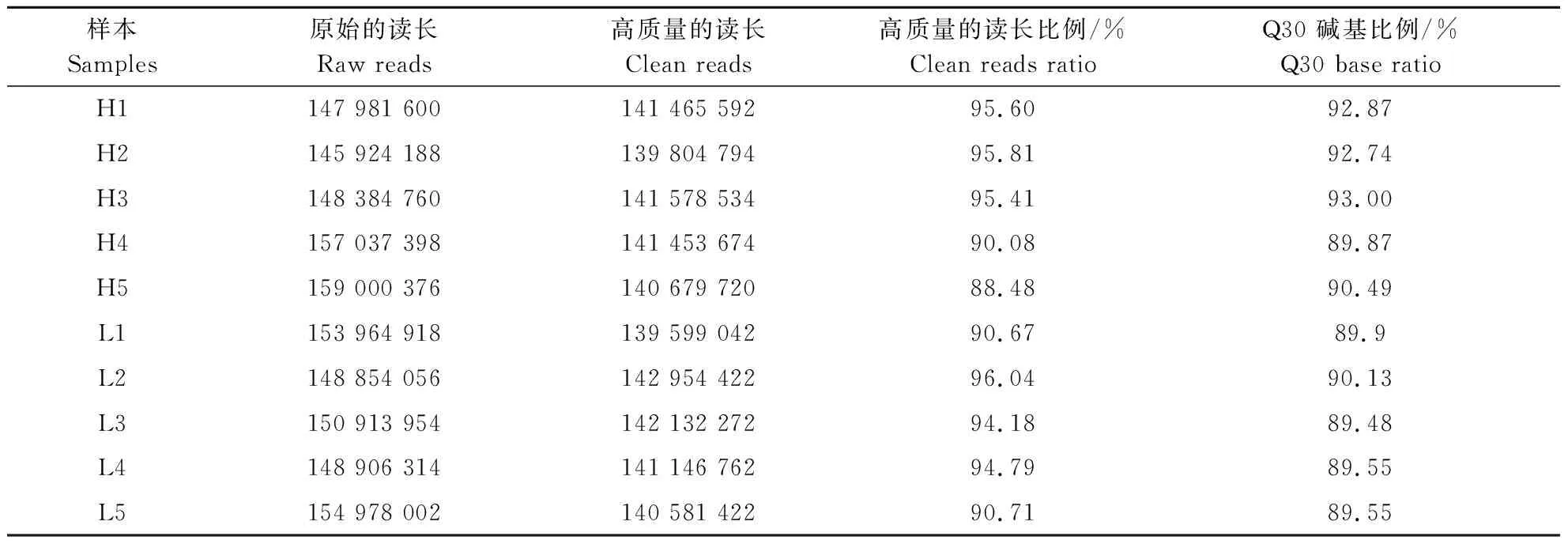
表2 高/低RFI水平安格斯牛下丘脑组织转录组测序数据评估统计Table 2 Statistics of sequencing data evaluation of Angus cattle with high/low RFI levels
2.2 高/低RFI水平安格斯牛SNP位点统计分析
使用变异检测软件GATK2检测各个样品潜在的SNP位点,同时进行SNP calling和InDel calling分析,结果如表3所示。根据测到的SNP位点,统计每组的突变类型的频率,对2个RFI水平(高/低)的安格斯牛10个样本进行SNP突变类型统计,结果如图2—图4所示。10个样本中,C/T和A/G的突变频率最高。所有样品中检测到的SNP数量为48 227~1 000 092个。分析2个RFI水平(高/低)的样本,发生频率最高的是转换型SNP,比例约为71%,而颠换型SNP位点比例约为29%,转换和颠换的比例为2.45,所有样品中发生InDel突变事件的数目为48 276~87 677个。
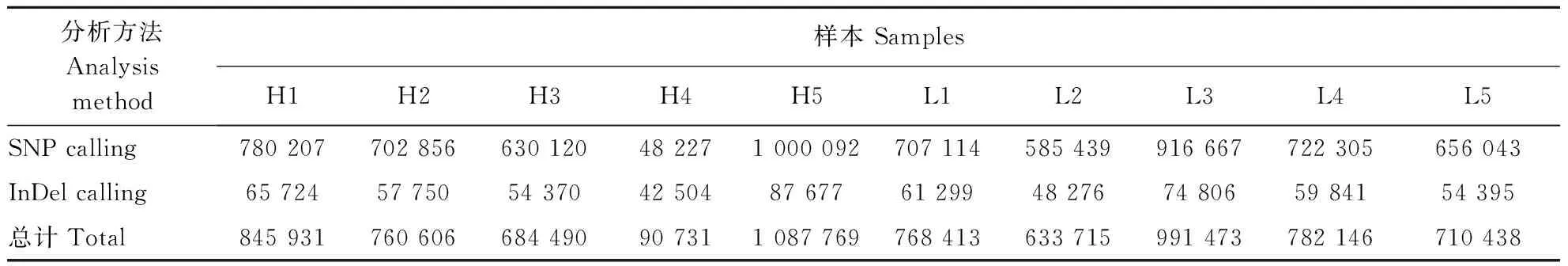
表3 高/低RFI水平安格斯牛SNP位点分析结果Table 3 Analysis results of SNP locus of Angus cattle with high/low RFI levels
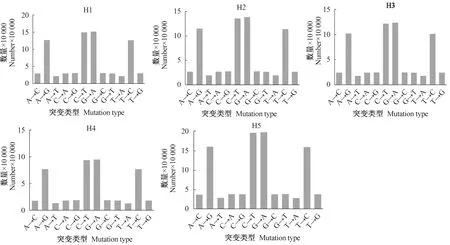
图2 高RFI水平安格斯牛SNP突变频率分布
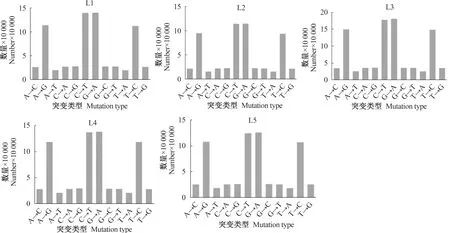
图3 低RFI水平安格斯牛SNP突变频率分布
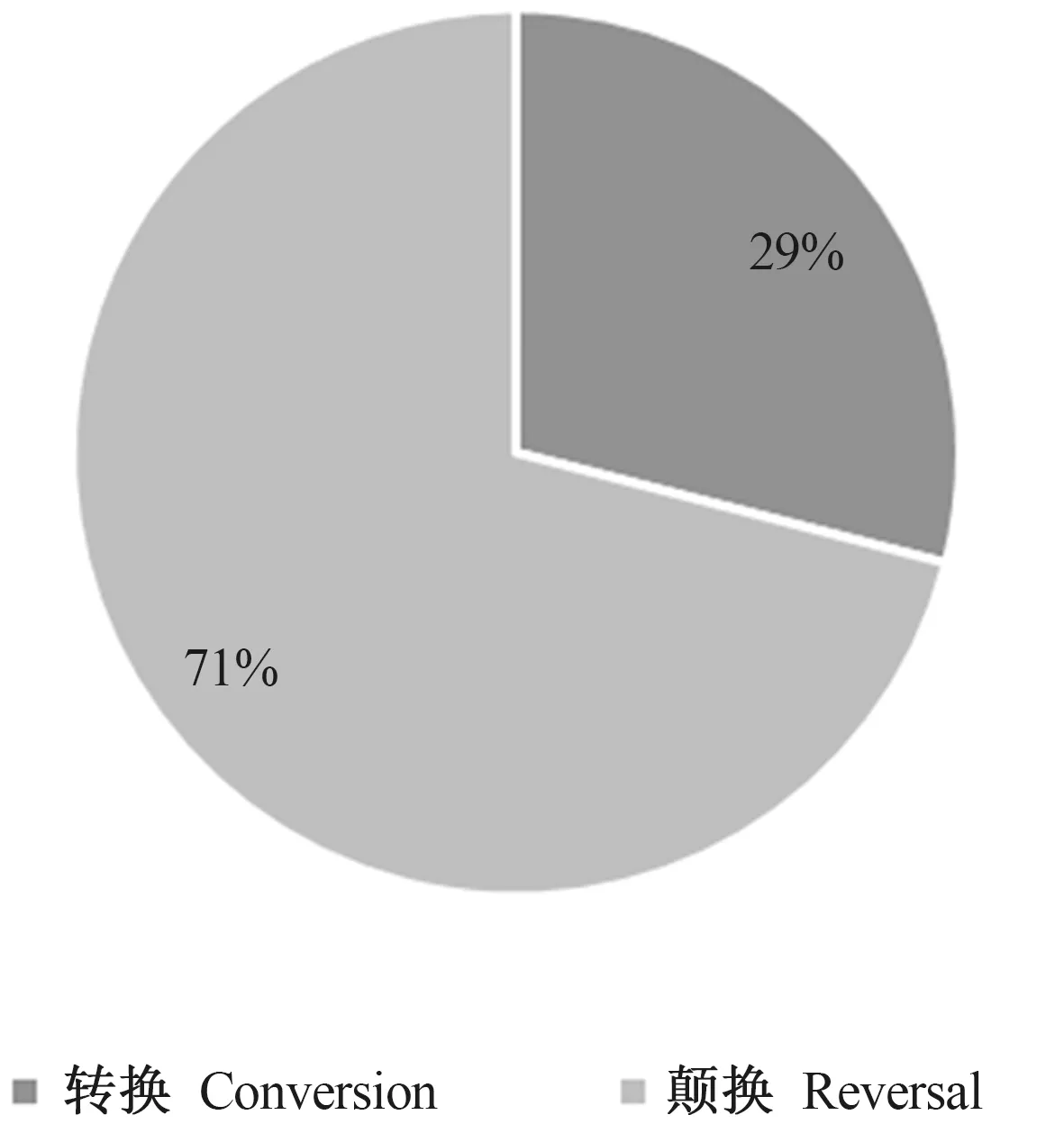
图4 高/低RFI水平安格斯牛转换和颠换SNP类型频率比例
2.3 高/低RFI水平安格斯牛AS差异分析
采用rMATS软件对HFRI组和LRFI组进行AS差异分析,共有12 385个基因对应35 463个AS事件,鉴定出差异表达AS事件498个(表4)。2个RFI水平(高/低)发生的AS事件中,SE类型最普遍,共30 262个,占85.33%;其次是MXE类型共5 057个,占14.26%;A5SS、A3SS和RI类型较少,共占0.41%,分别为33、78和33个。进一步对AS事件进行显著性分析发现,2个RFI水平(高/低)之间差异表达SE类型有416个,包括199个上调和217个下调。此外,差异表达MXE类型有79个,包括43个上调和36个下调;差异表达A3SS类型有1个下调;差异表达RI事件有2个,包括1个上调和1个下调;没有差异表达A5SS类型。对发生AS事件的外显子长度进行分析发现(表5),RI类型的外显子长度最长,平均数为887.9 bp,中位数为512.0 bp;另外4种类型的AS事件外显子长度较短,平均数在136.9~186.0 bp之间。
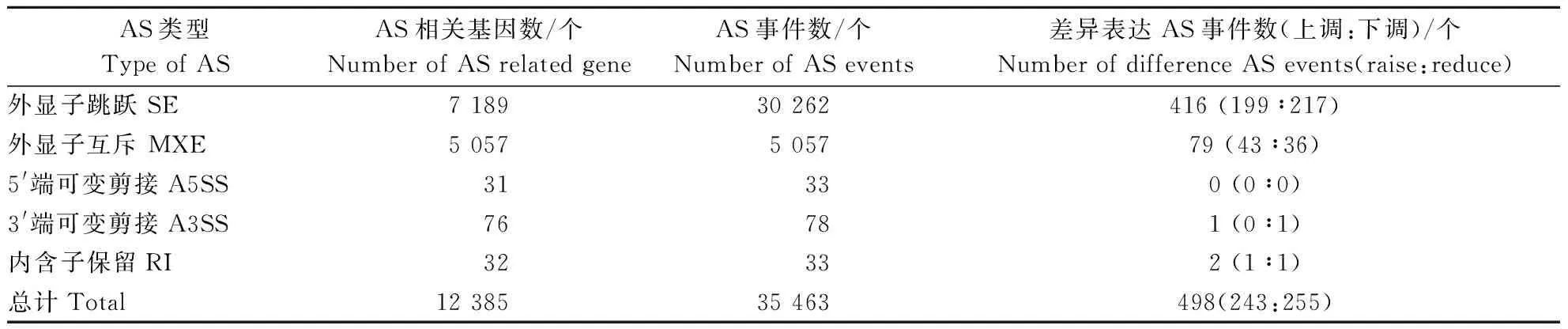
表4 高/低RFI水平安格斯牛AS差异分析统计Table 4 Differential AS statistics of Angus cattle with high/low RFI levels
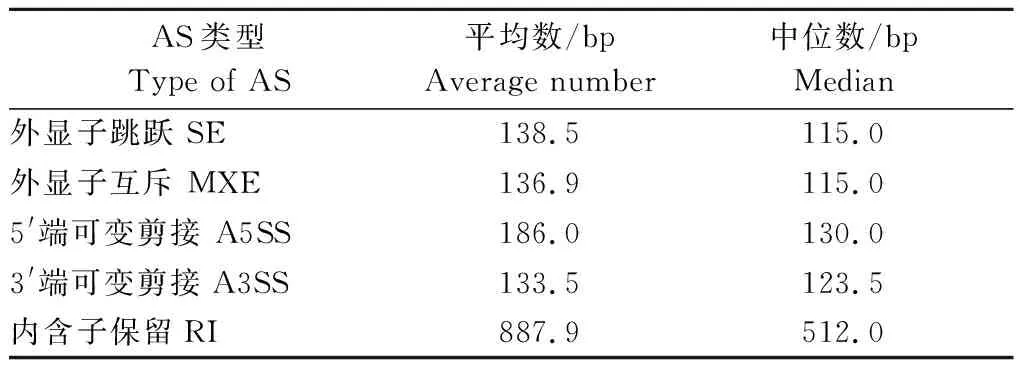
表5 高/低RFI水平安格斯牛AS外显子长度Table 5 AS exon length of Angus cattle with high/low RFI levels
2.4 高/低RFI水平安格斯牛差异表达AS相关基因GO和KEGG通路富集分析
对获得的高/低RFI水平安格斯牛差异表达AS相关基因进行GO通路富集分析(图5),共检索到34条GO条目,包括分子功能6条,细胞组成11条,生物学过程17条。在分子功能中有35个差异表达AS相关基因富集在ATP结合方面,生物学过程中分别有11个和8个差异表达AS相关基因富集在光感受器连接体和突触纤毛组装。对显著差异表达的AS相关基因进行KEGG通路富集分析发现(图6),共有82个基因显著富集在13条通路上,包括胰岛素信号通路、催乳激素信号通路、胆碱代谢、哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)信号通路、环磷酸腺苷(cyclic adenosine monophosphate,cAMP)信号通路、酪氨酸激酶受体(erbB)和昼夜节律等。

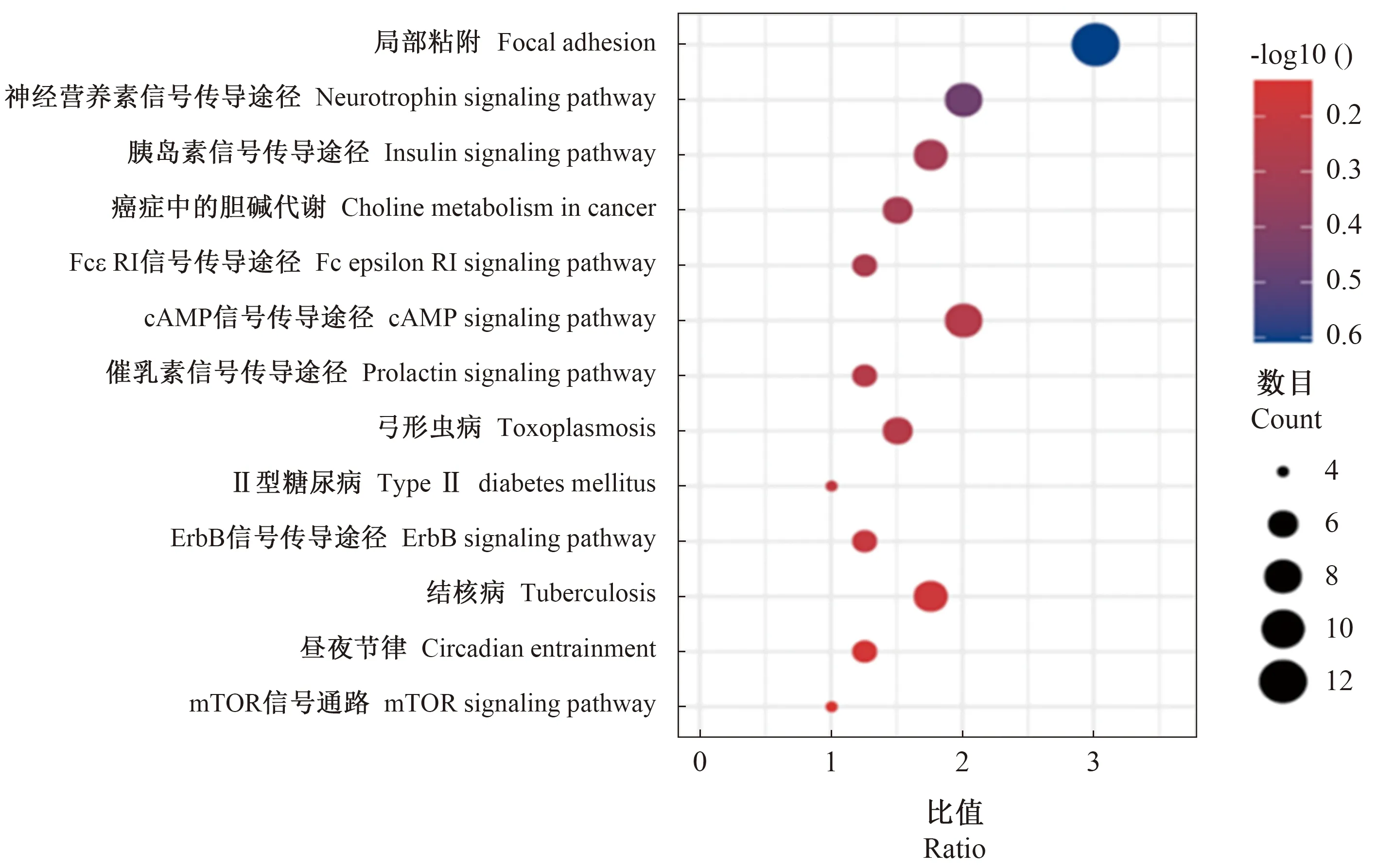
图6 高/低RFI水平安格斯牛差异表达基因KEGG通路富集散点图Fig.6 Enrichment distribution point diagram of differentially expressed gene KEGG pathway of Angus cattle with high/low RFI levels
2.5 差异表达AS相关基因测序结果验证
通过qRT-PCR验证PARP2、ITGB1、BPIFBI、LPR11、USP34、MGAT1和KCNN2共7个差异表达AS相关基因的表达情况(图7)。经qRT-PCR得到的基因相对表达量和RNA-seq测序结果的差异倍数基本一致,通过计算2个结果的皮尔逊相关性,相关系数r=0.95,表明RNA-seq数据相对可靠。
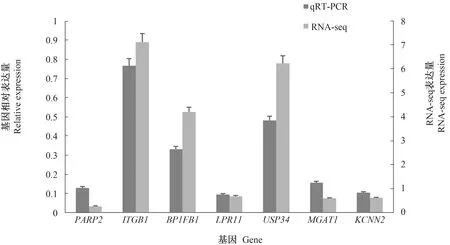
图7 qRT-PCR 对RNA-Seq检测出的差异表达基因验证
3 结论与讨论
下丘脑内存在复杂的调节食欲和能量平衡的神经元网络[12],通过促进或抑制摄食神经肽的合成作用调节采食量[13]。本研究通过对安格斯牛下丘脑的转录组测序分析,分析不同RFI水平的AS相关基因。这些基因均与食欲调节相关,如阿黑皮素原(proopiomelanocortin, POMC)相关基因和生长激素释放激素受体(growth hormone-releasing hormone receptor , GHRHR)相关基因均发生了AS事件[14-15]。本研究通过对2个RFI水平(高/低)安格斯牛下丘脑组织样品测序数据进行SNP检测分析发现,A/T和C/G的突变频率最高,约占总数的71%;而颠换型SNP位点比例为29%,转换和颠换的比例为2.45。这与其他动物如鸭、鸡和牛的研究结果一致[16-18]。SNP是基因组中最常见的遗传变异之一,但是转换偏差现象使得每个SNP 位点发生变异形式由4种变为只有2种,即转换型和颠换型,且比例为1∶2[19-21]。该现象可能是物种长期进化的结果,且这种变异在一定程度上有利于保持编码蛋白原有的结构,减少不利突变的概率[22]。因此,在SNP数据集中观察到的转换和颠换的比例相对较高。此外,本研究结果显示,共有12 385个基因发生35 463个AS事件,这也说明了单个基因通过AS可以形成不同的mRNA剪接异构体。对AS事件的类型进行统计,其中SE类型比例最高,达85.33%;其次是MXE类型,比例为14.30%。王旭平等[23]对番鸭下丘脑转录组数据分析发现,有5 034个基因发生7 528个AS事件,其中SE类型占比92.76%,而A5SS类型和RI类型仅占0.027%。鲍晶晶等[24]对绵阳不同发育时期的背最长肌组织的AS事件进行统计,其中SE类型比例最高,为70.69%,这与本试验的研究结果一致。本研究统计了各类型AS事件可变外显子的长度,其中发生频率较高的SE和MXE类型的AS事件的外显子长度平均数较小,而A5SS和RI类型的可变外显子平均长度较大,这表明较小的外显子更容易发生AS事件,该结果与STAMM等[25]的研究结果一致。
在本研究中,从2个RFI水平(高/低)安格斯牛中检测出35 463个AS事件,将差异表达的AS相关基因比对到KEGG数据库中,共筛选到13条AS相关基因显著富集的通路。其中,显著富集基因最多的是胰岛素信号通路,其次是催乳激素信号通路,还有较多的基因富集在胆碱代谢、mTOR信号通路、cAMP信号通路、erbB信号通路和昼夜节律等通路,这些通路广泛参与调节能量代谢[26-28]。其中,mTOR信号通路在细胞的增殖、生长和分化过程起到重要的调控作用[29-30]。研究表明,mTOR可监测下丘脑中营养和激素传输信号,当外界刺激增强下丘脑mTOR信号通路活性时能够抑制动物的采食,但当在脑室注射mTOR信号通路的抑制剂则显著增加禁食动物的短期采食量[31]。同时,本研究结果显示,有8个差异表达AS相关基因显著富集于cAMP信号通路。cAMP作为第二信使在脂肪分解中具有重要作用,其表达受Ca2+水平的影响。在猪脂肪组织中,cAMP磷酸二酯酶作为中间体,影响脂解过程[28]。此外,喂养治疗试验证实了肥胖和cAMP途径之间的可能联系[32]。本研究结果揭示AS事件及表达基因的SNPs在安格斯牛采食量调控中的潜在功能,表明不同剩余采食量水平的差异表达AS相关基因可能通过能量代谢相关的信号通路和影响食欲的通路来调节能量代谢,进而参与肉牛采食量性状,但具体生物学机制有待进一步的深入研究与探讨。
