豫农黑猪体尺性状遗传参数估计的模型研究
2022-03-11李聪段栋栋李梦雨周本丽李秀领王克君韩雪蕾王献伟乔瑞敏李凯李新建
李聪, 段栋栋, 李梦雨, 周本丽, 李秀领, 王克君, 韩雪蕾, 王献伟, 乔瑞敏, 李凯, 李新建
(1.河南农业大学动物科技学院,河南 郑州 450046;2.河南省畜牧总站,河南 郑州 450002)
豫农黑猪是利用中国优质地方猪中的南阳黑猪、莱芜黑猪、二花脸猪和外来品种杜洛克猪杂交选育的新品系,具有繁殖力高、耐粗饲、抗病力强和肉质好等优良特性[1-2],因此,对其经济性状的选择十分重要。对数量性状遗传参数的估计是豫农黑猪选育过程中制订育种计划的前提,主要包含遗传力、重复力、遗传相关以及亲缘相关等。传统估计方法采用最佳线性无偏预测(best linear unbiased prediction, BLUP),即利用个体的表型和系谱信息估计育种值,但该方法受系谱记录准确性的影响较大。随着分子标记在育种领域的发展,利用基因组信息矫正个体间亲缘关系的基因组最佳线性无偏预测(genomic best linear unbiased prediction, GBLUP)已应用到生产中[3]。利用BLUP和GBLUP方法对猪的繁殖、生长以及重要经济性状进行遗传参数估计已经是育种领域研究的热点与重点[4-5]。OGAWA等[6]使用单性状动物模型估计了纯种杜洛克的体尺性状遗传参数,体长、体高和胸围的遗传力为0.38、0.30和0.27。NIKKIL等[7]利用混合线性模型估计长白猪的体高、体深和体宽性状,遗传力分别为0.26、0.31和0.24。体尺性状反映了家畜的载肉量和生长发育,直接影响养猪业的经济效益[8]。受动物遗传背景差异的影响,体尺性状的遗传参数往往具有群体特异性。近交系数是衡量群体近交水平的特异化数值,在计算遗传参数的模型中添加近交系数更能准确反映出群体的真实状态,提高模型预测的准确性[9]。因此,本研究以702头豫农黑猪的系谱记录、Illumina PorcineSNP50 BeadChip(50 K)育种芯片数据和生长记录,构建3个单性状动物模型,并比较了添加近交系数对遗传参数的影响,以期提高豫农黑猪体尺性状遗传参数估计的准确性,优化该群体的遗传评估体系,加快群体的遗传进展。
1 材料与方法
1.1 试验动物
试验动物选自河南省三门峡牧阳农牧有限公司,采用260日龄、体重在95~105 kg的豫农黑猪702头,其中母猪635头,公猪67头;春、夏、秋、冬季分别出生114、330、211、47头;共9个批次,分别来自牧阳农牧的3个育种核心场,其中核心1区237头,核心2区358头,核心3区107头。试验群体按不同生理阶段依照中华人民共和国农业行业标准《猪饲养标准》(NY/T 65—2004)[10]进行配比日粮营养,猪场均按照统一流程饲养管理。
1.2 体尺性状测定
采集702头个体的系谱信息,根据王明宇等[11]的方法测定体尺性状,具体指标包括体长(body length,BL)、体高(body height,BH)、胸围(chest circumference,CC)、管围(cannon bone circumference,CBC)、腿臀围(legs buttocks circumference,LBC)、背膘厚(backfat thickness,BF)和眼肌深度(loin muscle depth,LMD)。对各性状的测定记录,选定“平均值±3倍标准差”作为正常值范围进行筛选,并计算各性状的基本统计量。
1.3 基因组DNA提取、基因分型及数据质控
采集耳组织,使用DNA提取试剂盒(DP304,天根生化科技有限公司)提取DNA,并使用琼脂糖凝胶电泳和微量核酸定量仪(Nanodrop 2000)检测质量和浓度。合格的基因组DNA利用Illumina PorcineSNP50 BeadChip(50 K)进行全基因组芯片分型。使用 PLINK(V1.90)软件[12]对芯片数据进行质量控制。具体参数:个体基因型检出率>90%,标记基因型检出率>95%,最小等位基因频率>1%,最小哈代温伯格平衡为10-6,位于常染色体。质控后剩余702头个体38 497个单核苷酸多态性位点,采用隐马尔科夫模型算法,在 BEAGLE(V5.2)软件[13]中执行对缺失基因型的填充。
1.4 体尺性状影响因素
本研究主要考虑影响体尺性状的环境因素为性别、出生季节、出生场区、批次和近交系数。使用PLINK(V1.90)软件对基因组纯合区域(region of homozygosity, ROH)进行检测。ROH是指子代与父母代相同单倍型中纯合基因型的连续片段。使用参数如下:20个SNP的滑动窗口沿染色体滑动,每个滑动窗口的杂合子个数不超过1个。ROH参数:最小纯合片段>10 kb,SNP个数/ROH个数>20,SNP的最大间隔>1 000 kb。
基于ROH的基因组近交系数(FROH):
(1)
式中:LROHi是个体i的ROH长度;Lauto是芯片中SNP覆盖的常染色体基因组长度。
1.5 体尺性状遗传参数估计模型
使用R软件(V3.6.0)处理数据,将原始数据整理成表型文件和系谱文件,表型文件按照个体号、父号、母号、固定效应(对表型具有显著影响的因素)、性状观测值排序,系谱文件需要包含子代和父母代的系谱记录。试验采用3个模型估计豫农黑猪体尺性状的遗传参数:模型1,利用系谱的BLUP法;模型2,利用基因组信息的GBLUP法;模型3,在模型2基础上额外考虑了个体基因组近交系数作为协变量。
模型1:y=Xb+V1a1+V2l+e
(2)
模型2:y=Xb+V1a2+V2l+e
(3)
模型3:y=Xb+V1a3+V2l+Wf+e
(4)
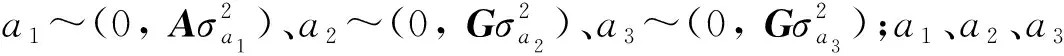
1.6 体尺性状遗传参数估计的准确性
本研究使用5折交叉验证法(5-fold cross validation):将702头样本数据分成5份,轮流将其中4份作为训练样本,1份作为验证样本。试验以10次5折交叉验证结果的均值用来评价模型估计遗传参数的准确性,计算公式如下:
(5)
式中:acc为模型估计遗传参数的准确性;y为表型观测值;h2为遗传力;(G)EBV分为2种情况,当不使用基因型数据时(模型1)为EBV即估计育种值(estimated breeding value),当使用基因型数据时(模型2和3)为GEBV即基因组估计育种值(genomic estimated breeding value);corr为(基因组)估计育种值与表型观测值的相关系数。
2 结果与分析
2.1 豫农黑猪体尺性状基本表型参数
702头豫农黑猪体尺性状表型参数如表1所示。体尺性状的标准差较小,表明数据的离散程度较为集中,群体体型的均匀度较好。体长、体高、胸围、管围、腿臀围的变异系数均在10.00%以下,其中体高的变异系数最低,为6.06%,表明该群体具有稳定的遗传基础;背膘厚和眼肌深度的变异系数较高,为28.69%和20.21%,表明针对背膘厚和眼肌深度性状,仍有较大的选育空间。

表1 豫农黑猪体尺性状统计Table 1 Statistics of body measurements traits in Yu’nong black pigs
2.2 豫农黑猪体尺性状表型和遗传相关
体尺性状间的表型和遗传相关性如表2所示。背膘厚和体高,眼肌深度和体长、体高性状表现为负遗传相关,其余各性状间均呈正遗传相关。体长和胸围的遗传相关最高,为0.985;眼肌深度和腿臀围的遗传相关最低,为0.070。体高与背膘厚,眼肌深度与体长、体高和腿臀围的遗传相关不显著,其余性状间呈不同显著水平的遗传相关。除背膘厚和体高性状表现为负表型相关,其余各性状间的表型相关均为正相关。胸围和背膘厚的表型相关最高,为0.576;体高和眼肌深度的表型相关最低,为0.007。除了体长和眼肌深度,体高和管围、背膘厚、眼肌深度,腿臀围和背膘厚、眼肌深度间的表型相关不显著,其余性状间呈不同显著水平的表型相关。

表2 豫农黑猪体尺性状间的相关性分析Table 2 Correlation analysis among body measurements traits in Yu’nong black pigs
2.3 豫农黑猪体尺性状影响因素的显著性检验
本研究通过R(3.6.0)的广义线性模型函数(generalized linear models, GLM)检验各因子对豫农黑猪体尺性状是否有显著影响,检验结果如表3所示。性别对眼肌深度的影响显著(P<0.05),对管围和背膘厚的影响极显著(P<0.01)。出生季节对管围的影响显著(P<0.05),对眼肌深度的影响极显著(P<0.01)。出生场区对腿臀围、背膘厚和眼肌深度的影响极显著(P<0.01)。批次对眼肌深度的影响显著(P<0.05),对体高、胸围、管围、腿臀围和背膘厚的影响极显著(P<0.01)。

表3 豫农黑猪体尺性状影响因素的显著性检验结果Table 3 Significance test results of factors affecting body measurements traits in Yu’nong black pigs
2.4 豫农黑猪体尺性状遗传力估计
本研究基于BLUP和GBLUP法估计了豫农黑猪体尺性状的方差组分和遗传力,结果如表4所示。BLUP法估计的体长为低遗传力性状,体高和腿臀围为中遗传力性状,胸围、管围、背膘厚和眼肌深度为高遗传力性状。GBLUP法估计的体长、胸围和管围为中遗传力性状,体高、腿臀围、背膘厚和眼肌深度为高遗传力性状。BLUP和GBLUP法估计的体长、体高、胸围、管围和腿臀围性状的遗传力存在差异,背膘厚和眼肌深度均为高遗传力性状。此时,模型3估计体高、腿臀围、背膘厚和眼肌深度的遗传力为0.566、0.302、0.467和0.652,属于高遗传力性状;体长、胸围和管围的遗传力为0.152、0.122和0.255,属于中遗传力性状。

表4 不同模型估计豫农黑猪体尺性状的方差组分和遗传力Table 4 Variance components and heritabilities estimation of body measurements traits in Yu’nong black pigs by different models
2.5 不同模型估计豫农黑猪体尺性状遗传参数的准确性
不同模型对豫农黑猪体尺性状进行遗传参数估计的准确性如表5所示。对于体长和背膘厚,模型2的准确性分别比模型1提高了20%和2%。对于体高,模型2和模型3分别比模型1的准确性提高了78%和69%。对于胸围,模型3的准确性比模型1提高了38%。对于管围,模型2和模型3分别提高了6%和5%的准确性。对于腿臀围,模型2和模型3分别提高了31%和36%的准确性。对于眼肌深度,模型2和模型3分别提高了231%和233%的准确性。综合结果表明,模型2估计体长、体高、管围和背膘厚的准确性最高,模型3估计胸围、腿臀围和眼肌深度的准确性最高。与利用个体系谱信息的BLUP法相比,使用基因组信息的GBLUP法提高了体尺性状遗传参数估计的准确性。使用添加个体近交系数的GBLUP法时,胸围、腿臀围和眼肌深度性状准确性最高。

表5 不同模型估计豫农黑猪体尺性状遗传参数的准确性Table 5 Accuracy of different models for estimating genetic parameters of body measurements traits in Yu’nong black pigs
3 结论与讨论
本研究中,GBLUP法估计的体尺性状遗传力为0.122~0.652,体高、腿臀围、背膘厚和眼肌深度属于高遗传力性状;体长、胸围和管围属于中遗传力性状,这与大白猪体长、体高和管围的遗传力分别为0.25、0.21和0.10[14]以及大白猪的体尺性状遗传力一般在0.11~0.53之间[15-16]的研究结果相似。谢保胜等[17]估计金华猪的体长、体高、胸围和腿臀围属中、高遗传力。韩国杂交猪的体长、体高遗传力为0.396和0.517[18],这略高于本研究结果,可能是由于猪的品种、饲养管理水平、遗传背景和动物模型的不同导致估计的遗传力有所差异。庄站伟等[19]估计美系杜洛克猪体高、管围的遗传力,分别为0.352和0.160。CHOI等[20]研究显示,杜洛克猪的背膘厚遗传力为0.38,低于本研究结果,推测是由于纯种猪与杂交群体的遗传结构存在差异。通过对不同猪种体尺性状遗传参数的比较发现,体长和体高的遗传力均为中等及以上。在育种工作中,对于遗传力高的性状可选留优秀个体,而对于遗传力低的性状可以利用杂交方式,发挥种间的杂种优势,进而改良部分性状。
本研究的模型1基于系谱亲缘关系矩阵的BLUP法,模型2与模型3则是基于基因组关系矩阵的GBLUP法。有研究报道,性别和出生季节对商品猪的产仔数、背膘、眼肌和能量利用率有显著影响[21-22],以及不同场区的猪舍环境对猪只的生长发育影响较大,直接导致育肥猪体尺性能的差异[23],这与本研究结果相似。此外,现代猪场多应用批次化生产技术,在模型中应综合考虑以上因素的影响。AGUILAR等[24]报道忽略近交系数会影响育种值估计的准确性。因此,本试验的模型3额外增加了个体的基因组近交系数作为协变量。近交系数的传统计算方法是根据系谱进行通径分析,但由于生产中各环节的系谱记录及人为因素,造成了近交系数的可靠性降低[25-26]。利用分子信息技术手段,基于ROH计算的基因组近交系数最接近群体真实的近交系数[27-28]。通常情况下,群体选育的世代越短,ROH片段重组的可能性越低,说明最近的世代可能发生过近交[29]。因此,鉴别和分析ROH是揭示繁育后群体的近交水平及个体间亲缘关系的有效手段[30-31]。
在各模型中,残差效应差异较大,可能是因为固定效应间的互作会增加残差效应的比重[32]。BLUP法估计的胸围和管围遗传力高于GBLUP,主要因为环境因素、亲属间的相似性使加性方差和遗传力偏高,而GBLUP法可以减弱环境效应造成的亲缘相似性[33]。模型估计的准确性表明,基于基因组信息的模型比利用系谱结构的模型估计准确性更高。在鸡和牛群体中的研究表明,应用GBLUP法估计个体育种值的准确性高于BLUP法[34-35]。这与本研究结果一致,是因为基因组信息的添加提高了没有系谱记录个体的育种值精度[36]。本研究中BLUP法对体尺性状遗传参数的估计准确性偏低,可能是由于试验群体系谱信息的准确性和完整性较低。与模型2相比,考虑基因组近交系数的模型3提高了胸围、腿臀围和眼肌深度的遗传参数估计准确性。增加了近交系数的GBLUP,其估计奶牛产奶量、乳脂和乳蛋白含量的基因组育种值准确性较高[9],这与本研究结果相似,可能是通过ROH可以检测到系谱记录之外的世代亲缘关系从而提高了遗传参数估计的准确性。模型3估计体长、体高、管围和背膘厚遗传参数的准确性略低于模型2,因为基因组育种值估计的准确性有一部分受限于群体规模的大小,可以在不断积累表型数据的同时扩大基因组测定群体,为生产实践中的早期选择建立基础[37-38]。综合模型估计的准确性和性状的遗传力来看,对于低遗传力性状,利用系谱和基因组数据估计遗传参数的准确性差别不大,对于中、高遗传力性状,GBLUP进行遗传参数估计的准确性更高[39-40]。因此,在生产实践中,可根据具体的体尺性状合理利用近交系数,以提高豫农黑猪体尺性状遗传参数估计的准确性。
综合分析表明,在豫农黑猪群体中,与BLUP法相比,GBLUP法提高了体尺性状遗传参数估计的准确性,利用个体的基因组近交系数,进一步优化了胸围、腿臀围和眼肌深度遗传参数估计模型的应用效果。
